Sr. Editor: La inmunidad por infección por SARS-CoV-2 es un campo en el que hay muchas interrogantes. Una de las más importantes es la duración de la protección después de una primera infección. La reinfección por SARS-CoV-2 se ha descrito en pocos países y es un asunto preocupante 1.
La reinfección por SARS-CoV-2 se ha definido como dos episodios clínicos diferentes en un paciente, con dos resultados de RT-PCR positivos, donde los linajes del SARS-CoV-2 se muestran diferentes a través de la secuenciación del genoma completo 2. Se presenta el primer caso confirmado de reinfección por SARS-CoV-2 y otros tres posibles casos de reinfección (Material suplementario), identificados a través de cultivo viral y secuenciación del genoma completo, procedentes de una base de datos nacional.
El primer caso de reinfección por SARS-CoV-2 se dio en una niña de 6 años, natural de Lima, con antecedentes de trisomía 21 y leucemia linfoblástica aguda. Los síntomas empezaron el 8 de abril del 2020, y se confirmó con prueba molecular positiva (RT-PCR +) el 11 de abril. Luego de 96 días, el 16 de julio, tuvo otra prueba molecular positiva; debido a los síntomas presentados, fue necesario usar oxígeno. Se le detecto luego bacteriemia por Klebsiella pneumoniae, productora de carbapenemasas, y falleció el 17 de octubre del 2020.
Se identificaron otros tres posibles casos de reinfección; de los cuales sus muestras no tuvieron crecimiento en el cultivo viral, ni hubo material genético suficiente para la secuenciación, probablemente debido a la degradación viral de la muestra. Sin embargo, el valor del umbral de ciclo del PCR indica una alta sospecha de reinfección (Material suplementario).
Existen pocos casos confirmados de reinfección por SARS-CoV-2 en el mundo, su seguimiento es importante ya que está directamente relacionado con la inmunidad generada tras la infección, y con las nuevas variantes de preocupación y mutaciones del virus. Estas variables son importantes para la generación futura de vacunas con adecuada inmunogenicidad, a fin de evitar nuevos brotes 3. Tillett et al. reportaron el primer caso de reinfección en un varón de 25 años en Estados Unidos, con diferencias genómicas en ambos episodios 4; luego se reportaron otros casos en China, Bélgica, Ecuador, etc. 1 En todos los reportes hubo variación en el episodio con síntomas más severos.
La paciente con reinfección confirmada presentó en la primera muestra el linaje B.1.1 (clado GR) y en la segunda muestra el linaje B.1 (clado G) (Figura 1), reportados previamente en el Perú 5. Los otros tres posibles casos de reinfección se produjeron en personal sanitario sin comorbilidades importantes y tuvieron el linaje B.1.1.1 (clado GR). Esto último posiblemente ocurre porque el personal de salud está más expuesto al contacto continuo con el SARS-CoV-2, lo que aumenta la probabilidad de reinfección; por lo que equipos de bioseguridad, uso de pruebas moleculares y más estrategias deben estar disponibles como estrategia de vigilancia en profesionales de salud.
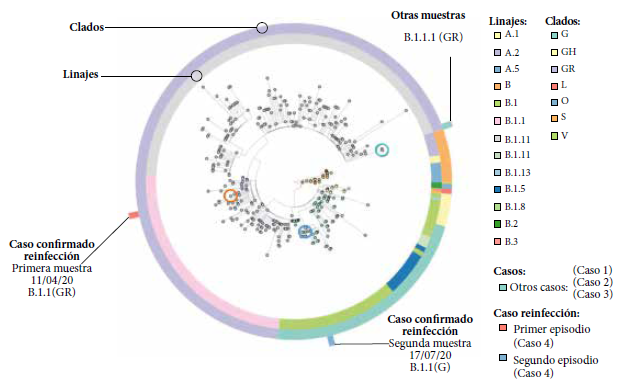
Figura 1: Análisis filogenético del primer y segundo aislamiento del SARS-CoV-2, del primer caso confirmado de reinfección por COVID-19 en el Perú.
El inicio de la inmunidad contra el SARS-CoV-2 ocurre días después de la infección, y del día 5 al día 10 se produce un fuerte aumento de la respuesta humoral mediada por células B y por los consiguientes anticuerpos contra la proteína espiga. Después de alcanzar un pico máximo entre el día 28 (IgM/A) y el día 49 (IgG), la inmunidad disminuye. La inmunidad celular (células T CD4+ y CD8+) también aumenta desde la primera semana de infección y alcanza su punto máximo en la segunda semana, solo para comenzar a disminuir al día 100 6. Aún se desconoce el tiempo que persiste la inmunidad de memoria, se sabe que su respuesta es variable entre los pacientes, según la cinética de los tipos de memoria y del cuadro clínico.
En el Perú la campaña de vacunación contra la COVID-19 debe continuar hasta lograr la inmunidad colectiva. Sin embargo, ante nuevos brotes o casos de reinfección es importante estudiar las mutaciones y las variantes de preocupación involucradas. Se debe construir un sistema de vigilancia genómica nacional que permita detectar las variantes (B.1.1.7, P1, B.1.35, B.1.43 y otras nuevas que se podrían presentar) del SARS-CoV-2 y de otras enfermedades (tuberculosis, VIH, infecciones intrahospitalarias resistentes, etc.). Nuestras capacidades de pruebas moleculares se han ampliado durante la epidemia, pero son necesarios más recursos públicos y privados para continuar estudiando su impacto en la dinámica epidemiológica del país.















