Introducción
Los caracoles dulceacuícolas del género Pomacea (Ampullariidae) son conocidos a nivel global como caracol manzana (apple snail), en cuanto que en la Amazonia Peruana como churos (Dourojeanni 1965; Llanos et al. 2018). Son consumidos por el poblador amazónico desde tiempos ancestrales (Alcántara & Nakagawa 1996; Lombardo et al. 2013), y están presentes en la culinaria regional (Vetter & Aliaga 2012). El consumo de churos se realiza todo el año, aunque en menor proporción en la época de vaciante (Cobos 1998; Manno et al. 2018), y son comercializados en mercados de Iquitos y otros de la Amazonia peruana, tanto por extractores, cultivadores como revendedores (Cobos 1998). Un proceso de globalización de la culinaria peruana iniciado hace unas décadas ha llevado a difundir la riqueza gastronómica amazónica más allá de los clubes departamentales y provinciales (Chirif 2005), llegándose a crear en Lima un restaurante gourmet dedicado completamente a la cocina amazónica donde uno de los platos más solicitados es el churo (Ferraro Vidal 2019).
La literatura regional correspondiente a los churos solo menciona a la especie Pomacea maculata, por ejemplo, en estudios sobre su biología (Mayta 1978; Alcántara et al. 1996; Alcántara & Nakagawa 1996; Cobos 1998; MINAM, 2014; Alcántara et al. 2015), cultivos (Padilla Pérez et al. 2000; Alcántara & Colace 2001; Álvarez & Ríos 2009; Ríos & Alava 2012), etnobiología (Gasché & Vela 2011; Huertas & Chanchari 2011; Zavaleta et al. 2018), industrialización (Padilla Pérez et al. 2000; López 2001; Sánchez Pinedo et al. 2008) y culinaria (Vetter & Aliaga 2012; Zocchi 2017).
Las especies de la familia Ampullariidae originalmente descritas para Perú son alrededor de 20 (Cowie & Thiengo 2003; Ramírez et al. 2003), y pocas fueron registradas con localidades específicas. Su taxonomía aún está pobremente dilucidada, porque muchas se conocen apenas por caracteres conquiológicos, y no existen revisiones taxonómicas completas que incluyan muestras peruanas (Ramírez et al. 2003; Ampuero & Ramírez 2017). Ello conduce a un gran número de nombres disponibles relacionados a Pomacea maculata [e.g., P. gigas (Spix in Wagner, 1827), P. crosseana (Hidalgo, 1871)] y P. urceus (O. F. Müller, 1774) [e.g., P. erythrostoma (Reeve, 1856), P. guyanensis (Lamarck, 1822), P. haemastoma (Reeve, 1856), P. nobilis (Reeve, 1856)] (Pain 1960; Tillier 1980; Cowie & Thiengo 2003). Sobre la base morfológica y genética, Hayes et al. (2012) propusieron a P. gigas y P. insularum (d’Orbigny, 1835) como sinónimos de P. maculata, con amplia distribución en América del Sur, y que junto con P. canaliculata (Lamarck, 1822) son especies altamente invasoras, reportadas en América del Norte, Europa y Asia (Rawlings et al. 2007; Hayes et al. 2008; Perez et al. 2017; Yang et al. 2018). Sin embargo, la distribución de P. maculata en Perú aún no está confirmada (Hayes et al. 2012).
Ante la ausencia de estudios que confirmen la presencia de P. maculata como la especie más usada comercialmente en el Perú, en el presente trabajo se identifica mediante filogenia molecular a los caracoles más expendidos en los mercados de la región Loreto (Perú), conocidos como churos negros, y los usados en un plato gourmet proveniente de un restaurante en Lima.
Material y métodos
Obtención del material biológico.- Se compraron churos en los años 2011, 2017 y 2018 (Tabla 1) en los mercados del departamento de Loreto, como Belén (3°46′01″S 73°14′48″W), Bellavista (3°42'12"S 73°14'54"W) y Yurimaguas (La Florida, 05°55´53.5”S, 076°06´50.0”W); además a pobladores de Nauta (río Marañón, cocha Mullaca, 4°35´10.93”S, 73°36´32.24”W) y río Itaya (4°5´14.75”S, 73°26´48.57”W), así como plato a la carta en la ciudad de Lima (Tabla 1). Los especímenes fueron relajados agregando pequeñas cantidades de etanol al agua que los contenía y sacrificados por shock térmico (4 °C); fueron preservados en etanol al 96 %.
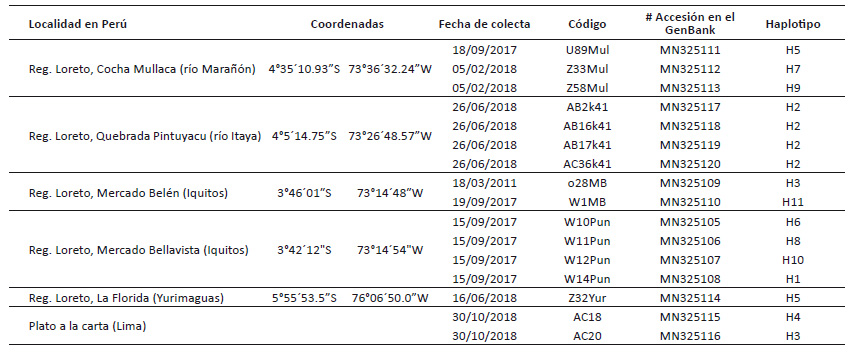
Tabla 1 Datos de las 16 secuencias COI de Pomacea nobilis generadas en el presente trabajo. Se indica el haplotipo al que pertenece cada muestra.
Para la identificación morfológica se contó con las descripciones originales de las especies descritas para Perú, así como fotografías de los ejemplares tipos de diferentes museos (NHMUK, Natural History Museum, London; NMW.Z., National Museum of Wales, Cardiff, UK). También se revisaron muestras de la colección del Departamento de Malacología del Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos. Los ejemplares utilizados en la presente investigación están depositados en el Departamento de Malacología del Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos.
Procesamiento molecular.- Para el análisis se consideraron un total de 16 ejemplares (Tabla 1), trabajados en el Laboratorio de Sistemática Molecular y Filogeografía de la Facultad de Ciencias Biológicas de la Universidad Nacional Mayor de San Marcos. La extracción de ADN total se realizó a partir de músculo de pie del caracol, mediante el kit de extracción E.Z.N.A. Mollusc DNA de OMEGA Bio-Tek. Un segmento del gen mitocondrial citocromo oxidasa subunidad I (COI) se amplificó por PCR con los primers de Folmer et al. (1994), LCO 1490 (5'-GGTCAACAAATCATAAAGATATTGG-3'), HCO 2198 (5'-TAAACTTCAGGGTGACCAAAAAATCA-3'). Cada reacción de PCR fue trabajada en un total de 30 µL, cuyo mix conformado por solución buffer PCR 1X, 0.2 mM dNTP, 0.3 µM de cada primer, 1.25 mM MgCl2, 1 unidad de Taq DNA polimerasa, 3 µL de DNA genómico, y agua bidestilada para completar el volumen. Para la amplificación se comenzó con una denaturación inicial de 96 °C por 1 min, seguida de 35 ciclos: Denaturación a 95 °C por 60 s, hibridación de los iniciadores a 40°C (40 - 54 °C) por 60 s y extensión a 72 °C durante 90 s, terminando con un paso de extensión final a 72 °C por 7 min. La visualización de los productos de PCR se realizó en electroforesis submarina, con gel de agarosa a 1% preteñida con Sybr Green, a 60 voltios por 20 min. Ambas hebras de los amplicones fueron secuenciadas por la compañía Macrogen Korea (https://dna.macrogen.com/eng/). Las 16 secuencias se encuentran depositadas en la base de datos GenBank (Códigos de Accesión MN325105-MN325120) (Tabla 1).
Análisis de datos moleculares. Las secuencias recibidas en formato AB1 fueron editadas en CHROMAS LITE (https://technelysium.com.au/wp/chromas/) y las secuencias consenso obtenidas con el programa CAP3 Sequence Assembly Program (http://doua.prabi.fr/software/cap3). La primera evaluación de las secuencias se realizó usando BLASTn (https://blast.ncbi.nlm.nih.gov/Blast.cgi) para descartar posibles contaminantes y hacer una identificación molecular preliminar. Secuencias (21) del gen COI de ocho especies del género Pomacea, incluyendo P. maculata, fueron obtenidas del GenBank para los análisis comparativos correspondientes (Tabla 2).
Tabla 2 Especies, procedencia, números de accesión y referencias de las secuencias COI obtenidas en el GenBank.
| Especie | Espécimen (haplotipo), País | # Accesión | Referencia |
|---|---|---|---|
| P. guyanensis | GF02, French Guiana | FJ710319.1 | Hayes 2009; Hayes et al. 2009b |
| P. guyanensis | BR400, Brazil | FJ710320.1 | Hayes 2009; Hayes et al. 2009b |
| P. guyanensis | BR100, Brazil | FJ710321.1 | Hayes 2009; Hayes et al. 2009b |
| P. glauca | SR05, Suriname | FJ710322.1 | Hayes 2009; Hayes et al. 2009b |
| P. glauca | VE07, Venezuela | FJ710323.1 | Hayes 2009; Hayes et al. 2009b |
| P. maculata | KM002, USA | KM107827.1 | Deaton et al. 2016 |
| P. maculata | KM013, USA | KR350499.1 | Deaton et al. 2016 |
| P. insularum | BR245, Brazil | EU528568.1 | Hayes 2009; Hayes et al. 2009b |
| P. insularum | Hap3, Argentina | AB728584.1 | Yoshida et al. 2014 |
| P. canaliculata | Hap2, Argentina | AB728586.1 | Yoshida et al. 2014 |
| P. canaliculata | Hap3, Argentina | AB728587.1 | Yoshida et al. 2014 |
| P. canaliculata | HI39, USA | EU523129.1 | Tran et al. 2008 |
| P. dolioides | BR140, Brazil | EU528555.1 | Hayes et al. 2008 |
| P. dolioides | BR143, Brazil | EU528556.1 | Hayes et al. 2008 |
| P. dolioides | BR217, Brazil | EU528565.1 | Hayes et al. 2008 |
| P. scalaris | AR209, Argentina | EU528532.1 | Hayes et al. 2008 |
| P. scalaris | BR56, Brazil | EU528545.1 | Hayes et al. 2008 |
| P. bridgesi | BR329, Brazil | EU528573.1 | Hayes et al. 2008 |
| P. bridgesi | BR331, Brazil | EU528574.1 | Hayes et al. 2008 |
| P. diffusa | BR214, Brazil | EU528564.1 | Hayes et al. 2008 |
| P. diffusa | BR72, Brazil | EU528547.1 | Hayes et al. 2008 |
Las secuencias fueron alineadas con CLUSTAL W (Thompson et al. 1994). La evaluación de los haplotipos, y los cálculos de la diversidad haplotípica y nucleotídica fueron realizados con el programa DnaSP (Librado & Rozas 2009). Para obtener las distancias genéticas se usó distancia p en el programa MEGA (Tamura et al. 2011), así como para el análisis filogenético por Neighbour-Joining (NJ), en el que se emplearon 1000 réplicas de bootstrap para estimar el soporte de los nodos. Las distancias genéticas se graficaron usando el programa R (R Core Team 2016).
El árbol filogenético de inferencia bayesiana (IB) fue estimado usando el programa BEAST (Bouckaert et al., 2014), usando un reloj relajado log normal, un modelo birth and death y el modelo de sustitución HKY+I+G, estimado en jModelTest (Posada 2008). El análisis fue implementado con 40 millones de cadenas de Markov (MCMC) y un burn-in de 10%. La convergencia de las cadenas fue evaluada usando el indicador ESS (Efective sample size) el cual fue superior a 200 en todos los priors.
Resultados
Variabilidad de la concha del churo negro.- La característica más saltante de las conchas del churo negro es que son gruesas y sólidas, con un color castaño variando hasta casi negro (Figs. 1, 2). La concha es de forma globosa, umbilicada; con espira algo expandida y acuminada; con opérculo córneo. En cuanto al color del periostraco, si bien predomina el color castaño oscuro que se confunde con negro, en una misma población se puede encontrar variación de tonalidades, hasta un castaño claro que permite percibir líneas espirales oscuras, las cuales varían en forma y tamaño. El interior de la concha es de color anaranjado, los hay de diversas tonalidades e inclusive casi rojo. Sin embargo, a diferencia del color de la concha, el color de la parte blanda del caracol es bastante uniforme: el pie es amarillento cremoso, con el dorso, tentáculos y sifón con pigmento de color granate (Fig. 2).

Figura 1 Pomacea nobilis. A: Posible sintipo de Ampullaria nobilisReeve, 1856 (NHMUK 20020669) © NHMUK. B: Pomacea urceus guyanensis de Perú de la colección de Pain (1960) (NMW.Z.1981.118.04851) © Amgueddfa Cymru - National Museum Wales. C, D: Ejemplares procedentes de Cocha Mullaca. E y F: Especímenes comprados en los mercados de Belén y Bellavista. Escala: 3 cm
Las características encontradas en las conchas del churo negro corresponderían a las descritas por Reeve (1856) para Pomacea nobilis, P. erythrostoma y P. haemastoma, las que fueron consideradas en la sinonimia de P. guyanensis (Lamarck, 1822) y P. urceus (Müller, 1774), (ver Pain 1960; Cowie & Thiengo 2003).
Distancias genéticas y relaciones evolutivas.- De las 16 secuencias COI obtenidas en el presente trabajo, dos proceden de un restaurante en Lima donde figuran como plato a la carta, material al que se aplicó las mismas condiciones seguidas para los especímenes completos, obteniéndose resultados óptimos. Ninguna de las 16 secuencias del marcador COI obtenidas de distintos morfotipos de churo negro, así como de un plato servido a la carta, corresponde a alguna especie de Pomacea depositada en el GenBank. Sin embargo, sí se observa cercanía a Pomacea guyanensis (Lamarck, 1822). Los más altos porcentajes (87.38% y 88.50%) de identidad en el BLASTn lo dieron con Pomacea guyanensis.
El alineamiento de las 16 secuencias resultó de 709 sitios, considerando los dos primers, que colapsaron en 11 haplotipos. Al agregar las 21 secuencias del GenBank (Tabla 2), y luego de recortar los extremos para ajustar las secuencias al mismo tamaño, el nuevo alineamiento quedó de 600 sitios, sin indels, y las 37 secuencias colapsaron en 32 haplotipos. Ninguna de las especies compartió haplotipos. Once haplotipos son del churo negro, que presentó diversidad haplotípica de h = 0.9333, y diversidad nucleotídica de π = 0.0184.
En la filogenia molecular, las 14 secuencias COI del churo negro y las dos que proceden del plato a la carta servido en la ciudad de Lima formaron un grupo monofilético relacionado con P. guyanensis y P. glauca, con soporte bootstrap de 86% (Fig. 3) y un valor de probabilidad posterior de 1.0 (Fig. 4), siendo hermano del clado de P. guyanensis, tanto en el árbol filogenético por NJ, con un soporte bootstrap de 99 % (Fig. 3) como por IB, con un valor de probabilidad posterior de 0.99 (Fig. 4).
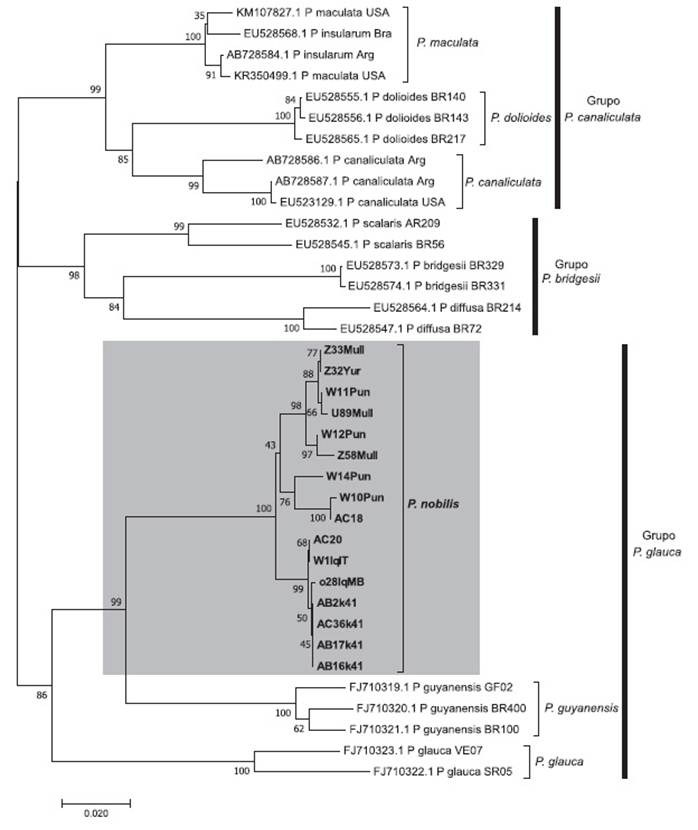
Figura 3 Árbol filogenético para el marcador mitocondrial COI obtenido usando distancias no corregidas, con Neighbour-Joining. Los números corresponden a valores de bootstrap. La escala corresponde a número de substituciones por sitio. La nominación de grupos según Hayes (2009) y Hayes et al. (2009a, b). Note que las 16 secuencias de Pomacea nobilis conforman un grupo monofilético hermano de Pomacea guyanensis.
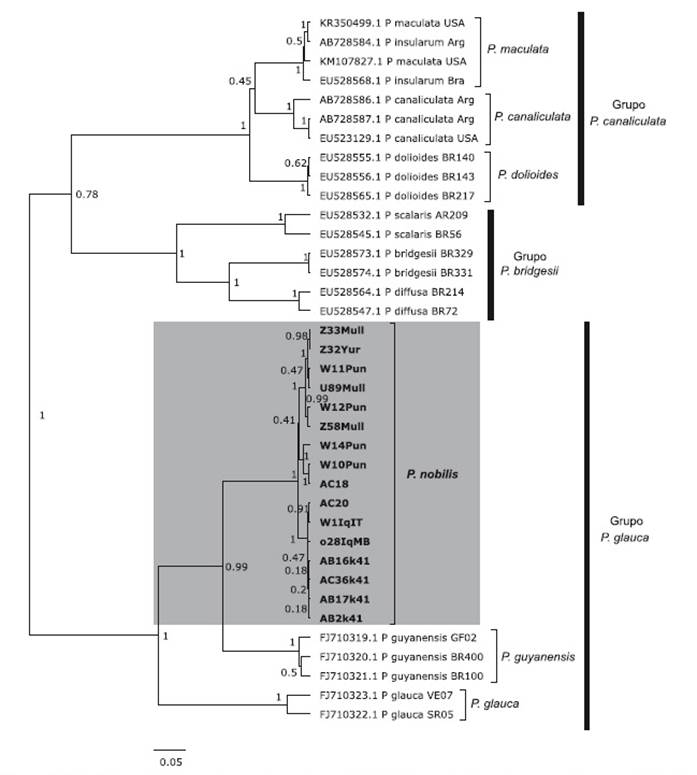
Figura 4 Árbol filogenético para el marcador mitocondrial COI obtenido utilizando el modelo de substitución nucleotídica HKY+I+G, por el método de Inferencia Bayesiana. Los números corresponden a valores de probabilidades posteriores bayesianas. La escala corresponde a número de substituciones por sitio. La nominación de grupos según Hayes (2009) y Hayes et al. (2009a, b). Note que las 16 secuencias de Pomacea nobilis conforman un grupo monofilético hermano de Pomacea guyanensis.
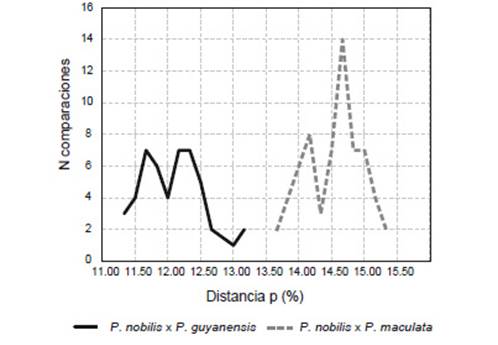
El análisis de distancia genética mostró una divergencia intraespecífica del churo negro de entre 0% y 3.17% (Fig. 5), en cuanto que la interespecífica con P. guyanensis entre 11.33% y 13.17%, y con P. maculata entre 13.67% y 15.33% (Fig. 6).
La variación morfológica de la concha no se reflejó en la filogenia molecular. Es así que, de las tres especies de Reeve (1856) antes mencionadas, aquí se identifica al churo negro como Pomacea nobilis, (ver discusión)
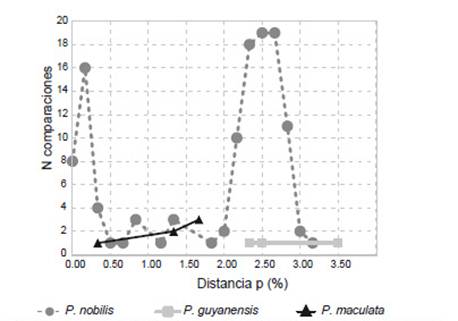
Figura 5 Distribución de distancia genética no corregida, intraespecífica, para Pomacea nobilis, P. guyanensis y P. maculata. N: número de comparaciones.
Discusión
El churo negro es el caracol dulceacuícola del género Pomacea (Mollusca, Ampullariidae) más comercializado en la Amazonia peruana, y utilizado en la cocina gourmet de Lima, y que fue identificado en este trabajo como Pomacea nobilis (Reeve, 1856).
Encontramos que la variación morfológica de la concha es intraespecífica, y que los morfotipos los podemos identificar como Pomacea nobilis (Reeve, 1856), P. erythrostoma (Reeve, 1856) y P. haemastoma (Reeve, 1856). Sin embargo, basados en el marcador mitocondrial COI, estos morfotipos se agrupan en el mismo clado y por lo tanto podrían representar variaciones de una misma especie. Debemos recalcar que P. nobilis, P. erythrostoma y P. haemastoma, han sido descritas solo sobre la base de sus conchas, y anteriormente ya han sido consideradas como una sola especie. Así, Sowerby (1909a, b) propuso a P. erythrostoma y P. haemastoma como sinónimos de P. guyanensis (Lamarck, 1822). Pain (1960) incluyó a P. erythrostoma y P. haemastoma en la sinonimia de P. (Pomacea) urceus guyanensis (Lamarck, 1819). Más tarde, Tillier (1980) propuso en la sinonimia de Ampullaria urceus (Mueller, 1774) a Pomacea (Pomacea) urceus guyanensis de Pain (1960), así como también a A. guyanensis Lamarck, 1819. Siguiendo a Tillier (1980), Cowie & Thiengo (2003) pasaron a la sinonimia de P. urceus a P. nobilis, P. erythrostoma y P. haemastoma. Entre las localidades tipo de las tres últimas especies, solamente la de P. haemastoma indica como “Perú”; este es el nombre que usaron Ampuero y Ramírez (2017) en su estudio anatómico descriptivo del churo negro, donde demuestran los aspectos morfológicos que la diferencian de P. guyanensis y de P. urceus.
En la filogenia molecular de la familia Ampullariidae (Hayes et al. 2009a, b) solo se encuentra a P. guyanensis, con material procedente de Guyana Francesa y de Brasil, sin hacer mención alguna a P. urceus. Al presente, P. guyanensis es considerada una especie válida (Hayes 2009; Massemin et al. 2009; Cowie & Héros 2012). En la filogenia molecular con el marcador COI, las 16 secuencias obtenidas en el presente estudio formaron un clado monofilético, con soporte bootstrap de 100% (Fig. 3) y un valor de probabilidad posterior de 1.0 (Fig. 4) y hermano al de P. guyanensis, con distancias intraespecíficas de 0 a 3.17% (Fig. 5) y distancias interespecíficas de 11.33% a 13.17% (Fig. 6), diferenciándose ampliamente ambos clados, cuyo nodo resultó fuertemente soportado con bootstrap de 99 % (Fig. 3) y 0.99 de probabilidad posterior (Fig. 4). Aquí nominamos al churo negro como Pomacea nobilis (Reeve, 1856: pl. 2, fig. 8), proponiendo una revalidación de esta especie, y por prioridad, quedando como sus sinónimos P. haemastoma (Reeve, 1856: pl. 7, fig. 34) y P. erythrostoma (Reeve, 1856: pl. 13, fig. 59). El posible sintipo de P. nobilis está depositado en el Natural History Museum, London (NHMUK 20020669) (Cowie & Thiengo 2003) (Fig. 1A), con localidad tipo “River Marañon”, en cuanto que el paradero del de P. erythrostoma es desconocido, con localidad tipo reconocida por Sowerby (1909a) también para Perú, luego de un error de Reeve (1856) al colocarla para Zanzibar (archipiélago de Tanzania). Pomacea nobilis conforma parte del grupo de P. glauca (Hayes 2009; Hayes et al. 2009a), como es evidenciado en nuestros resultados por métodos filogenéticos de NJ (Figura 3) e IB (Fig. 4).
El churo negro ha sido referido en la literatura para Perú como Pomacea maculata. Sin embargo, al revisar las fotografías o dibujos identificables en esa literatura, se pudo notar que corresponden a Pomacea nobilis (Reeve, 1856) (e.g. Mayta 1978; Cobos 1998; Alcántara & Colace 2001; López 2001; Alcántara et al. 2015; Zocchi 2017). En el caso de Cobos (1998), que compara la mención de Pain (1960) para P. maculata (por encima de 155x135 mm), indica que la “longitud” y “ancho” máximo de los especímenes observados en el río Marañón fueron de 91.6x74.9mm, lo que está en concordancia con lo encontrado por Ampuero y Ramírez (2017) (entre 75 y 100 mm). Kobelt (1913) da las siguientes medidas para el tipo de Ampullaria nobilis Reeve: “Alt. 110, diam. cca. 100 mm.” En cuanto al material de Pomacea maculata estudiado por Pain (1960), éste no guarda ninguna semejanza con P. nobilis, mas sí el ejemplar que menciona como “Pomacea urceus guyanensis (Lamarck)”, cuyo espécimen procedente de “Peru: Rio Marañon, nr. Iquitos (Pain)” se encuentra depositado en el National Museum of Wales (NMW.Z.1981.118.04851) (Fig. 1B).
Los ejemplares de Pomacea nobilis aquí trabajados proceden de mercados de las ciudades de Iquitos y Yurimaguas de la región Loreto, así como de zonas inundables de los ríos Marañón e Itaya. Esta especie es ampliamente comercializada y puede ser encontrada fácilmente siendo vendida en grandes cantidades (R. Ramírez obs. pers.), así como también lo mencionan Alcántara et al. (2015) que en diciembre del 2011 adquirieron dos lotes de 95 individuos. Cobos (1998) encontró un promedio de hasta 26.85 individuos por m2 en su hábitat natural en una zona inundada por el río Marañón, y menciona que “la venta se realiza por cientos en el lugar de captura, a embarcaciones que se dirigen a la ciudad de Iquitos” o “transportados por los mismos pobladores hacia puertos de los pueblos más grandes (Santa Rita de Castilla y Nauta) y mercados de la ciudad de Iquitos”. Pero también lo venden fuera de la Amazonia, en platos a la carta servidos en la ciudad de Lima.
Con el presente trabajo queda identificada la especie de churo negro que llega a los mercados de Iquitos, Pomacea nobilis (Reeve, 1856), mediante su código de barras de ADN (Hebert et al. 2003; Ardura et al. 2010; Marín et al. 2018), que es el respaldo molecular fehaciente para autenticar productos procesados que refieren como base al churo negro, antes mal determinado como Pomacea maculata. Asimismo, el tener claramente identificada a la especie de churo (caracol manzana, apple snail) más comercializada de la Amazonia Peruana, Pomacea nobilis, es un paso importante para su eventual exportación, pues el etiquetado correcto es requerido por seguridad alimentaria (Wong et al. 2011; Wallace et al. 2012; Böhme et al. 2019; Calegari et al. 2020).












 uBio
uBio