Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica v.28 n.1 Lima mar. 2011
SECCIÓN ESPECIAL
Experiencias en la vigilancia epidemiológica de agentes patógenos transmitidos por alimentos a través de electroforesis en campo pulsado (PFGE) en el Perú
Experiences in the epidemiological surveillance of foodborne pathogens by pulsed field gel electrophoresis (PFGE) in Peru
María Luz Zamudio1,a, Ana Meza1,b, Henri Bailón2,a, Jaime Martinez-Urtaza3,c, Josefina Campos4,d
1 Laboratorio de Referencia Nacional de Enteropatógenos, Centro Nacional de Salud Pública, Instituto Nacional de Salud, Lima, Perú.
2 Laboratorio de Biotecnología y Biología Molecular, Centro Nacional de Salud Pública, Instituto Nacional de Salud, Lima, Perú.
3 Instituto de Acuicultura, Universidad de Santiago de Compostela, Santiago de Compostela, España.
4 Servicio de Enterobacteria. ANLIS. Instituto Carlos G. Malbrán, Buenos Aires, Argentina.
a Bióloga; b Técnico Especializado en Laboratorio; c Doctor en Ciencias Biológicas; d Bioquímica.
RESUMEN
Las enfermedades transmitidas por alimentos (ETA) y otras enfermedades entéricas infecciosas ocurren a menudo como brotes y son causa de morbilidad y mortalidad en todo el mundo. En el Perú, son un importante problema de salud pública y son causados por una gran variedad de agentes infecciosos. Para la investigación epidemiológica se utiliza una variedad de métodos de tipificación. Una de las herramientas más importantes en la subtipificación molecular de patógenos bacterianos es la técnica de la electroforesis en campo pulsado (PFGE), que es un método altamente resolutivo que permite la discriminación entre diferentes aislamientos bacterianos epidemiológicamente relacionados. El Instituto Nacional de Salud (INS) del Perú integra las redes WHO Global Foodborne Infections Network y la Red PulseNet América Latina y Caribe, con quienes comparte los perfiles genéticos de las cepas patógenas aisladas, permitiendo comparar los genotipos de cepas semejantes halladas en diferentes países y reconocer la ocurrencia de brotes epidémicos en la región, fortaleciendo el sistema de vigilancia epidemiológica regional y generando una rápida respuesta conjunta entre países. Se presenta la experiencia de los dos últimos años sobre los avances en la utilización de estas herramientas estratégicas que nos ha permitido caracterizar patrones de genotipo de principales patógenos implicados en ETA a partir de aislamientos recuperados de la red de laboratorios del Perú.
Palabras clave: Vigilancia Epidemiológica; Enfermedades Transmitidas por Alimentos, Electroforesis en Gel de Campo Pulsado; Brotes; Perú (fuente: DeCS BIREME).
ABSTRACT
Foodborne diseases and other enteric infections often occur as outbreaks and cause morbidity and mortality all over the world. In Perú, they represent a serious public health problem, and are caused by a great variety of infectious agents. For epidemiological research, a wide array of typification methods are used. One of the most important tools for the molecular subtyping of bacterial pathogens is the Pulsed Field Gel Electrophoresis (PFGE), which is a highly precise method that allows the discrimination between different bacterial isolates which are epidemiologically related. The Instituto Nacional de Salud del Perú (INS) is part of the WHO Global Foodborne Infections Network (WHO-GFN) and of the PulseNet Latin American and Caribbean Net (PN-AL & C), with whom it shares the genetic profiles of the isolated pathogenic strains, so that it is possible to compare de genotypes of similar strains found in different countries and to identify the occurrence of epidemic outbreaks in the region, strengthening the regional system of epidemiological surveillance and generating a rapid, coordinated response between the countries. We present the two last years´ experience including the advances in the use of these strategic tools that have allowed us to characterize genotype patterns implicated in foodborne diseases from isolates recovered in the laboratory network of Peru.
Key words: Epidemiologic Surveillance; Foodborne Diseases, Electophoresis, Gel, Pulsed-Field; Outbreaks; Peru (source: MeSH NLM).
INTRODUCCIÓN
Las enfermedades transmitidas por alimentos (ETA) son enfermedades producidas por la ingestión de alimentos o agua contaminados con agentes químicos o microbiológicos. La contaminación puede deberse a la deficiencia en el proceso de elaboración, manipulación, conservación, transporte, distribución o comercialización de alimentos y agua, las cuales pueden clasificarse en infecciones o intoxicaciones alimentarias sin incluir las reacciones de hipersensibilidad a los alimentos (1-3).
Las ETA son una causa importante de morbilidad y mortalidad en todo el mundo, además de producir un gran impacto económico tanto por los gastos en salud producidos como en las actividades económicas relacionadas con la producción de alimentos (2-5). Para el año 2005 se estimó que 1,5 millones de personas murieron a causa de enfermedades diarreicas a nivel mundial y el 70% de ellas son atribuidas a las ETA (5). Meads et al. (6) estimaron que durante el año 1996 en Estados Unidos se produjeron 76 millones de casos, 325 mil hospitalizaciones y cinco mil muertes.
En el Perú, donde solo el 38% de hogares tiene acceso a agua libre de coliformes fecales (7) y se ha demostrado la presencia de patógenos bacterianos y parasitarios en alimentos tanto en la capital como provincias (8,9), las ETA son, indudablemente, un importante problema de salud pública.
Las ETA son causadas por una gran variedad de agentes infecciosos y, a menudo, ocurren como brotes, por lo que la vigilancia epidemiológica es de vital importancia, para ellos se utiliza una variedad de métodos de tipificación (3). Una de las herramientas más importantes en la subtipificación molecular de patógenos bacterianos es la técnica de la electroforesis en campo pulsado (PFGE, por sus siglas en inglés), que es un método altamente resolutivo que utiliza enzimas de restricción que cortan el ADN bacteriano generando unos patrones de macrorrestricción genómicos o huellas digitales que permite establecer el grado de identidad genética entre diferentes aislamientos bacterianos epidemiológicamente relacionados, de forma que puede realizar una discriminación genotípica de las distintas poblaciones bacterianas de una especie (10-12).
El Instituto Nacional de Salud del Perú es la institución encargada del diagnóstico microbiológico referencial de las ETA en nuestro país, y forma parte de las redes WHO Global Foodborne Infections Network (WHO-GFN) y la Red PulseNet América Latina y Caribe (PN-ALyC), esta última es una red regional de subtipificación molecular para la vigilancia epidemiológica de los patógenos transmitidos por alimentos y estudio de brotes por PFGE, que tiene establecida una Base de Datos Regional (BDR) con sede en la Organización Panamericana de la Salud (OPS), Centro Regional de Salud Pública Veterinaria PANAFTOSA (www.panaftosa.org) en Río de Janeiro Brasil. En esta BDR se registran los perfiles genéticos de las cepas patógenas que circulan en los países que integran la Red PN-ALyC, para comparar los genotipos de cepas semejantes halladas en diferentes países, y reconocer la ocurrencia de brotes epidémicos en la región, fortaleciendo el sistema de vigilancia epidemiológica regional y generando una rápida respuesta conjunta entre países. En el Perú, el Sistema Nacional de Vigilancia Epidemiológica en Salud Pública del Ministerio de Salud (MINSA) tiene como objetivo prevenir, controlar daños y reducir la carga de morbilidad y mortalidad en el país. Entre los años 2003 y 2007, este sistema detectó 134 brotes de enfermedades transmitidas por alimentos (ETA); 57 (42,5%) se relacionaron clínicamente con casos agudos de salmonelosis (13). El INS confirmó durante ese periodo 1 228 cepas de Shigella spp., 781 Campylobacter spp., 379 Salmonella spp., nueve Vibrio cholerae non O1 non O139 y un caso de Vibrio cholerae O139 no toxigénico (13).
De esta forma, el INS ha usado el PFGE para subtipificar los aislamientos relacionados con diferentes brotes acontecidos en el Perú. En este artículo se presenta la experiencia en los dos últimos años de los avances en la utilización del PFGE que nos ha permitido caracterizar patrones de genotipo de principales patógenos implicados en ETA a partir de aislamientos recuperados de la Red de Laboratorios del Perú.
BROTE DE Vibrio parahaemolyticus EN EL NORTE DE PERÚ
La dinámica epidémica de V. parahaemolyticus en Perú ha estado caracterizada por la existencia de casos de infección durante la época estival cuando las temperaturas ambientales son más altas. Este patrón estacional cambió drásticamente en 1997 cuando en el invierno austral fue detectado el clon pandémico asiático de V. parahaemolyticus causando un aumento en el número de infecciones a lo largo de la costa de Perú. La aparición y dispersión de casos en Perú en 1997 siguió el mismo patrón de dispersión que la epidemia de cólera en 1991. La aparición de estas dos epidemias de V. parahaemolyticus y V. cholerae coincidieron con el inicio de los eventos del Fenómeno del Niño de 1991 y 1997 (14).
En marzo de 2009, el Laboratorio de Referencia Nacional de Enteropatógenos de Perú (INS) confirmó la identificación de aislamientos clínicos asociados a un brote de infección causado por Vibrio parahaemolyticus en la provincia de Cajamarca, que estuvo relacionado al consumo de pescado. El origen del brote se asoció con el consumo de productos hidrobiológicos procedentes del puerto de Santa Rosa en la ciudad de Lambayeque.
De forma paralela, un total de 17 cepas de Vibrio parahaemolyticus fueron aisladas de casos clínicos de diarreas acuosas detectados en las provincias de Lambayeque, Piura y Lima. Un total de 24 cepas fueron analizadas por la técnica de reacción en cadena de la polimerasa (PCR) para la identificación confirmatoria de Vibrio parahaemolyticus mediante la detección del gen específico toxR. Adicionalmente, se investigó la presencia de los genes asociados con los dos factores de virulencia más importante de esta bacteria: la hemolisina directa termoestable (TDH) y la hemolisina relacionada con la TDH (TRH), que están codificadas respectivamente por los genes tdh y trh. Todas las cepas identificadas como V. parahaemolyticus presentaron el gen tdh, que es el factor de virulencia predominante en esta especie, mientras que fueron negativas para el gen trh.
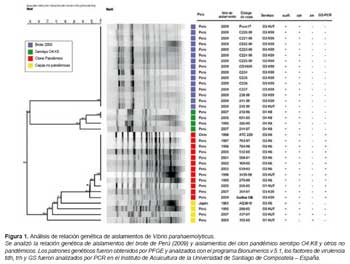
En el marco del convenio entre INS-Perú y la Universidad de Santiago de Compostela (España) durante el desarrollo del proyecto Investigación ecológica de las especies de Vibrio en la costa del Perú, se procedió a la realización de las pruebas de serotipificación y subtipificación molecular por PFGE de 16 cepas obtenidas durante el mes de marzo de 2009 . El protocolo de PFGE que se utilizó para el análisis de las cepas del brote empleó la enzima NotI para generar los patrones de restricción que se muestran en la figura 1 (15) .
La caracterización molecular de las cepas del brote de Cajamarca y los aislamientos clínicos de Lambayeque y Piura reveló que todas las cepas presentaban el mismo perfil genético, evidenciando una conexión epidemiológica entre los aislamientos del brote y los aislamientos de las otras ciudades de la costa norte. Estos resultados sugieren que el pescado contaminado causante del brote podría haber sido también causante de los casos clínicos en Lambayeque y Piura. Además, en al análisis se incluyó un aislamiento procedente de la ciudad de Lima (Guillén-136) que no presentó relación genética con las cepas del brote del norte de Perú.
La comparación de los perfiles genéticos de la cepa de V. parahaemoliticus del brote del año 2009 con otras cepas causantes de brotes en Perú en diferentes años (1997- 2009) (14) muestra que las cepas de este brote forman un grupo genético único y totalmente diferenciado de los otros grupos detectados en los casos clínicos de V. parahaemolyticus en Perú durante los últimos 15 años (Figura 1). Las cepas del brote 2009 fueron incluidas en un cluster diferente del grupo del clon pandémico y claramente diferenciadas del grupo dominante en los casos clínicos en Perú perteneciente al serotipo O4:K8. La cepa (Guillén-136) aislada en Lima fue identificada como perteneciente al clon pandémico. La mayoría de los aislamientos del brote Perú 2009 fueron asignadas al serotipo O3:K59, a excepción de dos aislamientos cuyo grupo K no pudo ser determinado y fueron incluidas dentro del serotipo O3:KUT.
La presencia de la región específica del gen toxR característica del clon pandémico fue analizada en todas las cepas por la técnica denominada GS-PCR. Ninguna de las cepas del brote presentó este marcador del clon pandémico. Otras dos cepas (437-07 y 155-05) presentaron el gen que codifica para el factor de virulencia trh; siendo ausente además en la cepa (155-05) el factor TDH presente en todas las otras cepas (16).
La investigación epidemiológica de este brote ha puesto de relieve el carácter emergente del serotipo 03:K59 entre los casos clínicos de V. parahaemolyticus en Perú. La detección de este serotipo en Perú es coincidente con la descripción de casos asociados a este serotipo en algunos países de Sudamérica en los últimos años (17), lo que podría significar la aparición de un nuevo clon patógeno de este microorganismo en la zona con una notable capacidad de expansión.
CASO DE Vibrio cholerae INABA NO TOXIGÉNICO
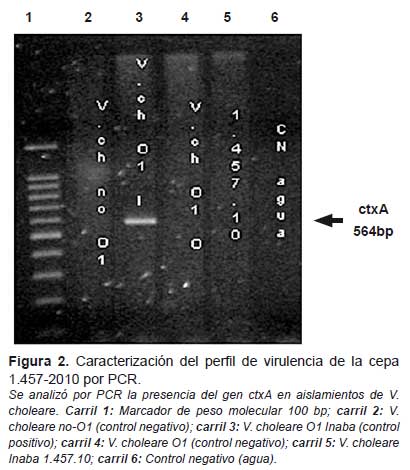
En mayo de 2010 la Unidad de Investigación Médica N.° 6 de la Marina de los Estados Unidos con sede en Perú (NAMRU-6) remite al INS un aislamiento de Vibrio cholerae el cual fue codificado como 1.457-2010 siendo una cepa de Vibrio cholerae serogrupo O1 serotipo Inaba. Las cepas patogénicas de Vibrio cholerae generalmente tiene en su genoma el gen de toxina colerica ctxA, que puede ser detectado por técnicas de biología molecular como la PCR, por lo tanto, esta técnica es muy útil en la identificación de cepas toxigénicas. La cepa no presentó el gen ctxA que codifica la toxina colérica, por lo cual no es productora de la toxina del cólera, responsable de la enfermedad del cólera (Figura 2).
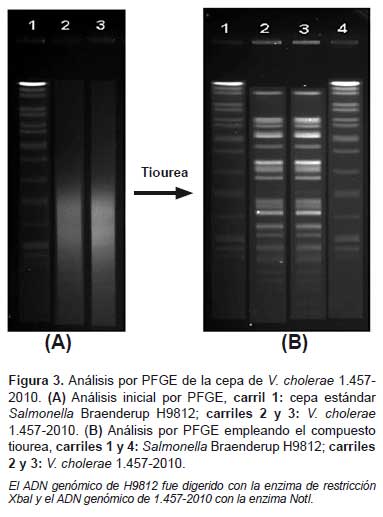
El análisis inicial de tipificación por PFGE de la cepa 1.457-2010 en el INS no presentó ningún perfil genético, obteniéndose únicamente degradación del ADN genómico hasta en tres análisis consecutivos; se decidió utilizar el compuesto tiourea, que es conocido por mejorar el análisis por PFGE de muchas cepas no tipificables por esta técnica (POE-PFGE, Tiourea), de donde se obtuvo una visible mejora del perfil genético (Figura 3). Para estas pruebas se aplicó los protocolos estandarizados de la Red PulseNet Internacional (18).
Se envió el archivo de la imagen del gel para ser analizado por el administrador de la BDR de Vibrio cholerae de la Red PN AL y C en el Instituto Nacional de Enfermedades Infecciosas- ANLIS Carlos G. Malbrán de Argentina. El análisis y la comparación del perfil PFGE del aislamiento de Perú con los patrones correspondientes a aislamientos de Vibrio cholerae de América Latina evidenció que se trataba de una cepa con un perfil genético nuevo y no presentó relación genética con los aislamientos de Vibrio cholerae O1 asociados a los casos de cólera durante los brotes epidémicos, ni con los aislamientos más recientes de V. choleare toxigénico de Argentina (2005) y Paraguay (2009). Tampoco se observó relación entre dicho aislamiento y otros del serogrupo O1 no portadores del gen ctxA de Argentina (Figura 4).

La cepa 1.457-2010 fue incluida en un grupo juntamente con los aislamientos de serogrupo no-O1/no-O139 de Argentina, aunque se identificaron más de siete bandas diferentes entre los perfiles electroforéticos, por lo que se considera que los aislamientos correspondientes no están relacionados genéticamente.
DISEMINACIÓN CLONAL DE Salmonella entérica serovar Infantis EN PERÚ
En el año 2010, a través de la vigilancia bacteriológica con la Red Nacional de Laboratorios en el Perú, se detectó un inusual aumento de casos de Salmonella en aislamientos de origen humano, en su mayoría pediátricos de diversos hospitales de Lima y aislamientos de alimentos. El INS identificó 33 aislamientos como Salmonella enterica serovar Infantis, 24 de casos clínicos y nueve de alimentos. Es la tercera serovariedad más frecuente en el Perú. Esta infección está asociada con el consumo de huevos contaminados y a productos cárnicos avícolas.
El INS realizó el análisis por PFGE con la enzima XbaI aplicando el protocolo estandarizado de la Red Internacional PulseNet (20) en siete aislamientos, cinco de casos clínicos provenientes de diferentes establecimientos de salud de Lima y dos aislamientos de carnes de aves evidenciando en los resultados que existe fuerte relación genética entre los aislamientos de Salmonella enterica serovar Infantis obtenidos de la vigilancia en el Perú (Figura 5).
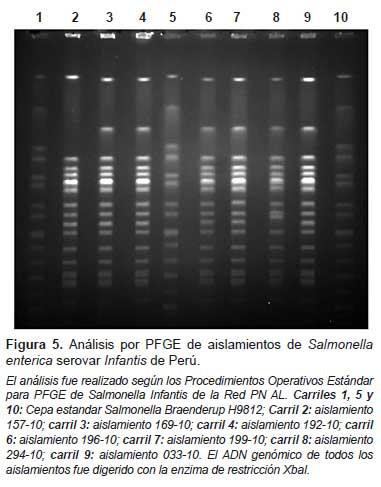
Se encontró que los dos aislamientos provenientes de productos cárnicos avícolas (192-10 y 196-10) presentaron un mismo patrón genético que el aislamiento de hemocultivo, evidenciando que se trataba de una misma cepa presente en tres diferentes distritos de la ciudad de Lima. Además estos tres aislamientos presentaron un mismo patrón de resistencia antimicrobiana, siendo resistentes al ácido nalidíxico, ciprofloxacina, tetraciclina, cotrimoxazol y nitrofurantoína. Así mismo, los aislamientos 033-10 y 294-10 tuvieron el mismo patrón genético pero presentaron diferente perfil de resistencia antimicrobiana (Figura 5).
También se observó relación genética cercana entre los aislamientos de Perú con respecto a aislamientos circulantes en Argentina; según el análisis realizado por comparación de subtipos de la BDR de Salmonella, Red PulseNet AL y C. En particular uno de los aislamientos de Perú (157-2010) estuvo más relacionado con aislamientos argentinos de Salmonella enterica serovar Infantis que con los de Perú (Figura 6).
Estos resultados demuestran que en general tanto en Perú como en Argentina existe poca diversidad genética entre los aislamientos clínicos de Salmonella enterica serovar Infantis que circulan en estos países. Es necesario estudiar un número mayor de brotes a fin de confirmar esta hipótesis, como también para investigar posibles nexos en las fuentes de infección presentes en ambos países.
CONCLUSIONES
El método PFGE es útil en la vigilancia epidemiológica de diferentes agentes patógenos transmitidos por alimentos y facilita la investigación de brotes en un país así como en diferentes países integrados a una Red Regional de Subtipificación PulseNet. Así mismo, permite reconocer aislamientos relacionados que se derivan de la expansión clonal (20,21).
La aplicación de la técnica de PFGE en la investigación epidemiológica permite rastrear la dispersión geográfica de las poblaciones causantes de enfermedades a un nivel local y supranacional, así como identificar las fuentes ambientales implicadas en la aparición de casos. Este conocimiento es básico para la intervención en caso de aparición de brotes de forma que se pueda conseguir contener las infecciones.
La Red PulseNet en América Latina y el Caribe permite la consolidación de la colaboración entre los laboratorios referenciales de cada país entre sí, esa acción articulada permite conocer en la región de América Latina la circulación de cepas genéticamente semejantes, detectar brotes y tomar acciones inmediatas de control y prevención.
AGRADECIMIENTOS
A Enrique Pérez, Norma Binsztein y Mariana Pichel coordinadores de la Red PulseNet ALyC al grupo administrador de la Base de Datos Regional de la red por el dendograma del caso Vibrio cholerae Inaba no toxigénico. A los trabajadores de los laboratorios de los hospitales San Bartolomé, Emergencias Pediátricas, Instituto Nacional de Enfermedades Neoplásicas, San José Villa El Salvador (Lima Sur), de los laboratorios de referencia regional de Lambayeque, Cajamarca y Piura, a la Dirección General de Salud Ambiental (DIGESA), Clínica San Borja y el NAMRU-6 por el envío de aislamientos para confirmación y tipificación. A la Universidad Santiago de Compostela por el financiamiento del Proyecto Vibrio Perú. Finalmente, a Susana Díaz, Oscar Escalante y Alfredo Guillén por su apoyo a la realización del presente artículo.
Conflictos de Interés
Los autores declaran no tener conflictos de interés en la publicación del presente artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. Kooper G, Calderon G, Schneider S, Dominguez W, Gutierrez G. Enfermedades transmitidas por alimentos y su impacto económico. Estudio de caso en Costa Rica, El Salvador, Guatemala, Honduras y Nicaragua. Roma: FAO; 2009. [ Links ]
2. Flint JA, Van Duynhoven YT, Angulo FJ, DeLong SM, Braun P, Kirk M, et al. Estimating the burden of acute gastroenteritis, foodborne disease, and pathogens commonly transmitted by food: a international review. Clin Infect Dis. 2005;41(5):698-704. [ Links ]
3. Newell DG, Koopmans M, Verhoef L, Duizer E, Aidara- Kane A, Sprong H, et al. Food-borne diseases – the challenges of 20 years ago still persist while new ones continue to emerge. Int J Food Microbiol. 2010;139(Suppl 1):S3-15. [ Links ]
4. Pires SM, Evers EG, van Pelt W, Ayers T, Scallan E, Angulo FJ, et al. Attributing the human disease burden of foodborne infections to specific sources. Foodborne Pathog Dis. 2009;6(4):417-24. [ Links ]
5. Buzby JC, Roberts T. The economics of enteric infections: human foodborne disease costs. Gastroenterology. 2009;136(6):1851-62. [ Links ]
6. Mead PS, Slutsker L, Dietz V, McCaig LF, Breese JS, Shapiro C, et al. Food-related illness and death in the United States. Emerg Infect Dis. 1999;5(5):607-25. [ Links ]
7. Miranda M, Aramburu A, Junco J, Campos M. Situación de la calidad de agua para consumo en hogares de niños menores de cinco años en Perú, 2007-2010. Rev Peru Med Exp Salud Publica. 2010;27(4):506-11. [ Links ]
8. Pérez-Cordón G, Rosales MJ, Valdez RA, Vargas- Vásquez F, Cordova O. Detección de parásitos intestinales en agua y alimentos de Trujillo, Perú. Rev Peru Med Exp Salud Publica. 2008;25(1):144-8. [ Links ]
9. Quispe JJ, Sánchez V. Evaluación microbiológica y sanitaria de puestos de venta ambulatoria de alimentos del distrito de Comas, Lima – Perú. Rev Peru Med Exp Salud Publica. 2001;18(1-2):27-32. [ Links ]
10. Goering RV. Pulsed field gel electrophoresis: a review of application and interpretation in the molecular epidemiology of infectious disease. Infect Genet Evol. 2010;10(7):866-75. [ Links ]
11. Vernile A, Giammanco G, Massa S. PFGE: importance in food quality. Recent Pat Food Nutr Agric. 2009;1(3):248-51. [ Links ]
12. Hyytia-Trees EK, Cooper K, Ribot EM, Gerner-Smidt P. Recents developments and future prospects in subtyping of foodborne bacterial pathogens. Future Microbiol. 2007;2(2):175-85. [ Links ]
13. Zamudio ML, Arias I, Luna MA, Valenzuela A, Segovia E, Villanueva E. Vigilancia de enfermedades transmitidas por alimentos en el Perú. Bol Inst Nac Salud (Perú). 2008;14(5- 6):103-4. [ Links ]
14. Martinez-Urtaza J, Huapaya B, Gavilan RG, Blanco- Abad V, Ansede-Bermejo J, Cadarso-Suarez C, et al. Emergence of asiatic Vibrio diseases in South America in phase with El Niño. Epidemiology. 2008;19(6): 829-37. [ Links ]
15. Martinez-Urtaza J, Lozano-León A, De Paola A, Ishibashi M, Shimada K, Nishibuchi M, et al. Characterization of pathogenic Vibrio parahaemolyticus isolates from clinical sources in Spain and comparison with Asian and North American pandemic isolates. J Clin Microbiol. 2004;42(10):4672-8. [ Links ]
16. Okuda J, Ishibashi M, Abobott SL, Janda JM, Nishibuchi M. Analysis of the termoestable direct hemolysin (tdh) gene and the tdh-related hemolysin (trh) genes in ureasepositive strains of Vibrio parahaemolyticus isolated on the West Coast of the United States. J Clin Microbiol. 1997;35(8):1965-71. [ Links ]
17. Garcia K, Torres R, Uribe P, Hernandez C, Rioseco ML, Romero J, et al. Dynamics of clinical and environmental Vibrio parahaemolyticus strains during seafood – related summer diarrhea outbreaks in southern Chile. Appl Environ Microbiol. 2009;75(23):7482-7. [ Links ]
18. Cooper KL, Luey CK, Bird M, Terajima J, Nair GB, Kam KM, et al. Development and validation of a PulseNet standardized pulsed-field gel electrophoresis protocol for subtyping of Vibrio cholerae. Foodborne Pathog Dis. 2006;3(1):51-8. [ Links ]
19. Ribot EM, Fair MA, Gautom R, Cameron DN, Hunter SB, Swaminathan B, et al. Standardization of pulsed-field gel electrophoresis protocols for the subtyping of Escherichia coli O157:H7, Salmonella, and Shigella for PulseNet. Foodborne Pathog Dis. 2006;3(1):59-67. [ Links ]
20. Centers for Disease Control and Prevention. Standardized molecular subtyping of foodborne bacterial pathogens by pulsed-field gel electrophoresis: training manual. Atlanta: CDC; 2000. [ Links ]
21. Swaminathan B, Gerner-Smidt P, Ng LK, Lukinmaa S, Kam KM, Rolando S, et al. Building PulseNet International: an interconnected system of laboratory networks to facilitate timely public health recognition and response to foodborne disease outbreaks and emerging foodborne diseases. Foodborne Pathog Dis. 2006;3(1):36-50. [ Links ]
Correspondencia: María Luz Zamudio Rojas.
Dirección: Laboratorio de Referencia Nacional de Entreropatógenos, Centro Nacional de Salud Pública, Instituto Nacional de Salud, Lima, Perú.
Cápac Yupanqui 1400 Jesús María, Lima, Perú.
Teléfono: (511) 617-6200; Anexo 2117.
Correo electrónico: mzamudio@ins.gob.pe
Recibido: 15-02-11
Aprobado: 09-03-11