Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista Peruana de Biología
On-line version ISSN 1727-9933
Rev. peru biol. vol.12 no.1 Lima Jan./jul. 2005
Mariela Romero-Carpio y Rolando Estrada-Jiménez1
1Laboratorio de Recursos Genéticos y Biotecnología. Facultad de Ciencias Biológicas. UNMSM.
Presentado: 21/04/2005
Aceptado: 02/08/2005
Resumen
A partir de la expresión diferencial de ARNm de plántulas in vitro de dos accesiones de Ullucus tuberosus Loz. «olluco» altamente tolerantes a estrés osmótico, fueron seleccionados 31 fragmentos diferenciales de ADNc relacionados con tolerancia a sequía.
Palabras clave: Estrés osmótico, tuberosa andina, despliegue diferencial, técnicas moleculares, expresión diferencial.
Abstract
Thirty-one differential fragments of cDNA related to drought tolerance have been selected from the mRNA differential expression of in vitro plantelets belonging to two accessions of Ullucus tuberosus Loz. «olluco» highly tolerant to osmotic stress.
Keywords: osmotic stress, Andean tuber, differential display, molecular techniques, differential expression.
Introducción
Aunque el agua es el compuesto más abundante de nuestro planeta, su escasez en el suelo es lo que más afecta la producción agrícola a nivel mundial. Aproximadamente la mitad de las comunidades vegetales terrestres sufren regularmente extensos períodos de déficit hídrico (Schulze et al., 1987), constituyéndose como la principal causa de pérdidas en el rendimiento de los cultivos económicamente importantes, deterioro de pastos y muerte de ganado. Incluso, por la gravedad de las consecuencias que origina, sobrepasa a las provocadas por otros factores bióticos y abióticos (Kramer, 1980). Es así que para incrementar la productividad vegetal se investiga el desarrollo de plantas mejoradas genéticamente, con mayor tolerancia al estrés hídrico; para ello es necesario seleccionar genes de especies con una historia evolutiva de alta tolerancia a factores abióticos adversos como es el caso de «olluco». Ullucus tuberosus es una Basellaceae andina, que al igual que Cactaceae, Chenopodiaceae y Aizoaceae pertenece a los Caryophyllales, orden que incluye a especies productoras de compuestos que, le confieren a la planta la tolerancia al estrés hídrico (Bohnert, 1998).
Inicialmente el estudio fisiológico se basó en análisis morfológicos, comparando plantas estresadas con sus respectivos controles, pero al observarse vacíos de información se crearon técnicas bioquímicas y moleculares que permiten explicar este complejo mecanismo.
El presente trabajo reporta la identificación de fragmentos diferenciales de ADNc relacionados con tolerancia al estrés hídrico, como paso previo a la determinación de genes involucrados en tolerancia a sequías, lo que conllevaría a incrementar la información existente en las bases de datos genómicos e ir entendiendo mejor las vías de transducción de señales que permitan la inducción de estos genes en especies andinas.
Material y métodos
Material vegetal
Las accesiones de Ullucus tuberosus «olluco» empleadas, pertenecen al Banco de Germoplasma de Raíces y Tuberosas Andinas del Laboratorio de Recursos Genéticos y Biotecnología (LRGB) de la Facultad de Ciencias Biológicas de la Universidad Nacional Mayor de San Marcos. Se seleccionaron tres ingresos tolerantes al estrés osmótico: UH-041, UH-423 y UH-335 (datos aún no publicados provenientes de un estudio realizado en el LRGB) y uno: UH-329 susceptible.
Propagación clonal
Con la finalidad de contar con un número adecuado de plántulas para iniciar el experimento, mediante el cultivo in vitro de nudos, se propagaron las accesiones en medio nutritivo estandarizado para olluco (Estrada et al., 1986); se los incubó bajo agitación continua (60 rpm); a 18+2 °C, 16 horas luz, y 23 mEm-2s.
Inducción de estrés hídrico
Después de 15 días de desarrollo, las plántulas fueron separadas en dos grupos: uno, que sería estresado a: 0 horas, 4 horas, 1 día, 3 días, 5 días, 10 días y 15 días, reemplazando al medio de propagación de olluco por 25 mL de la solución estresante -Medio Murashige y Skoog (MS) más Polietilenglicol (PEG) 8000 al 20%, mientras que el otro, sirvió de control para cada uno de los tiempos establecidos.
Extracción de ARN total
La extracción de ARN total de plántulas completas se realizó tomando como base el protocolo de Schuler y Zielinski (1989). El producto se resuspendió en 25 mL de agua tetra-destilada y se mantuvo a 15 °C.
Análisis de la calidad de ARN
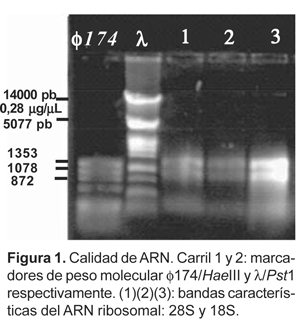
Se preparó un gel de agarosa 0,7%, empleando buffer de corrida TPE al 1X y 0,7 mL de bromuro de etidio (10 mg/mL).
Todas las corridas electroforéticas se realizaron a 80 V, voltaje constante, por un lapso de 40 minutos a temperatura ambiente, colocando como marcador referencial de peso molecular, el ADN del fago f174 digerido con Haemophilus aegypticus III (Hae III) y/o ADN del fago l digerido con la enzima PstI.
Al cabo de la corrida, con el empleo de un transiluminador se evidenciaron las bandas características del ARN total, imagen captada por una cámara fotográfica digital conectada por una interfase a una computadora, en donde con ayuda del software ScionImage, se pudieron editar y almacenar.
Despliegue diferencial (DDRT-PCR)
Esta técnica se trabajó según lo reportado por Liang y Pardee (1997) y Verástegui (1999). Los primers empleados: oligonucleótidos-dT: 1143 (dT11CT), 1144(dT11CG), 1145 (dT11CC), y cuatro decámeros: 1137 (5´-CTGATCCATT-3`, 1138 (5`-CTTGATTGCC-3´, 1139 (5´GGTACTAAGG-3´) y 1140 (5`TACCTAAGCG-3`) fueron preparados en el Departamento de Ingeniería Genética del Centro de Investigación y de Estudios Avanzados del Instituto Politécnico Nacional en Irapuato, Guanajuato, México.
El DDRT-PCR involucra la transcripción reversa del ARN total seguido por una amplificación PCR y la visualización de los ADNc amplificados en geles de acrilamida marcados/teñidos con aP32, aS35, y AgNO3 (se realizó empleando el protocolo de tinción con nitrato de plata de Promega) y en geles de agarosa con bromuro de etidio (10mg/mL).
En los geles resultantes, la idea era distinguir los ADNc de las plantas sometidas a estrés, para ello se compararon las bandas de los carriles en donde se sembró la muestra correspondiente a la plántula estresada y la plántula control. Más aún, para aumentar el grado de confiabilidad de la técnica, se optó por cargar las muestras de acuerdo a la siguiente secuencia:
(1) Muestra control sin transcriptasa reversa (TR).
(2) Muestra control con TR.
(3) Muestra estresada con PEG 8000 al 20% con TR.
(4) Muestra estresada con PEG 8000 al 20% sin TR.
De modo que el carril (1) sirva de control del carril (2), indicándonos si se amplificaron contaminantes de ADN provenientes de la extracción de ARN; de forma análoga a lo que el carril (4) representa para el (3) y así, comparar sólo los fragmentos diferenciales encontrados en los carriles (2) y (3) correspondientes a los ADNc de plántulas sin estresar y estresadas respectivamente.
Selección de fragmentos diferenciales
Sólo se seleccionaron aquellos fragmentos diferenciales que estaban presentes en las muestras estresadas y ausentes en el control, es decir, sólo aquellas que se expresaron por efecto de la inducción de estrés hídrico. Los fragmentos fueron aislados del gel con ayuda de una navaja, almacenados en tubos eppendorf debidamente rotulados, y guardados a 15 ºC.
Resultados
Inducción del estrés
Sólo las plántulas de las accesiones UH-423 y UH-335 sobrevivieron en un 100% al estrés impuesto durante quince días.
Extracción de ARN total
El abundante mucílago que presentaron algunos genotipos de olluco fue eliminado con la aplicación de acetato de potasio después de la precipitación del ARN total. Al cuantificarse en geles de agarosa se observaron 2 bandas correspondientes al ARN ribosómico (28S y 18S) (Fig. 1).
Identificación de fragmentos diferenciales de ADNc.
De los geles de agarosa al 0,4% teñidos con bromuro de etidio, se seleccionaron 4 bandas, aunque éstas fueron bastante tenues a pesar de la alta concentracion de la muestra; de los geles de acrilamida marcados con aP32 se aislaron 9 bandas, marcados con aS35 15 y teñidos con AgNO3 tres.
La mayor sensibilidad de detección y resolución de bandas, definitivamente se observó en los casos que se emplearon radioisótopos seguido por nitrato de plata y luego bromuro de etidio (Romero, 2001).
Discusión
Desde su invención en 1992, la técnica de Despliegue Diferencial, ha sido ampliamente usada para clonar genes expresados diferencialmente. De hecho, 1215 de las 1810 publicaciones reportadas en Medline hasta 1999, emplearon dicha técnica, a pesar de la existencia de otras como Hibridación sustractiva, fingerprinting de ARN con una PCR cebada arbitrariamente (RAP-PCR), ADNc-AFLP´s, Análisis de Diferencias Representativas y Análisis Seriales de la Expresión Diferencial (SAGE). En la actualidad, una técnica que ha acelerado enormemente la ubicación de ADNc es la técnica de Hibridación de Microarreglos (microarrays), pero el costo de adquisición de los equipos necesarios hace inaccesible su uso en países en vías de desarrollo.
Estandarización de la técnica
a) Extracción de ARN total
Para realizar el Despliegue diferencial, inicialmente, se debe contar con ARN de buena calidad, para ello, se extrajo ARN total y no ARNm porque no es lo recomendable para DDRT-PCR. El ARNm puede aislarse del ARN total por métodos tradicionales (Liang y Pardee, 1995), pero la probabilidad de que la cola poliadenilada de ARNm se contamine o se degrade es muy alta y es difícil de confirmarlo en geles de agarosa (Colonna-Romanno et al., 1998).
Para extraer ARN total, se empleó el protocolo de Schuler y Zielinsky, metodología basada en la extracción con fenol, adecuado a las condiciones del LRGB. Las adaptaciones estuvieron referidas a:
1. Utilización de pequeñas cantidades, ya que para la técnica de Despliegue Diferencial se requieren sólo algunos nanogramos de RNA (Liang y Pardee, 1992).
2. Para la eliminación de residuos de ARNasas, todo el material a ser utilizado, incluida el agua destilada, fueron esterilizados por 15 min. y manipulados con guantes, en lugar de utilizar DEPC, usualmente empleado para eliminar cualquier residuo enzimático. (Sambrook et al., 1989).
La calidad del ARN extraído con estas modificaciones del protocolo fue verificada en geles de agarosa 0,7%, encontrándose dos bandas correspondientes a las moléculas que sintetizan las subunidades 28S y 18S del ARN ribosómico 45 S, que alcanza el 75% del ARN total a diferencia del ARNm que sólo constituye del 1-5%.
Finalmente, el ARN mantenido a 15 ºC se conservó en buen estado incluso 6 meses después de haberla extraído, comprobándose la inexistencia de ARNasas endógenas (degradadas con fenol desde el primer momento de la extracción de ARN).
En la extracción de ARN total se emplearon a plántulas completas de olluco para evaluar la respuesta de este organismo ante las condiciones de sequía, ya que in vivo, las propiedades que determinan el comportamiento del agua a nivel celular y que se encargan de las respuestas metabólicas frente a deficiencias hídricas, no se restringen a un determinado órgano, sino que se dan en toda la planta (Hanson y Hitz, 1982).
b) Separación de fragmentos de ADNc
Luego de realizar la transcripción reversa de los ARN mensajeros, se evaluó la separación de productos de la PCR en geles de agarosa teñido con bromuro de etidio (Sokolow y Prockop, 1994), constituyendo una modificación rápida y sencilla, pero por su baja resolución para identificar ADNc se recurrió al empleo de geles denaturantes de poliacrilamida al 6% teñidos con AgNO3 y a geles marcados con radioisótopos, obteniéndose mejores resultados con éstos últimos.
Análisis de la expresión diferencial de olluco ante el estrés hídrico
La sola disminución del potencial hídrico en 0,1 MPa, produce una disminución perceptible en el crecimiento celular de muchas plantas (Hsiaio, 1973). Por ello, no es raro esperar que ciertos clones de la especie U. tuberosus, que como promedio se muestran tolerantes a sequía (Calzada, 1954), presenten susceptibilidad a 1,5 MPa, coincidente con el punto de marchitez permanente. Cuando una planta se somete por primera vez a un factor de estrés, el organismo crea una reacción de alarma, por lo cual algunas de sus funciones metabólicas se alteran, provocando una respuesta (Larcher, 1995). Posteriormente vendrá la fase de resistencia en la cual la planta se adaptará al factor que ocasiona el estrés, retornando a la normalidad. Si este factor de estrés continúa por un largo período, la planta llegará a un estado de agotamiento, en el cual su comportamiento puede modificarse nuevamente o se producirá la muerte.
Eso precisamente es lo que se observó al estresar plántulas de olluco; las accesiones UH-335, UH-423, y UH-041 toleran las condiciones impuestas, sus actividades fisiológicas y moleculares están en plena actividad, pero no ocurre lo mismo con la accesión UE-329, que ya al tercer día presentó una visible deformación biológica de tipo plástica.
Además es lógico que exista variabilidad en la respuesta de diferentes individuos de una especie a determinado factor estresante puesto que no todos recibieron la misma presión de selección en su evolución y eso sirve para mantener la variabilidad y con ello la supervivencia de la especie.
Al analizar la expresión diferencial se observó una alteración en el patrón de bandas, algunas veces reguladas positiva- y otras negativamente, pero nunca un cambio completo, tal como lo reporta Bray (1989).
Dependiendo de la combinación de cebadores, el despliegue diferencial de bandas se observa incluso a partir de pocos minutos de someter la planta bajo estrés, tiempo suficiente para que el olluco reconozca su estado y empiece a sintetizar ARNm.
Por ejemplo, en estudios realizados en proteínas sintetizadas por causa del estrés hídrico reportado en semillas (Misra y Bewley, 1986), raíces (Damerval et al., 1988), hojas (Bray, 1988), y yemas (Lalonde y Bewley, 1986) de diferentes especies, incluidos: maíz, frijol, cebada, soya, arroz, tomate, pino y sorgo; se observó acumulación de proteínas de novo, así como la acumulación y la reducción de muchas proteínas existentes. Y esta variación en la síntesis de ARNm puede ser muy rápida, después de producirse el estrés en las hojas de tomate (Bray, 1988).
El presente trabajo, que constituye el primer trabajo a nivel molecular para el estudio de estrés hídrico en plantas nativas en nuestro país, demuestra que la técnica de Despliegue Diferencial (DDRT-PCR) con las adaptaciones realizadas es eficiente para seleccionar ADNc diferenciales de individuos sometidos a estrés hídrico, el uso de radiactividad es el método de marcaje de ADNc con mejor resolución y finalmente quedó estandarizada la extracción de ARN total de «olluco» sin el empleo de DEPC ni ADNasas.
Agradecimientos
(*) Este trabajo formó parte de la tesis para optar el Título Profesional de Biólogo de Mariela Romero. Contó con el financiamiento de la Fundación McKnight. Proy. The Andean Root and Tuber Crops: Realizing the promise of forgotten Foods (Perú-USA).
A Milusqui Verástegui por su valiosa asesoría en la investigación, al Dr. Marc Ghislain y Mg. Carmen Herrera del Centro Internacional de la Papa por brindarnos todas las facilidades en el trabajo con radiactividad y nitrato de plata.
Literatura citada
Bohnert H. 1995. Adaptations to Environmental Stresses. The Plant Cell, Vol. 7, 1099-1111. American Society of Plant Physiologists. [ Links ]
Bray E.A. 1988. gene expression during environmental stress and its regulation by abscisic acid. Quarterly. 17: 113-126. The Plant Growth Regulator Society of America.
Bray E.A. 1988. Drought and ABA induced changes in polipeptide and mRNA accumulation in tomato leaves. Plant Physiology. 86:1210-1214.
Colonna-Romano S., A. Leonte, B. Maresca. 1998. Differential Display Reverse Transcription PCR (DDRT-PCR). Edit Springer-Verlag. Berlin-Alemania. Pp 108.
Damerval C., N. Vartanian & De Vienne. 1988. Differential two-dimmnensional protein patterns as related to tissue specificity and water conditions in Brassica napus var oleifera root system. Plant Physiol. 86:1394-1309.
Hanson A.D. & W.D. Hitz. 1982. Metabolic responses of mesophytes to plant water deficits. Ann. Rev. Plant Physiol. 33:163-203.
Hsiao T.C. 1973. Plant response to water stress. Annual Review of Plant Physiology. 24: 519-570.
Kramer P.J. 1980. Drought stress and the origien of adaptations». EN: Water Relations of Plants Academic Press, Nueva York. 390-415.
Lalonde L & J. Bewley. 1986. Desiccation during germination of pea axes and its effects on the messenger RNA population. J. Exp. Bot. 37:754-764.
Liang P. & A.B. Pardee. 1992: Differential display of eukariotic messenger RNA by means of the polymerase chain reaction. Science. 257: 967-971.
Estrada R., W. Manya, C. Pulache, H. Sánchez, & T. Yonamine. 1986. Maintenance, micropropagation and seed production of the andean tuber crops: Oca, olluco and mashua. Abstracts of the International Congress of Plant Tissue and Cell Culture. University of Minnesota, Minneapolis, USA. August 3-8.
Misra S., & J. Bewley. 1986. Desiccation of Phaseolus vulgaris seeds during and following germination, and its effect upon the traslatable mRNA population of the seed axes. J. Exp. Bot. 1767:362-374.
Romero M. 2001. Selección de fragmentos diferenciales de cDNAs relacionados con estrés hídrico en Ullucus tuberosus «olluco». Tesis, título profesional de Bióloga con mención en Biología Celular y Genética. Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos, Lima, Perú.
Sambrook J, E.F. Fritsch, & T. Maniatis.1989. Molecular cloning. 2° ed. Cold Spring Harbor Laboratory Press. EE.UU.
Schuler M.A. & R.E. Zielinski. 1989. RNA isolation from light- and dark-growth seed lings. En: Methods in Plant Molecular Biology. Academic Press. San Diego. California. 88-95.
Schulze E.D., R. Robichaux, J. Grace, P.W. Rundel, & J.R. Ehleringer. 1987. Plant water balance. BioScience 37:30-37.
Sokolow B.P. & D.J. Prockop. 1994. A rapid and simple PCR-based method for isolation of cDNAs from differentially expressed genes. Nucleic Acids Res 22:4009-4015.
Verástegui, M. 1999. Expresión diferencial de mRNAs de células de «chile» (Capsicum annum L.) tolerantes a polietilenglicol (PEG). Tesis para obtener el grado de Maestra en Ciencias con especialidad en Biotecnología de Plantas. Centro de Investigación y de Estudios Avanzados del Instituto Politécnico Nacional. Unidad de Biotecnología e Ingeniería Genética de Plantas. Irapuato-México.
Correspondencia
1Laboratorio de Recursos Genéticos y Biotecnología. Facultad de Ciencias Biológicas. UNMSM.
Email: Rolando Estrada: restradaj@unmsm.edu.pe
Email: Mariela Romero: mromero_01@hotmail.com













