INTRODUCCIÓN
En diciembre del 2019, China reportó la presencia de varios casos de neumonía atípica en la ciudad de Wuhan 1 que ocasionaba la muerte en pocos días. El agente etiológico fue identificado como un nuevo coronavirus que se denominó SARS-CoV-2 y a la enfermedad se le llamó COVID-19 (del inglés, Coronavirus Disease 2019). Para enero del 2020, y a pesar de las medidas de contención adoptadas por el gobierno chino, se reportaron más casos en los países fronterizos y en Europa. El incremento de casos de COVID-19 fue tan rápido que la Organización Mundial de la Salud (OMS) declaro a la enfermedad como una pandemia e instó a los países a adoptar medidas de control para evitar su diseminación 2. Para fines de febrero del 2020, la enfermedad ya había llegado a América. A inicios de marzo del 2020, en Perú se reportó el primer caso de COVID-19, y a la semana siguiente el gobierno adopto medidas de control cerrando las fronteras y declarando cuarentena a nivel nacional.
Una de las estrategias esenciales para controlar la enfermedad es la identificación rápida del virus SARS-CoV-2. La implementación de las pruebas moleculares en el Perú estuvo a cargo del Laboratorio de Referencia Nacional de Virus Respiratorio del Instituto Nacional de Salud (LRNVR-INS) siguiendo recomendaciones de la Organización Panamericana de Salud (OPS) y de la OMS. Estas recomendaciones, para el diagnóstico molecular, se basaban en el protocolo de Corman et al 3. Este protocolo describe el uso de cebadores y sondas para la detección de los genes E y RdRP utilizando la técnica de RT-PCR en tiempo real en dos etapas: primero la amplificación del gen E y las muestras que resulten positivas serian confirmadas en una segunda reacción amplificando el gen RdRP. Con el propósito de mejorar el sistema de detección para hacerlo más rápido y sencillo sin perder la sensibilidad y especificidad se planteó estandarizar un protocolo RT-PCR en tiempo real en formato duplex. El presente estudio tuvo como objetivo validar y evaluar un RT-PCR en tiempo real dúplex, el cual consiste en amplificar de manera simultánea (en un solo tubo) el marcador diagnóstico RdRP y un control interno endógeno GAPDH humano. Además, se presenta la evaluación de su utilidad en la detección del virus SARS-CoV-2 en muestras clínicas.
MENSAJES CLAVE
Motivación para realizar el estudio: Perú fue uno de los países más afectado por la pandemia de la COVID-19, registrándose un gran número de casos y fallecidos.
Principales hallazgos: esta prueba estandarizada es una buena alternativa para el diagnóstico de COVID-19; además, la prueba fue aplicada de manera exitosa en personas sospechosas de dicha enfermedad, permitiéndose controlar el número de falsos negativos.
Implicancias: el diagnóstico precoz y exitoso de la COVID-19 ayudará a implementar medidas de control para disminuir la velocidad de transmisión del virus SARS-CoV-2 en el Perú.
EL ESTUDIO
Se realizó un estudio de validación y evaluación de una prueba diagnóstica.
Muestras biológicas
Las muestras utilizadas en este estudio son de hisopado nasal y faríngeo (HNF) obtenidas de la viroteca del LRNVR-INS, obtenidas de pacientes sintomáticos y sospechosos de tener la COVID-19 en el periodo de marzo del 2020 a junio del 2021.
Desarrollo del ensayo
El ARN viral fue purificado usando el kit QIAamp viral RNA Mini (QIAGEN, Hilden, Alemania) siguiendo las instrucciones del fabricante. El ensayo RT-PCR real time dúplex se realizó usando los cebadores y sondas reportados (Tabla 1). El volumen final de la reacción fue de 20 µL, este contenía el Mix QPCR Probe 4x (BiotechRabbit), enzima RTase, agua de PCR, cebadores (RdRP_F, RdRP_R, GAPDH_F y GAPDH_R) y sondas (RdRp_P2, GAPDH_P). Los tubos de reacción fueron colocados en un termociclador en tiempo real RotorGene Q (QIAGEN, Alemania) y se utilizó un ciclaje de 50 °C por 10 minutos para la transcripción reversa, 95 °C por 3 minutos de denaturación inicial seguido de 45 ciclos de 95 °C por 10 segundos y 58 °C por 30 segundos (con adquisición para FAM y ROX), terminando con 40 °C por 30 segundos. Los resultados positivos se definieron con valores de Ct (Cycle Threshold) < 37 para el gen RdRP (FAM) y Ct < 40 para el gen GAPDH (ROX).
Tabla 1 Secuencia de cebadores y sondas del RT-qPCR dúplex.
| GEN | Cebadores y sondas | Secuencia 5´->3´ | Concentración por reacción | Volumen por reacción |
|---|---|---|---|---|
| RdRP | RdRp_F | GTGARATGGTCATGTGTGGCGG | 0,4 µM | 0,8 µL |
| RdRp_P2 | FAM-CAGGTGGAACCTCATCAGGAGATGC-BBQ | 0,4 µM | 0,8 µL | |
| RdRp_R | CARATGTTAAASACACTATTAGCATA | 0,2 µM | 0,4 µL | |
| GAPDH | GAPDH_F | GTGAAGGTCGGAGTCAACGG | 0,4 µM | 0,8 µL |
| GAPDH_R | TCAATGAAGGGGTCATTGATGG | 0,4 µM | 0,8 µL | |
| GAPDH_P | ROX - CGCCTGGTCACCAGGGCTGC- BHQ2 | 0,2 µM | 0,4 µL |
Validación del ensayo
Para evaluar los parámetros de sensibilidad, especificidad, valor predictivo positivo (VPP) y valor predictivo negativo (VPN) del ensayo RT-qPCR duplex se utilizaron 55 casos positivos diagnosticados con el método recomendado por la OPS (Corman et al.) 3 confirmadas por secuenciamiento y 50 casos negativos. Estas muestras fueron seleccionadas por conveniencia dado que era una enfermedad nueva y no se contaban con un número suficiente de muestras confirmadas por secuenciamiento.
Para evaluar el límite de detección del ensayo se usó un control positivo (ARN sintético del virus SARS-CoV-2 cuantificado) donado por la OPS. Para evaluar la especificidad diagnóstica se usaron muestras clínicas de HNF positivas para influenza A (n=3), influenza B (n=3), virus sincitial respiratorio (n=3), adenovirus (n=3), metapneumovirus (n=3), rhinovirus (n=3), virus dengue (n=2) y Streptococcus pneumoniae (n=2); dichas muestras provinieron de la viroteca del LRNVR-INS.
La repetibilidad fue evaluada con cuatro muestras positivas a SARS-CoV-2 durante cinco días, manteniendo las mismas condiciones: analista, ambiente, muestra y equipo. La reproducibilidad fue evaluada con 10 muestras (5 positivas y 5 negativas) por 2 analistas. La robustez fue evaluada con 4 muestras positivas a SARS-CoV-2 usando 3 concentraciones diferentes (0,6 µM, 0.4 µM y 0,2 µM) de los cebadores y sondas. La comparabilidad fue evaluada con 10 muestras (5 muestras positivas SARS-CoV-2 y 5 muestras negativas) en dos termocicladores diferentes. Los valores de Ct obtenidos fueron empleados para obtener el promedio, la desviación estándar y el coeficiente de variación. El límite de detección fue evaluado realizando diluciones seriadas desde 10-1 hasta 10-7 por triplicado determinando el valor del coeficiente de eficiencia para el gen RdRP, parámetro calculado por el software RotorGene (QIAGEN).
Evaluación de muestras clínicas
Se evaluaron 3006 muestras clínicas usando las condiciones optimizadas previamente, estas muestras fueron colectadas entre el 17 al 27 de marzo del 2020 y fueron enviadas al LRNVR-INS para confirmación diagnóstica en el marco de la vigilancia laboratorial de las infecciones respiratorias en el Perú.
Para evaluar si la prueba detectaba variantes de preocupación (VOC, por sus siglas en inglés) o de interés (VOI, por sus siglas en inglés) se evaluaron 31 muestras colectadas entre abril y agosto del 2021 derivadas al LRNVR-INS para confirmación diagnóstica y caracterizadas previamente mediante secuenciamiento del genoma completo por Next Generation Sequencing (NGS): 10 muestras de Lambda (C.37), 4 de Gamma (P.1), 4 de Alpha (B.1.1.7), 2 de Epsilon (B.1.429), 8 de Delta (AY.12), y 3 de Mu (B.1.621).
Evaluación bioinformática
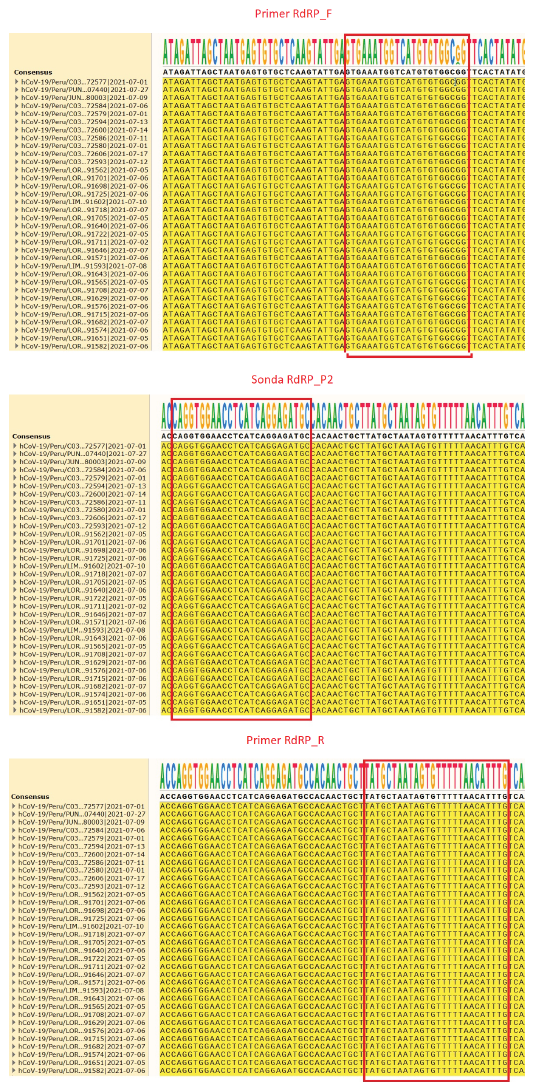
Para evaluar la conservación de los sitios de unión de los cebadores y sondas del marcador RdRP se realizó un alineamiento de 5600 secuencias del genoma completo de aislamientos peruanos de SARS-CoV-2 colectados entre marzo del 2020 y junio del 2021 (descargados de la base de datos GISAID [https://www.gisaid.org/]) usando el software MAFFT versión 7 (https://mafft.cbrc.jp/alignment/server/). Este alineamiento fue visualizado usando el programa SnapGene Viewer versión 5.3.2 (https://www.snapgene.com/snapgene-viewer/).
Análisis estadístico
La comparación de los valores de Ct se realizó con el programa Stata versión 16 (Stata Corporation, College Station, Texas, EE. UU.). Se calculó el índice kappa usando el programa Statistical Package for the Social Sciences (SPSS). Se determinó el grado de concordancia entre los resultados de la prueba de RT-qPCR dúplex y RT-PCR en tiempo real según Cormann et al. 3. Además, se calculó la sensibilidad, especificidad, VPN y VPN; y se consideró el coeficiente menor que 1% (próximo al 0) para indicar la existencia de poca variabilidad en los datos. Las medidas fueron obtenidas a través de estimadores puntuales e intervalos de confianza al 95% (IC95%).
Criterios éticos
La estandarización y validación en laboratorio no requirió evaluación por el Comité Institucional de Ética en Investigación (CIEI), ya que las muestras empleadas fueron obtenidas en las actividades rutinarias establecidas dentro del Plan de acción del Instituto Nacional de Salud para la prevención, diagnóstico y control de la COVID-19, aprobado con RJ-103-2020-J-OP-INS-2020.
HALLAZGOS
El ensayo RT-qPCR dúplex para la detección del virus SARS-CoV-2 presentó resultados de 100% de sensibilidad, 100% de especificidad, 100% de VPP y 100% de VPN (Tabla 2), 100% de especificidad diagnóstica (material suplementario, tabla S4), 100% de concordancia en los parámetros de repetibilidad (CV=0,00975, DS=0,232), reproducibilidad (CV=0,638, DS=0,126), robustez (CV=0,00575, DS=0,141) y comparabilidad (CV=0,52, DS=0,124) (material suplementario, tabla S5-S8), con un límite de detección de 100 copias/µL (Figura 1) (material suplementario, tabla S9) y una eficiencia del 93%, se obtuvo un valor de índice kappa de 1 en comparación con el método de Corman et al. según los resultados cualitativos. Los valores de Ct obtenidos para el marcador específico RdRP no fueron afectados cuando fue utilizado en el formato dúplex (material suplementario, tabla S1).
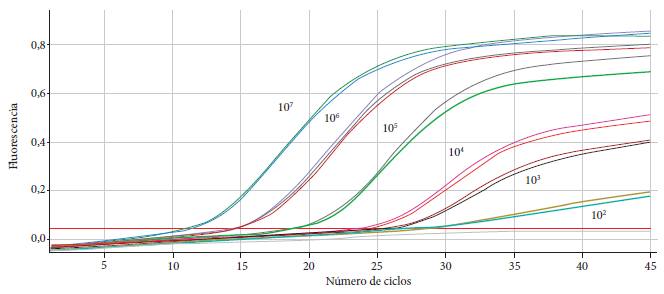
Figura 1 Curvas de fluorescencia obtenidas durante la evaluación del límite de detección de la prueba RT-PCR en tiempo real dúplex para la detección de SARS-CoV-2.
Tabla 2 Determinación de la sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo.
| RT-PCR en tiempo real dúplex | Prueba de referencia (Corman et al.) | Total | |
|---|---|---|---|
| Positivo | Negativo | ||
| Positivo | 55 | 0 | 55 |
| Negativo | 0 | 50 | 50 |
| Total | 55 | 50 | 105 |
Sensibilidad: 100%
Especificidad: 100%
Valor predictivo positivo: 100%
Valor predictivo negativo: 100%
Esta prueba ha sido utilizada para evaluar 3006 muestras clínicas obteniéndose una positividad de 9,2%, las muestras pertenecen a todos los grupos etarios y a todas regiones del país (material suplementario, tabla S3).
Adicionalmente, esta prueba detecta las VOC Delta y Gamma, y las VOI Mu, Epsilon y Lambda reportados durante el 2021 (Material suplementario, tabla S10). El análisis bioinformático que se realizó con 5600 genomas de aislamientos peruanos de SARS-CoV-2 reportados en GISAID indicó que las zonas de uniones de los cebadores y las sondas son conservados, excepto para el primer forward del gen RdRP que presenta un cambio nucleotídico al 2,9% de los genomas analizados (Figura 2).
DISCUSIÓN
Este estudio estandarizó y validó una prueba de RT-PCR en tiempo real dúplex usando cebadores y sondas específicas para el gen RdRP de SARS-CoV-2, y para el gen GAPDH humano como control endógeno. El gen GAPDH, que produce una proteína constitutiva involucrada en muchas funciones celulares como la glicolisis, la exportación del ARN nuclear, la replicación y reparación del ADN, y la apoptosis 4; este mismo gen fue descrito por Wong et al. 5 como un buen control endógeno interno para disminuir la posibilidad de obtener resultados falsos negativos en el diagnóstico de SARS-CoV-1. Esta estrategia permitió asegurar (en una sola reacción de PCR en tiempo real) la calidad de la muestra y del proceso de extracción de ARN. El uso de control endógeno interno ha sido recomendado por la OPS 6, y es usado en diferentes metodologías por el LRNVR-INS, como en el diagnóstico de virus de la influenza 7.
Los parámetros de validación obtenidos fueron de 100% en especificidad, sensibilidad y VPP y VPN al ser comparados con el método reportado por Corman et al. 4; adicionalmente, estas muestras fueron caracterizadas por secuenciación del genoma completo. El mismo logro fue obtenido para otros parámetros de validación en laboratorio como la robustez, la repetibilidad y la reproducibilidad. Los parámetros de validación fueron seleccionados y establecidos a partir de la experiencia del equipo de investigadores del LRNVR-INS y según estándares sugeridos por la OMS, descrito por Kosak et al. 8.
Dado que la OMS ha sugerido que el diagnóstico de la COVID-19 se debe realizar considerando la amplificación de, por lo menos, dos genes de SARS-CoV-2, en dos reacciones consecutivas de RT-PCR, esta estrategia no sería ampliamente factible de implementación en países en desarrollo como en Latinoamérica. Otros investigadores han descrito protocolos o adaptaciones de protocolos como Ishige et al. 9, quienes incluyeron al gen abl1 como control interno endógeno en las reacciones de identificación de SARS-CoV-2. Sin embargo, este gen produce una tirosina-quinasa presente en grandes cantidades en la célula humana, con riesgo de reducir la sensibilidad analítica de la prueba. Por otro lado, Pezzi y su equipo 10 indicaron que el gen MS2 (para la expresión de la enzima metionina sintetasa 2) podría ser utilizado como control endógeno junto al marcador RdRp reportado de Corman et al. 3 reduciendo los falsos negativos, presentando resultados similares a los obtenidos en este estudio, sin problemas con el rendimiento de las reacciones.
En Brasil, un grupo de investigadores de la Universidad de São Paulo 11 desarrolló un protocolo basado en otros métodos más económicos de detección usando un RT-PCR convencional. Estos métodos no son tan específicos como aquellos que usa las sondas; sin embargo, podrían ser una alternativa aplicable en los casos de emergencia sanitaria y de crisis en la provisión de reactivos.
Los resultados del presente estudio fueron exitosos toda vez que los ensayos cumplieron con todos los criterios de aceptación. Además, los análisis de eficiencia indicaron bajo coeficiente de variación, menor que 1%. Todos estos logros indicaron que los criterios para identificación de las muestras como verdaderamente positivas o negativas fueron adecuados para una prueba cualitativa. Adicionalmente, esta prueba ha sido utilizada para el diagnóstico de muestras clínicas de hisopado nasal y faríngeo, garantizando la constancia de las pruebas moleculares de los pacientes sintomáticos.
Las pruebas moleculares de diagnóstico deben ser continuamente actualizadas para evitar que sean afectadas por las mutaciones de los genomas virales como el caso de SARS-CoV-2 y otros virus patógenos, dado que se ha detectado una mutación en el uno de los cebadores sugerimos su actualización para evitar pérdida de la sensibilidad de la prueba.
Entre las limitaciones del estudio se encuentra la poca disponibilidad de muestras confirmadas para la validación debido a que se trataba de una enfermedad nueva, afortunadamente se contaba con un set de muestras con resultado de secuenciamiento. Otra limitación es que este sistema usa solo un marcador diagnóstico; sin embargo, el análisis bioinformático indica que los cebadores son específicos y sensibles para la detección del SAR-CoV-2.
En conclusión, la validación de la prueba de RT-PCR en tiempo real dúplex indica que la prueba es sensible y específica para la detección de infecciones causadas por el SARS-CoV-2 y ha sido evaluada de manera exitosa con muestras clínicas.











 text in
text in