Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista Peruana de Biología
On-line version ISSN 1727-9933
Rev. peru biol. vol.16 no.2 Lima Dec. 2009
COMENTARIO
Los virus Influenza y la nueva pandemia A/H1N1
Influenza virus and the new Influenza A/H1N1 pandemics
Miguel Talledo1,2 y Kattya Zumaeta2
1 Laboratorio de Fagotipia y Virología General. Facultad de Ciencias Biológicas. Universidad Nacional Mayor de San Marcos. Email Miguel talledo: mtalledor@unmsm.edu.pe
2 Instituto Peruano de Biología Molecular. Email Kattya Zumaeta: kattya_zumaeta@ipbiomol.com
Sumario
Los virus Influenza pertenecen a la familia Orthomyxoviridae, virus con genoma RNA de sentido negativo segmentado. Los virus influenza tipo A infectan a humanos y otros organismos, y son los agentes causantes de influenza en humanos. Resaltan entre sus principales proteínas la Hemaglutinina y la Neuraminidasa, que son utilizadas en la clasificación de los miembros de este grupo. Estos virus mutan continuamente, exhibiendo patrones muy estudiados, como el cambio y la deriva antigénica, siendo uno de los principales eventos de recombinación el reordenamiento. Todos los subtipos se encuentran en aves acuáticas silvestres, aunque se han encontrado otros hospederos, como equinos, visones, ballenas, focas, cerdos, gallinas y pavos, entre otros. Tanto las aves salvajes, las aves domésticas y el cerdo juegan un rol fundamental en la adaptación progresiva del virus al hospedero humano. Aunque los subtipos H2N2 y H3N2 han sido muy comunes, el subtipo H1N1 ha reemergido con mutaciones que le han permitido alcanzar el estado de pandemia en 2009. Este nuevo virus surge de un virus generado por triple reordenamiento con el virus humano, porcino norteamericano y aviar, conteniendo a su vez segmentos génicos de virus influenza porcina euroasiática. Esto ha hecho que el virus presente una enfermedad humana moderada y solamente severa y hasta letal en casos de individuos con condiciones médicas previas. A nivel mundial ha causado más de 134,510 casos y en el Perú alcanza cerca de 3,700 casos. El estado actual indica que la pandemia está por llegar a su pico máximo en el Perú, debido a la alta morbilidad del virus coincidente con la estación más fría del año. Es importante contener al máximo la dispersión del virus, ya que cuanto mayor sea el número de personas que infecte, el mismo estará sometido a un mayor número de eventos de recombinación genética por reordenamiento con virus influenza humanos previos y esto puede condicionar a la aparición todavía de nuevas cepas, para las que el sistema inmune podría no estar preparado a nivel poblacional.
Summary
The Influenza virus belongs to the Orthomyxoviridae family, viruses with a negative sense segmented RNA genome. The influenza virus type A infects humans and other organisms, and is the causative agent of human influenza. Hemagglutinin and Neuraminidase stand out among other proteins, and are used in the classification of the members of this group. These viruses mutate continuously, with patterns long studied, the antigenic shift and the antigenic drift, with one major event of recombination called reassortment. All subtypes exist in wild aquatic birds, although other hosts can be found, such as horses, minks, whales, seals, pigs, hens and turkeys, among others. As part of its progressive adaptation to the human host, wild birds and poultry play a fundamental role as well as swine. Although H2N2 and H3N2 subtypes have been very common among the human population, H1N1 subtype has re-emerged with mutations that have allowed it to reach the pandemics state in 2009. This new virus has a close ancestor in a triple reassortant virus from a human influenza virus, a classic influenza swine virus and an avian influenza virus, and contains as well genetic segments from a Euroasian swine influenza virus. This has caused that the virus displays a mild disease, only severe or lethal in individuals with previous medical history. At worldwide level it has caused more than 134.510 cases and in Peru they are close to 3.700. The current state indicates that in Peru the pandemics is about to reach its peak due to the high morbidity of the virus and coldest season of the year. The containment of this virus is important, since the greater the number of people infected, the greater the number of reassortment events the virus will be subjected to, with previous human influenza viruses, and may determine the appearance of new strains, for which the immune system might not be prepared at the population level.
Introducción
En abril de 2009, los Centros para el Control y Prevención de Enfermedades de los Estados Unidos (CDC) a través de su reporte Semanal de Morbilidad y Mortalidad, informaron de la ocurrencia de dos casos de enfermedad respiratoria febril en dos niños del estado de California, en los Estados Unidos, ambos por infección con virus Influeza tipo A/H1N1 de origen porcino y genéticamente similares. Estos virus contenían una combinación única de segmentos génicos no reportados entre los virus Influeza que afectan a porcinos ni humanos, ni en los Estados Unidos, ni en ningún otro lugar (OMS 2009a). De esta forma se hacía de conocimiento público un fenómeno cíclico y repetitivo en la historia de la humanidad: el desarrollo de una nueva pandemia.
En Perú, hasta julio de 2009, se contaban ya más de 3,500 casos de Influeza A/H1N1. En el mundo, la Organización Mundial de la Salud reporto más de 134,510 casos y 816 muertes, y desde junio es declarada como una nueva pandemia. Debido a la capacidad de mutar y de adaptarse a casi cualquier circunstancia, este virus representa una amenaza para la humanidad.
Este trabajo revisa las características más importantes de los virus Influeza, en particular del tipo A, con especial énfasis en la nueva cepa A/H1N1 responsable de la actual pandemia.
Los virus Influeza
Clasificación y estructura
Los virus Influenza pertenecen a la familia Orthomyxoviridae, la cual comprende 5 géneros: Influenzavirus A, Influenzavirus B, Influenzavirus C, Thogotovirus e Isavirus. Los virus Influeza A se clasifican en subtipos basados en la antigenicidad de sus moléculas de superficie: hemaglutininas (16 subtipos, de H1 a H16) y neuraminidasas (9 subtipos, de N1 a N9).
Los virus Influenza A son virus esféricos o pleomórficos (a veces se presentan algunas formas filamentosas) de hasta 120 nm de diámetro; presentan una envoltura derivada de la célula huésped. Es precisamente esta envoltura la que alberga la hemaglutinina (HA), la neuraminidasa (NA), y la proteína M2. Estas moléculas se encuentran muy dispersas en la envoltura. La HA se encuentra en una proporción de 4-5 a 1 con respecto a la NA, tiene de 4 a 6 nm de diámetro y se proyecta por encima de la envoltura hasta 10 a 14 nm.
El espacio entre la envoltura y la cápside viral es ocupado por la proteína matriz (M1). El centro mismo de la partícula viral lo conforman el complejo de ribonucleoproteína (RNP), que está compuesto por los 8 segmentos de RNA del genoma viral (Influenza A), las proteína polimerasa (PB1, PB2 y PA: polimerasa básica 1, polimerasa básica 2 y polimerasa ácida, respectivamente) y la nucleoproteína (NP) (Fig. 1). Adicionalmente, en preparaciones virales purificadas se puede encontrar la proteína NEP/NS2 (proteína nuclear de exportación/proteína no estructural 2) (Richardson y Akkina 1991). La composición global de la partícula viral es 1% de RNA, 5-8% de carbohidratos, 20% de lípidos y cerca de 70% de proteína (Knipe et al. 2007).
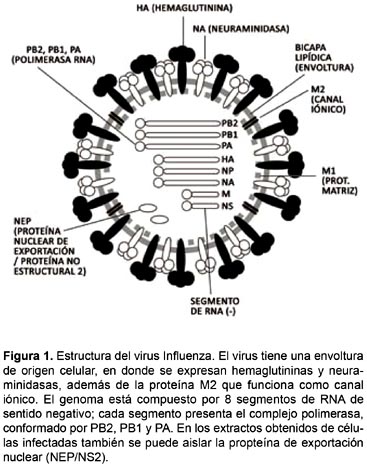
La partícula viral tiene una masa de 250 x 106 D. Es muy sensible al calor, solventes de lípidos, detergentes no iónicos, formaldehido y a los agentes oxidantes. Su infectividad se reduce luego de la exposición a la radiación. Los virus Influeza se clasifican formalmente en tipos, subtipos y cepas (Fig. 2).
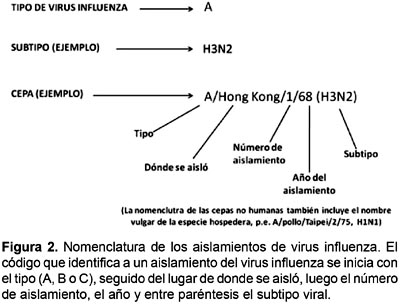
Debido a que en las aves acuáticas silvestres coexisten los 16 subtipos de HA y 9 de NA, en ellas se pueden presentar cerca de 144 permutaciones. En los humanos hay 3 subtipos que están actualmente en circulación: H1N1, H3N2 y H5N1.
Características del genoma viral
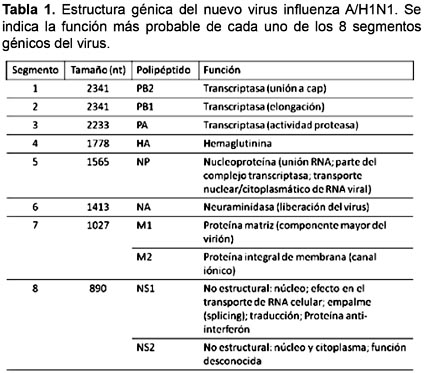
El genoma de los virus Influenza es segmentado. El genoma de los virus Influenza A está compuesto de 8 segmentos, otros tipos presentan 7 y algunos hasta 6 segmentos (Fauquet et al. 2005). Estos segmentos están compuestos de RNA lineal de una sola cadena de sentido negativo y contienen de 10 a 11 marcos de lectura abierta (ORFs).
El genoma completo tiene 13600 nucleótidos (Tabla 1). Los 3 segmentos más grandes contienen los genes que codifican para el complejo polimerasa, responsable de la replicación y la transcripción, y formado por PB2, PB1 y PA (Steinhauer y Skehel 2002). Esta polimerasa tiene actividad endonucleasa. Otros dos genes codifican las proteínas en la envoltura viral, HA y NA, las cuales juegan un rol crucial en la interacción entre el virus, la célula hospedera y el sistema inmune del individuo. Dos genes del mismo segmento codifican a dos proteínas que forman la cápside (M1 y M2). Las otras proteínas son la ribonucleoproteína (RNP) y NS1, NS2 y PB-1-F2 (Rabadan y Robins 2007). Al parecer, NS1 Y NS2 tienen función reguladora en la síntesis de los componentes virales de la célula infectada. PB1-F2 es una proteína proapoptótica que no está presente en todos los virus Influenza A (Rabadan y Robins 2007; Vega y Reyes 2007; Nicholson et al. 2003; Wong y Yuen 2006).
La actividad de la polimerasa RNA viral es proclive al error. La HA del virus Influenza está sujeta a una tasa de mutación muy alta, estimada en cerca de 2 x 10-3 sustituciones de bases por posición por generación viral, o alrededor de una sustitución de bases en el gen de la HA por generación viral (Webster et al. 1992).
Evolución de los virus Influeza y su relación con el hospedero
¿Qué mecanismos gobiernan la capacidad de cambio de este virus? Los virus Influenza generan nuevas cepas, es decir, virus antigénicamente distintos, de dos formas, que se explican a continuación.
Deriva antigénica (antigenic drift)
La forma más común de variación para estos virus se da mediante cambios de nucleótidos individuales en los genes que codifican a los sitios antigénicos de la HA y la NA. Este tipo de variación se conoce como deriva antigénica (antigenic drift). La deriva antigénica que se observa en los virus Influenza A se debe casi por completo a mutaciones en la molécula de HA (Wilson y Cox 1990). Estos virus se unen a los ácidos siálicos en la superficie de las células hospederas vía una depresión en la superficie distal de la cabeza globular de la molécula de HA (Weis et al. 1988; Wilson et al. 1981). Debido a su posición en la estructura tridimensional de la HA, las mutaciones en los residuos de las bolsas de unión al receptor pueden alterar la antigenicidad de un virus, además, o en vez de, modificar la especificidad o afinidad por el receptor (McDonald et al. 2007). Este tipo de fenómeno es muy importante para la patogénesis del virus y no sólo se restringe a modificaciones de la HA, ya que en algunos casos por virus Influeza A H3N2 estacional, la deriva antigénica ha llevado a modificaciones de la nucleoproteína viral y su consiguiente escape del reconocimiento por parte de los linfocitos T citotóxicos (Voeten et al. 2000). En la práctica, una variante por deriva antigénica que cause enfermedad significativa aparece en promedio cada 4 años y tiene 4 a más sustituciones de aminoácidos en dos o más sitios antigénicos (Dimmock et al. 2001) (Fig. 3).
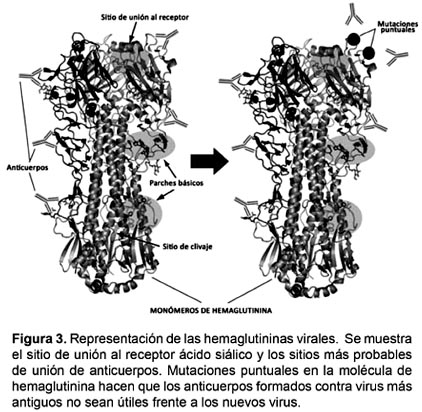
Los eventos de deriva antigénica epidemiológicamente relevantes, como los que ocurren en el caso de la H3, son acompañados por un inicio relativamente temprano de una epidemia de Influeza más severa. La deriva antigénica se toma muy en cuenta cuando se consideran cepas para su inclusión en vacunas de Influeza (Treanor 2004).
La selección de las sustituciones de aminoácidos es dirigida, al menos en parte, por la presión inmunológica, ya que la HA es el principal blanco de la respuesta inmune del hospedero. Aunque los aminoácidos que conforman el sitio de unión al receptor, así como residuos de cisteína y prolina, están muy conservados, el resto de la molécula de HA es altamente mutable.
Cambio antigénico (antigenic shift)
El segundo mecanismo, muy significativo desde el punto de vista molecular, se da cuando dos virus Influenza diferentes infectan a una misma célula. Durante la replicación, el genoma es replicado y enviado al citoplasma para su ensamblaje en nuevas partículas virales. Debido a que este genoma es segmentado, y habiendo dos cepas virales en proceso dentro de la misma célula, es relativamente frecuente que se introduzcan segmentos genómicos en la cápside del virus no correspondiente, lo que, por lo general, genera partículas no viables. Pero producto de esta recombinación pueden aparecer cepas con características nuevas. Este fenómeno se ha llamado reordenamiento (reassortment) (Brown et al. 1998; Downie 2004) (Fig. 4).
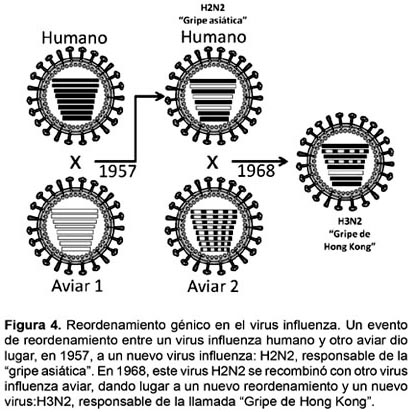
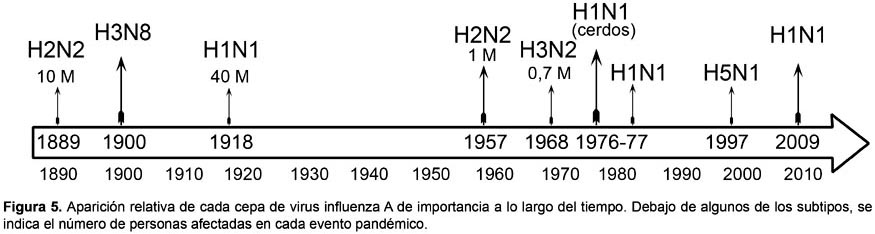
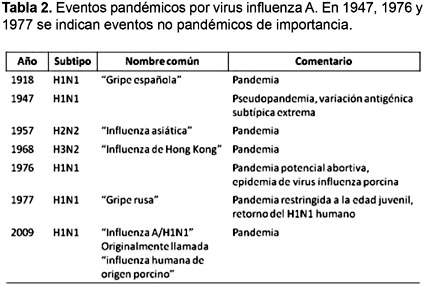
El reordenamiento génico ha sido el verdadero causante de la aparición de los diferentes subtipos virales a lo largo de la historia conocida de los virus Influeza en humanos y animales (Fig. 5) y de las diferentes pandemias que ha sufrido la humanidad (Tabla 2) (Nicholls 2006; Carter y Saunders 2007), los animales inferiores y las aves. El agente causal de la pandemia de Influeza española de 1918 pudo haberse derivado de virus clásicos H1N1 de porcinos (Taubenberger et al. 1997; Chowell et al. 2008), pero los que causaron las pandemias de 1957 y 1968 resultaron de un reordenamiento genético entre virus humanos y aviares (Brown et al. 1998). El causante de la pandemia actual, A/H1N1, resulta también de un complejo reordenamiento entre un virus porcino euroasiático y un virus Influenza A porcino norteamericano, este último procedente a su vez de un triple reordenamiento (Trifonov et al. 2009a; Trifonov et al. 2009b; Nava et al. 2009; Garten et al. 2009).
En principio, se piensa que el cambio antigénico ocurre cuando un virus de un ave silvestre infecta a un humano. No obstante, los virus Influeza, al igual que la mayoría de virus, tienen un rango de hospedero limitado a individuos de la misma especie, lo que hace que ni siquiera pueda infectar a las aves de otras especies, menos aún a miembros de otras clases. Por lo tanto, el virus tiene que pasar obligadamente por un proceso de mutación progresiva antes de dar el salto a los humanos. Nótese que en los reservorios naturales de este virus, las aves acuáticas silvestres, la infección ocurre a nivel del intestino, donde causa una infección subclínica y el virus es evolutivamente estable. Al migrar grandes distancias, estas aves se comportan como dispersoras de los virus en el mundo. Las aves domésticas que entran en contacto con aves migratorias o sus deposiciones son el enlace mediante el cual el virus pasa, de generar una infección intestinal subclínica, a una infección avirulenta del tracto respiratorio en aves de corral. La infección pasa de ave a ave, al mismo tiempo que ocurren mutaciones sucesivas en el sitio de clivaje de la proteasa del polipéptido precursor de la HA. Esto es fundamental para la infectividad del virus, ya que la HA sólo puede ser clivada por una proteasa presente sólo en el tracto respiratorio, pero, luego de un incremento importante en los residuos básicos de la molécula la HA puede ser clivada por una proteasa bastante distribuida en los tejidos del cuerpo y esto le permite al virus replicarse a lo largo de todo el cuerpo, aumentando su virulencia y patogenicidad en aves. Llegado este punto, el virus se vuelve capaz de saltar de las aves a los mamíferos y multiplicarse en humanos (Fig. 6).
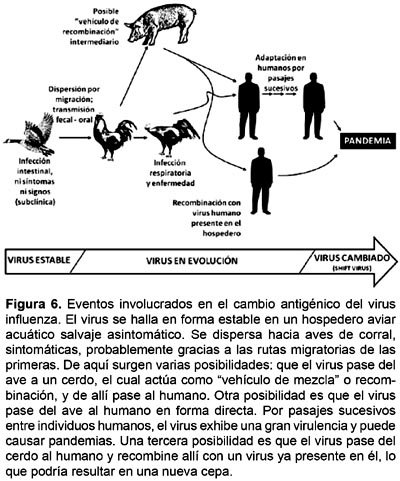
Es así que el virus tiene dos opciones evolutivas: seguir acumulando mutaciones y adaptarse mejor al humano o puede recombinarse con una cepa del virus Influeza humana ya existente. De hecho puede hacer las dos cosas al mismo tiempo. En este escenario el fenómeno anteriormente mencionado, el reordenamiento o reassortment, adquiere importancia relevante.
Este reordenamiento, en algunos casos, puede llevar a la aparición de resistencia frente a fármacos, tal es el caso de los virus Influeza A/H3N2 (Simonsen et al. 2007; Gubareva 2004) y últimamente A/H1N1 (OMS, 2009b). Desde la epidemia de 1918, también causada por un virus H1N1, el virus ha sufrido múltiples reordenamientos génicos. Algunos de ellos han sido relacionados con epidemias particularmente severas como las de 1947 y 1951. Inclusive el virus asociado a la epidemia de 1947 estaba compuesto por segmentos genómicos con diferentes historias filogenéticas sugiriendo un evento de reordenamiento entre subtipos del virus (Nelson et al. 2008). Ya desde finales de la década de los noventa se había comprobado la presencia de nuevos virus en cerdos, producto del reordenamiento entre virus Influenza A de origen porcino, aviar y humano (Brown et al. 1998; Zhou et al. 1999).
Base molecular de la patogénesis por el virus Influenza
El ciclo de replicación del virus Influeza se inicia con la unión del virus a las moléculas receptoras ácido siálico en la superficie de la célula hospedera a través de las glicoproteínas de superficie, las HA. Este paso es determinante para la patogénesis, transmisión y estrechez del rango de hospedero (Salomon y Webster, 2009). La replicación de los 8 segmentos de RNA de sentido negativo que comprenden el genoma del virus es fundamental para la patogénesis viral y podría ser un blanco terapéutico potencial. Cada segmento del genoma viral debe ser transcrito en RNAm, lo que da oportunidad para el control transcripcional de la expresión génica. La mayoría de virus con genomas segmentados y de sentido negativo se replican en el citoplasma (traen sus propias enzimas para la transcripción), sin embargo, el virus Influenza se replica en el núcleo celular, y utiliza la RNA polimerasa II, dependiente de DNA, celular y funcional, aunque esta enzima no transcriba su genoma (Dimmock et al. 2001). El virus genera dos tipos de RNA de sentido positivo: un RNAm y una plantilla para la replicación (RNA antigenómico).
El proceso de replicación viral dispara los factores de transducción de señales que inducen a la expresión de los genes de citoquinas proinflamatorias (p.e. Factor de Necrosis Tumoral - alfa, TNF-alfa), iniciando la comúnmente llamada "tormenta de citoquinas", y que se piensa que juega un rol prominente en la morbilidad y mortalidad de este virus (Cheung et al. 2002; de Jong et al. 2006; Kash et al. 2006) La síntesis de proteínas no estructurales del virus activa a la producción de Interferón alfa y beta (IFNa/b) activando la regulación de la respuesta inmune (Salomon y Webster 2009).
La infección por el virus Influeza en su fase primaria causa la enfermedad respiratoria febril y puede conducir a neumonía viral, síndrome de distrés respiratorio agudo y muerte. Las infecciones bacterianas por Streptococcus pneumoniae, Staphylococcus aureus, Streptococcus pyogenes, Neisseria meningitidis y Haemophilus Influezae son causas secundarias de neumonía, empiema, abceso pulmonar, choque tóxico, meningitis, sepsis y en un número de casos, de muerte. La infección combinada de virus y bacterias produce efectos fisiopatológicos determinantes en la neumonía viral-bacteriana que conduce a la muerte en individuos que se hallan en grupos de riesgo; estos efectos incluyen barreras físicas destruidas o dañadas, adherencia incrementada (mediada por anticuerpos neutralizantes, actividad mucociliar disminuida, disfunción de células del sistema inmune, desregulación del sistema inmune y hasta expresión génica sobre regulada (Brundage 2006).
Es evidente que tanto la inmunidad humoral como la inmunidad mediada por células juegan un rol de suma importancia en el control de la infección por el virus Influeza. Los anticuerpos producidos en respuesta a la infección reducen la carga viral y restringen ostensiblemente la reinfección, mientras que las células T citotóxicas destruyen activamente las células del epitelio respiratorio infectadas y hasta podrían provocar cierta supresión por citoquinas. En la inmunidad mediada por células se carece de especificidad elevada, lo cual se correlaciona con las respuestas inmunes de amplio espectro frente a antígenos centrales del virus (Hilleman 2002).
Ecología de los virus Influeza tipo A
Los virus Influeza A tienen una ecología muy dinámica y compleja, involucrando a muchas especies hospederas y genes virales. En base a su historia natural, los virus Influenza A tienen como hospedero primario a las aves acuáticas silvestres (patos, gaviotas, aves de orilla) (Webster et al. 1992). Todos los subtipos A/H1-16 se han aislado de aves acuáticas en Eurasia, los subtipos H14, H15 y H16 no han sido detectados en América (Fig. 7). Además, los virus predominantes en aves salvajes (H3, H4 y H6) muestran un patrón de periodicidad de 2 años (Webster et al. 2007). También han surgido los virus Influeza aviar altamente patógenos (Highly Pathogenic Avian Influenza Virus, HPAI), basados principalmente en los subtipos H5 y H7, y que emergen en aves domésticas. Sin embargo, los virus HPAI no se perpetúan en reservorios acuáticos silvestres y evolucionan ya sea por inserción (Perdue et al. 1997), mutación (Ludwig et al. 1995) o recombinación (Suárez et al. 2004; Pasick et al. 2005).
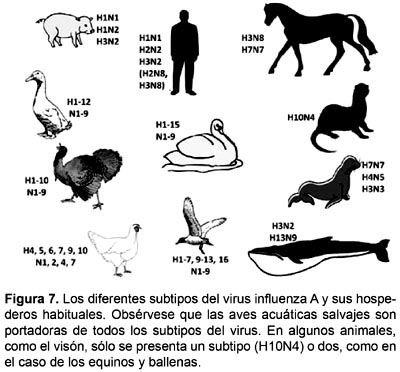
El sitio que el virus usa para replicación contrasta en aves silvestres y en humanos. En patos silvestres es asintomática, y el virus prefiere replicarse en el epitelio del tracto gastrointestinal (Webster et al. 1978). En los humanos, no obstante, se asocia frecuentemente con síntomas clínicos, y el virus se replica primariamente en el tracto respiratorio (Causey y Edwards 2008; Eccles 2005). Luego de la transferencia entre especies aviares y el humano, la infección resultante causa enfermedad extensiva y se puede observar una tasa de mutación viral incrementada mientras que el virus se va adaptando al nuevo hospedero (Suárez 2000). No obstante que todos los subtipos de HA de Influeza A han sido detectados en aves, en humanos solo encontramos HA 1, 2 y 3 y NA 1 y 2.
La separación que se observa en los reservorios para Influeza no solo depende del hospedero, sino también de la ubicación geográfica. Es interesante notar que la separación de las diferentes rutas migratorias de aves acuáticas ha resultado en la formación de reservas de genes de los virus Influeza geográficamente definidos de los linajes norteamericanos y euroasiáticos (Gorman et al. 1992; Suárez et al. 1998). La dinámica evolutiva de los virus Influeza en aves es muy alta, probablemente por la variedad de hospederos que puede alcanzar (Chen y Holmes 2006).
Esta dinámica se puso a prueba durante la aparición de la nueva cepa Influeza A/H5N1, que en 2004 inició una epidemia de HPAI entre aves de corral en países asiáticos, causando enfermedad humana en 108 personas, de las cuales 54 murieron en Tailandia, Vietnam y Camboya (Campitelli et al. 2006; Lu et al. 2006).
La evolución y ecología de los virus Influeza en hospederos como humanos, pollos y porcinos es relativamente comprobable porque las nuevas apariciones de estos virus son detectadas con cierta facilidad y los virus que se derivan de ello se pueden atribuir a un cierto linaje. En este sentido la transmisión de animal a animal parece mantener al virus Influeza en estos hospederos alternativos (Hay et al. 2001; Rambaut et al. 2008).
Otros reservorios mamíferos no humanos se pueden presentar para el virus Influenza A, como porcinos y caballos. El número de subtipos virales aislados de porcinos (H1N1, H3N2, H1N2) y de caballos (H7N7, H3N8) es limitado (Webster et al. 1992; Webby y Webster 2001). También se ha hallado este virus en mamíferos marinos y semiacuáticos (ballenas, focas y visones). Desde el punto de vista filogenético y antigénico, los virus de mamíferos marinos parecen ser de origen aviar y probablemente resultan de la transmisión independiente desde aves acuáticas. Por lo anteriormente mencionado, es probable que todos los virus Influenza A de mamíferos tengan enlaces a linajes aviares ancestrales (Ito y Kawaoka 1998).
El virus Influenza A en aves
Hay una gran diferencia entre la respuesta de las aves silvestre y las domésticas. Como ya se mencionó, en aves silvestres es casi invariablemente asintomática. La mayoría aquí son virus de baja patogenicidad (LP) también para las aves de corral. En aves silvestres es muy inusual la presencia de HPAI y solo se presenta en la vecindad de los lugares donde hay aves de corral. Las cepas LP causan entonces cuadros asintomáticos y muy suaves. Sin embargo, hay algunas cepas que producen infección sistémica con invasión del sistema nervioso central y alta mortalidad. Estos virus (HPAI) son sin excepción miembros de los subtipos H5 y H7. Los signos clínicos en aves de corral también dependen de otros factores, como la edad, ambiente, infecciones concurrentes y hasta la especie particular de ave.
Infección humana con Influeza aviar
El hombre se encuentra en un bajo riesgo de infección por virus Influenza aviar; solo unos pocos casos, todos por contacto directo con aves de corral infectadas, han sido originados por virus altamente patogénicos. Se han hallado cuatro subtipos de Influeza aviar en humanos: H5N1, H7N3, H7N7 y H9N2 (Van Reeth 2007). La mayoría se ha dispersado solo a una o unas cuantas personas, pero los subtipos H7N7 y H5N1 han infectado a decenas de personas y han demostrado ser excepcionalmente virulentos para los humanos (Van Reeth 2007). En humanos el pulmón parece ser el principal sitio de replicación. Casi todos los pacientes desarrollan una neumonía viral primaria. Se han reportado signos no respiratorios, por lo que es posible que el virus tenga un tropismo amplio en tejidos humanos. Sin embargo, no es definitivo que la detección de virus H5N1 u otro virus Influeza fuera del tracto respiratorio refleje infección genuina de otros tejidos.
Virus Influenza Porcina (VIP)
El VIP es uno de los pocos patógenos respiratorios primarios en porcinos. El virus puede inducir a enfermedad y lesiones pulmonares por sí mismo. En los porcinos, se presenta con un rápido inicio de fiebre alta, apatía, pérdida de apetito, respiración abdominal laboriosa y tos; pérdida de peso considerable, mortalidad baja y recuperación en 7 a 10 días (Van Reeth 2007).
Tres subtipos son comunes en porcinos (H1N1, H3N2 y H1N2) y circulan alrededor del mundo (Olsen et al. 2006; Sreta et al. 2009). Los virus de Influeza porcina (VIP) H1N1 que predominan en Europa son completamente de origen aviar y fueron introducidas por patos salvajes en porcinos en 1979 (Van Reeth 2007). En Estados Unidos circulan dos cepas de VIP H1N1, los virus "clásicos" H1N1, que permanecen desde inicios del siglo XX y también virus producto de reordenamiento con glicoproteínas de superficie del virus clásico y proteínas internas del más actual virus H3N2 o del VIP H1N2. Hay que resaltar que la mayoría de los VIP son virus reordenados con mezclas de genes humanos, aviares y porcinos. De ahí la noción general acerca de la susceptibilidad de los cerdos tantos a cepas virales humanas y aviares, comportándose, por lo tanto, como "vehículos de mezcla".
La hipótesis del "vehículo de mezcla" fue deducida por Scholtissek (1990) quien razonó que los porcinos infectados con virus Influeza porcina, podrían a su vez ser doblemente infectados con virus Influenza A aviar o humana. Esta infección dual podría producir reordenamientos entre virus porcinos y aviares/humanos. Estos virus reordenados podrían ser transmitidos al hombre resultando en la introducción de virus nuevos únicos en la población humana. Ocasionalmente, la combinación de genes porcinos, aviares y humanos produce una cepa pandémica humana. Esto no es de extrañar, ya que a nivel molecular ha sido probado que los virus Influenza A aviares y humanos llevan a cabo una unión preferente por tipos específicos de receptor: los virus aviares se unen a receptores ácido siálico con enlaces a2,3-Gal y los virus humanos a receptores ácido siálico con enlaces a2,6-Gal (Fig. 8) (Jung et al. 2002; Auewarakul et al. 2007). Estos receptores se expresan de forma distinta entre el epitelio humano y aviar. Sin embargo, ambos tipos de receptores pueden hallarse en el tracto respiratorio de porcinos (Ito et al. 1998).
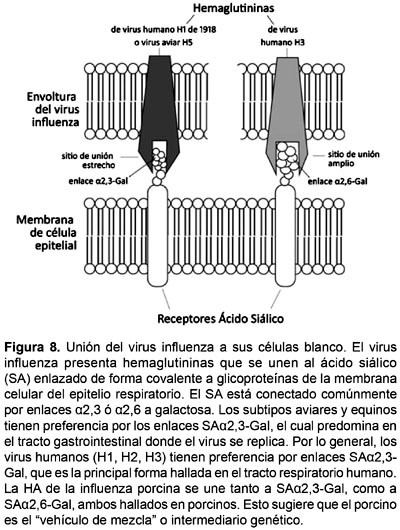
La hipótesis tiene tres partes claramente separadas: 1) los porcinos son susceptibles a los virus Influenza A aviar/humana; 2) el reordenamiento de genes de virus porcino/aviar/humano ocurren en el porcino y 3) los porcinos pueden transmitir los virus Influeza a la gente. La primera parte ya ha sido probada con el descubrimiento en diferentes partes del mundo, de virus H1N1 aviar a cerdos en China (Guan et al. 1996), H4N6, H1N1 y H3N3 aviares en cerdos canadienses (Karasin et al. 2004) y hasta los virus Influeza aviar altamente patogénicos H5N1 (HPAIV-H5N1) han sido aislados de porcinos aunque no han logrado establecerse en ellos (Zhu et al. 2008).
La segunda parte también ha sido debidamente comprobada. El análisis genómico completo arroja que el reordenamiento de virus aviares, humanos y/o porcinos ocurre en estos últimos. Por ejemplo, en cerdos europeos ha ocurrido reordenamiento entre virus aviar H1N1 y humano H3N2 (Castrucci et al. 1992; Gatherer 2009); también se han aislado, a partir de niños holandeses, virus con HA y NA de origen mamífero, pero genes internos de origen aviar (Claas et al. 1994). Muy recientemente, virus H3N2 de doble reordenamiento (dos fuentes génicas) con genes de origen humano (HA y NA) y genes de origen aviar (PB2, PB1, PA, NP, M y NS) y los virus H3N2 de reordenamiento triple (tres fuentes génicas) portando genes humanos (HA y NA), un gen porcino (NP) y genes de origen aviar (PB2, PB1, PA, M y NS), han aparecido en cerdos de China (Yu et al. 2008).
La transmisión de virus Influeza a humanos también se ha verificado recientemente por varios autores (Van Reeth 2007; Myers et al. 2007). No parece ser un evento aislado o raro, ni para virus Influeza porcina H3N2 ni H1N1 (Ma et al. 2009).
Infección humana con Influeza porcina
La mayoría de las infecciones por Influeza porcina son clínicamente indistinguibles de la Influeza humana, pero se han visto casos fatales en humanos infectados con virus Influeza porcina H1N1. En 1976, cuando ocurrió el llamado "incidente New Jersey" en los Estados Unidos, cerca de 500 humanos se infectaron con un virus H1N1 idéntico al aislado de cerdos en ese momento. No se pudo probar que los cerdos fueran la fuente del virus y además la cepa causaba una enfermedad muy suave o asintomática en humanos y en todo caso no era pandémica. También se han encontrado individuos seropositivos a VIP H1N2 entre granjeros y veterinarios en Iowa, Estados Unidos (Gaydos et al. 2006; Karasin et al. 2002).
El nuevo virus Influeza A/H1N1 y la nueva pandemia
Entre los años 1930 y los 1990s, el virus Influeza porcina más común en circulación, llamado virus Influeza A porcina clásico (H1N1), pasó por muy pocos cambios. Sin embargo, a finales de los 1990s, han surgido varias cepas y subtipos (H1N1, H3N2 y H1N2) de virus Influeza A porcina producto de triple reordenamiento, con genomas que incluyen combinaciones de segmentos genómicos de virus Influeza de origen aviar, humano y porcino (Shinde et al. 2009; Newman et al. 2008). Hasta Abril de 2009, solo se había reportado transmisión del virus Influeza de origen porcino de humano a humano de manera irregular. Antes del 2005, los Centros para el Control y Prevención de Enfermedades de los Estados Unidos (CDC) habían estado recibiendo uno o dos reportes de casos de infecciones humanas por virus Influeza de origen porcino clásico por año. En 2005 se reporta el primer caso de infección humana con Influeza A (H1) de origen porcino producto de triple reordenamiento en los Estados Unidos. Desde Diciembre 2005 hasta Febrero 2009, la CDC recibió 11 notificaciones de infección humana con virus Influeza A (H1) de origen porcino con reordenamiento triple.
El 24 de abril de 2009, La Organización Mundial de la Salud (OMS) lanzó una primera alerta indicando la ocurrencia de casos confirmados de Influeza A (H1N1) de origen porcino en Norteamérica (OMS 2009a). Unos días después, la CDC en los Estados Unidos confirmó que estos casos de Influeza humana eran causados por el mismo nuevo virus Influeza A (H1N1) (CDC 2009a). En unas semanas, se propuso que la epidemia de Influeza era causada por un nuevo virus Influenza A (H1N1) generado de un virus de reordenamiento triple a partir de virus Influenza humana, aviar y porcina (Cohen y Enserink 2009; CDC 2009b; Butler 2009).
La CDC desarrolló en 2008 una prueba basada en la Reacción en Cadena de la Polimerasa con retrotranscripción (rt-PCR) en tiempo real aprobada por la Food and Drug Administration (FDA) de los Estados Unidos y que sirvió para identificar los primeros casos en Norteamérica. El análisis de los 49 primeros aislamientos del entonces llamado "virus Influeza humana de origen porcino" se realizó primero por secuenciamiento en la CDC el 5 de mayo de 2009 y dio como resultado que todos virus aislados tenían una similitud del 99 al 100% (se trataba del mismo virus).
El análisis genómico del virus Influeza A/H1N1 en humanos indica que el virus se halla muy relacionado a los virus de Influenza A porcina generados por reordenamiento que han sido aislados con cierta frecuencia en Norteamérica, Europa y Asia. Los segmentos que codifican al complejo polimerasa, HA, proteína nuclear y proteínas no estructurales muestran alta similaridad con los virus Influeza A porcina H1N2 aislados en Norteamérica en la década de los años 90. H1N2 y otros subtipos son descendientes de los virus H3N2 porcinos de triple reordenamiento aislados en Norteamérica (Trifonov et al. 2009a). Estos virus se han dispersado en hospederos porcinos por todo el mundo y se ha demostrado que infectan a humanos (Garten et al. 2009).
Los segmentos génicos que conforman el nuevo virus Influenza A/H1N1 tienen distintos años de introducción y han pasado por varios hospederos, como se muestra en la Figura 9. Al parecer, los segmentos génicos del virus Influeza A humana H1N1 han coexistido en cepas del virus Influeza A porcina por más de 10 años; los ancestros de la NA no se habían observado en casi 20 años (Trifonov et al. 2009b).
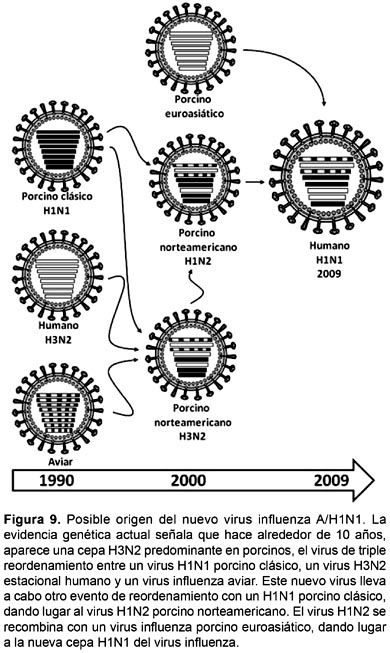
Hasta el momento, la investigación llevada a cabo sobre el virus circulante alrededor del mundo ha arrojado resultados muy claros sobre lo que ha ocurrido con este virus desde el punto de vista genético: en los años 90 circulaban tres cepas del virus Influeza A de diferentes orígenes (H1N1 porcino clásico, H3N2 humano estacional y virus Influeza aviar), las cuales entre 1998 y 2000 dan origen a un virus de reordenamiento triple, el virus Influeza A H3N2 porcino norteamericano; casi al mismo tiempo, este último virus se recombina por reordenamiento con el virus predominante hasta ese momento, H1N1 porcino clásico, apareciendo el virus H1N2 porcino norteamericano, que finalmente, por otro evento de reordenamiento con un virus Influenza A porcino euroasiático, genera la nueva cepa Influenza A/H1N1 humana 2009 (Trifonov et al. 2009a; Trifonov et al. 2009b; Nava et al. 2009; Wang y Palese 2009; Peiris et al. 2009).
En los casos presentados en los Estados Unidos y en otras partes del mundo, la infección por el virus de Influenza A de origen porcino se caracteriza por una enfermedad respiratoria febril autolimitada y con síntomas similares a los de la Influeza estacional, como fiebre (94%), tos (92%), inflamación de garganta (66%), rinorrea, dolor de cabeza, y dolor muscular, pero de forma interesante, también involucra vómitos (25%) y diarreas (25%), ninguno de los cuales es un síntoma típico de la Influeza estacional (Belshe 2005). Algunos pacientes han presentado un cuadro de neumonía severa con infiltrados multifocales y síndrome de distrés respiratorio agudo y falla multiorgánica (Peiris et al. 2009; Ong et al. 2009). La tasa de mortalidad en México (lugar donde el virus se expandió muy rápidamente al principio) se estimó en 0,4% y en otros países fue cercana al 0,15% (OMS 2009a), es decir, es muy baja. En el Perú, se calcula en 0,62% (Ministerio de Salud - Perú 2009).
Es muy útil evaluar el riesgo individual. Los factores de riesgo clásicos de la Influeza son enfermedad cardíaca, enfermedad pulmonar, diabetes, enfermedad renal, enfermedad reumatológica y demencia, entre otros. Está bien establecido que la Influeza es causa de morbilidad y mortalidad entre poblaciones con sistema inmune alterado, así como los pacientes receptores de trasplantes (Rothberg et al. 2008).
El ataque contra virus Influeza mediante el uso de antivirales tiene una historia de larga data (Hayden 2006; Jefferson et al. 2006). La OMS ha verificado que las personas que fueron afectadas en México y Estados Unidos por el nuevo virus H1N1, no respondían bien a antivirales antiguos, pero sí a Oseltamivir (nombre comercial Tamiflu®) y Zanamivir (nombre comercial Relenza®) (OMS 2009a). Ya hay reportes acerca de resistencia de este los virus Influenza A/H1N1 frente a Oseltamivir y Amantadina (Cheng et al. 2009, Weinstock y Zuccotti 2009), aunque para el virus pandémico actual aún no se reporta este fenómeno.
En cuanto a medidas de protección frente este virus, todavía no se dispone de una vacuna contra la cepa actual, aunque la CDC, la OMS y otros están trabajando en el desarrollo de una. Aunque la evidencia científica es incompleta todavía, sugiere que las vacunas de Influeza estacional disponibles actualmente no ofrecerán protección contra el virus. La OMS y la CDC están trabajando con algunos virus candidatos a vacunas y estiman que una vez que el virus haya sido modificado, tomará entre 5 a 6 meses producir en masa una vacuna contra H1N1. Una vez desarrollada, la OMS calcula que anualmente se podrían producir de una a dos mil millones de vacunas (OMS 2009c).
Influenza A/H1N1 en el Perú - Perspectivas a futuro
Aunque las epidemias gripales son tan comunes en el Perú como en el resto del mundo, ha habido algunos estudios realizados sobre brotes de cepas específicas, estacionales (Influenza A H3N2) con patogenicidad variable, pero sin mayor trascendencia para la salud poblacional (Mayca et al. 2003; Saldarriaga et al. 2007; Torres de Yon et al. 2004).
Luego de un inicio gradual, a fines de julio de 2009, las cifras oficiales del Ministerio de Salud informaban de 3686 casos confirmados y 23 muertes por Influenza A/H1N1, mostrando una letalidad del 0,62%. La mayoría de casos se han presentado en Lima (63,81%), y en individuos de 0 a 14 años (51,38%). Es interesante observar que la mayoría de afectados en Perú son aquellos que menos se han enfrentado al virus Influenza. Los individuos de 60 años a más, es decir, los que más tiempo han estado expuestos a virus de este tipo, tiene el porcentaje de casos más bajo (2,45%).
Existen en el Perú y a nivel mundial, varios protocolos moleculares, basados en la Reacción en Cadena de la Polimerasa (PCR), particularmente la PCR en tiempo real con transcripción en reversa (por tratarse de un virus RNA), que por su altísima sensibilidad y precisión se usan para el diagnóstico rápido de la Influenza A/H1N1 (Mahony et al. 2009; Chan et al. 2006). Aunque los pasos varían ligeramente, la mayoría detecta las mismas secuencias génicas: una correspondiente a la HA1 de esta nueva cepa 2009, otra para confirmar que se trata de un virus Influenza A y otro más que confirma que se está detectando un virus Influeza de origen porcino (Carr et al. 2009; Whiley et al. 2009)
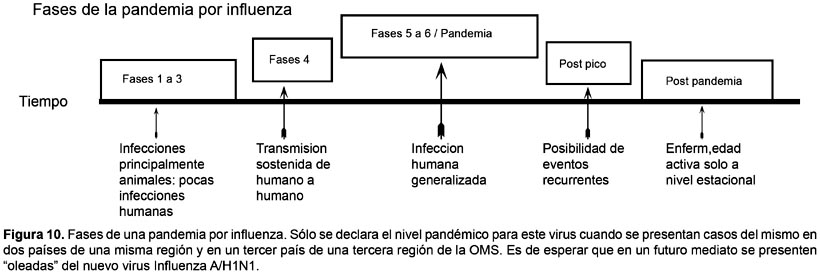
Pero, ¿qué es lo que va a ocurrir con esta pandemia? En este momento, según la OMS, el proceso se encuentra en fase 6. Esto quiere decir que el virus se propaga de persona a persona en dos países de una región de la OMS, y además han ocurrido brotes comunitarios en al menos un tercer país de una región distinta. Es decir, está en marcha una pandemia mundial. Con posterioridad, en un tiempo que no se puede determinar con exactitud, se ingresará a un período posterior al de máxima actividad, en el que la intensidad de la pandemia en la mayoría de los países que cuenten con una vigilancia adecuada habrá disminuido por debajo de la intensidad observada en el momento más álgido (Fig. 10). Las estadísticas indicarán que la pandemia remite, pero no se puede descartar que aparezcan nuevas oleadas. Estas oleadas de actividad de la infección se pueden alternar por varios meses. Ante la disminución del número de casos, las políticas de salud deben estar muy alertas, puesto que no hay períodos de actividad e inactividad previstos. Finalmente, cuanto se ingrese al período postpandémico, los casos de Influeza se podrán comparar a los de los habituales casos de gripe estacional. El virus pandémico actual posiblemente se comportará como un virus Influeza estacional de tipo A.
Hay muchos elementos que quedan por conocer. Por ejemplo, no se sabe con exactitud hasta dónde llegará este nuevo virus H1N1 y de qué manera va a competir con los virus H1N1 estacionales derivados de la cepa de 1918. Recordemos que el linaje actual (2009) lleva tres segmentos génicos que comparten una ascendencia remota pero común del virus de 1918 con el virus estacional humano, entre ellos, el que codifica a la HA. Será muy importante determinar cómo las poblaciones en las diferentes regiones del mundo respondan tanto a nivel de anticuerpos neutralizantes, como a través de la inmunidad mediada por células. Algunos reportes señalan que aunque los linfocitos T citotóxicos no confieren de una protección clínicamente efectiva contra la infección en humanos, pueden mediar de alguna forma en la protección cruzada y heterotípica en respuesta a proteína virales conservadas, al menos en modelos roedores y hasta se ha observado liberación viral reducida en humanos, aún en ausencia de anticuerpos contra HA y NA.
Aunque en este momento la enfermedad causada por el nuevo virus Influenza A/H1N1 es moderada, y solamente se complica en pacientes con condiciones médicas previas, debemos recordar que esta es una nueva forma del virus de la Influenza A, a la cual la mayoría de seres humanos tiene poca o ninguna inmunidad pre-existente. Por una parte, el virus todavía puede causar enfermedad a una proporción tan grande de personas, que en algunos lugares podría causar un efecto económico notable.
El cierre de colegios, de universidades, de espectáculos deportivos y otros eventos masivos se ha tomado como una medida de contención a la dispersión del virus, pero en algunos escenarios estas medidas resultan excesivas y resulta siendo mayor (y peor) el efecto sobre la actividad productiva del lugar que sobre la dispersión de la enfermedad.
Por otra parte, y quizás más importante, sea el hecho de que el nuevo virus Influenza A/H1N1, cuanto más personas infecte, y mayor el número de lugares que alcance, va a estar sometido a numerosos y novedosos eventos de recombinación por reordenamiento con virus pre-existentes, tanto en humanos como en animales, lo que representa no sólo un fenómeno natural para el virus, que trata de persistir en el ambiente, sino también una amenaza latente para la salud humana, ya que así como este nuevo virus presenta una alta morbilidad y muy baja mortalidad, ello no descarta que nuevas cepas virales, emergentes a muy corto plazo, presenten características mucho más severas que las que estamos atestiguando en la pandemia actual.
Es recomendable estudiar molecularmente los aislados virales que están apareciendo en cada región y correlacionarlos con las características clínicas que presentan, es decir hacer un seguimiento molecular del progreso biológico de este virus en la población humana, con el objeto de estar mejor preparados frente a una nueva oleada y mejor aún, a una nueva pandemia.
Literatura citada
Auewarakul P., O. Suptawiwat, A. Kongchanagul, et al. 2007. An Avian Influenza H5N1 Virus That Binds to a Human-Type Receptor. Journal of Virology 81(18):9950-9955. [ Links ]
Belshe R. B. 2005. The origins of pandemic Influeza - Lessons from the 1918 virus. The New England Journal of Medicine 353(21):2209-2211.
Brown I. H. , P. A. Harris, J. W. McCauley & D. J. Alexander. 1998. Multiple genetic reassortment of avian and human Influeza A viruses in European pigs, resulting in the emergence of an H1N2 virus of novel genotype. Journal of General Virology 79:2947-2955.
Brundage J. F. 2006. Interactions between Influeza and bacterial respiratory pathogens: implications for pandemic preparedness. Lancet Infectious Diseases 6:303-12.
Butler, D. 2009. Swine flu goes global. Nature. 458(7242):1082-3.
Campitelli L., M. Ciccozzi, M. Salemi, et al. 2006. H5N1 Influeza virus evolution: a comparison of different epidemics in birds and humans (1997-2004). Journal of General Virology 87:955-960.
Carr M. J., R. Gunson, A. Maclean, et al. 2009. Development of a real-time RT-PCR for the detection of Swine-lineage Influenza A (H1N1) virus infections. Journal of Clinical Virology 45:196-199.
Carter J. & R. Saunders. 2007. Virology Principles and Applications. John Wiley & Sons Ltd. West Sussex, 383 pp.
Castrucci, M. R., I. Donatelli, L. Sidoli, et al. 1993. Genetic reassortment between avian and human Influeza A viruses in Italian pigs. Virology 193:503-506.
Causey D. & S. V. Edwards. 2008. Ecology of avian Influeza virus in birds. J Inf Dis 197:S29-33.
CDC. Centers for Disease Control and Prevention. 2009a. Swine Influeza A(H1N1) infection in two children - Southern California, March-April 2009. MMWR Morbidity Mortality Weekly Report 2009; 58(15):400-2.
CDC. Centers for Disease Control and Prevention. 2009b. Update: swine Influeza A(H1N1) infections - California and Texas, April 2009. MMWR Morbidity Mortality Weekly Report 2009; 58(16):435-7.
Chan C.-H., K.-L. Lin, Y. Chan, et al. 2006. Amplification of the entire genome of Influeza A virus H1N1 and H3N2 subtypes by reverse-transcription polymerase chain reaction. Journal of Virological Methods 136:38-43.
Chen R. & E. C. Holmes. 2006. Avian Influeza virus exhibits rapid evolutionary dynamics. Molecular Biology and Evolution 23(12):2336-2341.
Cheng P. K. C., T. W. C. Leung, E. C. M. Ho, et al. 2009. Oseltamivir- and amantadine-resistant Influeza viruses A (H1N1). Emerging Infectious Diseases. 15(6):966-968.
Cheung C.Y., L. L. Poon, A. S. Lau, et al. 2002. Induction of proinflammatory cytokines in human macrophages by Influeza A (H5N1) viruses: a mechanism for the unusual severity of human disease? Lancet 360:1831-1837.
Chowell G., L. M. A. Bettencourt, N. Johnson, et al. 2008. The 1918-1919 Influeza pandemic in England and Wales: spatial patterns in transmissibility and mortality impact. Proceedings of the Royal Society B 275:501-509.
Claas, E. C., Y. Kawaoka, J. C. de Jong, et al. 1994. Infection of children with avian-human reassortant Influeza virus from pigs in Europe. Virology 204:453-457.
Cohen, J. & M. Enserink. 2009. Infectious diseases. As swine flu circles globe, scientists grapple with basic questions. Science. 324(5927):572-3.
de Jong M. D., C. P. Simmons, T. T. Thanh, et al. 2006. Fatal outcome of human Influeza A (H5N1) is associated with high viral load and hypercytokinemia. Nature Medicine 12:1203-1207.
Dimmock N. J., A. J. Easton & K. N. Leppard. 2001. Introduction to Modern Virology. 5th edition. Blackwell Science, Oxford. 449 pp.
Downie J. C. 2004. Reassortment of Influeza A virus genes linked to PB1 polymerase gene. International Congress Series 1263:714-718.
Eccles R. 2005. Understanding the symptoms of the common cold and Influeza. Lancet Infectious Diseases 5:718-725.
Fauquet C. M., M. A. Mayo, J. Maniloff, U. Desselberger & L. A. Ball (eds). 2005. Virus Taxonomy. Classification and Nomenclature of Viruses. Elsevier Academic Press, Amsterdam, 1273 pp.
Garten R. J., C. T. Davis C. A. Russell et al. 2009. Antigenic and Genetic Characteristics of Swine-Origin 2009 A(H1N1) Influenza Viruses Circulating in Humans. Science 325:197-201.
Gatherer D. 2009. The 2009 H1N1 Influeza outbreak in its historical context. Journal of Clinical Virology 45:174-178.
Gaydos J. C., F. H. Top, Jr., R. A. Hodder & P. K. Russell. 2006. Swine Influenza A Outbreak, Fort Dix, New Jersey, 1976. Emerging Infectious Diseases 12(1):23-28.
Gorman O. T., W. J. Bean & R. G. Webster. 1992. Evolutionary processes in Influeza viruses: divergence, rapid evolution and stasis. Current Topics in Microbiology and Immunology 176:75-97.
Guan, Y., K. F. Shortridge, S. Krauss, et al. 1996. Emergence of avian H1N1 Influeza viruses in pigs in China. Journal of Virology 70:8041-8046.
Gubareva L. V. Molecular mechanisms of Influeza virus resistance to neuraminidase inhibitors. Virus Research 103:199-203.
Hay A. J., V. Gregory, A. R. Douglas & Y. P. Lin. 2001. The evolutiomn of human Influeza viruses. Philosophical Transactions of the Royal Society B. 356:1861-1870.
Hayden F. G. 2006. Antivirals for Influeza: Historical perspectives and lessons learned. Antiviral Research 71:372-378.
Hilleman M. R. 2002. Realities and enigmas of human viral Influeza: pathogenesis, epidemiology and control. Vaccine 20:3068-3087.
Ito, T. & Y. Kawaoka. 1998. Avian Influeza. En: Textbook of Influenza. K.G. Nicholson, R.G. Webster y A.J. Hay, editores, pp. 126-136. Blackwell Science. Oxford.
Ito, T., J. N. Couceiro, S. Kelm, et al. 1998. Molecular basis for the generation in pigs of Influeza A viruses with pandemic potential. Journal of Virology 72:7367-7373.
Jefferson T., V. Demicheli, D. Rivetti, et al. 2006. Antivirals for Influeza in healthy adults: systematic review. Lancet 367:303-313.
Jung T., C. Choi & C. Chae. 2002. Localization of Swine Influenza Virus in Naturally Infected Pigs. Veterinary Pathology 39:10-16.
Karasin A. I., J. Landgraf, S. Swenson, et al. 2002. Genetic Characterization of H1N2 Influenza A Viruses Isolated from Pigs throughout the United States. Journal of Clinical Microbiology 40(3):1073-1079.
Karasin, A. I., K. West, S. Carman & C. W. Olsen., 2004. Characterization of avian H3N3 and H1N1 Influeza A viruses isolated from pigs in Canada. Journal of Clinical Microbiology 42:4349-4354.
Kash J. C., T. M. Tumpey, S. C. Proll, et al. 2006. Genomic analysis of increased host immune and cell death responses induced by 1918 Influeza virus. Nature 443:578-581.
Knipe D. M. & P. M. Howley, eds. 2007. Fields Virology. 5th edition. Lippincott Williams & Wilkins, Philadelphia, 3177 pp.
Lin Y. P., M. Shaw, V. Gregory, et al. 2000. Avian-to-human transmission of H9N2 subtype Influeza A viruses: Relationship between H9N2 and H5N1 human isolates. Proceedings of the National Academy of Sciences 97(17):9654-9658.
Lu J., D. Zhang & G. Wang. 2006. Highlight the significance of genetic evolution of H5N1 avian flu. Chinese Medical Journal 119 (17):1458-1464.
Ludwig S., l. Stitz, O. Planz, et al. 1995. European swine virus as a possible source for the next Influeza pandemic? Virology 212: 555-561.
Ma W., R. E Kahn & J. A Richt. 2009. The pig as a mixing vessel for Influeza viruses: Human and veterinary implications. Journal of Molecular Genetics and Medicine. 3(1):158-166.
Mahony J. B., T. Hatchette, D. Ojkic, et al. 2009. Multiplex PCR tests sentinel the appearance of pandemic Influeza viruses including H1N1 swine Influeza. Journal of Clinical Virology 45:200-202.
Mayca J., Y. Torres de Yon, S. Capristano & M. Farfán. 2003. Brote de Influeza en Pucallpa, Ucayali, Perú. 2002. Revista Peruana de Medicina Experimental y Salud Pública 20(4).
McDonald N. J., C. B. Smith & N. J. Cox. 2007. Antigenic drift in the evolution of H1N1 Influeza A viruses resulting from deletion of a single amino acid in the haemagglutinin gene. Journal of General Virology 88:3209-3213.
Ministerio de Salud. Dirección General de Epidemiología. Situación Actual de la Influenza A (H1N1) - 28 de Julio de 2009. <http://www.dge.gob.pe/ Influeza/AH1N1/sala/Sala_pandemia_28-07-2009.pdf> (acceso 28/07/09).
Myers K.P., C. W. Olsen, S. F. Setterquist, et al. 2006. Are swine workers in the United States at increased risk of infection with zoonotic Influeza virus? Clinical Infectious Diseases 42:14-20.
Myers, K. P., C. W. Olsen & G. C. Gray. 2007. Cases of swine Influeza in humans: a review of the literature. Clinical Infectious Diseases 44:1084-1088.
Nava G. M., M. S. Attene-Ramos, J. K. Ang & M. Escorcia. 2009. Origins of the new Influeza A(H1N1) virus: time to take action. Eurosurveillance 14(22):1-22.
Nelson, M. I., C. Viboud, L. Simonsen, et al. 2008. Multiple reassortment Events in the Evolutionary History of H1N1 Influenza A Virus Since 1918. PLos Pathogens 4(2):1-12.
Newman A. P., E. Reisdorf, J. Beinemann, et al. 2008. Human case of swine Influeza A (H1N1) triple reassortant virus infection, Wisconsin. Emerging Infectious Diseases 14:1470-2.
Nicholls H. 2006. Pandemic Influeza: the inside story. PLos Biology 4(2):0156-0160.
Nicholson K.G., J. M. Wood &M. Zambon. 2003. Influenza. The Lancet. 362:1733-1745.
Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team. 2009. Emergence of a novel swine-origin Influeza A (H1N 1) virus in humans. The New England Journal of Medicine 360:2605-15.
Olsen C. W., I. H. Brown, B. C. Easterday & K. Van Reeth. 2006. Swine Influeza. En: Disease of swine. Straw B. E., J. J. Zimmerman, S. d'Allaire & D. J. Taylor, Editores. Blackwell Publishing, Oxford, pp. 469-482.
Ong C. W. M., K. Y. Ho, L. Y. Hsu, et al. 2009. Reacting to the emergence of swine-origin infl uenza A(H1N1). The Lancet Infectious Diseases 9:397-398.
Organización Mundial de la Salud (OMS) 2009a. World Health Organization (WHO). Influenza-like illness in the United States and Mexico. Epidemic and Pandemic Alert and Response (EPR). 24 April 2009. <http://www.who.int/csr/don/2009_04_24/en/index.html> (acceso 10/07/09).
Organización Mundial de la Salud (OMS). 2009b. Update: drug susceptibility of swine origin Influeza A (H1N1) viruses, April 2009. MMWR Morbidity Mortality Weekly Report 2009; 58:433-5.
Organización Mundial de la Salud (OMS). 2009c. Influenza A(H1N1)—update 42. <http://www.who.int/csr/don/2009 06 01a/en/index.html> (acceso 12/07/09).
Pasick J., K. Handel, J. Robinson, et al. 2005. Intersegmental recombination between the haemagglutinin and matrix genes was responsible for the emergence of a highly pathogenic H7N3 avian Influeza virus in British Columbia. Journal of General Virology 86:727-731.
Peiris J. S. M., L. L. M. Poon & Y. Guan. 2009. Emergence of a novel swine-origin Influeza A virus (S-OIV) H1N1 virus in humans. Journal of Clinical Virology 45:169-173.
Perdue M. l., M. Garcia, D. Senne & M. Fraire. 1997. Virulence-associated sequence duplication at the hemagglutinin cleavage site of avian Influeza viruses. Virus Research 49:173-186.
Rabadan R. & H. Robins. 2007. Evolution of the Influenza A virus: Some new advances. Evolution and Bioinformatics Online 3:299-307.
Rambaut A., O. G. Pybus, M. I. Nelson, et al. 2008. The genomic and epidemiological dynamics of human Influeza A virus. Nature 453:615-620.
Richardson J. C. & R. K. Akkina. 1991. NS2 protein of Influeza virus is found in purified virus and phosphorylated in infected cells. Archives of Virology 116(1-4):69-80.
Rothberg M. B., S. D. Haessler & R. B. Brown. 2008. Complications of Viral Influenza. The American Journal of Medicine 121:258-264.
Saldarriaga T., V. A. Laguna-Torres, J. Arrasco, et al. 2007. Características clínicas y moleculares de un brote de Influeza en dos bases militares, Tumbes-Perú,. Revista Peruana de Medicina Experimental y Salud Pública. 25(1): 35-43.
Salomon R. & R. G. Webster. 2009. The Influeza virus enigma. Cell 136:402-410.
Scholtissek C. 1990. Pigs as the `mixing vessel' for the creation of new pandemic Influeza A viruses. Medicine Principles Practice, 2:65-71.
Shinde V., C. B. BridgesT. M. Uyeki et al. 2009. Triple-Reassortant Swine Influenza A (H1) in Humans in the United States, 2005-2009. New England Jornal of Medicine 361:1-20.
Simonsen L., C. Viboud, B. T. Grenfell, et al. 2007. The Genesis and Spread of Reassortment Human Influenza A/H3N2 Viruses Conferring Adamantane Resistance. Molecular Biology and Evolution 24(8):1811-1820.
Sreta D., R. Kedkovid, S. Tuamsang, et al. 2009. Pathogenesis of swine Influeza virus (Thai isolates) in weanling pigs: an experimental trial. Virology Journal 6:34
Steinhauer D. A. & J. J. Skehel. 2002. Genetics of Influeza viruses. Annual Review of Genetics 36: 305-332.
Suárez D. L. 2000. Evolution of avian Influeza viruses. Veterinary Microbiology 74:15-27.
Suarez D. L., D. A. Senne, J. Banks, et al. 2004. Recombination resulting in virulence shift in avian Influeza outbreak, Chile. Emerging Infectious Diseases 10:693-699.
Suárez D. L., M. L. Perdue, N. Cox, et al. 1998. Comparison of highly virulent H5N1 Influeza A viruses isolated from humans and chickens from Hong Kong. Journal of Virology 72:6678-6688.
Taubenberger J. K., A. H. Reid, A. E. Krafft, et al. 1997. Initial genetic characterization of the 1918 Spanish Influenza virus. Science 275:1793-1796.
Torres de Yon Y., J. Mayca, F. Llanos-Zavalaga, et al. 2004. Virus Influeza y el diagnóstico diferencial de sintomáticos febriles en la costa norte del Perú (Mayo, 2001). Revista Peruana de Medicina Experimental y Salud Pública 21(1).
Treanor, J. 2004. Influenza vaccine - outmaneuvering antigenic shift and drift. The New England Journal of Medicine 350(3):218-220.
Trifonov V., H. Khiabanian & R. Rabadan. 2009a. Geographic Dependence, Surveillance, and Origins of the 2009 Influenza A (H1N1) Virus. The New England Journal of Medicine 10.1056/NEJMp0904572.
Trifonov V., H. Khiabanian, B. Greenbaum & R. Rabadan. 2009b. The origin of the recent swine Influeza A(H1N1) virus infecting humans. Eurosurveillance 14(17):1.
Van Reeth K. 2007. Avian and swine Influeza viruses: our current understanding of the zoonotic risk. Veterinary Research 38:243-260.
Vega R. S. & G. Reyes-Terán. 2007. El virus de la Influeza. Neumología y Cirugía de tórax 66(S1):S12-S14.
Voeten J. T. M., T. M. Bestebroer, N. J. Nieuwkoop, et al. 2000. Antigenic Drift in the Influenza A Virus (H3N2) Nucleoprotein and Escape from Recognition by Cytotoxic T Lymphocytes. Journal of General Virology 74(15):6800-6807.
Wang T. T. & P. Palese. 2009. Unraveling the mistery of swine Influeza virus. Cell 137: 983-985.
Webby R. J. & R. G. Webster. 2001. Emergence of Influeza A viruses. Philosophical Transactions of the Royal Society B 356:1817-1828.
Webster R. G., M. Yakhno, V. S. Hinshaw, et al. 1978. Intestinal Influeza: replication and characterization of Influeza viruses in ducks. Virology 84:268-278.
Webster R. G., S. Krauss, D. Hulse-Post & K. Sturm-Ramirez. 2007. Evolution of Influeza A viruses in wild birds. Journal of Wildlife Diseases 43(3):S1-S6.
Webster R. G., W. J. Bean, O. T. Gorman, et al. 1992. Evolution and ecology of Influenza A viruses. Microbiological Reviews 56(1):152-179.
Weinstock D. M. & G. Zuccotti. 2009. The evolution of Influeza resistance and treatment. Journal of the American Medical Association (JAMA) 301(10):1066-1069.
Weis W., J. H. Brown, S. Cusack, et al. 1988. Structure of the Influeza virus haemagglutinin complexed with its receptor, sialic acid. Nature 333:426-431.
Weis W., J. H. Brown, S. Cusack, et al. 1988. Structure of the Influeza virus haemagglutinin complexed with its receptor, sialic acid. Nature 333:426-431.
Whiley D. M., S. Bialasiewicz, C. Bletchly, et al. 2009. Detection of novel Influeza A(H1N1) virus by real-time RT-PCR. Journal of Clinical Virology 45:203-204.
Wilson I. A. & N. J. Cox. 1990. Structural basis of immune recognition of Influeza virus hemagglutinin. Annual Reviews of Immunology 8:737-771.
Wilson I. A., J. J. Skehel & D. C. Wiley. 1981. Structure of the haemagglutinin membrane glycoprotein of Influeza virus at 3 Å resolution. Nature 289:366-373.
Wong S. S. & K. Y. Yuen. 2006. Avian Influeza virus infections in humans. Chest 129:156-168.
Yu H., R. H. Hua, Q. Zhang, et al. 2008. Genetic evolution of swine Influeza A (H3N2) viruses in China from 1970 to 2006. Journal of Clinical Microbiology 46:1067-1075.
Zhou N. N., D. A. Senne, J. S. Landgraf, et al. 1999. Genetic Reassortment of Avian, Swine, and Human Influenza A Viruses in American Pigs. Journal of Virology 73(10): 8851-8856.
Zhu Q., H. Yang, W. Chen, et al. 2008. A naturally occurring deletion in its NS gene contributes to the attenuation of an H5N1 swine Influeza virus in chickens. Journal of Virology 82:220-228.
Presentado: 03/08/2009
Aceptado: 17/11/2009
Publicado online: 12/01/2010













