Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista Peruana de Biología
On-line version ISSN 1727-9933
Rev. peru biol. vol.22 no.2 Lima Aug. 2015
TRABAJOS ORIGINALES
Evaluación del gen que codifica la enzima βHPMEH para la inhibición de la marchitez bacteriana causada por Ralstonia solanacearum
Evaluation of the gene encoding the enzyme βHPMEH for the bacterial wilt inhibition caused by Ralstonia solanacearum
Elizabeth Fernandez1; Liliam Gutarra1; Jan Kreuze1
1 Laboratorio de Bacteriología y Biotecnología Aplicada, Centro Internacional de la Papa, Apartado 1558, Lima 12, Perú.
Resumen
La marchitez bacteriana causada por Ralstonia solanacearum E.F. Smith es una de las enfermedades bacterianas más importantes que ataca a cultivos agrícolas como papa, tomate, banana, entre otros, causando grandes pérdidas en la producción. Desafortunadamente, su control ha sido difícil por su amplio rango de hospederos alternativos, su supervivencia en el suelo y su variación biológica y genética; así como porque no hay variedades con altos niveles de resistencia y porque no existe un control químico efectivo. Quorum sensing (percepción de quorum) es el fenómeno mediante el cual la acumulación de unas moléculas permite a una bacteria saber el número de bacterias que se encuentran en el medio es decir la densidad poblacional. La bacteria R. solanacearum posee un sistema quorum sensing para la regulación de la expresión de genes de virulencia, y en la cual la molécula 3-OH-PAME es el autoregulador de esta señal. Se conoce que la molécula ΒHPMEH hidroliza a 3-OH-PAME, anulando así la señal de autorregulación y por tanto la comunicación quorum sensing en R. solanacearum. Con el objetivo de evaluar el gen βhpmeh, se diseñaron dos vectores que expresen este gen bajo el control de dos diferentes promotores, los cuales fueron verificados por análisis de restricción, secuenciamiento y posteriormente mediante técnicas de agroinfiltración, se observó su expresión y su efecto frente a R. solanacearum en hojas de papa de la variedad Desiree. Los resultados de la expresión transitoria, muestran que el gen βhpmeh retrasó la aparición de síntomas de la marchitez bacteriana y sería un candidato potencial para transformación genética de la planta entera.
Palabras clave: Expresión transitoria; marchitez bacteriana; quorum sensing; Ralstonia solanacearum; quorum quenching.
Abstract
Ralstonia solanacearum is the causal agent of the devastating bacterial wilt disease that attacks important agricultural crops such as potato, tomato, banana, among others, causing serious yield losses. Control of R. solanacearum is difficult because of its wide range of alternate hosts, its long survival in soil, its biological and genetic variation, the lack of natural resistance sources and the insufficiency of the appropriate chemical control measures. Quorum sensing is the term that describes the phenomenon whereby the accumulation of molecules allows bacteria to know the number of bacteria found in the environment (population density). R. solanacearum has a quorum sensing system for the regulation of the expression of virulence genes; the molecule 3-OH-PAME is the self-regulatory signal. The molecule ΒHPMEH hydrolyzes 3-OH-PAME nullifying the signal of virulence, and thus, the quorum sensing communication in R. solanacearum. In order to evaluate the βhpmeh gene we designed two vectors that express this gene under the control of two different promoters. Both vectors were verified by restriction analysis and sequencing. Agroinfiltration assays were used to analyze gene expression and the effect against R. solanacearum in potato (Solanum tuberosum) leaves. The results of the transient expression experiments showed that the expression of gene βhpmeh caused a delay in the appearance of symptoms of bacterial wilt and thus is a good candidate for whole genetic plant transformation.
Keywords: Transient expression; bacterial wilt; quorum sensing; Ralstonia solanacearum; quorum quenching.
Introducción
La marchitez bacteriana causada por la bacteria Ralstonia solanacearum E.F. Smith, es la principal enfermedad bacteriana que limita la producción de papa en la mayoría de las regiones tropicales y subtropicales del mundo. La enfermedad es causada principalmente por la raza 3, bv 2A, Filotipo II y sequevar 1, adaptada tanto a climas calurosos como fríos (menor a 18 °C); así como a grandes altitudes (mayor a 2500 m). En climas fríos, la bacteria se desarrolla muy lentamente o permanece en estado de latencia dentro de la planta y tubérculos infectados, sin que los síntomas sean notados (Boletín fitosanitario 2006), convirtiéndose en un peligro potencial, porque la semilla de papa infectada constituye el medio principal para la diseminación de R. solanacearum cuando los tubérculos infectados con esta bacteria se siembran en lugares cálidos (Aley et al. 1999). Es así, que pueden producirse severos brotes de la enfermedad ocasionando una reducción de hasta el 90% del rendimiento del cultivo y pérdidas en almacenamiento hasta un 98% (SENASA 2005). Además de provocar la pérdida del tubérculo y la planta, la bacteria sobrevive en el suelo, por lo que el campo debe ser abandonado para este cultivo (Borba 2008). Por otro lado, este microorganismo presenta una gran diversidad genética (Yu et al. 2003), un amplio rango de hospederos (Allen et al. 2004), una extensa distribución geográfica y una alta patogenicidad lo cual ha permitido la clasificación de la bacteria en razas y biovares (French 1965), sin embargo un nuevo sistema de clasificación para R. solanacearum fue propuesto por Fegan y Priorenel 2005, basado en el análisis de la secuencia espaciadora interna (ITS por sus siglas en inglés) dividiéndolo en cuatro grupos genéticos, o Filotipos, cada uno de los cuales refleja el origen geográfico de la cepa. (Fegan & Prior 2005).
La virulencia y los factores de patogenicidad de R. solanacearum son transcripcionalmente controlados por una extensa red de caminos distintos, que interactúan en la vía de transducción de señales. El núcleo de esta red es el sistema sensorial de cinco genes phc (phenotype conversion) (Schell 2000). PhcA es el factor central en una compleja cascada de regulación que activa la expresión de numerosos genes que codifican factores de virulencia tales como EPS I y varias exoproteínas, causando marchitez al restringir el flujo de agua a través del xilema y facilitando la invasión y la colonización vascular. Ralstonia solanacearum posee un sistema quorum sensing para la regulación de la expresión de estos genes de virulencia, siendo la molécula 3-hydroxy-palmitic acid methyl ester (3-OH-PAME) el auto regulador de esta red (Flavier et al. 1997). Esta característica convierte a la molécula 3-OH-PAME, en un potencial blanco para el desarrollo de inhibidores de los factores de virulencia (Shinohara et al. 2007). El sistemade quorumquenching (interceptación del quorum) actúa bloqueando distintos pasos implicados en quorum sensing, tales como la generación del agente autoinductor, la acumulación del agente señal, o bien la recepción de la señal (Dong et al. 2007). Shinohara et al. (2007) purificaron y caracterizaron la molécula β-Hydroxypalmitate methyl ester hydrolase (βHPMEH), aislada a partir de Ideonella sp. La enzima βHPMEH hidroliza a la molécula 3-OH-PAME en 3-OH ácido palmítico y metanol, y anula así la señal quorum de R. solanacearum. Debido a que se conoce poco acerca del gen βhpmeh y la resistencia que potencialmente puede conferir este gen expresado en plantas de papa, el presente trabajo evaluó la expresión del gen βhpmeh in vivo, cuya enzima del mismo nombre es inhibidora de la señal quórumsensing 3-OH-PAME, señal responsable de la inducción de genes de virulencia en R. solanacearum.
Materiales y métodos
Material vegetal.- Las plántulas de papa variedad Desiree fueron obtenidas del banco de germoplasma del Centro Internacional de la Papa (CIP) (accesión CIP800048). Las plántulas de Nicotiana benthamiana, fueron suministradas por el área de virología del CIP para la estandarización de la técnica de agroinfiltración.
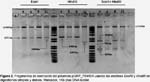
Diseño de plásmidos.-Elgen βhpmeh y el promotor GRP1.8 fueron sintetizados por la compañía Intelechon (The Synthetic Genes Company, Alemania), ambas secuencias fueron clonadas en el sitio de clonamiento múltiple del vector pENo8H. El gen βhpmeh fue sintetizado basado en codones optimizados para plantas; hacia el extremo 5’ del gen se agregaron secuencias reguladoras como la secuencia potenciador PVA 5’UTR que proviene del virus A de papa (PVA por sus siglas en inglés, género Potyvirus) la cual potenciará la traducción del gen βhpmeh. Adicionalmente en el extremo 5’ del gen se agregó la secuencia del péptido señal GRP y que posteriormente dirigirá la proteína hacia el espacio extracelular. El gen optimizado βhpmeh seclonó en el sitio SmaI del vector pBIN61 (Bendahmane et al. 2002) el cual contiene el promotor 35S del virus del mosaico de la coliflor (CaMV, género Caulimovirus) y el gen nptII que codifica para una neomicina fosfotransferasa responsable de la resistencia al antibiótico kanamicina usado como agente de selección, la construcción final se denominó pPAMEH (Fig. 1).
Para la construcción del segundo plásmido se removió el promotor 35S del vector pBIN61 con la enzima EcorI, para clonar en aquel sitio la secuencia híbrida GRP1.8-PAMEH obtenida por PCR de extensión por solapamiento, el cual consistió en amplificar independientemente el promotor GRP1.8 usando los iniciadores EcoRV-GRP1.8 Fw: GATATCGCTTCCCTCTTAGG y GRP1.8 Rv: GTAGTTTGTTTATTTTTATCCCACTTAAAG y el gen optimizado βhpmeh usando los iniciadores GRP1.8_ PVA_SP_PAME Fw: CTTTAAGTGGGATAAAAATAAACAAAC y EcoRV_PAME Rv: GATATCCACCTTATTGGGCG y luego unirlas en una segunda reacción de PCR usando los iniciadores EcoRV-GRP1.8 Fw y EcoRV_PAME Rv (Sambrook & Russel 2006), a este segundo plásmido se le denominó pGRP_PAMEH (Fig. 1). La verificación del clonamiento y la orientación de los fragmentos se llevó a cabo mediante digestión con las enzimas EcoRI y HindIII y se usó el secuenciamiento de ADN para comprobar la integridad de las secuencias clonadas.
Agroinfiltración.-Se sembró Agrobacterium tumefaciens conteniendo el plásmido pPAMEH en caldo Luria Bertani (LB) y se incubó a 28 °C por 2 días. Seguidamente, se inoculó 300 µL del cultivo en 50 mL de medio LB suplementado con 5 µL de acetosiringona (200mM) y se incubó toda la noche a 28°C. Posteriormente, las células fueron precipitadas por centrifugación y resuspendidas en solución tampón de agroinfiltración (MgCl210 mM, acetosiringona 150 µM); ajustando la concentración final a una Densidad Óptica (DO) de 0.25 a 620 nm. Los cultivos fueron incubados a temperatura ambiente por 3 horas antes de la infiltración. La infiltración con las suspensiones bacterianas se hizo con la ayuda de una jeringa estéril (sin aguja) y en el envés de toda la hoja.
Infección con R. solanacearum.-La cepa 204 (raza 3 biovar 2A, Filotipo II y sequevar 1) de R. solanacearum fue sembrada en medio Kelman e incubada a 28 °C por 2 días, al cabo de los cuales se agregó agua destilada estéril y se removió la masa bacteriana con la ayuda de un hisopo estéril. Las células bacterianas fueron resuspendidas en solución tampón de agroinfiltración hasta llegar a una concentración final correspondiente a una DO de 0.1 a 600 nm, que corresponde a 2x108 UFC por mL. La infiltración en la hoja fue similar a la agroinfiltración con la única diferencia de que sólo se infiltró una porción de la hoja. Las plantas después de la infiltración fueron mantenidas a 22 ±1 °C y la evaluación se realizó a partir de la aparición de síntomas.
Determinación de la población de R. solanacearum.- A las 0, 12 y 24 horas post infección de cada tratamiento (Rs control, Agro-pPAMEH+Rs, Agro-pBIN61+Rs,yAgro+Rs)secolectaron dos hojas, tomando un disco de aproximadamente 0.5 cm de diámetro por hoja con un tubo eppendorf de 1.5 mL, usándolo a manera de sacabocado. Se añadió a cada tubo 500 µL de agua destilada estéril y se trituró los discos de hoja con un asa de siembra estéril, luego se realizaron cinco diluciones seriadas (1:10). Se sembró en el medio Kelman 5 µL de cada una de las diluciones por cuadriplicado y se incubó a 28 °C por dos días, al cabo de los cuales se contó el número de colonias y se reportó como unidades formadoras de colonias (UFC).
Aislamiento de ARN.- El ARN total fue aislado a partir de hojas colectadas al tercer día de la agroinfiltración usando el método de extracción con Trizol (Reagent-INVITROGEN)y digiriendo el ADN con DNase TURBO (Ambion). La calidad del ARN extraído fue visualizada bajo luz UV sobre un gel de agarosa al 2% teñido con GelRed (Biotium). El ARN aislado fue convertido en ADNc por la transcriptasa reversa M-MLV (Promega)de acuerdo a las instrucciones del fabricante. Se realizó un RT-PCR para la detección del ARNm del gen βhpmeh, para ello se usaron los iniciadores PAHME Fw: GGGCGAAGTT-GGCAAAATATCC y pameh Rv: GATCAGAGCTACCCAGGGAAGTG. El volumen final de la reacción de PCR fue de 25 µL, conteniendo 25 ng de ADNc, 1X PCR buffer, 0.2 µM de cada cebador, 0.2 µM dNTPs y 1 U de la enzima GoTaq DNA polimerasa (Promega). El programa de PCR usado fue: 95 °C por 2 min; 35 ciclos de 94 °C por 30seg, 55 °C por 30 segundos y 72 °C por 15 segundos; y una elongación final de 72 °C por 5 minutos. La amplificación se realizó en el termociclador Veriti (Applied Biosystems).
Extracción de proteínas totales.- Se extrajo las proteínas totales a partir de hojas de N. benthamiana a las 72 h después de la agroinfiltración. Se infiltró las hojas con tampón de extracción (Tris-HCl 0.05, DDT 1 mM, EDTA 1 mM), en seguida las hojas fueron colocadas en una jeringa sin émbolo de 5 mL y esta dentro de un tubo Falcon de polipropileno de 14 mL, luego se centrifugó a 3000 rpm por 10 minutos. El líquido intercelular que se colectó en los tubos fue inmediatamente transferido a tubos de 1.5 mL y congelados a –20 °C. Para el análisis de SDS-PAGE se agregó a cada muestra tampón de carga (Glicerol 10%, Tris-HCl 50mM, azul de bromofenol 0.2%, SDS 2%, β-Mercaptoetanol), se hirvieron por 5 minutos y finalmente fueron inmediatamente colocados en hielo para evitar su renaturación. El gel de acrilamida está compuesto de dos sectores: el gel concentrador (4% de acrilamida) y el gel separador (15% de acrilamida). La corrida se realizó entre 60 – 70 Voltios(V) durante dos horas hasta que las muestras se apilaron y llegaron todas al gel separador se subió el voltaje a 100 V y se dejó correr aproximadamente 3 horas, terminada la corrida se realizó la tinción del gel con una solución de azul de coomassie (0.025%).
Resultados y discusión
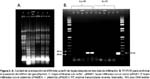
Verificación de los vectores de transformación.- La digestión del plásmido pPAMEH con la enzima HindIII generó los fragmentos esperados de tamaños 12145pb y 2398pb. De igual forma, el plásmido pGRP_PAMEH generó dos fragmentos de 12359 pb y 2402 pb al ser cortado con la enzima de restricción HindIII, mientras que la digestión doble con EcoRI y HindIII generó 4 fragmentos de restricción de 11726, 1638, 764 y 633 pb (Fig. 2). La integridad de los promotores y la secuencia del gen βhpmeh fueron confirmadas por secuenciación. Las secuencias fueron ensambladas y posteriormente analizadas por un alineamiento local con BLASTX, los resultados del alineamiento muestran que nuestra secuencia peptídica tiene una identidad del 99% con respecto a la secuencia de la proteína βHPMEH de Ideonella sp. FERM BP-08660 (accesión: BAF64544.1) esto debido a que nuestra secuencia tiene una sustitución de nucleótido que llevó a la codificación de una Prolina (P) a una Serina (S), sin embargo esta no se situó dentro del sitio activo y por lo tanto puede que no afecte su actividad enzimática (datos no mostrados). Además, se comprobó que la secuencia de la proteína βHPMEH conserva el motivo consenso G-X-S-X-Gentretodas las serine esterasas (Brenner 1988, Shinohara et al. 2007) y que el motivo conservado se encuentra ubicado cercano al centro de toda la secuencia proteíca. Además ésta es una característica importante en enzimas que poseen motivos conteniendo Serina (Akoh et al. 2004).
Expresión del gen βhpmeh.-Se detectó en las muestras infiltradas con pPAMEH un transcrito de 241 pb (Fig. 3B). Las hojas infiltradas con el vector pBIN61 (control negativo) no mostraron ninguna señal. El resultado por lo tanto indicaría que el gen clonado está siendo expresado apropiadamente a nivel transcripcional. No obstante, no se pudo evidenciar expresión a nivel traduccional pues en el SDS-PAGE no se pudo observar claramente un fragmento del peso molecular esperado correspondiente al de la proteína βHPMEH. Debido al uso de un extracto crudo de proteínas (proteínas totales que se encuentran en la hoja), posiblemente no se pudo diferenciar el péptido esperado debido a la gran cantidad de proteínas extraídas que enmascararon su visualización en el gel, debido al uso de un crudo de proteínas (proteínas totales que se encuentran en la hoja). Por otra parte, la ausencia de la banda esperada pudo deberse al bajo nivel de expresión del producto final pues expresión transitoria de la proteína cesa al cabo de 2 ó 3 días (Menassa et al. 2012).
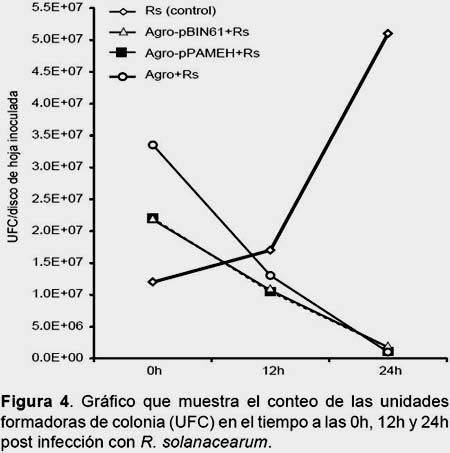
Determinación de la población de R. solanacearum.- En la Figura 4 se puede observar que la población de R. solanacearum se incrementó en el transcurso del tiempo, sin embargo, no se pudo apreciar si pPAMEH tuvo algún efecto en la población de R. solanacearum ya que en los tres tratamientos que incluían Agrobacterium se apreció una marcada disminución de la población, por lo tanto no se le puede atribuir esta disminución de la población de R. solanacearum al gen βhpmeh, lo que podría ser un efecto que está ejerciendo la bacteria A. tumefaciens sobre la bacteria R. solanacearum, probablemente debido a algún tipo de competencia por espacio y nutrientes o de cierta interacción bacteria-bacteria (Cano 2011).
Infección con R. solanacearum.- En las hojas infiltradas con Agrobacterium conteniendo el plásmido pPAMEH y que fueron infectadas a los tres días con R. solanacearum, se observó un retardo en la aparición de síntomas. La clorosis y la marchitez aparecieron a los 10 y 12 días respectivamente; en cambio, en el control inoculado con R. solanacearum éstos síntomas aparecieron a los 4 días y 6 días respectivamente (Fig. 5). Este retraso, de 6 días en la aparición de síntomas de marchitez bacteriana, nos lleva a concluir un cierto nivel de tolerancia conferida por el gen βhpmeh, debido a que la acción enzimática de esta proteína interfiere con la comunicación intercelular entre las células de R. solanacearum, anulando así la señal de virulencia y evitando por tanto la conversión fenotípica al modo patogénico. Esto explicaría la tardía aparición de los síntomas característicos de la enfermedad. Según los resultados del conteo de las UFC de R. solanacearum, el gen por sí solo no afectaría el crecimiento de la población bacteriana, es decir no afectaría directamente a la supervivencia del patógeno (Romero & Otero 2010), pero el gen sí afectaría a la expresión de los factores de virulencia, lo cual concuerda con el retraso de la sintomatología de la marchitez bacteriana hallado en el presente trabajo.
El uso de la estrategia quorum quenching ha sido aplicada con éxito por diversos autores para la interferencia de la comunicación bacteriana (Dong & Zhang 2005, Park et al. 2005, Dong et al. 2007, Williams et al. 2007), ésta estrategia tendría cierta ventaja ya que no debería ejercer presión selectiva sobre el patógeno, evitando así la aparición de resistencias (Romero & Otero, 2010). Sin embargo, sería recomendable combinar este mecanismo con otros genes de resistencia que sí afecten el crecimiento de la bacteria, ya que como se demostró en este trabajo el gen βhpmeh inhibió parcialmente la patogenicidad, pero no afectó el crecimiento de la bacteria, quedando esta como infección latente en el tejido del hospedero. Por lo tanto, la combinación con genes de resistencia constituiría una estrategia mucho más interesante en el tratamiento de la marchitez bacteriana. Genes como el de la lactoferrina que expresado en plantas de tabaco (Nicotiana tabacum) retrasa significativamente los síntomas de R. solanacearum e inhiben la multiplicación de la bacteria (Mitra & Zhang 1994, Zhang et al. 1998) podría ser usado como candidato. De igual forma estudios previos demostraron que la proteína ferredoxina incrementa la resistencia a la marchitez bacteriana en tomate (Lycopersicum esculentum) y Arabidopsis (Arabidopsis thaliana); y que la población bacteriana fue mucho más baja en los tratamientos con ferredoxina que en los controles (Huang et al. 2007, Lin 2010). Así mismo estudios con el gen cecropina b en tomate han mostrado que la expresión de este péptido aumenta la resistencia a la marchitez bacteriana y además inhibe el crecimiento del patógeno in vitro (Jan et al. 2010). La expresión del EFR, un receptor de reconocimiento de patrones de patógenos, en plantas de Nicotiana (N. benthamiana) y tomate igualmente mostraron una reducción drástica de los síntomas de marchitez (Lacombe et al. 2010). Por consiguiente, los resultados demuestran que la actividad Quorum quenching de la enzima βHPMEH es una estrategia viable para contrarrestar parcialmente la patogenicidad de R. solanacearum y su expresión estable en plantas combinado con otros genes de resistencia ofrecería oportunidades interesantes para el control de la enfermedad.
Literatura citada
Aley P., E. Chujoy, E. French, B. Lemaga & S. Priou. 1999. Control integrado de la marchitez bacteriana de la papa: Guía de diapositivas. Centro Internacional de la Papa. Series de Diapositivas del CIP para Capacitación IV: 3.
Allen C., P. Prior & A. Hayward. 2004. Bacterial wilt disease and the Ralstonia Solanacearum. Species complex. American Phytopathological Society eds., St. Paul (Minnesota) Pp.29-38.
Bendahmane A., G. Farnham, P. Moffett & D. Baulcombe. 2002. Constitutive gain-of-function mutants in a nucleotide binding site–leucine rich repeat protein encoded at the Rx locus of potato. The Plant Journal 32: 195–204.doi: http://dx.doi.org/10.1046/j.1365-313X.2002.01413.x
Boletín Fitosanitario. 2006. Podredumbre parda de la patata o marchitez bacteriana (Ralstonia Solanacearum (Smith) Yabuuchi et al). Junta de Castilla y León, Valladolid. Pp.2-7.
Borba N. 2008. La papa un alimento básico. Posibles impactos frente a la introducción de papa transgénica. RAP-AL eds. Montevideo, Uruguay. Pp. 1-11.
Brenner S. 1988. The molecular evolution of genes and proteins: a tale of two serines. Nature 334: 528-530.doi: http://dx.doi.org/10.1038/334528a0
Cano M. 2011. Interacción de microorganismos benéficos en Plantas: Micorrizas, Trichoderma spp. y Pseudomonas spp. U.D.C.A Actualidad & Divulgación Científica 14 (2): 15–31.
Akoh C., L. Guan-Chiun, L. Yen-Chywan, H. Tai-Huang & Jei-Fu. 2004. GDSL family of serine esterases/lipases. Progress in Lipid Research 43(6): 534-552.doi: doi:10.1016/j.plipres.2004.09.002
Dong Y. & H. Zhang. 2005. Quorum Sensing and Quorum-Quenching Enzymes. Journal of Microbiology 43: 101-109.
Dong Y., L. Wang, & L. Zhang. 2007. Quorum quenching microbial infections: mechanisms and implications. Philosophical transactions of the Royal Society of London. Series B, Biological sciences 362: 1201-1211. Doi: http://dx.doi.org/10.1098/rstb.2007.2045
Fegan M. & Prior P. 2005. How complex is the ‘Ralstonia solanacearum’ species complex? American Phytopathological Society 449-461.
Flavier A., S. Clough, M. Schell & T. Denny. 1997. Identification of 3-hydroxypalmitic acid methyl ester as a novel autoregulator controlling virulence in Ralstonia solanacearum. Molecular Microbiology 26(2):251-259. doi:http://dx.doi.org/10.1046/j.1365-2958.1997.5661945.x
French, E. 2006. Interaction between strains of Ralstonia solanacearum, its host and the environment. International Workshop PCARRD. Proceedings The Philippines. Pp. 94-104.
Jan P., H. Hsu-Yuang & C. Hueih-Min. 2010. Expression of a Synthesized Gene Encoding Cationic Peptide Cecropin B in Transgenic Tomato Plants Protects against Bacterial Diseases. Applied and Environmental Microbiology 76(3):769.doi: http://dx.doi.org/10.1128/AEM.00698-09
Huang H., C. Liu, M. Lee, et al. 2007. Resistance enhancement of transgenic tomato to bacterial pathogens by the heterologous expression of sweet pepper ferredoxin-I protein. Phytopathology 97:900-906.doi: http://dx.doi.org/10.1094/PHYTO-97-8-0900
Lacombe, S., A. Rougon-Cardoso, E. Sherwood, et al. 2010. Interfamily transfer of a plant pattern-recognition receptor confers broad-spectrum bacterial resistance. Nature Biotechnology. 28: 365–369. doi:http://dx.doi.org/10.1038/nbt.1613
Lin Y., H. Hsiang-En, W. Fang-Sheng, et al. 2010. Plant ferredoxin-like protein (PFLP) outside chloroplast in Arabidopsis enhances disease resistance against bacterial pathogens. Plant Science 179: 450–458.doi: http://dx.doi.org/10.1016/j.plantsci.2010.07.006
Menassa R., A. Ahmad & J. Joensuu. 2012. Transient expression using Agroinfiltration and its aplications in molecular farming. Molecular Farming in Plants: Recent Advances and Future Prospects, Springer, Netherlands. Pp.183–198. doi: 10.1007/978-94-007-2217-0_9
Mitra A. & Z. Zhang. 1994. Expression of a human lactoferrin cDNA in tobacco cells produces antibacterial protein(s). Plant Physiology. 106:977-981.doi: http://dx.doi.org/10.1104/pp.106.3.977
Patron N. 2014. DNA assembly for plant biology: techniques and tools. Current Opinion in Plant Biology 19:14–19.doi: http://dx.doi.org/10.1016/j.pbi.2014.02.004
Park S., H. Kang, O. Jang, et al. 2005. Identification of extracellular N-acylhomoserine lactone acylase from a Streptomyces sp. and its application to quorum quenching. Applied and Environmental Microbiology 71: 2632-2641.doi: http://dx.doi.org/10.1128/AEM.71.5.2632-2641.2005
Romero M. & A. Otero. 2010. Interceptación de señales de comunicación bacteriana en bacterias aisladas del medio marino. Revista de la Real Academia de Ciencias Exactas, Físicas y Naturales (España) XXIX: 129-206.
Sambrook J. & D. Russel. 2006. Molecular Cloning: A Laboratory Manual. Cold Spring Harbour Laboratory Eds. New York. Tercera edicion. 628 pp.
Schell M. A. 2000. Control of Virulence and pathogenicity genes of Ralstonia solanacearum by an elaborate sensory network. Annual Review of Phytopathology 38:263–92. doi: http://dx.doi.org/10.1146/annurev.phyto.38.1.263
SENASA, Servicio Nacional de Sanidad Agraria. 2005. (en línea) Marchitez bacteriana de la papa en línea. Peru. http://www.senasa.gob.pe/sanidad_vegetal/programas_%20fitosanitarios/cip_papa/aspectos_generales.html
Shinohara M., N. Nakajima & Y. Uehara. 2007. Purification and characterization of a novel esterase (b-hydroxypalmitate methyl ester hydrolase) and prevention of the expression of virulence by Ralstonia solanacearum. Journal of Applied Microbiology 103: 152–162.doi: http://dx.doi.org/10.1111/j.13652672.2006.03222.x
Williams P., K. Winzer, W. Chan & C. Cámara. 2007. Look who’s talking:communicationandquorumsensinginthebacterial world. Philosophical transactions of the Royal Society of London. Series B, Biological sciences 362: 1119-1134.doi: http://dx.doi.org/10.1098/rstb.2007.2039
Yu Q., M. Alvarez, P. Moore, F. Zee, et al. 2003. Molecular Diversity of Ralstonia solanacearum Isolated from Gingerin Hawai. Bacteriology, 93 (9): 1124-1130. doi: 10.1094/ PHYTO.2003.93.9.1124.
Zhang Z., D. Coyne, A. Vidaver, & A. Mitra. 1998. Expression of human lactoferrin cDNA confers resistance to Ralstonia solanacearum in transgenic tobacco plants. Phytopathology 88:730-734. doi:http://dx.doi.org/10.1094/PHYTO.1998.88.7.730
Información sobre los autores:
EF, LG, JK: realizaron el diseño experimental; EF, LG, JK: realizaron los experimentos; EF, LG, JK: analizaron los datos; EF, LG, JK: redactó el manuscrito; EF, LG, JK: revisaron y aprobaron el manuscrito. Los autores no incurren en conflictos de intereses.
Autor para correspondencia
E-mail Elizabeth Fernandez: e.fernandez@cgiar.org
E-mail Liliam Gutarra: l.gutarra@cgiar.org
E-mail Jan Kreuze: j.kreuze@cgiar.org
Presentado: 09/02/2015
Aceptado: 23/04/2015
Publicado online: 14/10/2015