Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Medica Herediana
versión impresa ISSN 1018-130Xversión On-line ISSN 1729-214X
Rev Med Hered vol.30 no.4 Lima oct./dic 2019
http://dx.doi.org/10.20453/rmh.v30i4.3659
INVESTIGACIÓN ORIGINAL
β-lactamasas de espectro extendido tipo CTX-M en aislamientos clínicos de Escherichia coli y Klebsiella pneumoniae en el Instituto Nacional de Salud del Niño-Breña, Lima, Perú
Extended spectrum type CTX-M β-lactamases in clinical infections caused by Escherichia coli y Klebsiella pneumoniae at the Instituto Nacional de Salud del Niño-Breña, Lima, Peru
Edgar Gonzales 1,2,a,b, Lilian Patiño 1,c, Elsa Ore 1,a,Violeta Martínez 1,a, Silvia Moreno 1,a, Norka Beatriz Cruzado 1,a, Roberto Rojas 1,3,a,d, María del Carmen Quispe 1,4,a,e,f, Ivonne Carbonell 1,a, Freddy Villarreal 1,a, Giovanna Maza1,a, José Olivo 1,a, Raúl Vicuña 1,a, Daniel Bustamante 1,a
1 Instituto Nacional de Salud del Niño-Breña. Lima, Perú.
2 Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Universidad Nacional Federico Villarreal. Lima, Perú.
4 Universidad Peruana Cayetano Heredia. Lima, Perú.
a Tecnólogo Médico, b Magister en Microbiología, c Medico Patólogo Clínico, d Magister en Gestión en Salud, e Magister en docencia, f Segunda Especialidad en Microbiología
RESUMEN
Objetivo: Determinar la frecuencia de β-lactamasas de espectro extendido (BLEE) en Escherichia coli y Klebsiella pneumoniae y la frecuencia de CTX-M en las productoras de BLEE en el Instituto Nacional de Salud del Niño - Breña (INSN-B). Material y métodos: Se analizaron enterobacterias productoras de BLEE del INSN-B entre los meses de agosto de 2012 y enero del 2013. Se incluyeron 724 aislamientos de Escherichia coli y 181 aislamientos de Klebsiella pneumoniae, consecutivos no repetidos, de pacientes hospitalizados y de la comunidad. La identificación se realizó por bioquímica convencional. la detección fenotípica de BLEE se hizo por el método de Jarlier y la detección genotípica de CTX-M mediante reacción en cadena de la polimerasa (PCR). Resultados: 281 (31%) de los aislamientos de ambas enterobacterias fueron productoras de BLEE; 207/724 (28,6%) E. coli y 74/181 (40,9%) K. pneumoniae. Se detectó el gen bla en 256 de los aislamientos productores de BLEE (91,1%). Conclusiones: Las BLEE de tipo CTX-M están presentes en nuestra institución, a pesar que nuestros datos representan una sola institución, brinda parte del panorama nacional sobre la resistencia a los antimicrobianos; por lo tanto, el enfoque de epidemiológico molecular es importante para desarrollar más y mejores estrategias de control y manejo de estos patógenos en nuestro país.
PALABRAS CLAVE: Beta-lactamasas, Escherichia coli, Klebsiella pneumoniae, pediatría. (Fuente: DeCS BIREME).
SUMMARY
Objective: To determine the frequency of extended spectrum β-lactamases (ESBL) in clinical infections caused by Escherichia coli y Klebsiella pneumoniae and the frequency of CTX-M among them at the Instituto Nacional de Salud del Niño-Breña, Lima, Peru. Methods: ESBL producing strains of E. coli and K. pneumoniae collected from August 2012 and January 2013 were analyzed; a total of 724 E. coli and 181 K. pneumoniae consecutive, non- repeated isolates from community and hospital acquired infections were included. Identification was performed by conventional biochemistry, ESBL phenotype was detected following the Jarlier´s method and PCR was used to detect CTX-M. Results: overall prevalence of ESBL was 31% (281 strains); 207/724 (28.6%) E. coli and 74/181 (40.9%) K. pneumoniae. The bla gene was detected in 256 of ESBL producing strains (91.1%). Conclusions: The CTX-M phenotype of ESBL producing strains is present in our institution. Despite of showing information of a single institution, these data bring a glance of what the antimicrobial resistance pattern may be at a national level and underscores the utility of molecular biology in designing preventing measures.
KEYWORDS: Beta-lactamases, Escherichia coli, Klebsiella pneumoniae, pediatrics. (Source: DeCS BIREME).
INTRODUCCIÓN
Las β-lactamasas de espectro extendido (BLEE) representan el mecanismo más importante para la resistencia a cefalosporinas de tercera generación en enterobacterias, especialmente en Escherichia coli y Klebsiella pneumoniae; estas son derivadas a partir de mutaciones puntuales de las β-lactamasas de espectro ampliado (BLEA) TEM-1, TEM-2 y SHV-1. Hasta la fecha, se han documentado más de 10 familias asociadas con BLEE, incluyendo CTX-M, SHV, TEM, PER, VEB, BES, GES, TLA, SFO y OXA. Sin embargo, son las enzimas CTX-M predominantes a nivel mundial; estas tienen principalmente actividad sobre la cefotaxima y no sobre la ceftazidima; aunque en los últimos años la emergencia de variantes de CTX-M (CTX-M-15, CTX-M-16, CTX-M-27 y CTX-M-19) están mejorando su actividad frente a ceftazidima (1,2).
Los elementos genéticos móviles son fuente importante en la diseminación de genes de resistencia (integrones, transposones y plásmidos). Son capaces de proporcionar plataformas estables para el mantenimiento y la propagación de genes responsables de la resistencia a los antimicrobianos y juegan un papel esencial en la diseminación mundial de resistencia a múltiples drogas entre microorganismos Gram-negativos, especialmente las enterobacterias (3).
La diseminación mundial de microorganismos resistentes a los antimicrobianos es un problema mayor en la atención en salud porque disminuye las opciones disponibles para el tratamiento apropiado.
Esto contribuye a una mayor mortalidad y morbilidad del paciente (4). Por lo tanto, no es sorprendente que la propagación mundial de la resistencia a los medicamentos haya sido reconocida como una gran amenaza para la salud humana (5).
El Clinical and Laboratory Standards Institute (CLSI) publicó directrices para detectar y confirmar fenotípicamente la producción de BLEE en K. pneumoniae, K. oxytoca, y E. coli (6). Para la detección genotípica se puede emplear la amplificación del gen codificante por Reacción en Cadena de la Polimerasa (PCR). La técnica de PCR permite la detección de genes específicos responsables de la producción de las BLEE conforme lo descrito en diversos estudios (7,8).
Ante el creciente aumento mundial y nacional de la resistencia bacteriana, en especial de enterobacterias productoras de BLEE; y el aumento de las infecciones por este tipo de gérmenes, no solo en el ámbito hospitalario sino también en la comunidad, se hace necesario conocer las características de epidemiología molecular de estos aislamientos.
El objetivo del estudio fue determinar la frecuencia de β-lactamasas de espectro extendido (BLEE) en Escherichia coli y Klebsiella pneumoniae y la frecuencia de CTX-M en las productoras de BLEE en el Instituto Nacional de Salud del Niño - Breña (INSN-B).
MATERIAL Y MÉTODOS
Estudio descriptivo sobre la presencia de enterobacterias productoras de BLEE en muestras de pacientes atendidos en el INSN-B entre los meses de agosto de 2012 a enero del 2013. Se incluyeron en el estudio 724 aislamientos de Escherichia coli y 181 aislamientos de Klebsiella pneumoniae, realizados de forma consecutiva, no repetidas, y recuperados de muestras de orina, sangre, secreciones respiratorias, secreciones de heridas, LCR y catéter, provenientes de pacientes hospitalizados en los servicios de cirugía, medicina y la unidad de cuidados intensivos (UCI); y pacientes ambulatorios (de la comunidad) de los servicios de emergencia y consultorios externos.
Detección fenotípica
Los aislamientos de E. coli y K. pneumoniae fueron obtenidos según el procedimiento estándar de cultivos. La identificación se realizó por pruebas bioquímicas convencionales y las pruebas de susceptibilidad por el método disco difusión, siguiendo los lineamientos del CLSI (6). La determinación fenotípica de BLEE se realizó mediante el método de Jarlier (método de sinergia del doble disco) (9).
Detección genotípica
A partir de cultivos de 24 horas de los microorganismos en estudio, se suspendieron de 3 a 4 colonias en 200 μl de agua para PCR Milli-Q. Se sometieron a ebullición durante 15 minutos y se centrifugó durante 2 minutos a 12 000 r.p.m. para descartar los restos celulares. Se conservó el sobrenadante a -20°C.
La detección genotípica de BLEE tipo CTX-M se realizó a partir de ADN total, mediante amplificaciones de PCR. Los cebadores usados para la obtención del gen blaCTX-M fueron; (5’-TTTGCGATGTGCAGTACCAGTAA-3’) y (5’-CGATATCGTTGGTGGTGCCAT-3’) correspondiente a 544 pares de bases; la amplificación se realizó en las siguientes condiciones; desnaturalización inicial a 94ºC por 7 min; seguido de 35 ciclos a 94ºC por 50 seg; 52ºC por 50 seg; 72ºC por 1min y extensión final a 72ºC durante 5 min (10).
Los productos de amplificación fueron resueltos por electroforesis en agarosa al 1% conteniendo bromuro de etidio (0,5 μg/ml). Para ello, se mezcló 9 μl de cada producto con 1μl de buffer de carga de DNA 10x y se sembró todo el volumen en el gel. Se incluyeron marcadores de peso molecular. Las bandas resultantes se visualizaron con un transiluminador UV. Se usaron como controles cepas de referencia del Laboratorio de Epidemiología Molecular y Genética del CITBM-UNMSM.
Análisis de datos
Se realizó un estudio descriptivo univariado de cada una de las muestras en estudio, clasificándolas en productoras o no productoras de β-lactamasas. Para la determinación de los porcentajes de resistencia y sensibilidad antimicrobiana de las especies de enterobacterias en estudio, los datos obtenidos fueron introducidos en MS Excel 2010 y en el programa WHONET (World Health Organization Net v. 5.6), programa estadístico utilizado por la Organización Panamericana de la Salud para la vigilancia de la resistencia bacteriana, que permitió analizar datos epidemiológicos y las características del germen en estudio.
Aspectos éticos
El protocolo del estudio fue aprobado por el Comité de Ética del INSN-B.
RESULTADOS
Se incluyeron 905 aislamientos, 560/905 (61,9%) de las muestras provenían de pacientes de la comunidad y 345/905 (38,1%) de pacientes hospitalizados. De los comunitarios 494/560 (88,2%) fueron E. coli y 66/560 (11,8%), K. pneumoniae. De los hospitalizados 230/345 (66,7%) fueron E. coli y 115/345 (33,3%), K. pneumoniae.
Escherichia coli y Klebsiella pneumoniae productoras de BLEE
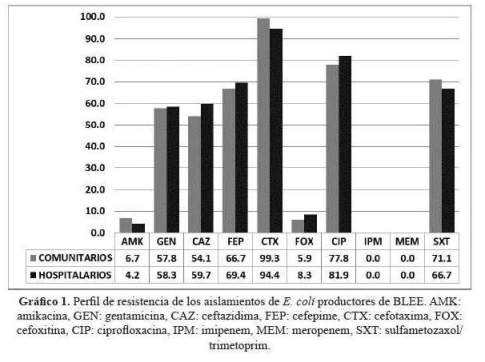
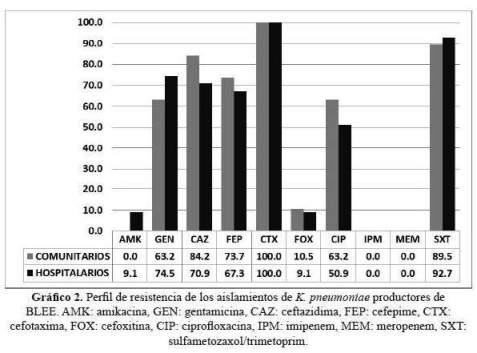
Doscientos ochenta y uno (31%) de los aislamientos de ambas enterobacterias fueron productoras de BLEE; 207/724 (28,6%) de E. coli y 74/181 (40,9%) de los aislamientos de K. pneumoniae fueron productores de BLEE. De las E. coli productora de BLEE 135/207 (65,2%) fueron de la comunidad y 72/207 (34,8%) hospitalarias; en el caso de K. pneumoniae productora de BLEE 19/74 (26,7%) fueron de la comunidad y 55/74 (74,3%) hospitalarias. Los perfiles de resistencia de E. coli y K. pneumoniae productores de BLEE de los aislamientos comunitarios y hospitalarios muestran resistencia acompañante a fluoroquinolonas, sulfametoxazol/trimetoprima y gentamicina, manteniéndose activos los carbapenémicos y amikacina (gráfico 1 y gráfico 2).
Análisis genotípico mediante PCR
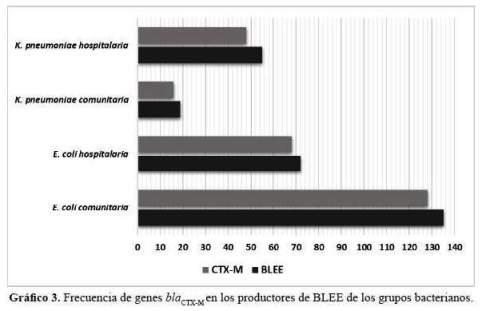
La caracterización molecular utilizando cebadores específicos blaCTX-M detectó su presencia en 256 de los aislamientos productores de BLEE (91,1%). En los aislamientos de E. coli de origen hospitalario 68/72 (94,4%) y en 124/135 (91,8%) de la comunidad presentaron el gen blaCTX-M. En el caso de K. pneumoniae hospitalarias 48/55 (87,2%) y en la comunitarias 16/19 (84,2%) evidenció la presencia del gen blaCTX-M (gráfico 3).
DISCUSIÓN
Las enzimas de tipo CTX-M son consideradas las BLEE más importantes que emergen en las enterobacterias en todo el mundo. La mayoría de aislamientos que producen enzimas de tipo CTX-M han sido implicadas en infecciones nosocomiales, pero a diferencia de lo que sucede con las BLEE tipo SHV y TEM, estas enzimas también se han informado en aislamientos clínicos de infecciones adquiridas en la comunidad (11).
En nuestro estudio, la frecuencia de productores de BLEE en aislamientos de muestras hospitalarias fue 34,8% en E. coli y 74,3% en K. pneumoniae; en 2005 en un estudio realizado en dos hospitales generales, se encontraron frecuencias de 2,9% y 44,4% para E. coli y K. pneumoniae, respectivamente (12). En el Instituto de Enfermedades Neoplásicas de Lima, en 2005 se reportó una frecuencia de 40,78% en aislamientos de E. coli y K. pneumoniae (13). En 2012 la publicación de García et al. (14), mostró que el 75,1% de E. coli y el 76,8% K. pneumoniae, de aislamientos recuperados de bacteriemias eran productores de BLEE.
La mayoría de estudios de prevalencia de BLEE en el Perú fueron realizados en aislamientos hospitalarios (12,13), no existen muchos datos referidos a aislamientos comunitarios y su resistencia a los antibióticos. Colquechagua et al. (15), en un estudio de colonización con muestras fecales en el INSN, encontraron que el 64,2% eran portadores de enterobacterias productoras de BLEE y de estas el 86% eran E. coli.
En otro estudio de portadores puede observarse un incremento de la resistencia bacteriana al paso de los años, según lo reportado por Pallechi et al. (16,17), en Perú y Bolivia donde detectaron enterobacterias productoras de BLEE en 0,1% de las muestras fecales de niños sanos de cuatro centros urbanos de América Latina; la caracterización molecular reveló la presencia del gen blaCTX-M. El año 2005 en la misma población de niños sanos en Perú y Bolivia reveló el incremento de portadores fecales de E. coli, en comparación al 2002 (0,1% el año 2002 y 1,7% en el 2005).
En 2013, Salles et al., encontraron que en América Latina más de un cuarto de aislamientos de E. coli (27%) y más de un tercio de K. pneumoniae (38%) fueron productores de BLEE. La prevalencia de productores de BLEE en E. coli y Klebsiella spp. asociado con infecciones adquiridas en la comunidad, en particular, fue 29% (18).
Hasta fines del siglo pasado, las enterobacterias albergaban variables de BLEE tipo TEM (Temoneira) y SHV (sulfhidrilo variable) en los aislamientos nosocomiales, y CTX-M (cefotaximasa) era rara vez aislado (19-21). Sin embargo, en los primeros años de este siglo, América Latina se convirtió en el primer continente donde CTX-M comenzó a desplazar a TEM y SHV como el tipo más común de BLEE, principalmente en E. coli (20).
Dentro del genotipo aislado en el presente estudio, las BLEE de tipo CTX-M se presentaron en 91,1% de los casos, resultados que difieren de los publicado por Arce et al. (22), quien en 2014 analizó 50 aislamientos de E. coli en un hospital de Chiclayo; los resultados mostraron que el gen mayormente encontrado fue SHV con 44%, seguido de CTX-M con 20% y TEM con 16%, cabe mencionar que esta investigación se realizó en aislamientos hospitalarios, dentro de este mismo grupo bacteriano (E. coli), resultados que difieren de los encontrados en nuestro estudio.
Referente a los resultados de los aislamientos comunitarios, la mayoría de estudios realizados muestran un predominio de BLEE tipo CTX-M presente en E. coli. En nuestra investigación las cepas de E. coli de la comunidad presentaban el gen blaCTX-M en el 91,8% de los casos, y en K. pneumoniae el 84,2% presentaron blaCTX-M. Galvan et al. (23), encontraron en su estudio una frecuencia del gen blaCTX-M en 55% y la coexistencia de gen blaCTX-M y blaTEM en 24% en aislamientos comunitarios de E. coli en orina; otro estudio realizado en Perú ha evidenciado la presencia de BLEE tipo CTX-M en aislamientos comunitarios sin determinar su frecuencia (24).
Una de limitaciones de nuestro estudio que debemos mencionar es que no se analizaron mecanismos de resistencia diferentes a las BLEE tipo CTX-M, lo que explicaría el 8,9% de aislamientos detectados fenotípicamente como productores de BLEE que no presentaron el gen blaCTX-M. Además, es necesario el secuenciamiento para conocer las variantes alélicas de los genes identificados por PCR, que nos permita saber si existen otros genes de resistencia relacionados.
En conclusión, las enterobacterias productoras de BLEE de tipo CTX-M están presentes en nuestra institución; sin embargo, la información de su distribución e impacto en el país no es bien conocida, a pesar que nuestro datos representan una sola institución, brinda parte del panorama nacional sobre la resistencia a los antimicrobianos; por lo tanto, el enfoque de epidemiológico molecular es importante para desarrollar más y mejores estrategias de control y manejo de estos patógenos en nuestro país.
REFERENCIAS BIBLIOGRÁFICAS
1.Sadeeq R, Tariq A, Ijaz A, Nazir AK, Bo H, Jian G. The Growing Genetic and Functional Diversity of Extended Spectrum Beta-Lactamases. BioMed Research International. 2018;1:14. doi: https://doi.org/10.1155/2018/9519718 [ Links ]
2.Cantón R, González-Alba J, Galán J. CTX-M enzymes: origin and diffusion. Front Microbiol. 2012; 3:110. doi: 10.3389/fmicb.2012.00110 [ Links ]
3.Woodford N, Turton JF, Livermore DM. Multiresistant Gramnegative bacteria: the role of high-risk clones in the dissemination of antibiotic resistance. FEMS Microbiol Rev. 2011; 35(5):736–755. doi: 10.1111/ j.1574-6976.2011.00268 [ Links ]
4. Pana ZD, Zaoutis T. Treatment of extended-spectrum β-lactamase-producing Enterobacteriaceae (ESBLs) infections: what have we learned until now? F1000Res. 2018 Aug 29;7:F1000 Faculty Rev-1347. doi:10.12688/f1000research.14822.1 [ Links ]
5. Infectious Diseases Society of America. The 10 x ‘20 initiative: pursuing a global commitment to develop 10 new antibacterial drugs by 2020. Clin Infect Dis. 2010; 50(8):1081–1083. doi: 10.1086/652237
6. Clinical and Laboratory Standard Institute. Performance Standards for Antimicrobial Susceptibility Testing. Twenty-eight Informational Supplement M100-S28. Wayne: Clinical and Laboratory Standard Institute; 2018. [ Links ]
7. Tofteland S, Haldorsen B, Dahl KH, Simonsen GS, Steinbakk M, Walsh T, Sundsfjord A. Effects of Phenotype and Genotype on Methods for Detection of Extended- Spectrum-β-Lactamase-producing clinical isolates of Escherichia coli and Klebsiella pneumoniae in Norway. J Clin Microbiol. 2007; 45(1):199–205. [ Links ]
8. Pitout J, Hossain A, Hanson D. Phenotypic and molecular detection of CTX-M-β-lactamases produced by Escherichia coli and Klebsiella spp. J Clin Microbiol. 2004; 42(12):5715-5721. [ Links ]
9. Jarlier V, Nicolas M, Fournier G, Philippon A. Extended broad-spectrum β-lactamases conferring transferable resistance to newer β-lactam agents in Enterobacteriaceae: hospital prevalence and susceptibility patterns. Rev Infect Dis. 1988;10(4):867–878. [ Links ]
10. Castro N, Salgado JFS, Ocampo RLO, Silva J, Ruiz MR. Caracterización de β -lactamasas de espectro extendido producidas por Escherichia coli de infecciones del tracto urinario adquiridas en la comunidad de Chilpancingo, Guerrero, México. Tlamati. 2014; 5(1):14-23. [ Links ]
11. Pallecchi L, Malossi M, Mantella A, el al. Detection of CTX-M-Type β-lactamase genes in fecal Escherichia coli isolates from healthy children in Bolivia and Peru. Antimicrob Agents Chemother. 2004; 48(12):4556-4561. doi: 10.1128/ AAC.48.12.4556-4561.2004 [ Links ]
12. Morales J, Reyes K, Monteghirfo M, Roque M, Irey J. Presencia de β-lactamasas de espectro extendidoen dos hospitales de Lima, Perú. An Fac Med. 2005; 66(1):24-32. [ Links ]
13. Cuéllar L, Vicente W, Silva M. Cepas de E. coli y Klebsiella spp. Productoras de betalactamasas de espectro extendido en aislamientos clínicos de infecciones intrahospitalarias en un Hospital Oncológico, en Perú. Caracas: XII Congreso de la Asociación Panamericana de Infectología; 2005. [ Links ]
14. García C, Horna G, Linares E, et al. Antimicrobial drug resistance in Peru. Emerg Infect Dis. 2012;18(3):520–521. doi: 10.3201/eid1803.100878 [ Links ]
15. Colquechagua F, Sevilla C, Gonzales E. Enterobacterias productoras de Betalactamasas de espectro extendido en muestras fecales en el Instituto Nacional de Salud del Niño. Perú. Rev Peru Med Exp Salud Pública. 2015; 32(1):26-32. [ Links ]
16. Luvsansharav UO, Hirai I, Nakata A, et al. Prevalence and risk factors associated with fecal carriage of CTX-M beta-lactamase Enterobacteriaceae that produce in rural communities of Thailand. J Antimicrob Chemother. 2012; 67(7): 1769-1774. doi:10.1093/jac/dks118 [ Links ]
17. Pallecchi L, Bartoloni A, Fiorelli C. Rapid dissemination and diversity of CTX-M Extended-Spectrum β-Lactamase genes in commensal escherichia coli isolates from healthy children from low-resource settings in Latin America. Antimicrob. Agents Chemother. 2007;51(8):2720-2725. doi: 10.1128/AAC.00026-07 [ Links ]
18. Salles MJ, Zurita J, Mejía C, Villegas MV, Latin America Working Group on Bacterial Resistance. Resistant Gram-negative Infections in the outpatient setting in Latin America. Epidemiol Infect. 2013; 141(12):2459–2472. doi: 10.1017/S095026881300191 [ Links ]
19. Minarini LA, Gales AC, Palazzo IC, Darini AL. Prevalence of community-occurring extended spectrum beta-lactamase-producing Enterobacteriaceae in Brazil. Current Microbiology. 2007; 54(5):335–341. [ Links ]
20. Cantón R, Coque TM. The CTX-M beta-lactamase pandemic. Current Opinion in Microbiology. 2006; 9(5):466–475. [ Links ]
21. Mattar S, Martinez P. Emergence of antimicrobial resistance to extended-spectrum-β-lactamases (ESBL): detection, clinic impact and epidemiology. Infection. 2007; 11(1):23–35. [ Links ]
22. Arce Z, Llontop J, Alarcón E, López E. Detección de los genes SHV, TEM Y CTX-M en cepas de Escherichia coli ß-lactamasas de espectro extendido procedentes de un Hospital de Chiclayo-Perú. Rev cuerpo med HNAAA. 2014; 7(3):13-16. [ Links ]
23. Galván F, Agapito J, Bravo N, Lagos J, Tamariz J.Caracterización fenotípica y molecular de Escherichia coli productoras de β-Lactamasas de espectro extendido en pacientes ambulatorios de Lima, Perú. Rev Med Hered. 2016; 27(1):22-29. [ Links ]
24. Gonzales E, Sevilla R, León S. β-lactamasas de espectro extendido tipo CTX-M en aislamientos de Shigella flexneri de pacientes pediátricos con diarrea aguda. Rev Peru Med Exp Salud Publica.2013; 30(3):527-8. [ Links ]
Los autores declaran no tener conflicto de interés.
Contribución de autoría: Todos los autores participaron en la idea de la investigación, concepción, la recolección de datos, material de estudio y redacción del artículo. Todos los autores aprobaron la versión final del manuscrito.
Declaración de financiamiento y de conflictos de interés: El estudio fue financiado por el Fondo a la investigación del Instituto Nacional de Salud del Niño (Estudio j.1574-6976.2011.00268.
Correspondencia:
Edgar Gonzales Escalante
Centro de Investigaciones Tecnológicas, Biomédicas y Medioambientales
Calle José Santos Chocano 199 Ciudad Universitaria
Bellavista, Callao, Perú.
Correo electrónico: egones_5@hotmail.com
Recibido: 09/04/2019
Aceptado: 04/08/2019