Introducción
La biolixiviación consiste en el uso de microorganismos para extraer metales a partir de minerales y concentrados. Este proceso utiliza la capacidad natural de los microorganismos para transformar los metales presentes en el mineral a una forma soluble (Brierley & Brierley 2013). Este tipo de tecnología ha captado considerablemente el interés debido a los beneficios económicos y por ser ecológicamente amigable. Es así como se aplica principalmente en la recuperación de cobre y oro, siendo responsable del 20 - 25% de la producción mundial de cobre (Watling 2006; Panda et al. 2015).
Varios estudios han evidenciado la alta eficiencia de la biolixiviación en la recuperación de metales, con tasas superiores al 80%, mediante el uso de bacterias quimiolitotróficas acidófilas, tanto en cepas individuales como en consorcios (cultivos mixtos) (Petersen & Dixon 2007; Rawlings & Johnson 2007; Brierley & Brierley 2013). En particular, las bacterias oxidantes de hierro y azufre son ampliamente utilizadas en estos procesos de biolixiviación, siendo las especies de Acidithiobacillus de las más eficientes y empleadas. Actualmente, el uso de consorcios bacterianos autóctonos de yacimientos, drenajes ácidos, pilas de sulfuro o depósitos de minerales ha tomado relevancia debido a su eficiencia como catalizadores de biolixiviación. Estos microorganismos al estar adaptados a un ambiente natural con altas concentraciones de metales responderían eficazmente al aumento progresivo de elementos principales durante los procesos de biolixiviación a gran escala (Watling 2006; Romo et al. 2013). Entre los consorcios de bacterias acidófilas más empleados y estudiados, destacan los conformados por Acidithiobacillus ferrooxidans y Acidithiobacillus thiooxidans, y también aquellos que incluyen a Leptospirillum ferrooxidans. Estos consorcios han mostrado resultados favorables para la extracción de cobre a partir de minerales de baja ley (Romo et al. 2013; Sajjad et al. 2018).
Los consorcios microbianos utilizados industrialmente en el procesamiento de sulfuros de cobre suelen operar a condiciones mesofílicas y termofílicas (Watling 2006). En Sudamérica, particularmente en Chile y Perú, la minería se concentra en zonas altoandinas, las cuales presentan condiciones ambientales desfavorables para la biolixiviación, tales como bajas temperaturas, baja presión atmosférica, escasez de oxígeno y altos índices de radiación. Bajo estas condiciones los procesos de biolixiviación a nivel industrial son afectados significativamente en su eficiencia. Especialmente las bajas temperaturas (<10 °C) tienen un efecto directo ya que los microorganismos mesófilos no son capaces de generar suficiente ion férrico (Fe3+) a estas temperaturas, el cual es el factor principal de lixiviación en estos procesos (Escobar et al. 2010). Por lo tanto, una opción prometedora para mejorar la biominería en las regiones altoandinas, sería el uso de microorganismos acidófilos autóctonos adaptados a estas condiciones extremas, como los psicrófilos o psicrotolerantes, y que sean capaces de lixiviar minerales sulfurados para la recuperación de metales de baja ley.
Existen algunos reportes de microorganismos lixiviantes en minas peruanas abandonadas a gran altitud, entre ellas, Acidithiobacillus ferrivorans PQ33 (Ccorahua-Santo et al. 2017) que destaca como una cepa psicrotolerante, capaz de realizar la biolixiviación de minerales sulfurados eficientemente a bajas temperaturas (< 5 °C) (Liljeqvist et al. 2011; Ccorahua-Santo et al. 2017; Peng et al. 2017). Sin embargo, las iniciativas de búsqueda de microorganismos lixiviantes autóctonos y eficientes a bajas temperaturas son pocas y en general se restringen al uso de cepas individuales a nivel de laboratorio, a pesar de que el diseño y uso de consorcios microbianos lixiviantes autóctonos de la minería altoandina mejoraría la extracción de metales de interés comercial, como el cobre, para su aplicación a escala industrial.
Por lo anterior, este trabajo describe el aislamiento y caracterización de seis consorcios microbianos psicrotolerantes lixiviantes de sulfuro de cobre, obtenidos de drenajes ácidos en localidades mineras del Perú. Además, la capacidad de estos consorcios es evaluada para la biolixiviación del sulfuro de cobre. Asimismo, la composición microbiana es determinada mediante secuenciación del gen ARNr 16S de la comunidad. Este estudio pretende ser una referencia útil para determinar consorcios adecuados para la extracción de cobre a partir de minerales sulfurados, con el fin de aplicarlo en la biominería altoandina.
Material y métodos
Área de estudio. El muestreo de drenajes ácidos de minas (DAM) se realizó en cuatro localidades mineras del Perú (Fig. 1). Tres de estas localidades se ubican en la provincia y departamento de Pasco, e incluyen a dos lagunas ácidas y un arroyo de DAM. Las lagunas ácidas de estudio corresponden a la laguna Yanamate y la laguna Quiulacocha, las cuales son usadas como depósitos de relaves mineros, y están ubicadas en los distritos de Tinyahuarco y Simón Bolívar, respectivamente. El arroyo de DAM se encuentra en la localidad de Huarón, en el distrito de Huayllay, en las cercanías de la relavera operada por la compañía Pan American Silver Perú S.A.C. La última localidad se encuentra ubicada en el centro poblado San José de Parac, en el distrito de San Mateo, provincia de Huarochirí, departamento de Lima. En esta localidad, se muestreó un arroyo de DAM proveniente de los pasivos ambientales mineros de la Compañía Minera Millotingo.
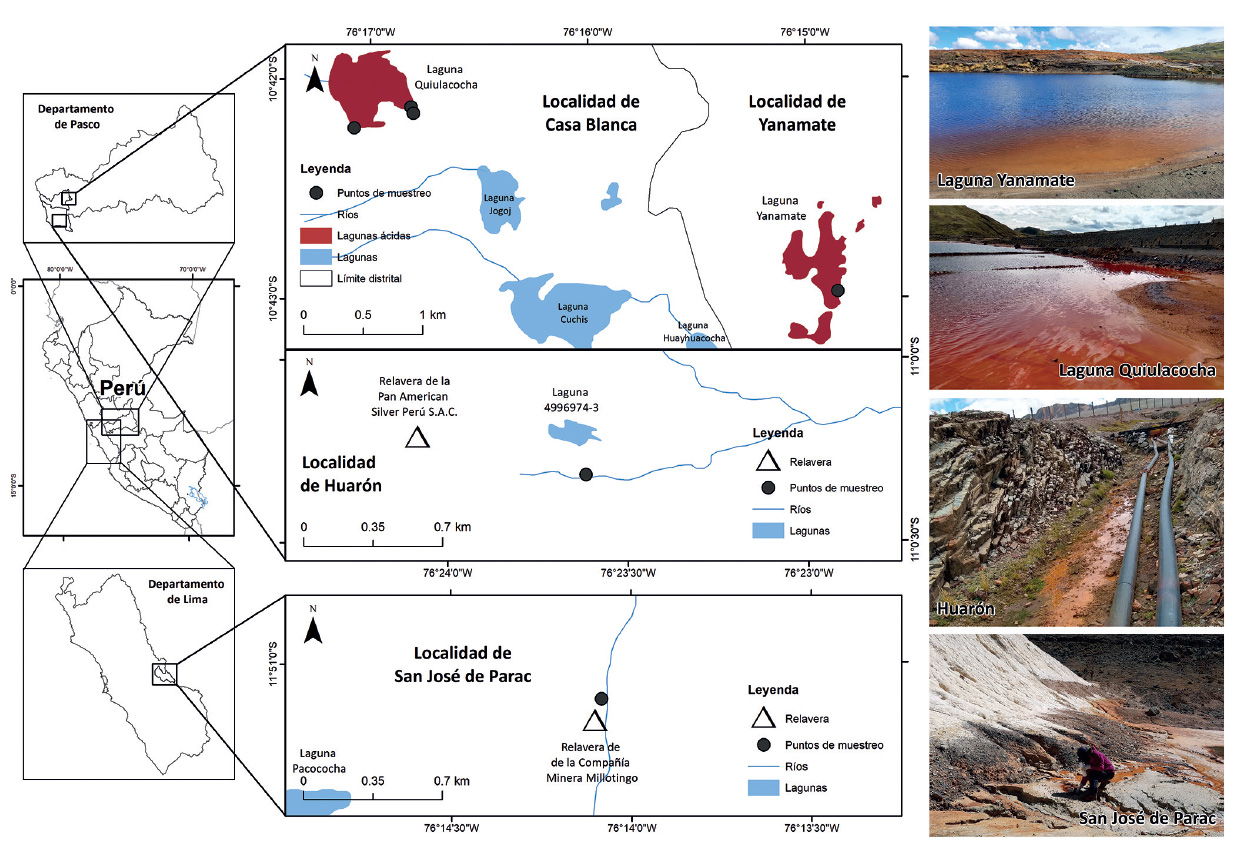
Figura 1 Mapa de ubicación de los sitios de muestreo de drenajes ácidos de mina en el departamento de Pasco y Lima. En Pasco, los sitios de muestreo se localizaron en la laguna Yanamate, laguna Quiulacocha y drenajes ácidos de Huarón. En Lima, se muestrearon en localidad de San José de Parac (provincia de Huarochirí).
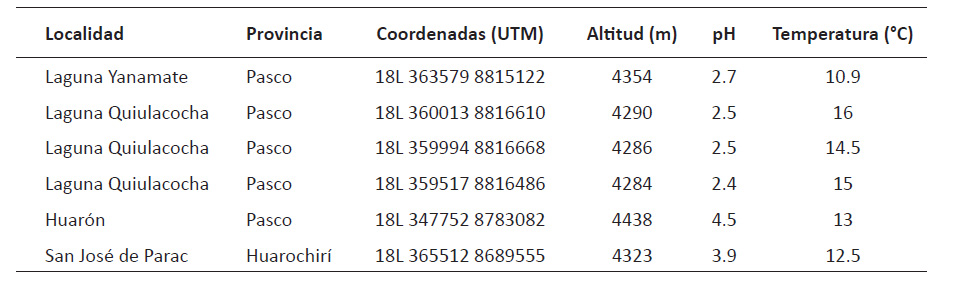
Muestreo. Las muestras de DAM se colectaron usando frascos de plástico estériles de 1 L y transportadas en una caja en frío hasta el laboratorio, donde fueron guardadas a 4 °C hasta su procesamiento. Se tomaron datos de las coordenadas de cada sitio de muestreo y se realizaron las mediciones in situ de las variables fisicoquímicas del agua, como el pH y temperatura. En total, se recolectaron seis muestras de DAM de las cuatro localidades mineras. Esto incluyó una muestra de la laguna Yanamate, tres muestras de la laguna Quiulacocha, y dos muestras de arroyos de DAM, uno en la localidad de Huarón (Pasco) y otro en San José de Parac (Huarochirí). En la laguna Quiulacocha, se tomaron en tres puntos diferentes, una en la entrada del agua proveniente de la relavera y dos en cada extremo opuesto de la laguna.
Aislamiento de consorcios microbianos psicrotolerantes lixiviantes (CMPL). Las muestras de DAM se sembraron al 20% v/v en medio de cultivo 9K modificado compuesto por 3.33% p/v FeSO47H2O, 0.04% p/v MgSO47H2O, 0.01% p/v (NH4)2SO4, 0.004% p/v KH2PO4 a pH 1.85 (Varela et al. 1998; Ramírez et al. 2004). Los cultivos se incubaron inicialmente a 28 °C con agitación constante de 180 rpm, hasta que se evidenció la oxidación del ion ferroso. Posteriormente, se realizaron resiembras y los cultivos fueron adaptados a una temperatura de 15 °C durante un máximo de 15 días de incubación a 180 rpm. Se evaluaron las cinéticas de crecimiento de los consorcios adaptados a 15 °C, mediante el recuento de células viables utilizando la cámara de recuento Petroff-Hausser y un microscopio Leica DM750.
Cultivo de CMPL en medio suplementado con sulfuro de cobre. A partir de los consorcios en fase exponencial en medio 9K modificado a 15 °C (~ 2.5x108 células/mL), se centrifugaron 100 mL del cultivo y las células recuperadas se lavaron tres veces con agua ácida (pH 2.5) para eliminar restos del medio de cultivo. Luego, se resuspendieron en 1 mL de agua ácida para obtener los inóculos iniciales. Estos inóculos se cultivaron en matraces Erlenmeyer de 250 mL con 100 mL de medio base 9K, compuesto por 0.04% p/v MgSO4.7H2O, 0.01% p/v NH4 (SO4)2 y 0.004% p/v KH2PO4, ajustado a pH 2.5; y con sulfuro de cobre (Sigma-Aldrich) al 0.5% p/v (Ramírez et al. 2004; Ccorahua-Santo et al. 2017). Los cultivos se incubaron a 15 °C a 180 rpm durante 30 - 60 días. Una vez adaptadas al medio de cultivo, se monitoreo la cinética de crecimiento cada 7 días durante 63 días.
Evaluación de la capacidad de oxidación del sulfuro de cobre. La evaluación de la cinética de liberación de cobre a 15 °C se realizó según el método propuesto por (Bernaldo 2016). Para este fin, se seleccionó uno de los CMPL con el mejor comportamiento de crecimiento. Los experimentos se llevaron a cabo por triplicado. Se utilizaron matraces Erlenmeyer de 250 mL, en los que se administraron 199 mL de medio base 9K suplementado con CuS al 0.5% p/v y 1 mL del inóculo de células resuspendido, obteniendo para el inicio de la prueba una concentración inicial de ~3.4x107 células/mL. Los matraces se incubaron durante 30 días a una temperatura de 15 °C y en agitación constante de 180 rpm. Se tomaron muestras del sobrenadante en los 0, 7, 15, 22 y 30 días, las cuales fueron filtradas utilizando filtros de jeringa estériles de 0.22 µm en tubos centrífuga de 50 mL. Además, se realizaron recuentos celulares por triplicado en la cámara de Petroff-Hauser en los días mencionados. Para la cuantificación de cobre liberado, se analizaron las muestras filtradas mediante un espectrofotómetro de absorción atómica de flama (Shimadzu AA-700).
Extracción de ADN y secuenciamiento de ARNr 16S. A partir de los seis consorcios cultivados en medio base 9K suplementado con CuS al 0.5% p/v, incubados a 15 °C durante un promedio de 30 días, se recuperaron las células mediante centrifugación a 7500 rpm por 10 minutos a 10 °C. Seguido de lavados celulares con agua ácida (pH 2.5) mediante centrifugaciones a 13000 rpm, para finalmente eliminar el sobrenadante y obtener solo el pellet. Se extrajo el ADN genómico usando el GenElute™ Bacterial Genomic DNA Kit (Sigma), siguiendo las instrucciones del fabricante. Se cuantificó la concentración mediante un fluorómetro Qubit (Thermo Fisher Scientific) y se evaluó la integridad mediante electroforesis en agarosa. Se extrajo el ADN de los seis consorcios por triplicado. La región V3-V4 de los genes ARNr 16S fue amplificada utilizando los siguientes cebadores: 341F (5′-CCTACGGGNGGCWGCAG-3′) y 805R (5′-GACTACHVGGGTATCTAATCC-3′). La preparación de bibliotecas genómicas de ARNr 16S y la secuenciación se llevaron a cabo siguiendo el protocolo estándar de Illumina (Illumina Inc 2013). La secuenciación se realizó en una plataforma Illumina MiSeq (Illumina, USA). Las bibliotecas se secuenciaron como lecturas de extremos emparejados de 2 x 250 pb. Las secuencias sin procesar de las bibliotecas genómicas se depositaron en la base de datos NCBI BioProject (PRJNA854680).
Análisis bioinformático. Las secuencias obtenidas se procesaron con el software QIIME2 v. 2021.8. La calidad de las secuencias sin procesar se comprobó mediante gráficos de calidad interactivos, con el fin de obtener valores para el recorte y el truncamiento de secuencias. La configuración predeterminada de DADA2 dentro de QIIME2 se utilizó para detectar, corregir errores de secuenciación, filtrar lecturas de phiX y secuencias quiméricas, unir lecturas de extremos de pares y secuencias desreplicadas. DADA2 se ejecutó utilizando el comando denoise-single, con muestras truncadas en 243 nucleótidos (trunc-len 243), para eliminar las bases con una puntuación de baja calidad (puntuación de calidad media <30). Todos los demás parámetros se establecieron por defecto. A continuación, las lecturas de extremos emparejados se fusionaron para producir variantes de secuencia de amplicón (ASV). La anotación taxonómica de los ASV se realizó con base en RESCRIPt (Robeson et al. 2021) en QIIME2 utilizando un clasificador Naive-Bayes entrenado en la región V3-V4 en la base de datos de referencia SILVA SSU NR99 (versión 138) (Quast et al. 2012). Se eliminaron las secuencias mitocondriales y de cloroplastos, así como los ASV con una abundancia relativa inferior al 0.01%.
Análisis de datos. El análisis de los ASV se realizó en R versión 4.1.2 utilizando los paquetes phyloseq, vegan y microbiome. Los datos fueron convertidos a un objeto phyloseq. La abundancia relativa de los diferentes taxones se determinó mediante la función de transform “compositional” incluida en el paquete de microbiome, después de la normalización mediante enrarecimiento. La diversidad alfa fue analizada usando los paquetes vegan y microbiome. Se calcularon los índices de diversidad alfa Chao 1 y Shannon. Se realizó una prueba de ANOVA y de post-hoc Tukey, para verificar diferencias significativas (p < 0.05) en los índices de diversidad alfa entre los consorcios. Adicionalmente, se realizó un análisis de correlación Pearson entre el índice de Shannon de los consorcios y el pH del DAM de donde fue aislado. La diversidad beta se estimó mediante la métrica de disimilitud de Bray-Curtis, para comparar las diferencias entre cada consorcio en función del número de secuencias por ASV y se visualizó a través del análisis de coordenadas principales (PCoA) usando el paquete vegan. El análisis estadístico se llevó a cabo mediante el análisis multivariante permutado de la varianza (PERMANOVA), utilizando la función adonis2 implementada en el paquete vegan.
Resultados
Características fisicoquímicas de los DAM. Todos los sitios de muestreo se ubicaron a sobre los 4200 m de altitud, y compartieron características comunes, como un pH ácido (<5) y temperaturas frías (<16 °C), tal como se muestra en la Tabla 1. Además, se observaron coloraciones naranja-rojiza características de los DAM (Fig. 1), debido a su estado de oxidación. En la provincia de Pasco, los niveles de pH oscilaron entre 2.5 y 4.5, siendo el valor más bajo registrado en la Laguna Quiulacocha y el más alto en la localidad de Huarón. Las temperaturas de los DAM variaron entre 10.9 °C y 16 °C, con el valor más bajo registrado en la Laguna Yanamate y el más alto en la Laguna Quiulacocha. En la provincia de Huarochirí, el DAM de la localidad de San José de Parac presentó un pH de 3.9 y una temperatura de 12.5 °C.
Tabla 1 Coordenadas (WGS84) y parámetros fisicoquímicos de los sitios de muestreo de aguas ácidas en las provincias de Pasco y Huarochirí, Perú.
Consorcios microbianos psicrotolerante lixiviantes (CMPL). Se obtuvieron seis CMPL: LQ.P1.1, LQ.P2.1, LQ.P3.1 de la laguna Quiulacocha, LY.P2.2 de la laguna Yanamate, RH.P1.2 de relaves de Huarón y SJ.P1.2 de los pasivos de San José de Parac. Todos los consorcios mostraron crecimiento a 28 y 15 °C en medio 9K modificado. Las muestras de la laguna Quiulacocha se adaptaron más rápido en comparación a otros sitios de muestreo, teniendo crecimiento inicial a los seis días de incubación a 28 °C. En cambio, las muestras de la relavera de Huarón presentaron un crecimiento inicial a los 18 días de incubación. Los consorcios adaptados al medio 9K a 15°C, alcanzaron su fase estacionaria a los 7 a 9 días de incubación y una concentración celular máxima entre 2.3x108 y 2.8x108 células/mL (Fig. 2A). Los CMPL con mejores patrones de crecimiento en medio 9K a 15 °C fueron: LQ. P1.1, LQ. P2.1 y LQ. P3.1.
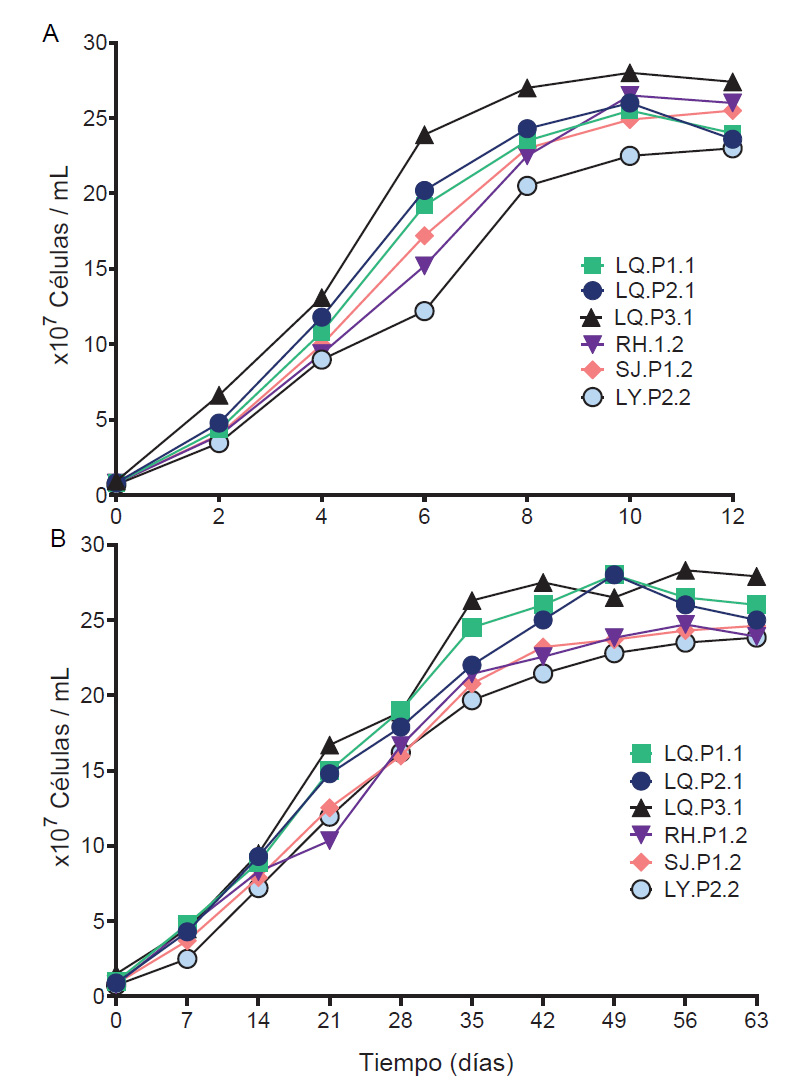
Figura 2 Cinética de crecimiento de los seis consorcios microbianos psicrotolerantes lixiviantes a 15 °C. (A) Crecimiento en medio modificado 9K. (B) Crecimiento en medio base 9K suplementado con CuS al 0.5% p/v.
Los seis CMPL presentaron crecimiento en medio base 9K suplementado con CuS al 0.5% p/v, a los 30 días de la incubación inicial, evidenciado por el viraje de transparente a color celeste. Los CMPL adaptados al medio a 15 °C, entraron en la fase logarítmica el día 14 y a la estacionaria entre el día 30 y 35 (Fig. 2B), alcanzando concentraciones celulares entre 2.3 x108 y 2.8 x108 células/mL. El consorcio LQ.P3.1 presentó el mejor comportamiento de crecimiento, con un menor tiempo de duplicación (18.25 días) y altas concentraciones celulares (2.8 x108 células/mL). En cambio, el consorcio LY.P2.2, presentó el mayor tiempo de duplicación (22.08 días) y menores concentraciones celulares (2.3 x108células/mL).
Recuperación de Cu soluble. El consorcio LQ.P3.1, fue usado para cinética de liberación de cobre (II). El rendimiento fue de 23.78 mg/L por cada 107 células/mL y la liberación de aproximadamente 562.68 mg/L de cobre por acción microbiana a los 30 días.
En la Figura 3, se muestra un aumento progresivo y significativo de la concentración de cobre soluble a partir del día 7 a medida que la concentración celular aumenta. Alcanzando una máxima concentración a los 30 días de evaluación, representando una recuperación de cobre de 12.47%.
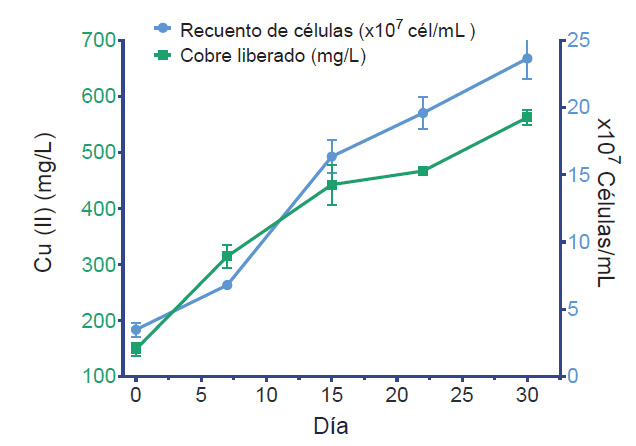
Figura 3 Cinética de liberación de cobre (II) del consorcio LQ.P3.1 en medio base 9K suplementado con CuS al 0.5 % p/v.
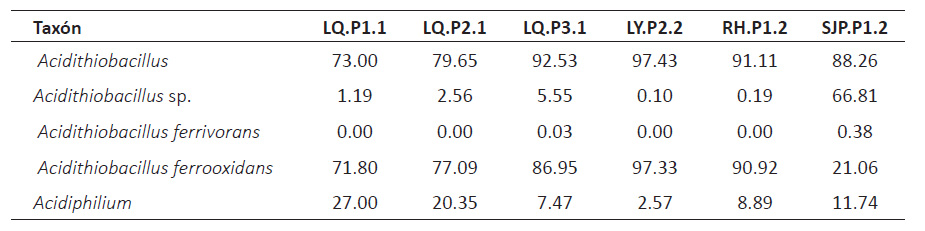
Estructura bacteriana de los CMPL. Se analizó el perfil taxonómico de los seis consorcios que desarrollaron en el medio con sulfuro de cobre, siguiendo un enfoque independiente del cultivo basado en la secuenciación de alto rendimiento de la región hipervariable V3-V4 de los amplicones de ARNr 16S. El secuenciamiento generó un total de 11517839 lecturas de extremo de par sin procesar de 250 pb para 18 muestras, con un promedio de 639879 (487009 - 876750) lecturas por muestra. Después de la eliminación de ruido usando DADA2 de QIIME2, quedaron un promedio de 448682 (308829 - 606665) lecturas. Una vez que se completó la asignación taxonómica, se eliminaron secuencias correspondientes de mitocondrias y cloroplastos, y las de baja abundancia relativa (<0.01%). En total se obtuvieron 40 ASV, entre 17 y 32 ASV por consorcio, compartiendo entre todos 9 ASV.
Se observó que Proteobacteria fue el único phylum presente en todos los consorcios (100%). Mientras que, a nivel de género, la mayor parte de la diversidad se capturó dentro de: Acidithiobacillus y Acidiphilium (Fig. 4). Acidithiobacillus presentó una abundancia superior al 72% en todos los consorcios, con un máximo de 97.43% en el consorcio LY.P2.2. En cambio, Acidiphilium presentó abundancias entre 2.57 y 27%, siendo mayor en los consorcios LQP1.1 y LQP2.1. Los ASV identificados como especie, mostraron un predominio de Acidithiobacillus ferrooxidans (21.06 - 97.33%) en todos los consorcios (Tabla 2). Además, se encontró Acidithiobacillus ferrivorans en los consorcios LQ.P3.1 y SJ.P1.2, en porcentajes bajos, 0.03 y 0.38% respectivamente. Otros ASV fueron identificados sólo hasta género (Acidithiobacillus), con una abundancia relativa entre 1.19% (LQ.P1.1) y 66.81% (SJ.P1.2). En el caso del género Acidiphilium, todos los ASV se clasificaron hasta género o como bacteria no cultivable.
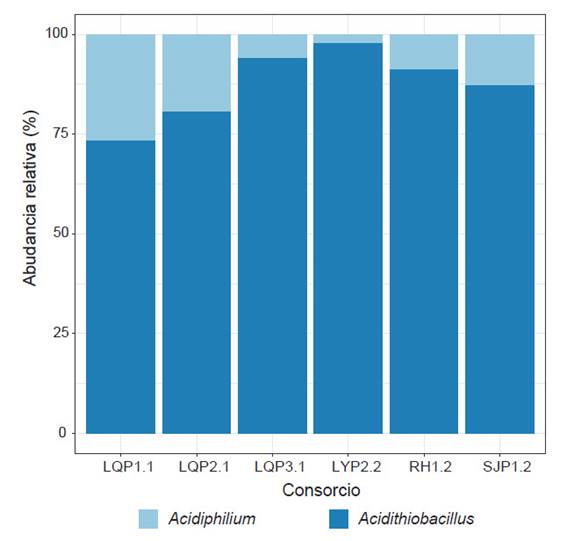
Figura 4 Composición de la comunidad bacteriana de los seis CMPL a nivel de género, expresada en abundancia relativa.
Un análisis de rarefacción realizado para cada conjunto de datos de secuencias de los consorcios, mostraron curvas que alcanzaron una meseta para todas las muestras, lo que sugiere que el tamaño de la muestra fue lo suficientemente grande para representar la diversidad bacteriana en cada muestra. La evaluación de diversidad alfa basada en los ASV totales encontrados en cada grupo de consorcios, presentó índices de diversidad de Shannon muy bajos (<2) y diferentes en los consorcios, con valores promedios entre 0.56 y 1.67 (Fig. 5).
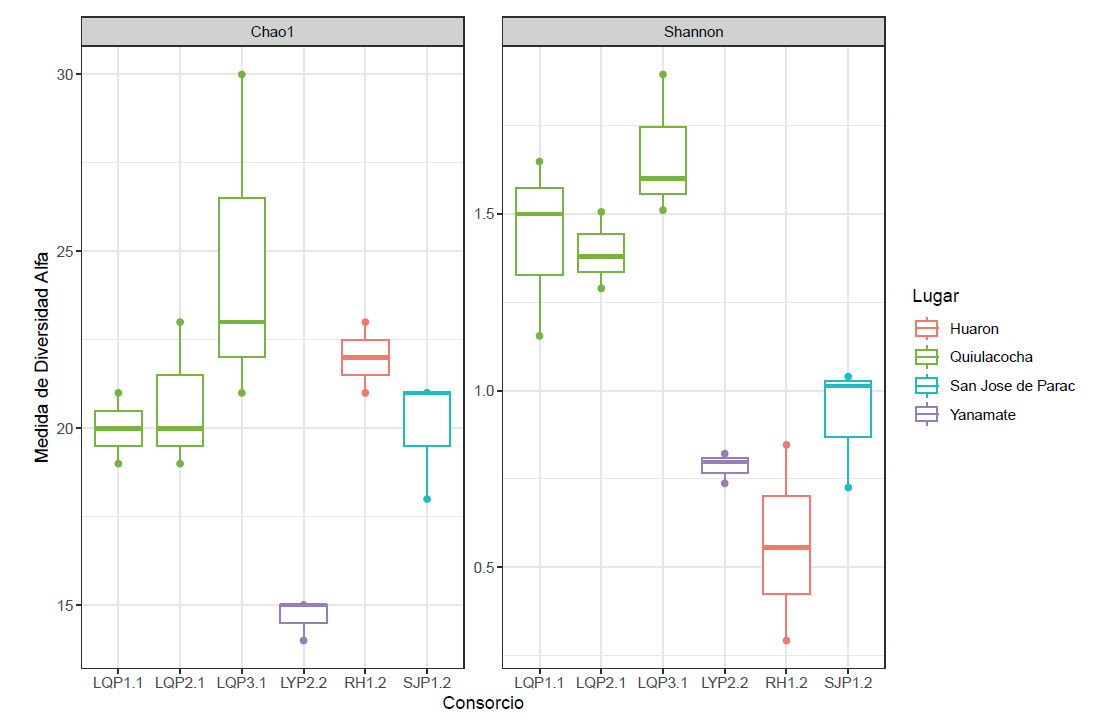
Figura 5 Análisis de la diversidad alfa de la comunidad bacteriana de los CMPL, de la laguna Quiulacocha en color verde, de la laguna Yanamate en morado, de los drenajes de Huarón en rojo, y de San José de Parac en celeste.
La diversidad del consorcio RH.P1.2 fue significativamente menor en comparación con los otros consorcios. En cambio, los consorcios aislados de la laguna Quiulacocha (LQ.P1.1, LQ.P2.1 y LQ.P3.1), presentaron mayor diversidad (1.39 - 1.67) que los otros consorcios, cuyos valores fueron significativamente diferentes (p <0.05) a los consorcios RH.P1.2 y LY.P2.2.
El índice de diversidad del consorcio LQ.P3.1 fue mayor a los otros consorcios y presentó diferencias significativas con los consorcios RH1.2, LY2.2 y SJP1.2. En relación al índice de Chao 1, se observa que el consorcio LQ.P3.1 presenta la mayor riqueza estimada de ASV, con un valor máximo de 30. Además, la mayoría de los consorcios tienen una riqueza estimada entre 19 y 23, excepto el consorcio LY.P2.2 que tiene una riqueza estimada de 14 - 15, significativamente menor en comparación a LY.P2.2 y LQ.P3.1. El análisis de correlación Pearson entre la diversidad y el pH del ambiente natural de los consorcios, mostraron una relación lineal inversa significativa (p = 0.00064) con un coeficiente de determinación (R2) de 0.53, lo que nos sugiere que la diversidad de los consorcios recuperados de ambientes de menor pH (<3), es mayor a comparación de los recuperados a un pH mayor de 3.
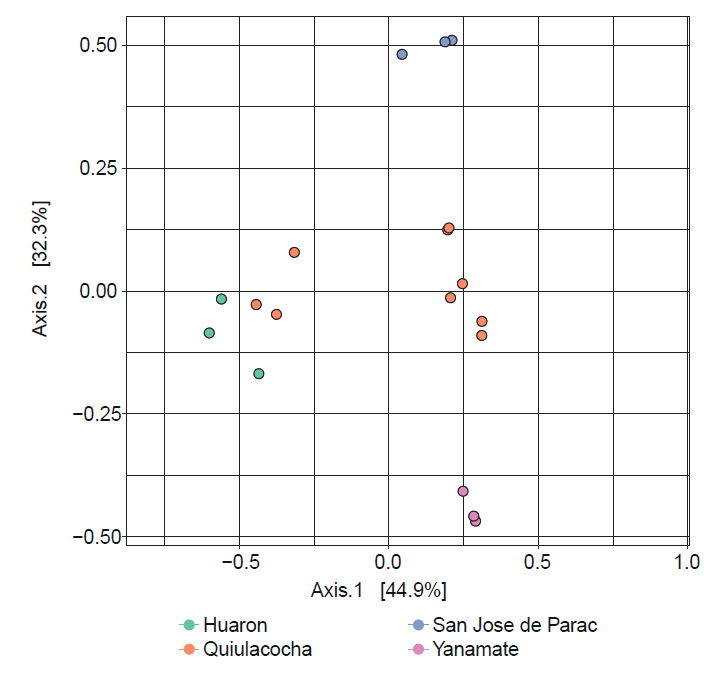
El análisis de coordenadas principales (PCoA) basado en las distancias de Bray-Curtis (Fig. 6) evidenció notables diferencias en la estructura de la comunidad bacteriana de los CMPL, en función a la localización geográfica de muestreo. Se observó una agrupación que incluyó a los CMPL de Quiulacocha (LQ.P1.1, LQ.P2.1, LQ.P3.1) y Huarón (RH.P1.2), mientras que separó a los de Yanamate (LY.P2.2) y San José de Parac (SJ.P1.2) en dos grupos distintos. Estos análisis revelaron que la comunidad bacteriana de los CMPL de Quiulacocha y Huarón difieren de los de Yanamate y San José de Parac. Esta diferencia fue respaldada por el análisis PERMANOVA, que confirmó la existencia de diferencias significativas (p = 0.001) entre las comunidades microbiana de los diferentes CMPL.
Discusión
Los drenajes ácidos de mina generalmente están habitados por microorganismos acidófilos oxidantes de hierro y azufre. Entre los géneros más comunes que habitan estos entornos, se encuentran Acidithiobacillus, Leptospirillum y Acidiphilium (Chen et al. 2015; Méndez-García et al. 2015). El medio base 9K suplementado con CuS, permitió la selección y crecimiento de especies de Acidithiobacillus y Acidiphilium presentes en los drenajes de las cuatro localidades muestreadas. Los seis CMPL formados principalmente por ambos géneros, presentaron crecimiento y lixiviaron sulfuro de cobre a una temperatura de 15 °C, permitiendo la recuperación de cobre de hasta el 12.47%.
Después de 30 a 35 días de evaluación, los CMPL alcanzaron concentraciones entre 2.3 x108 y 2.8 x108 células/mL, valores cercanos a lo obtenido por el consorcio MHJ evaluado por Peng et al. (2021) para la biolixiviación de calcopirita a 6 °C. Estos consorcios, conformados principalmente por Acidithiobacillus spp. y Sulfobacillus spp., presentaron concentraciones celulares máximas entre 2.2 × 108 células/mL en 60 días de evaluación. Además, presentaron una recuperación de cobre de 11.2% en 44 días, resultado cercano al obtenido por LQ.P3.1 de 12.47% en 30 días a 15 °C. En otro estudio, utilizaron el consorcio T7 para lixiviar un concentrado de calcopirita, donde se logró recuperar el 16% del cobre en 62 días, cuya composición estuvo dominada por A. ferrivorans (Liljeqvist et al. 2013). Reportes muestran que A. ferrivorans tiene la capacidad de oxidar el Fe2+ con más eficacia a 5 °C que A. ferrooxidans (Dopson et al. 2007); por ende, se obtiene una mayor tasa de lixiviación a bajas temperaturas. Es importante destacar que la temperatura y el tipo de mineral pueden ser factores determinantes en la recuperación de cobre durante la lixiviación (Watling 2006). En el caso de los estudios previos mencionados, estos se llevaron a cabo a temperaturas más bajas, en tiempos más prolongados y con diferentes tipos de minerales en comparación con las evaluaciones realizadas en este estudio, lo que podría explicar las diferencias en la recuperación de cobre obtenidas. No obstante, en el caso de LQ.P3.1 se evidenció una mejora significativa en la recuperación de cobre (12.47%), lo que sugiere que los microorganismos presentes podrían estar mejorando la disolución del sulfuro de cobre.
La diversidad alfa fue menor en los CMPL aislados de DAM con pH mayor a 3. El pH es uno de los principales factores ambientales involucrados en el ensamblaje de las comunidades microbianas asociadas a los DAM, por lo tanto, impulsan las variaciones de la diversidad en estos sitios (Kuang et al. 2013; Méndez-García et al. 2015). Esto fue evidenciado en la estructura de la comunidad de los CMPL, los cuales fueron diferentes a SJP1.2, aislado de un DAM mayor a pH 3. Asimismo, la composición química del DAM suele influir en la composición de microbiana en estos entornos. El crecimiento de todos los CMPL en el medio suplementado con CuS al 0.5% p/v, podría haberse facilitado por la exposición previa a concentraciones elevadas de diferentes metales, incluyendo el cobre, en su hábitat natural ubicadas en zonas de relaveras mineras. Es probable que estos microorganismos hayan desarrollado mecanismos de resistencia al estrés del cobre, tal como se ha reportado para A. ferrivorans (Ccorahua-Santo et al. 2017; Barahona et al. 2020). La laguna Yanamate, ha recibido por muchos años desechos de la actividad minera, generando que sus aguas presenten pH extremadamente ácido (<3) y altas cantidades de metales pesados como el arsénico, cadmio y plomo, que sobrepasan los límites máximos permisibles de los estándares nacionales de calidad ambiental (ECAs) (Wißkirchen et al. 2005; Baylón Coritoma et al. 2018). La calidad mala del agua puede ser perjudicial para la biodiversidad, como lo reportado en las comunidades fitoplanctónicas en esta laguna (Baylón Coritoma et al. 2018). De la misma forma, estaría sucediendo con la comunidad bacteriana, permitiendo sólo el desarrollo de microorganismos quimiolitotróficos acidófilos. Esto se reflejó en el consorcio recuperados de esta laguna, el cual presentó la mayor abundancia de Acidithiobacillus, y una baja diversidad microbiana.
Los análisis metagenómicos previos realizados en DAM muestran una predominancia del género Acidithiobacillus en ambientes fríos (Bernaldo 2016). Liljeqvist et al. (2015), evidenciaron en DAM de baja temperatura, secuencias similares a especies de Acidithiobacillus, una especie similar a Acidobacteria y una especie similar a Gallionellaceae, siendo el microorganismo más abundante, A. ferrivorans. La presencia de estos taxones, en su mayoría, fue evidenciada en los CMPL analizados, teniendo al género Acidithiobacillus como el más abundante. Acidiphilium fue el segundo género más abundante en todos los consorcios. Este género también se encuentra ampliamente representados en entornos de DAM, son heterotróficos metabolizadores de hierro y puede prosperar en consorcio con A. ferrooxidans, promoviendo su crecimiento (Liu et al. 2011). Los heterótrofos crean ambientes adecuados para el crecimiento de oxidantes de hierro mediante la eliminación de componentes orgánicos (lisados, exudados) que pueden ser tóxicos para los autótrofos (Bacelar-nicolau and Johnson 1999) y, por lo tanto, afecten en la tasa de lixiviación de los sulfuros metálicos (Johnson 2001). Peng et al. (2008) reportaron que Acidiphilium sp. PJH, posee la capacidad de lixiviar calcopirita a 40 °C y puede mejorar la tasa de lixiviación hasta en un 10.3% cuando se incorpora con Leptospirillum ferriphilium YTW315, después de 35 días de evaluación. Por otro lado, Yang et al. (2013) evaluaron la capacidad de biolixiviación de mineral de cobre de baja ley a temperatura ambiente durante 117 días, por consorcios de Acidiphilium sp. DX1-1 y A. ferrooxidans GF, obteniendo una mayor tasa de extracción de cobre (20.11%) que en cultivos puros de A. ferrooxidans (14.87 %), probablemente debido a que el pH del consorcio se mantenía más bajo y presentó una mayor concentración de Fe3+ durante todo el proceso. También, se ha reportado que los consorcios en los que está presente Acidiphilium pueden tener una mayor resistencia a ciertos metales como Cd2+ y Zn2+ (Cabrera et al. 2005), sin embargo, no se conocen con certeza los mecanismos de resistencia.
Con respecto a los trabajos anteriores, han enfatizado la importancia de las interacciones mutualistas entre microorganismos acidófilos autótrofos y heterótrofos para mejorar la disolución de minerales de sulfuro (Okibe & Johnson 2004; Peng et al. 2021). Los consorcios conformados por Acidiphilium, Acidithiobacillus y Leptospirillum, son empleados generalmente a condiciones mesofílicas y/o termofílicas. Por ende, el presente estudio es uno de los primeros reportes de consorcios constituidos por Acidiphilium y Acidithiobacillus, con capacidad de lixiviar sulfuro de cobre a temperaturas moderadamente bajas, y obtenidas por selección mediante medios de cultivo enriquecidos con sulfuro de cobre. Por lo que podemos deducir que, consorcios conformados Acidiphilium spp., A. ferrooxidans y A. ferrivorans , serían excelentes candidatos para utilizarlos en la biolixiviación de minerales sulfurados de cobre a bajas temperaturas. Además, un posterior aislamiento y caracterización de especies puras a partir de estos consorcios podrían permitir seleccionar cepas y diseñar consorcios más eficientes para la minería altoandina.












 uBio
uBio