1. Introducción
La biodiversidad se comprende en diferentes niveles (la variedad de ecosistemas, especies y genes) y la complejidad de sus interacciones conforma el sistema de soporte de la vida (De Andrade, 2013; Rawat & Agarwal, 2015), sus muchos beneficios han sido confirmados en múltiples estudios y aún en la actualidad continúan revelándose, llegando incluso a proponerse a la biodiversidad como controlador en la manifestación de enfermedades emergentes por zoonosis (Ezenwa et al., 2006; Johnson & Thieltges, 2010; Keesing et al., 2010).
No obstante, a pesar de todos sus beneficios, se registra una alarmante pérdida de la biodiversidad, ligado a un marcado efecto antropogénico sobre todos los niveles de variación biológica (Hoffmann et al., 2010), reportándose tasas de extinción entre 100 y 1000 veces superiores a las tasas de los cincos episodios de extinción masiva anteriores a la aparición del ser humano (Pimm et al., 1995). En este contexto, es una prioridad global acelerar la descripción de la biodiversidad y gestionar su conservación (Marchese, 2015; Martins et al., 2020).
La diversidad de especies y ecosistemas deriva de procesos evolutivos a nivel genético, por ello, esta es la base de la biodiversidad y un requisito primordial para la evolución (Collada & Jiménez, 2008; Piñero, 2008). Si la diversidad genética de una población se reduce, indefectiblemente disminuye su capacidad de adaptarse ante potenciales cambios ambientales, por lo que, mantener la variabilidad genética es vital para responder exitosamente a las presiones ambientales de selección.
El estudio de la diversidad genética se basa en el análisis de potentes marcadores moleculares de naturaleza proteica y de ADN (nuclear y citoplasmático), en particular para el grupo de las plantas, los marcadores del cloroplasto (ADN cp), destacan en la investigación de la historia de las poblaciones, flujo genético, hibridación e identificación de áreas con elevada diversidad genética (Caruso et al., 2015; Collada & Jiménez, 2008; Salvador et al., 2000). El grupo de trabajo de plantas del “Consortium for the Barcode of Life” (CBOL), recomienda el uso de los genes cloroplastidícos matK + rbcL, para desarrollar códigos de barras (Little, 2014), también se ha reportado que el marcador matK tiene una tasa evolutiva rápida y provee una alta señal de filogenia para resolver la sistemática en plantas (Hilu & Liang, 1997).
El género Cinchona está representado por 24 especies botánicas, comúnmente conocidas como “quina” o “cascarilla”, de importancia en el tratamiento contra la malaria por su composición de alcaloides de quinina en la corteza (Aymard, 2019; Cueva-Agila et al., 2019; Zevallos, 1989; Nair, 2010). Las especies de este género, usualmente, habitan en los bosques de montaña de los sectores central y noreste de cordillera andina (Antonelli et al., 2009; Aymard, 2019).
El Perú registra 18 de las 24 especies reconocidas, siendo el país con mayor cantidad de especies para el género Cinchona (Albán, 2013; Aymard, 2019; Huamán, Albán, & Chilquillo, 2019). Este género reviste importancia para el país, más allá de los niveles forestal y cívico, por representar la riqueza del recurso vegetal (Huamán et al., 2019; Zevallos, 1989). A pesar de esto, actualmente sus poblaciones se encuentran fuertemente amenazadas por actividades, como la sobre explotación del recurso, deforestación y degradación del hábitat por crecimiento urbano (Huamán et al., 2019; López, 2016; Mendoza, Ramírez, & Jiménez, 2014).
A nivel internacional diversos estudios moleculares en el género Cinchona y taxones relacionados emplearon el marcador matK, obteniendo información representativa en sistemática y filogenia (Andersson & Antonelli, 2005; Chilquillo, 2016; Maldonado et al., 2017). En contraparte, para el Perú, si bien los datos biológicos existentes sobre el género Cinchona registran aspectos históricos, taxonómicos, farmacológicos y ecológicos (Albán, 2013; Huamán et al., 2019; Nair, 2010; Zevallos, 1989), se carece de estu dios moleculares para este género y del conocimiento de su historia evolutiva, datos fundamentales para el correcto establecimiento de actividades de restauración sin perjuicio de la diversidad local (Albán-Castillo et al., 2020).
El PNC, establecido el 20 de setiembre de 1961, es la primer ANP de esta categoría en el Perú y se enfoca en la conservación del ave “guácharo” Steatornis caripensis y ecosistemas de tipo páramo, bosque seco y bosque húmedo (SERNANP, 2014;SERNANP 2017). Adicionalmente, es considerado una de las cuatro unidades de conservación para el género Cinchona en el Perú (Zevallos, 1989), pese a ello, hasta la actualidad se carece de investigaciones para este género en el PNC. Por consiguiente, el objetivo del presente estudio fue determinar la diversidad genética y las relaciones filogenéticas de las especies del género Cinchona que habitan en el PNC, con la finalidad de generar información que permita la conservación de este recurso.
2. Materiales y métodos
Área de estudio y recolección de la muestra: Corresponde al PNC, el cual se encuentra localizado en el área deno minada Cordillera de Tarros (2200-3500 m.s.n.m.), se ubica en los andes del norte del Perú, protegiendo bos ques montanos, páramos, y principalmente los bosques de neblina de las nacientes más altas de la cuenca del Marañón en la Región Cajamarca.
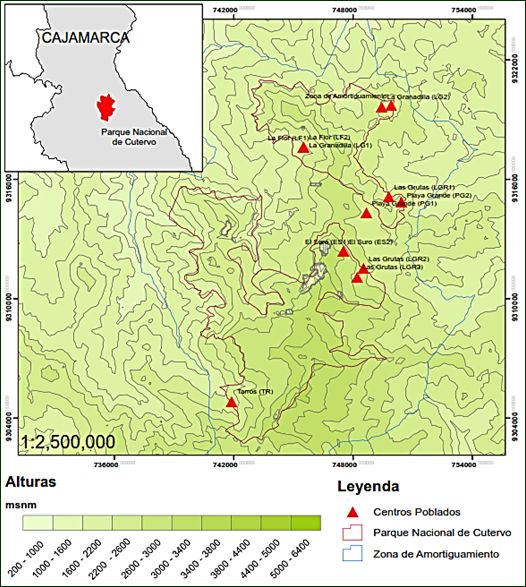
El material botánico fue colectado durante los meses de febrero a marzo del 2016, en siete centros poblados (Figura. 1), distribuidos en los distritos de: San Andrés de Cutervo, Santo Tomás, Santo Domingo de la Capilla, ubicados en el departamento de Cajamarca, Perú (Tabla 1).
La identificación preliminar en campo de los individuos del género Cinchona, se basó en caracteres morfológicos, siguiendo las claves establecidas Zeballos (1989) y bibliografía científica especializada (Aymard, 2019; Gómez, Beraun, Gómez, & Llatas, 2016; Killeen, García & Beck, 1993; Mostacero, Mejía & Gamarra, 2009). Con la finalidad de evaluar hojas sanas, se colectaron dos muestras de hojas maduras por individuo, extraídas de la parte superior de la planta, a una altura aproximada de 2,5m, solo se procesó una hoja por individuo, la segunda hoja fue almacenada como material de respaldo. Durante la colecta se registraron las coordenadas geográficas, el material biológico fue rotulado y transportado en bolsas herméticas estériles al laboratorio de la empresa Inca´Biotec SAC para su posterior procesamiento.
Tabla 1 Coordenadas de muestreo en el Parque Nacional Cutervo, Cajamarca-Perú
| Código de la muestra | Centro Poblado | Este (X) | Norte (Y) | Altitud (m.s.n.m.) |
| 1 | Tarros (TR) | 741883.00 | 9304831.00 | 2308.00 |
| 8 | La Flor (LF1) | 745508.00 | 9317600.00 | 2138.00 |
| 11 | La Flor (LF2) | 745505.00 | 9317605.00 | 2142.00 |
| 12 | La Granadilla (LG1) | 745508.00 | 9317605.00 | 2141.00 |
| 14 | Zona de Amortiguamiento (Puente El Suro-límite entre el distrito de San Andrés de Cutervo y Santo Tomás) (ZA) | 749914.00 | 9319693.00 | 2498.00 |
| 15 | La Granadilla (LG2) | 749433.00 | 9319636.00 | 2405.00 |
| 17 | Playa Grande (PG1) | 748665.00 | 9314309.00 | 2411.00 |
| 18 | Playa Grande (PG2) | 750428.00 | 9314864.00 | 2393.00 |
| 21 | Las Grutas (LGR1) | 749795.00 | 9315125.00 | 2318.00 |
| 23 | Las Grutas (LGR2) | 748514.00 | 9311518.00 | 2682.00 |
| 25 | Las Grutas (LGR3) | 748202.00 | 9311092.00 | 2771.00 |
| 30 | El Suro (ES1) | 747514.00 | 9312366.00 | 2850.00 |
| 32 | El Suro (ES2) | 747511.00 | 9312365.00 | 2850.00 |
Datum WGS84, Zona 17S.
Extracción de ADN cloroplastidial (ADN cp): Se realizó la extracción de ADN cp, siguiendo el protocolo establecido por Doyle & Doyle (1987), adaptado para el género Cinchona, mediante ensayos preliminares en el laboratorio. La superficie de las hojas fue desinfectada con alcohol al 70%, consecutivamente, se extrajo un corte de 2 cm2, evitando las nervaduras. Las muestras fueron trituradas en un mortero, con nitrógeno líquido, transfiriéndose 0,1g del triturado a un microtubo, en el cual se agregaron 590 µl del buffer de extracción Bromuro de Cetiltrimetilamonio (CTAB 2X) previamente calentado a 60 °C, y 10 µl de b-Mercaptoetanol. Seguidamente, la mezcla fue homogeneizada en Vortex (GB Lab®) durante 1 minuto, mantenida a una temperatura de 60°C por 60 minutos (uniformizando cada 15minutos), luego se adicionaron 300 µl de fenol concentrado y 300 µl de Cloroformo/Alcohol isoamílico (24:1).
La mezcla fue homogeneizada y centrifugada a 12 mil rpm por 10min a 4 °C, posteriormente se transfirió el sobrenadante a un nuevo microtubo, evitando tomar la interfaz, y se adicionó un volumen igual de Cloroformo /Alcohol Isoamílico (24:1), para ser centrifugada secuencialmente a 13 mil rpm durante 10 min a 4 °C y a 12 mil rpm por 10 minutos a 4 °C, recuperándose el sobrenadante a un nuevo microtubo. Rápidamente, se agregó Isopropanol helado (6/10) y se incubó a -20 °C por un lapso de 30-60 minutos. Se centrifugó secuencialmente la muestra a 10 mil rpm a 4 °C por 10 y 5 minutos, eliminando el sobrenadante y agregando 500 µl de etanol al 75% después del primer centrifugado, posteriormente se eliminó el sobrenadante.
Finalmente, el pellet se dejó secar a temperatura ambiente, se suspendió en 50µl de T.E (Tris HCL 10mM y EDTA 1Mm pH 8.0) y se agregó 1 µl de RNasa (10 mg/ml) e incubó a 37 °C por 30 minutos, el ADN se conservó a -20 °C. La cuantificación del ADN se realizó por espectrofotometría, con un volumen total de 60 μl de la muestra y una dilución de 5 μl ADN + 55 μl buffer TE pH: 8.
Amplificación del gen actiplant: Se amplificó la región específica del gen de actina del ADNcp mediante PCR, empleándose los siguientes primers: Forward (5’-CAC TAC TAC TGC TAA ACG GG AAA-3’) y Reverse (5’-ACA TCT GCT GGA AGG TGC TG-3’); y los volúmenes de reacción: 2,5 µL de Buffer Premix 10X, 0,5 µL de dNTP’s, 1 µL de MgCl₂ a 25 mM, 1 µL de Primer ACTplant F, 1 µL de Primer ACTplant R, 0,2µL de Taq DNA Polymerase (PlatinumR), 16,8µL de agua libre de nucleasas y 2 µL de ADNcp. Los ciclos térmicos para la amplificación consistieron en una desnaturalización inicial a 95 °C por 5 minutos, seguido por 35 ciclos compuestos por una desnaturalización a 95 °C por 5 segundos, hibridación a 54 °C por 45 segundos, elongación a 72 °C por 45 segundos, concluyendo con una extensión final a 72 °C por 6 minutos.
Amplificación del gen matK: La amplificación de la región del gen matK del ADNcp se realizó con los primers Forward (5’-CCT TCG ATA CTG GGT RAA AGA T-3’) Y Reverse (5’-CGT ACA GTA YTY TTG TGT TTA CGA-3’), para lo cual se emplearon los siguientes volúmenes de rección: 2,50µl Buffer Premix 10X, 0,50 µL de dNTPs (10mM), 0,50 µL de BSA (100X), Taq Polimerasa (0,5 U / μl) 0,10 µL, Primer Reverse matK3B (15 pMol / μl) 0,60 µL, Primer Forward matK2A (15 pMol / μl) 0,60 µL, agua ultra pura 18,20 µL, ADN 2,00 µL. Los ciclos térmicos para la amplificación consistieron en una desnaturalización inicial a 94 °C por 5 min, seguido por 34 ciclos compuestos por una desnaturalización 94 °C por 30 segundos, hibridación a 49 °C por 45 s; extensión 72 °C por 45seg, concluyendo con una extensión final a 72 °C durante 6min.
Validación de extracción de material genético: La validación de la extracción de ADN se llevó a cabo mediante la migración del ADN genómico en un gel de agarosa al 1%, agregándose 0,5 g de agarosa y 50 ml de TAE 1X; mientras que para los productos de los genes actiplant y matK se migraron geles de agarosa al 1,5%, agregándose 0,9 g de agarosa y 60 ml de TAE 1X. Ambos productos emplearon Blue/Orange Loading Dye 6X como tampón de depósito a una relación 5 : 1 μl por cada muestra. La migración del ADN se llevó a 90V por 15 min, colocando posteriormente el gel de agarosa en el transiluminador UV.
Secuenciamiento y análisis filogenético: Los productos de la PCR fueron preparados para posteriormente ser enviados a secuenciar a MACROGEN®. Las secuencias fueron editadas y alineadas con el algoritmo del software MAFFT (https://mafft.cbrc.jp/alignment/software/), en el software Mr Bayes 3,2 (Ronquist et al., 2012). Para la identificación de especies se consideró un límite máximo de divergencia genética de 1%.
Las secuencias del gen matK obtenidas por PCR fueron comparadas con las secuencias disponibles en la base de datos del GenBank usando el Basic Local Alignment Search Tool (BLAST) de la National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/BLAST)
El análisis filogenético se efectúo empleando el método de máxima verosimilitud (ML), la prueba de Filogenia Bootstrap se efectuó con 1000 repeticiones. La raíz del del filograma se infirió empleando el haplotipo generado de la secuencia del gen matK para la especie Remijia pedunculata como grupo de referencia.
El análisis diversidad genética se realizó mediante el polimorfismo de secuencias de ADN (DnaSP), evaluando como indicadores el número de haplotipos (h), la diversidad haplotípica (Hd) y la diversidad nucleotídica (π), los valores de los indicadores se calcularon con el software DnaSP (Librado & Rozas, 2009).
3. Resultados y discusión
Identificación de secuencias mediante el gen matK: Por medio del gen matK en la base de datos del GenBanK, se logró identificar 4 especies con una alta similitud: C. pitayensi, C. pubescens, C. calisaya y Remijia pedunculata (99%), con un índice de cobertura (Query Cover) promedio de 100%, corroborando una identificación precisa para todos los individuos (Tabla 2).
Tabla 2 Diversidad genética de especies del género Cinchona mediante el gen matK (En todos los casos se registró un índice de cobertura del 100% y similitud al 99%)
| Centro poblado | Código de Muestra | Identificación más cercana | Max puntuación | Código de Acceso |
| TR | 1 | Cinchona pitayensis | 375 | AY538382.1 |
| LF1 | 8 | Cinchona pitayensis | 713 | AY538382.1 |
| LF2 | 11 | Cinchona pitayensis | 791 | AY538382.1 |
| LG1 | 12 | Cinchona pubescens | 747 | LN680344.1 |
| ZA | 14 | Cinchona pubescens | 780 | LN680344.1 |
| LG2 | 15 | Remijiia pedunculata | 616 | AY538417.1 |
| PG1 | 17 | Cinchona calisaya | 675 | AY538379.1 |
| PG2 | 18 | Cinchona calisaya | 700 | AY538379.1 |
| LGR1 | 21 | Cinchona pubescens | 675 | LN680344.1 |
| LGR2 | 23 | Cinchona pubescens | 647 | LN680344.1 |
| LGR3 | 25 | Cinchona pubescens | 708 | LN680344.1 |
| ES1 | 30 | Cinchona pubescens | 750 | LN680344.1 |
| ES2 | 32 | Cinchona pubescens | 798 | LN680344.1 |
Descripción de centros poblados: Tarros (TR); La Flor (LF1); La Flor (LF2); La Granadilla (LG1); Zona de Amortiguamiento (Puente El Suro-límite entre el distrito de San Andrés de Cutervo y Santo Tomás) (ZA); La; Granadilla (LG2); Playa Grande (PG1); Playa Grande (PG2); Las Grutas (LGR1); Las Grutas (LGR2); Las Grutas (LGR3); El Suro (ES1) y El Suro (ES2).
Análisis filogenético: El árbol filogenético muestra que las muestras identificadas como C. pitayensis se agrupan en un solo clado con un elevado valor de soporte de bootstrap (<98%); por otra parte, destaca que las muestras identificadas como C. pubescens se agrupan en distintas ramas, algunas muestras de C. pubescens se agrupan en un solo clado con un valor elevado de soporte de Bootstrap (100% y 90%), sin embargo, son disimiles entre grupos. Finalmente, otro gran grupo conformado por C. pubescens, C. calisaya y R. pedunculata se encuentran agrupados (bootstrap 79), mientras que C. calisaya, se encuentra en una politomía sin resolver (Figura 2).
Distribución de sitios polimórficos: Se registraron 45 sitios polimórficos para el grupo de secuencias, descritas como: 56, 59, 114, 122, 123, 180, 183, 199, 205, 216, 44, 45, 47, 58, 112, 123, 180, 183 ,199, 205, 216, 55, 124, 158, 182, 46, 138, 140, 152, 154, 156, 159, 160, 179, 200, 201, 210, 211, 212, 213, 214, 215, 217, 218, 219, 110, 153, 204.
Análisis de diversidad genética: Entre los 13 organismos analizados se registró un total de 8 haplotipos (h), estando el primer haplotipo conformado por las muestras 1, 8, 11, 30 y 32; el segundo haplotipo, por las muestras 17 y 18; mientras que el resto de los haplotipos son correspondientes de manera individual a las muestras 12, 14, 15, 21, 23 y 25 (Tabla 3); finalmente se comprobó que los resultados muestran un alto nivel de diversidad haplotípica (Hd = 0,859) y nucleotídica (π = 0,35 %) para las poblaciones de las especies del género Cinchona dentro del PNC.
El centro poblado de Las Grutas (LG) registró la mayor cantidad de haplotipos por localidad, correspondiente a la especie C. pubescens, por otra parte, el centro poblado de Playa Grande registro el único haplotipo de la especie C. calisaya con dos muestras analizadas; C. pitayensis se identificó en tres muestras de un mismo haplotipo para los centros poblados de Tarros y La Flor; de manera resaltante, la especie C. pubescens se identificó en siete muestras para todos los centros poblados con excepción de Tarro, La Flor y Playa Grande, sin embargo, se agrupó en cinco haplotipos, siendo las secuencias de las muestras del centro poblado El Suro (30 y 32) compartidas con el haplotipo de C. pitayensis.
Para el PNC se logró identificar a la especie R. pedunculata y tres especies del género Cinchona: C. calisaya, C. pubescens y C. pitayensis; representando aproximadamente el 17% de la riqueza de especies reconocidas para el género Cinchona en el Perú, la cual asciende a 18 especies (Albán, 2013; Aymard, 2019; Huamán et al., 2019). De acuerdo con el Plan Maestro del PNC y publicaciones científicas referentes, se reconoce para esta ANP la presencia de C. humboldtiana, C. offinalis y C. pubescens (Sánchez & Calderón, 2010; SERNANP, 2014), por lo que nuestros resultados representan el primer registro de las especies C. pitayensis y C. calisaya para el PNC.
Nuestros resultados muestran conflicto con la idea ampliamente extendida de que R. pedunculata es la especie hermana del clado monofilético conformado por el género Cinchona (Andersson & Antonelli, 2005; Andersson, 1995; Aymard, 2019; Chilquillo, 2016). Cabe mencionar que la presencia de caracteres mixto entre Ladenbergia y Cinchona, determinó la reclasificación de Remijia pedunculata dentro del nuevo género Ciliosemina (Andersson & Antonelli, 2005; Aymard, 2019; Chilquillo, 2016; Manns & Bremer, 2010).
Tabla 3 Distribución de haplotipos identificadas con el marcador matK
| Haplotipos | Muestra | Individuos | Centro Poblado |
| 1 | 1-8-11-30-32 | 5 | TR, LF1, LF2, ES1, ES2 |
| 2 | 12 | 1 | LG1 |
| 3 | 14 | 1 | ZA |
| 4 | 15 | 1 | LG2 |
| 5 | 17-18 | 2 | PG1, PG2 |
| 6 | 21 | 1 | LGR1 |
| 7 | 23 | 1 | LGR2 |
| 8 | 25 | 1 | LGR3 |
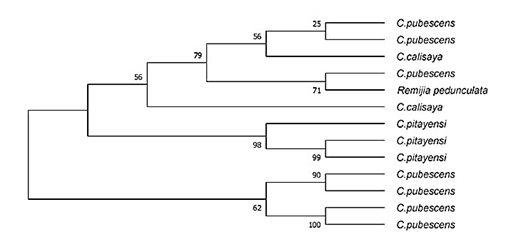
Figura 2 Árbol filogenético obtenido por Máxima verosimilitud de individuos del género Cinchona del PNC (haplotipos citoplasmáticos del gen matK). Los números en los nodos representan el valor de soporte de Bootstrap. Grupo hermano (“outgroup”): Remijia pendunculata.
El árbol filogenético reporta que las muestras identificadas como C. pitayensis se agrupan en un solo clado con un elevado valor de soporte de bootstrap (<98%); por otra parte, destaca que las muestras identificadas como C. pubescens se agrupan en distintas ramas, algunas de estas muestras se agrupan en un solo clado con un valor elevado de soporte de bootstrap (100% y 90%), sin embargo, estas mismas son disimiles entre grupos. Finalmente, otro gran grupo conformado por C. pubescens, C. calisaya y R. pedunculata se encuentran vinculados (bootstrap 79%), mientras que C. calisaya se encuentra en una politomía sin resolver (Figura 2).
La robusta agrupación de C. pitayensis demuestra que esta especie se encuentra conservada en el PNC. Por otra parte, la distribución de C. pubescens en diversos clados independientes y en conjunto con C. calisaya y R. pedunculata, indican que esta especie tendría variedades hibridando con otras especies en determinadas zonas y conservadas en otras.
Patrones similares a los reportados en el presente estudio fueron reportados en el árbol filogenético de la tribu Cinchoneae, generado través del método de máxima parsimonia empleando cinco marcadores (región ITS, gen matK, gen rbcL, intrón rps16 y la región trnL-F) (Andersson & Antonelli, 2005); y para el árbol filogenético generado para Cinchoneae e Isertieae a través del método de inferencia bayesiana empleando siete marcadores (ITS1, ITS2, la región espaciadora atpB-rbcL, ndhF, rbcL, el intrón rps 16 y la región trn-T-L-F) (Manns & Bremer, 2010); esto a pesar de que el uso de diferentes marcadores moleculares y métodos de evaluación pueden generar diferentes valores de diversidad.
El registro de una mayor cantidad de haplotipos de C. pubescens sería un indicador de la presencia de variedades originadas por la presión ambiental, la cual ocasiona el desarrollo de caracteres adaptativos con tendencia a la especiación (García, 2012). Lo anterior mencionado sería correspondiente con lo manifestado por Albán-Castillo et al. (2020), quienes mencionan que la alta plasticidad morfológica presente en varias especies de Cinchona puede ser atribuida a gradientes ambientales, así como a eventos de hibridación o duplicación de sus genomas.
La diversidad haplotípica (Hd = 0859) y nucleotídica (π =0,35%) del género Cinchona en el PNC es elevada respecto del área del PNC (8,214.23 hectáreas) (SERNANP, 2017). Se conoce que la diversidad genética está afectada por eventos demográficos históricos y contemporáneos que pueden ser atribuibles a un contacto secundario entre linajes alopátricos previamente diferenciados, o bien a una gran población estable con una larga historia evolutiva (Grant & Bowen, 1998). A nivel local, la distribución de la diversidad genética registró al centro poblado de “Las Grutas” con la mayor cantidad de haplotipos (C. pubescens) y a “Playa Grande” con un único haplotipo (C. calisaya); mientras que la especie C. pitayensis con un único haplotipo se registró en los centros poblados de “Tarros” y “La Flor” y C. pubescens del centro poblado “El Suro” (30 y 32), esto se relacionaría con el comportamiento diferencial de las poblaciones, debido a factores actuales como la agricultura y ganadería local; y factores históricos como la formación de la geografía presente.
Los resultados aquí expuestos respaldan la hipótesis de que la distribución biogeográfica de C. calisaya, al sur del Perú y en Bolivia, con un reciente registro puntual de ampliación hacia el norte, representaría un caso de vicarianza respecto de la especie C. pubescens (bootstrap 79%), de amplia distribución, tal como lo propusieron Andersson & Antonelli (2005). No obstante, para respaldar esta hipótesis se recomienda complementar los estudios con el uso de más marcadores moleculares.
Con respecto a C. calisaya, la cual se encuentra en categoría de conservación “Vulnerable” para la legislación nacional (MINAGRI, 2006) y representa endemismo para el Perú y Bolivia, esta es reconocida como la especie con mayor cantidad de alcaloides (Nair, 2010), mientras que, recientes investigaciones indican que su composición de alcaloides anti malaria a nivel local estaría siendo dirigida por su filogenia (Maldonado et al., 2017). En consecuencia, el haplotipo de C. calisaya descrito para el PNC podría ser una variante particular de su diversidad genética, por lo que continuar con estudios en esta especie es preponderante.
4. Conclusiones
Mediante este estudio, se incrementó el número de especies del género Cinchona en el PNC con la identificación molecular de C. calisaya, C. pubescens y C. pitayensis, asimismo se confirmó la presencia de R. pedunculata. La resolución de la topología del árbol filogenético confirma que C. pitayensis se encuentra conservada dentro del PNC, mientras que la identificación de 7 haplotipos distribuidas en tres grupos de Cinchona demuestra la elevada diversidad genética presente en el PNC. La especie C. pubsecens se encuentra presente en distintos grupos con similitud variable. Los valores de la diversidad haplotípica (Hd = 0,859) y nucleotídica (π = 0,35%) del género Cinchona y el registro del haplotipo de la especie C. calisaya, endémica para el sur andino y con categoría de conservación nacional de “vulnerable”, registrada para el PNC permitirá diseñar y detectar nuevas especies y comprender su distribución, además de concientizar a la población sobre la importancia en la conservación y expansión de esta especie amenazada.