INTRODUCCIÓN
Hepatozoon canis es un protozoario apicomplexo de la clase Conoidasida (Cavalier-Smith, 2003; Adl et al., 2005). Este hemoparásito es transmitido por la garrapata Rhipicephalus sanguineus, la cual parasita comúnmente a cánidos (Baneth et al., 2007). Otras garrapatas como Amblyomma ovale, Haemaphysalis flava, Haemaphysalis longicornis, Rhipicephalus microplus y Rhipicephalus turanicus también han sido descritas como vectores de H. canis (Murata et al., 1995; de Miranda et al., 2011; Giannelli et al., 2017; Kwon et al., 2017).
La transmisión de H. canis ocurre por la ingesta de oocistos maduros contenidos en alguno de sus vectores (Baneth et al., 2007), y mediante la transmisión intrauterina (Murata et al., 1993). La hepatozoonosis puede causar manifestaciones clínicas severas en los perros (anorexia, fiebre, letargia y linfadenomegalia) (Otranto et al., 2011).
H. canis se ha reportado en África (Harris et al., 2017), Asía (Kwon et al., 2017), Europa (Ebani et al., 2015) y Oceanía (Greay et al., 2018). En Sudamérica se ha documentado ampliamente en perros de áreas rurales y urbanas (Rey-Valeiron et al., 2012; Da Silva et al., 2016; Vezzani et al., 2017). En Colombia fue reportada en 2012 emplean- do técnicas moleculares (Vargas-Hernandez et al., 2012), aunque existen otros reportes por frotis sanguíneo (Arcila et al., 2005; Ardila et al., 2007; Acevedo et al., 2009; Castellanos, 2009; Correa, 2013; Cala et al., 2018).
Dentro de las técnicas empleadas para el diagnóstico se encuentran el extendido de sangre periférica, así como pruebas serológicas e histopatológicas; además de la reacción en cadena de la polimerasa (PCR) que presenta mayor especificidad y sensibili- dad para el diagnóstico definitivo (Oyamada et al., 2005). El propósito del presente traba- jo fue la evaluación y aplicación de una PCR en tiempo real empleando la tecnología de sondas de hibridación con detección dúplex y control interno de amplificación GADPH para la detección de H. canis en perros de la ciu- dad de Cúcuta (Colombia).
MATERIALES Y MÉTODOS
Muestras
Entre mayo y junio de 2016 se recolec- taron 350 muestras de sangre de perros que fueron llevados a consulta a clínicas veteri- narias de la ciudad de Cúcuta (Colombia) con sospecha clínica de hemoparasitosis. Se tomó 1 ml de sangre con EDTA de los pacientes con previa autorización de sus propietarios. La muestra fue distribuida en dos alícuotas de 500 µl en viales de 1.5 ml (SSI, USA) y almacenadas a -20 °C para la extracción de ADN en las 72 horas siguientes a la recolecta.
Cebadores para PCR Tiempo Real
Se utilizaron secuencias del gen 18S rRNA para H. canis (AF176835.1, LC331055.1, HQ829448.1, KP715303.1, FJ497022.1, KJ605145.1, KY965141.1) y secuencias para el gen GADHP (AB0382- 41.1, NM_001003142.2) disponibles en GenBank (www.ncbi.nlm.nih.gov). Las se- cuencias recuperadas se alinearon para ge- nerar secuencias consenso, con base a las cuales se diseñaron los cebadores y sondas empleando el programa Primer3Plus (http://www.bioinformatics.nl/cgi-bin/primer3plus/ primer3plus.cgi) (Untergasser et al., 2007).
Extracción de ADN y Amplificación de Genes
Se utilizó la técnica de Salting out mo- dificada. Se tomó 200 µl de sangre y se agre- gó 1000 µl de ddH O, se centrifugó a 13 400 x g durante 2 min y se descartó el sobrenadante. Se agregó 500 µl de buffer de lisis de glóbulos rojos (10 mM Tris [Biobasic, USA]- HCl pH 8.0, Triton al 1% [VWR Amresco], sacarosa al 11% [Carlo Erba]), se agitó en el vórtex (fuerte) y se centrifugó a 13400 x g durante 2 min, descartándose el sobrenadante y se repitió este paso una vez más. Luego se agregó 500 µl de ddH O, se centrifugó a 13 400 x g durante 2 min, des- cartándose el sobrenadante. Seguidamente, se agregó 180 µl de buffer de lisis de glóbulos blancos al sedimento (10 mM Tris - HCl pH 8.0, 400 mM NaCl [Emsure Merck], 2 mM EDTA p VWR Amresco]), más 10 µl de SDS al 20% y 20 µl de proteinasa K (1 mg/ml proteinasa K [Thermo Fischer], 1% SDS, EDTA 2 mM). Se incubó a 37 °C durante 1 h (vórtex cada 15 min).
Las proteínas se precipitaron con 90 µl de 5M NaCl centrifugándose a 13 400 x g durante 5 min. Luego se agregó 20 µl de 5M NaCl al sobrenadante y se centrifugó a 13400 x g durante 5 min. Al sobrenadante se le agre- gó 300 µl de alcohol al 99% y se centrifugó a 16 700 x g durante 5 min. Seguidamente se realizaron dos lavados con 300 µl de alcohol al 70%. Las muestras se hidrataron con 150 µl de ddH O para su posterior cuantificación con el Biofotómetro de Eppendorf Plus (Eppendorf, Alemania).
Las reacciones en cadena de la polime- rasa en tiempo real se realizaron en un termociclador Rotor-Gene Q 2PLEX HRM de Qiagen empleando los cebadores y son- das diseñadas en este trabajo (Cuadro 1) en un volumen final de 12 µl, constituido por el master mix Quantinova probe de Qiagen, 400 µM por cebador, 200 µM de sonda y 90 ng de DNA. Para cada PCR en tiempo real se usaron controles positivos de H. canis, controles negativos (pacientes sanos) y un control interno de amplificación del gen GADPH.
Las condiciones de amplificación se establecieron en 95 °C durante 5 min (acti- vación), seguido de 30 ciclos a 95 °C durante 10 s (desnaturalización) y 60 °C durante 30 s (anillamiento - extensión). La fluorescencia se midió en cada ciclo de anillamiento - ex- tensión. Las reacciones fueron evaluadas con el software Rotor-Gene Q v. 2.3, conside- rándose como positivas aquellas donde la cantidad de fluorescencia excedía el valor del umbral (threshold) (emisión basal de 0.05 delta florescence) y seguido de la curva en forma sigmoide.
RESULTADOS
Los cebadores fueron diseñados para generar fragmentos conservados de 192 pb (control positivo -18S rRNA) y 101 pb (con- trol interno GADPH) (Cuadro 1). El tamaño de los fragmentos se verificó mediante geles de electroforesis teñidos con RedGel y visualizados bajo luz ultravioleta. El ciclo umbral (Cq) para el control positivo fue de y para el control interno GADPH fue de 16.98 (Figura 1).
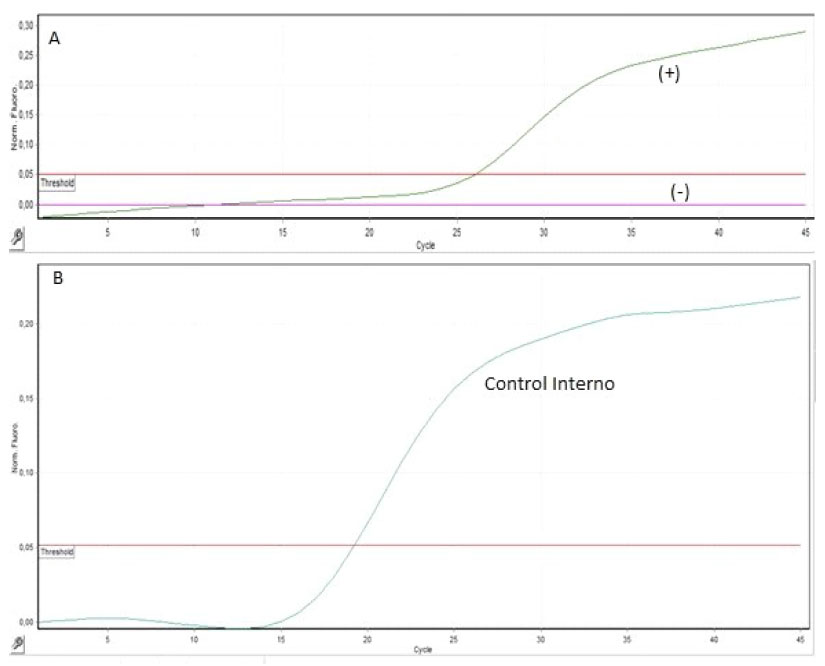
Figura 1 Valores del ciclo umbral (Cq) del control positivo y negativo para Hepatozoon canis y del control interno. El control positivo con un Cq de 26.47 y el control negativo que no supera el umbral, mientras que el Cq del control interno fue de 16.98
La muestra positiva secuenciada por Macrogen Inc. (Corea) con los cebadores HEPAFW y HEPARV arrojó en el BLAST(r) un 99% de identidad con la secuencia MG758124.1 de Hepatozoon canis y verifi- có el tamaño de 192 pb. Por otro lado, el con- trol interno GADPH de 101pb presentó un 96% de similitud con la secuencia XM_025438124.1 de Canis lupus familiaris.
Un total de 30/350 (8.6%) muestras de caninos pacientes de las clínicas veterinarias atendidos con sospecha de hemoparasitosis fueron positivas para H. canis.
DISCUSIÓN
La documentación sobre Hepatozoon canis y la hepatozoonosis canina en Colom- bia es escasa. La primera mención del hemo- parásito y su patología la realizaron Arcila et al. (2005), quienes hallaron gametocitos de H. canis en un Pitbull. Castellanos (2009) catalogó la enfermedad como emergente en Colombia basándose en reportes propios y anteriores (Arcila et al., 2005; Ardila et al., 2007); sin embargo, la primera caracteriza- ción molecular de H. canis fue realizada por Vargas-Hernandez et al. (2012) en perros de tres localidades del país.
Los escasos reportes existentes en el país no permiten establecer la distribución de la parasitosis y los roles epidemiológicos (Castellanos, 2009). No obstante, se relacionan con reportes de Rhipicephalus sanguineus, Rhipicephalus microplus y Amblyomma ovale parasitando perros (Paternina et al., 2009; Rivera-Páez et al., 2018), además de la amplia distribución de Rhipicephalus sanguineus en Colombia (López, 2017), su principal especie vector (Baneth et al., 2007).
El presente trabajo es el primer reporte de Hepatozoon canis mediante PCR en tiempo real en la ciudad de Cúcuta (Colombia) y el segundo reporte de H. canis des- pués del trabajo de Cala et al. (2018), quie- nes detectaron el hemoparásito por extendi- do sanguíneo y PCR. El presente estudio re- porta 8.6% (30/350) de casos positivos, una frecuencia baja respecto a los 29/91 perros (31.8%) positivos con H. canis reportada por Vargas-Hernández et al. (2012), aunque su- perior al 2.3% (14/614) de frecuencia docu- mentada por Li et al. (2008). Se recomienda realizar un muestreo aleatorio de caninos, idealmente representativo de la población, con el fin de generar valores de prevalencia que se puedan comparar con otros estudios.
Por otro lado, se logró una extracción eficiente de ADN, además de la detección de H. canis a través de la amplificación de un fragmento conservado de 192 pb del gen 18S rRNA, fragmento menor al amplificado con PCR en tiempo real por Li et al. (2008) con 198 pb para Hepatozoon spp y por Inokuma et al. (2002) con 625 pb para H. canis.
CONCLUSIONES
Se logró la detección de Hepatozoon canis en sangre de caninos domésticos en la ciudad de Cúcuta, Colombia, me- diante la técnica de PCR en tiempo real empleando sondas de hibridación con detección dúplex, tanto de H. canis como del control interno de amplificación GADPH.
Se reporta una frecuencia de 8.6% (30/350) de casos positivos de pacientes atendidos en clínicas veterinarias con sospe- cha de hemoparasitosis












 uBio
uBio 


