INTRODUCCIÓN
Salmonella spp es una bacteria zoonótica distribuida mundialmente y ha sido reconocida como una de las bacterias de mayor relevancia en la generación de enfermedades transmitidas por alimentos (ETA) (Scallan et al., 2011), siendo la carne de cerdo uno de los vehículos de transmisión al humano (Boyen et al., 2008; Baer et al. 2013). En 2018 se presentaron 60 999 casos de salmonelosis en Estados Unidos (CDC, 2019) y 91 857 casos en Europa, donde el 5.4% de los casos estuvieron asociados a productos a base de carne de cerdo (EFSA y ECDC, 2019). Los cerdos pueden adquirir la bacteria durante su estancia en granja, ya que es un contaminante del agua, concentrados y diferentes superficies, entre otros (Baer et al., 2013).
Salmonella spp en el cerdo puede causar infecciones sintomáticas, asintomáticas (Boyen et al., 2008) y subclínicas, lo que genera pérdida de peso (aprox. 3 kg por animal) durante el ciclo de producción (Wong et al., 1997), causando severas pérdidas económicas. Los animales portadores de Salmonella spp durante el proceso de beneficio presentan mayor riesgo de contaminar la canal (De Busser et al., 2011); siendo esta la razón del diseño de estrategias de implementación de medidas de control en las plantas de beneficio que incluyen etapas de desinfección e implementación del sistema de análisis de peligros y puntos críticos de control (HACCP) (FSIS, 2018).
En Colombia, según el Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA), existían 80 plantas de beneficio registradas en 2019, siendo 23/80 (28.8%) de autoconsumo (plantas autorizadas para comercializar el producto en el municipio de ubicación) (DANE, 2015), 55/80 (68.7%) plantas clasificadas como nacionales (venta en todo el país) y 2/80 (2.5%) habilitadas para exportar (INVIMA, 2019).
El boletín epidemiológico del Instituto Nacional de Salud (INS) del año 2018, reportó que el 6% de los brotes alimentarios ocurridos en Colombia se relacionaban con el consumo de carne contaminada de res o cerdo, aunque no se determinó en todos los casos el agente causal (INS, 2018). Por otra parte, a nivel mundial la presencia de serotipos de Salmonella spp resistentes a antibióticos ha incrementado la tasa de hospitalización (Barco et al., 2014); siendo los aislamientos de origen animal los más resistentes (European Food Safety et al., 2020), y que pueden estar asociados al uso indiscriminado de antibióticos en granjas (Rincón-Gamboa et al. 2021a,b; Mora-Gamboa et al., 2022).
En Colombia, la información disponible sobre la prevalencia de Salmonella spp en plantas de beneficio porcícolas es escasa, ya que los muestreos oficiales se iniciaron en la última década (INVIMA, 2019). En este orden de ideas, los estudios realizados en el país también son escasos, habiéndose estimado prevalencias de Salmonella spp en canales de cerdo entre 10 y 28% (Carrascal et al., 2015). Por lo tanto, para conocer la prevalencia de Salmonella spp reportada en plantas de beneficio porcícola en Colombia entre los años 2009-2020 y tener un panorama actualizado al respecto, se realizó una revisión sistemática de literatura y un metaanálisis sobre la prevalencia de Salmonella spp en plantas de beneficio en Colombia.
MATERIALES Y MÉTODOS
Se realizó la revisión de estudios que reportaban la prevalencia de Salmonella spp en plantas de beneficio de cerdo en Colombia. Se definió la prevalencia mediante la fórmula P = (N.° muestras positivas para de Salmonella spp / N.° muestras analizadas) * 100, expresado en porcentaje.
Estrategia de Búsqueda
La búsqueda bibliográfica se realizó en las bases datos Science Direct, PubMed, SciELO, EBSCO host, Redalyc, ProQuest y Google Scholar. Se utilizaron palabras clave como: Salmonella, Colombia, cerdo, planta de beneficio, canal, prevalencia; asó como los pares boleanos AND y OR en la búsqueda. Se tuvieron en cuenta todos los estudios realizados que incluyeran criterios de inclusión como: prevalencia de Salmonella spp en plantas de beneficio de cerdos publicados entre los años 2009 al 2020, en inglés o español, en todas las regiones de Colombia. Como criterios de exclusión se descartaron resultados redundantes. Adicionalmente, se incluyeron informes técnicos del gobierno, trabajos de grado y presentaciones de reuniones técnicas.
Extracción de Resultados
Se construyó un archivo en Microsoft Excel con la siguiente información: autor, año de publicación, tipo de muestra (canales o muestras ambientales), región, número de muestras analizadas, técnica o metodología utilizada para la detección del patógeno, prevalencia, serotipos y resistencia a antimicrobianos. Luego, dos investigadores independientes revisaron los resúmenes de los documentos encontrados, y los procedimientos y resultados obtenidos de todos los documentos bajo los criterios de inclusión y a partir de ellos se excluyeron o incluyeron los documentos. En la Figura 1 se muestra el procedimiento para la selección y eliminación de los artículos seleccionados.
Metaanálisis
Para el cálculo de la prevalencia combinada se incluyeron 11 documentos obtenidos del análisis previo, usando el programa MetaXL (Epigear International) (Barendregt et al., 2013). Para ello, se usaron tres modelos de metaanálisis (efectos fijos, efectos aleatorios y heterogeneidad de la varianza inversa) para estimar la heterogeneidad de los datos y elegir el modelo adecuado para estimar la prevalencia combinada de Salmonella spp.
RESULTADOS
Se recolectaron 3007 documentos relacionados con las palabras clave. Mediante los criterios de exclusión y basados en el Resumen de los trabajos se preseleccionaron 51 documentos que cumplieron con los criterios de inclusión. Después, al revisar detalladamente los resúmenes se eliminaron otros 16 (31.4%) por no estar del todo relacionados. Los 35/51 artículos restantes fueron seleccionados para lectura completa, y que al revisarlos se encontró que 23/35 (71.4%) estaban duplicados y 5/35 (14.28%) artículos no reportaban datos de prevalencia, eran revisiones o las muestras provenían de otros puntos de la cadena (granja y puntos de venta). De esa forma, solo 7/51 (13%) artículos fueron incluidos en el metaanálisis.
Adicionalmente, se incluyeron cuatro documentos obtenidos de la literatura gris, los cuales eran reportes suministrados por el Instituto Nacional de Vigilancia de Medicamentos y Alimentos (INVIMA, 2019) y reportes de proyectos realizados en el país (Ramón et al., 2015), para un total de 11 documentos.
Aunque el periodo de inclusión comenzaba en 2009, el primero de los 11 documentos fue del 2012. En el Cuadro 1 se presentan los resultados más relevantes de los 11 estudios incluidos en la revisión. Se observa que las regiones en las que se realizó la mayor cantidad de estudios fueron el Departamento de Antioquia y en Bogotá. Los principales serotipos reportados (Cuadro 1) fueron S. Typhimurium, S. Javiana y S. Derby.
Cuadro 1. Estudios incluidos en la revisión de literatura 2009-2020
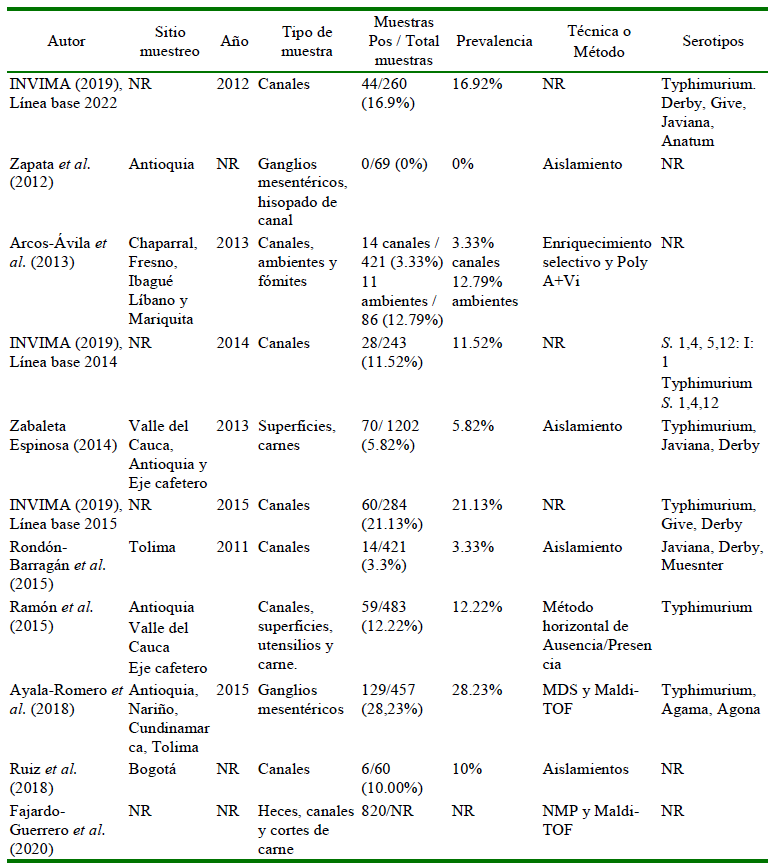
NR: No reportado; NMP: Número más Probable
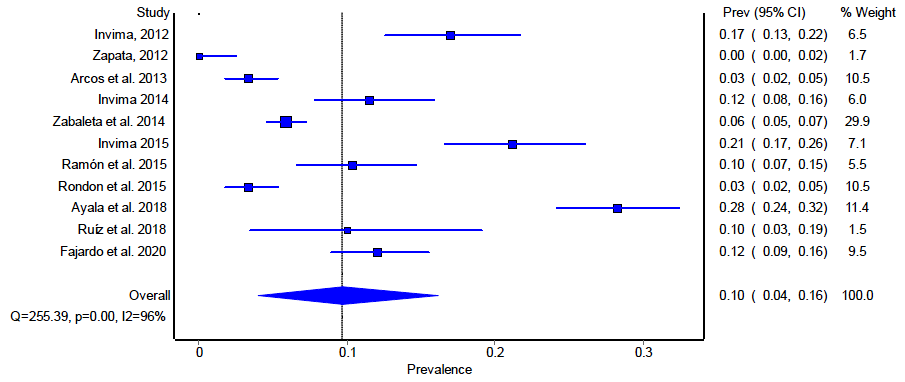
En el Cuadro 2 se muestra la prevalencia combinada estimada utilizando los tres modelos. El valor del test de heterogeneidad de Cochran’s Q (Q) y el índice del inverso de la varianza (I2) fue mayor al 75 %, indicando una alta heterogeneidad en los estudios. Según Barendregt et al. (2013), en el caso de datos con alta heterogeneidad se recomienda el uso del modelo heterogeneidad de la varianza inversa. El valor de prevalencia combinada fue de 9.7% (4.0-16.2%).
Cuadro 2. Prevalencia combinada e intervalos de confianza para los modelos empelados
| Tipo de modelo | Prevalencia combinada | LCI | HCI | Rango1 | Q | I2 |
|---|---|---|---|---|---|---|
| Modelo de efectos fijos | 0.097 | 0.088 | 0.106 | 0.018 | 255.39 | 96 |
| Modelo de efectos aleatorios | 0.097 | 0.053 | 0.152 | 0.098 | 255.39 | 96 |
| Modelo heterogeneidad de la varianza inversa | 0.097 | 0.040 | 0.162 | 0.122 | 255.39 | 96 |
LCI: Intervalo de confidencia bajo (2.5 %); HCI: interval de confidencia alto (97.5 %)
1 Diferencia entre el valor mínimo y máximo
La Figura 2 muestra la prevalencia estimada de cada estudio y el peso de los datos de cada estudio en la estimación de la prevalencia combinada. En el estudio de Zabaleta et al. (2014) se obtuvo el mayor peso relativo respecto al cálculo de la prevalencia combinada con 29.9%. Sin embargo, el análisis de sensibilidad mostró que ningún estudio afectó significativamente (p=0.000) el cálculo de la prevalencia combinada (Zabaleta, 2014).
DISCUSIÓN
La revisión de literatura permite inferir que Salmonella spp ha estado circulando en las plantas de beneficio en Colombia, encontrándose en muestras de canales porcinas (Zabaleta, 2014; Ramón et al., 2015), ganglios mesentéricos (Ayala-Romero et al., 2018) y en muestras ambientales (utensilios, corrales, piso, por equipos) (Rondón-Barragán et al., 2014). En estos casos las fuentes de contaminación estuvieron asociadas a la presencia de cerdos infectados desde la granja y a la contaminación cruzada con equipos y utensilios dentro del proceso de sacrificio (Arcos-Ávila et al., 2013; Zabaleta, 2014; Sánchez-Rodríguez et al., 2018). Los datos presentaron variaciones entre 0 a 28% de prevalencia de Salmonella en las canales de cerdo, y al estabilizar las varianzas de los respectivos estudios se obtuvo una prevalencia combinada del 9.7%; datos similares a los obtenidos enWisconsin, Estados Unidos (11.414.7%) (Algino et al., 2009) y en España (11%) (Sánchez-Rodríguez et al., 2018).
La prevalencia de Salmonella spp en las plantas procesadoras de carne de cerdo puede variar debido a factores como las condiciones geográficas, las prácticas de cría en las granjas y las estaciones del año (Berends et al., 1997); sin embargo, en el caso de Colombia, no se presentan estaciones, salvo dos épocas de lluvia y dos de sequía. Al revisar los datos de los estudios incluidos, se encontró que la variabilidad en la prevalencia reportada obedecía a factores como la técnica de muestreo (tipo de esponja empleada, tamaño del área muestreada, método de recolección de la muestra, tipo de diluyente empleado y protocolos de aislamiento, entre otros) (Arcos-Avila et al., 2013; Zapata et al., 2012; Rondón-Barragán et al., 2015), las características de la planta de beneficio (tamaño, equipos empleados, protocolos de higiene, procesos de desinfección de canales, entre otros) (Fajardo-Guerrero et al., 2020; Ayala-Romero et al., 2018), así como el origen de los lotes de cerdos que ingresaron a las plantas (INVIMA, 2019; Ruiz et al., 2018). Las plantas de beneficio porcino colombianas prestan el servicio de sacrificio a los productores, así la mayoría de ellas son privadas y reciben cerdos de diferentes granjas, de tal manera que ingresan cerdos procedentes de granjas tecnificadas, semi-tecnificadas y de pequeños productores, que cuentan con normas de bioseguridad diferentes, además, de recibir cerdos de diferentes zonas geográficas del país; por tanto, el riesgo de contaminación cruzada en este tipo de establecimientos se incrementa.
Al analizar los métodos de detección e identificación (Cuadro 1) se observaron diferencias en las técnicas empleadas, incluidos los métodos microbiológicos de cultivo e identificación, NMP y detección e identificación molecular; lo que puede afectar la capacidad de recuperación del microorganismo. Por ejemplo, Zapata et al. (2012) utilizaron un método que no corresponde a los métodos estandarizados por entes internacionales como el Departamento de Agricultura de Estados Unidos y la Comunidad Europea, entre otros, para la detección de Salmonella spp, a partir de canales de cerdo, pues no contó con pre-enriquecimiento. Estas diferencias en los métodos utilizados pueden explicar, en parte, la ausencia de resultados positivos, pues la ausencia de pre-enriquecimiento puede afectar la recuperación de la Salmonella (Wesche et al., 2009; Doyle et al., 2012).
Los serotipos más frecuentes encontrados por Zabaleta (2014) fueron S. Typhimurium (70%), S. Javina (9%) y S. Derby (6%), mientras Ayala-Romero et al. (2018) reportaron S. Typhimuriun (12.5%), S. Javiana (12.5 %) y S. Agona (12.5 %), sugiriendo una estrecha relación entre el serotipo de Salmonella proveniente del cerdo y la contaminación de la canal. Sin embargo, Rondón-Barragán et al. (2015) reportaron S. Muester en 4/9 (44.4%), seguido de S. Derby (22.2%), en Tolima, Colombia, como el serotipo principal aislado de canales, aunque el número de cepas fue bajo, en comparación con los estudios anteriores (Arcos-Ávila et al., 2013), lo que puede generar una subestimación en la información.
Los resultados reportados en Colombia coinciden con los obtenidos en Europa entre los años 2006-2007, donde S. Typhimurium y S. Derby fueron los serotipos más comúnmente aislados de canales porcinas, representando el 49.4 y el 24.3%, respectivamente (EFSA, 2008). Asimismo, en España se reportaron como serotipos predominantes a S. Typhimurium (35.6%) y S. Derby (31.1%) (Sánchez-Rodríguez et al., 2018). En el caso de Brasil, Dos Santos Bersot et al. (2019) indican que 63.1% de las cepas de Salmonella spp aisladas de cerdos corresponden a S. Typhimuriun.












 uBio
uBio