Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Anales de la Facultad de Medicina
versão impressa ISSN 1025-5583
An. Fac. med. vol.78 no.2 Lima abr./jun. 2017
http://dx.doi.org/10.15381/analesv78i2.13183
ARTÍCULO ORIGINAL
Polimorfismo Val/Met en gen OCT1 de respuesta a metformina en muestras de Lima y Puno. Enfoque farmacogenético de la diabetes
Val/Met polymorphism in metformin response gene OCT1 in Lima and Puno samples. Pharmacogenetic approach of diabetes
Luis Olivares1,a, María Villanueva1,a, Elvira Aparicio1,a, Aracelli Yauri 1,a, Rosa Medina1,a, Noelia De la Sota1,a,Doris Huerta2,b, Luisa Negrón 1,b, María Luisa Guevara3,c, José Sandoval3,d, Ricardo Fujita3,e, Oscar Acosta1,f
1 Facultad de Farmacia y Bioquímica, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Facultad de Medicina, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 Centro de Genética y Biología Molecular, Facultad de Medicina, Universidad de San Martín de Porres, Lima, Perú
a Estudiante de Farmacia y Bioquímica
b Doctora en Biología
c Doctora en Ciencias
d Doctor en Genética
e PhD en Genética Molecular
f Magister en Bioquímica, Biólogo Genetista
Resumen
Introducción: La farmacogenética tiene utilidad clínica para evaluar los efectos de los fármacos según perfil genético y aporta a la medicina poblacional y personalizada. La diabetes mellitus tipo 2 (DMT2) es una enfermedad prevalente en el Perú y el mundo. Para su tratamiento existen varios fármacos, entre ellos la metformina. La respuesta individual puede estar influenciada por el polimorfismo Val/Met en el gen octT1 y las frecuencias varían según grupo étnico. Se ha relacionado al alelo Met con una menor respuesta a la metformina. Objetivo: Evaluar la distribución del polimorfismo Val/Met en el gen OCT1 en muestras de sangre de sujetos de Lima y Puno, e inferir su impacto en la farmacogenética de la DMT2. Diseño: Estudio descriptivo, transversal. Lugar: Facultades de Farmacia y Bioquímica, Medicina, Universidad Nacional Mayor Lima, Perú; Centro de Genética y Biología Molecular, Facultad de Medicina, Universidad de San Martín de Porres, Lima, Perú. Participantes: 56 individuos de Puno y 57 de Lima-ciudad. Intervenciones: Análisis del polimorfismo Val/Met en el gen OCT1 con la técnica PCR-RFLP. Principales medidas de resultados: Frecuencias genotípicas y alélicas. Resultados: Las frecuencias de los genotipos, en general,fueron: Val/Val=85,0% y Val/Met=15,0%. La frecuencia del alelo Val, en general, fue mayor al 93%; el alelo Met, asociado con una menor respuesta a metformina, se encontró presente en Amantaní (8,3%) y Lima (9,6%), y ausente en Taquile. Conclusiones: Para el alelo Val del gen OCT1, se ha encontrado la más alta frecuencia registrada en el mundo. Respecto al alelo Met, aunque es menos frecuente, existen diferencias entre las subpoblaciones peruanas evaluadas, y ese conocimiento puede ayudar en la farmacogenética y la toma de decisiones en el tratamiento con los antidiabéticos orales como la metformina.
Palabras clave: Farmacogenética; Gen OCT1; Polimorfismo Val/Met; Diabetes; Metformina; Lima, Puno, Perú.
Abstract
Introduction: Pharmacogenetics can be used in clinical analysis to assess the efficiency of drugs according to the patient’s genetic profile, and it is becoming important for population genetics and precision medicine. The type 2 diabetes mellitus (T2DM) is highly prevalent all over the world, including Peru. Among the different drugs for T2DM, metformin is used the most and patient’s response to it can be influenced by the Val/Met polymorphism of the OCT1 (SLC22) gene, where Met is associated with a lower response. The frequencies of these polymorphisms vary according to ethnic origin. Objective: To evaluate the distribution of the Val/Met polymorphism in the OCT1 gene in samples of Lima and Puno, and to assess their impact on pharmacogenetics of T2DM. Design: Descriptive, cross-sectional study. Settings: Faculties of Pharmacy and Biochemistry, and Medicine, Universidad Nacional Mayor de San Marcos, Lima, Peru; Centro de Genética y Biología Molecular, Facultad de Medicina, Universidad de San Martín de Porres, Lima, Peru. Participants: DNA samples of 56 non-selected subjects from Puno and 57 from Lima regions. Interventions: Analysis of the Val/Met polymorphism in OCT1 gene using the PCR-RFLP technique. Main outcome measures: Phenotypic and allelic frequencies. Results: Genotype frequencies were Val/Val=85,0% and Val/Met=15,0%. The Val allele frequency was higher than 93%, the Met allele was associated with a lower response to metformin and was present in Amantaní (8.3%) and in Lima (9.6%), and absent in Taquile. Conclusions: We found the highest Val allele frequency in the world. Regarding the Met allele, less frequent, we found differences among the Peruvian subpopulations tested, and this knowledge can help in the pharmacogenetics and decision making about oral treatment of metformin against diabetes.
Keywords: Pharmacogenetics; OCT1 Gene; Val/Met Polymorphism; Type 2 Diabetes mellitus; Metformin; Lima, Puno, Peru.
INTRODUCCIÓN
La farmacogenética, cuyo origen se remonta a los inicios del siglo veinte con el surgimiento de la bioquímica genética, en los últimos años ha tomado gran relevancia debido a la variabilidad de la respuesta individual frente a un fármaco, que puede incluir una respuesta nula o producir toxicidad (1). Esta diferencia individual de asimilar, metabolizar, transportar y eliminar los principios activos de los fármacos es determinada por la genética de las proteínas (enzimas, transportadores, factores reguladores, otros). La respuesta individual depende del perfil genético de la persona y es influenciada por su origen étnico. Por tanto, son factores importantes para la toma de decisiones en la clínica y tratamiento de las enfermedades (2).
Caracterizar las variantes farmacogenéticas del paciente, incluso antes de la administración del fármaco, permitirán recomendar la dosis adecuada para optimizar el tratamiento. Los aspectos farmacocinéticos y/o farmacodinámicos son altamente variables en la respuesta y la sensibilidad a las drogas entre los individuos. Los polimorfismos en diversos genes pueden explicar la variabilidad en la respuesta farmacológica. Por lo tanto, si se conoce la farmacogenética, las evaluaciones de riesgo serán más precisas y mejora la toma de decisiones (1,2).
La diabetes mellitus tipo 2 (DMT2) es una enfermedad multifactorial, es decir, intervienen factores ambientales y genéticos predisponentes (3). La estadística mundial de la diabetes es alarmante. Según la Organización Mundial de la Salud (OMS), en el año 2013 a nivel mundial hubo más de 347 millones de personas con diabetes, en la cual el tipo 2 representó el 90% de los casos mundiales y se debe en gran medida a un peso corporal excesivo y a la inactividad física, además de factores genéticos (4).
En el Perú, la DMT2 es una enfermedad que afecta a casi 2 millones de personas y es la decimoquinta causa de mortalidad en el Perú, según informes de la Oficina de Estadística e Informática del Ministerio de Salud. Según proyecciones de la OMS, la diabetes será la sétima causa de mortalidad en el año 2030 (5-7). La morbilidad de la DMT2 además se asocia a la ceguera, insuficiencia renal y cardiaca, amputaciones y otros problemas discapacitantes (8).
Según la OMS, los factores condicionantes para la adherencia al tratamiento de la DMT2 son los relacionados directamente con el paciente, el medio ambiente (estilo de vida), los medicamentos, relación del personal de salud-paciente, la edad y el género, siendo el factor efecto del medicamento importante en la poca adherencia al tratamiento antidiabético (9).
Los fármacos, según su mecanismo de acción, actúan sobre los diversos factores causantes de la hiperglicemia. En el tratamiento, puede incluirse una combinación fija de dos o más agentes terapéuticos con mecanismos de acción complementaria. Existen varios grupos de fármacos antidiabéticos orales, entre los que destacan, las sulfonilureas como la glibenclamida, y las biguanidas como la metformina (10).
La metformina es un antihiperglicemiante utilizado para el tratamiento de la DMT2 desde 1959 y sigue siendo importante en la terapéutica de la enfermedad. Es el fármaco de primera línea para la DMT2 por su eficacia, seguridad, múltiples beneficios cardiovasculares, metabólicos y además puede utilizarse con cualquier otro fármaco antidiabético, incluyendo la insulina. La metformina mejora la sensibilidad y disminuye la resistencia a la insulina presente en esta patología. Asimismo, tiene efectos beneficiosos como la disminución de los niveles de lípidos, prevención de algunas complicaciones vasculares y mínimo riesgo de producir hipoglucemia (11).
La acción principal de la metformina es disminuir la producción de glucosa hepática inhibiendo la gluconeogénesis sin producir hipoglucemia. A nivel periférico, en los tejidos musculares y grasos, facilita el transporte de glucosa mediante el aumento de la actividad tirosina quinasa en los receptores de insulina y optimizando la función de los transportadores. Además, no se metaboliza en el cuerpo, siendo eficientemente excretado en la orina (11, 12).
Los transportadores de cationes orgánicos (OCTs), proteínas de la familia de genes SLC22 (solute carrier family 22), movilizan moléculas polares a través de la membrana celular en distintos órganos, y han sido implicados en la distribución de la metformina. Los OCTs son los transportadores poliespecíficos más importantes en el hígado y el riñón, trabajan en conjunto con las enzimas metabolizadoras de xenobióticos en vías de desintoxicación hepática, eliminando cationes orgánicos de la circulación sistémica. Utilizando ratones knockout, se ha demostrado que el OCT1 tiene un papel importante en la absorción de la metformina en el hígado, y por tanto tiene un impacto en su efecto terapéutico (12,13).
La metformina, en los humanos, es movilizada en los hepatocitos y en el epitelio renal por el transportador OCT1, el cual es codificado por el gen SLC22A1 (llamado también OCT1, OMIM#602607) localizado en la región cromosómica 6p25.3 (14). El transportador OCT1 también moviliza compuestos endógenos como la colina, histamina, epinefrina, adrenalina, noradrenalina y dopamina. Entre los xenobióticos, aparte de la metformina, también se conoce que el OCT1 transporta quinina y su actividad es afectada por varios compuestos, incluyendo cocaína, cimetidina, antagonistas de receptores NMDA, prazosina y otros (13,14).
En la actualidad, se consideran importantes los factores genéticos y epigenéticos en la farmacocinética y la farmacodinámica, destacándose la alta variación interindividual en la respuesta y la sensibilidad a las drogas. En ese sentido, si se conoce el perfil farmacogenético, las evaluaciones de riesgo serán más precisas y de esa manera mejorará la toma de decisiones. En particular, estudios previos en humanos han demostrado que la variación genética en el OCT1 y también factores epigenéticos, puede contribuir en la respuesta al fármaco metformina, pudiendo alterar su efecto terapéutico (15-17).
Los polimorfismos en el gen OCT1 podrían afectar los parámetros farmacocinéticos y/o farmacodinámicos de los medicamentos y, por tanto, aumentar el riesgo de las reacciones adversas, considerando que puede excederse la concentración mínima tóxica o los niveles del medicamento pueden disminuir por debajo de la concentración adecuada(18,19).
A pesar de que el gen OCT1 puede tener un papel importante en la respuesta a los antidiabéticos orales como la metformina y existir variabilidad en las diferentes regiones del mundo, los estudios genéticos en el país son escasos y se desconocen las frecuencias en la población peruana, caracterizada por su mestizaje y diversidad poblacional. El objetivo de la investigación fue evaluar la distribución del polimorfismo Val/Met en el gen OCT1 en muestras de Lima y Puno, e inferir su impacto en la farmacogenética de la DMT2.
MÉTODOS
Se trabajó con muestras que forman parte de bancos de ADN de la facultad de Medicina de la UNMSM y de la facultad de Medicina de la USMP, como parte de investigaciones en poblaciones nativas y mestizas del Perú. Los ADNs fueron obtenidos por metodología convencional a partir de muestras de sangre o de frotis bucal (20,25). Se evaluó 56 muestras correspondientes a voluntarios sanos de poblaciones nativas de la región Puno (Uros, Taquile y Amantaní) y 57 de Lima (Lima ciudad).
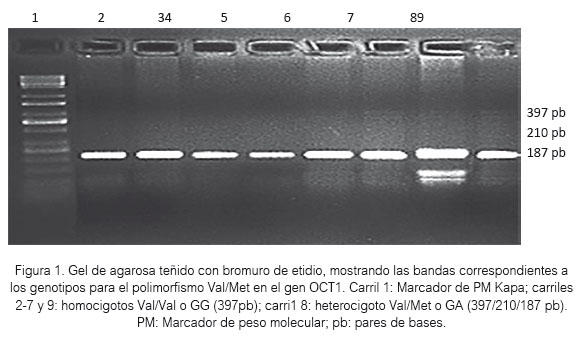
En este estudio, se analizó la variante Val/Met (G/A, rs 628031) ubicado en el exón 7 del gen OCT1 mediante la técnica PCR-RFLP (14). Las amplificaciones de ADN se llevaron a cabo en un termociclador Veriti (Applied biosystems). Se mezcló 11,5 uL de mezcla de reacción (7,8uL de H2O PCR; 1,25uL de Buffer 10X; 0,25uL dNTPs; 0,75uL MgCl2; 0,625uL primer directo: 5’-TTTCTTCAGTCTCTGACTCATGCC-3’; 0,625uL primer reverso: 5’-AAAAAACTTTGTAGACAAAGGTAGCACC-3’, 1 U Taq polimerasa) con 1 uL de ADN (10 ng/ uL). Se las incubó inicialmente a 94°C por 5 minutos y después fueron sometidas a 32 ciclos de amplificación. Cada ciclo consistió de una etapa de desnaturalización a 94°C por 30 segundos, una etapa de alineamiento de 58°C por 1,5 minutos y una etapa de extensión a 72°C por 1 minuto; la etapa de extensión final fue a 72°C por 5 minutos. Los productos de PCR fueron sometidos a electroforesis en gel de agarosa al 1%, teñidos con bromuro de etidio y observados bajo luz ultravioleta para evaluar el tamaño esperado de 397 pares de bases (pb).
Para identificar los genotipos, se preparó 5 uL de la solución de restricción (3,0 uL de H2O PCR; 1,5uL de Buffer R y 0,5uL de enzima MscI), se mezcló con 5,0 uL del amplificado y se incubó a 37 ºC por 16 horas. Se realizó una electroforesis en gel de agarosa al 2%, tinción con bromuro de etidio y observación bajo luz UV. La enzima de restricción MscI permite identificar el alelo Met (alelo A), es decir el cambio 1222A>G en el gen OCT1, generando el corte del amplificado de 397 pb en 2 segmentos de 210 y 187pb, mientras que la presencia del alelo Val (alelo G) impide el corte y se mantiene el fragmento de 397 pb (figura 1).
Las frecuencias genotípicas y alélicas se determinaron mediante conteo directo. Se ha evaluado las frecuencias genotípicas observadas, según lo esperado bajo la hipótesis del equilibrio de Hardy-Weinberg. Para comparar y establecer diferencias de las frecuencias genotípicas y/o alélicas del polimorfismo Val/Met en el gen OCT1 entre los grupos peruanos y con otras poblaciones del mundo, se utilizaron las pruebas X2 o la prueba exacta de Fisher según el caso (α=0,05). Para los cálculos se utilizó el paquete estadístico SPSS v21,0 y programas de genética poblacional.
RESULTADOS
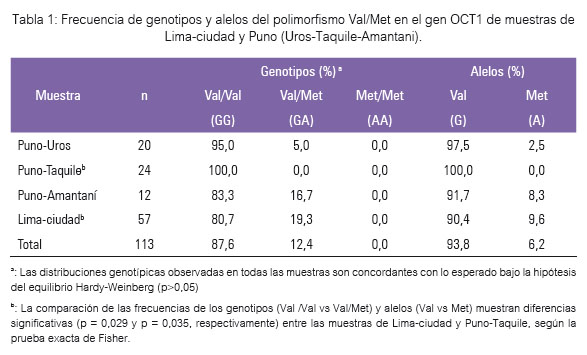
La distribución de las frecuencias genotípicas y alélicas del gen OCT1 en las muestras de los 4 grupos subpoblacionales peruanos, se muestra en la tabla 1. Las frecuencias genotípicas observadas en 3 grupos siguen una distribución consistente con el equilibrio de Hardy-Weinberg (p>0,05).
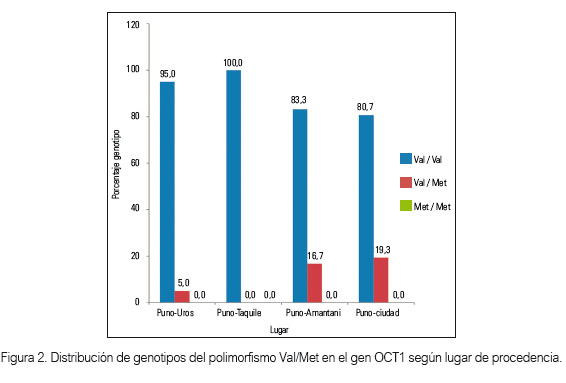
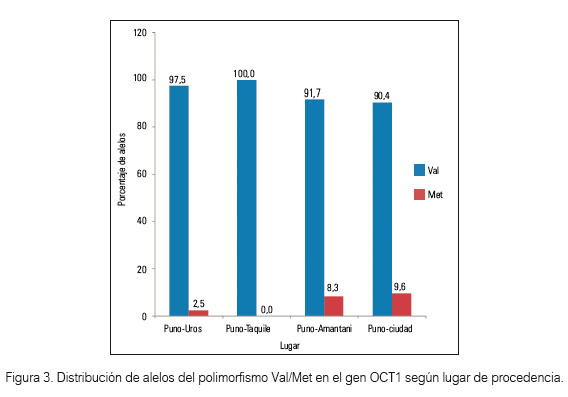
El alelo Val fue el más común (93,8%) en el conjunto de muestras evaluadas (n=113), siendo muy frecuente en Puno-Taquile (100,0%), mientras que el alelo Met, en general, tuvo una frecuencia menor (6,2%). En el grupo de Lima-ciudad predominó el alelo Val (90,4%). Los genotipos que se encontraron fueron Val/Val y Val/Met, siendo el más frecuente el genotipo Val/Val (87,6%). No se encontraron genotipos Met/Met en los 4 grupos evaluados.
La comparación de las frecuencias genotípicas y alélicas mostró diferencias significativas (p<0,05) solo cuando se comparó la muestra de Lima-ciudad (la más diversa, considerada como mestizos) con la de Puno-Taquile, y no fue significativa (p>0,05) cuando se comparó con los demás grupos (tabla 1 y figuras 2 y 3).
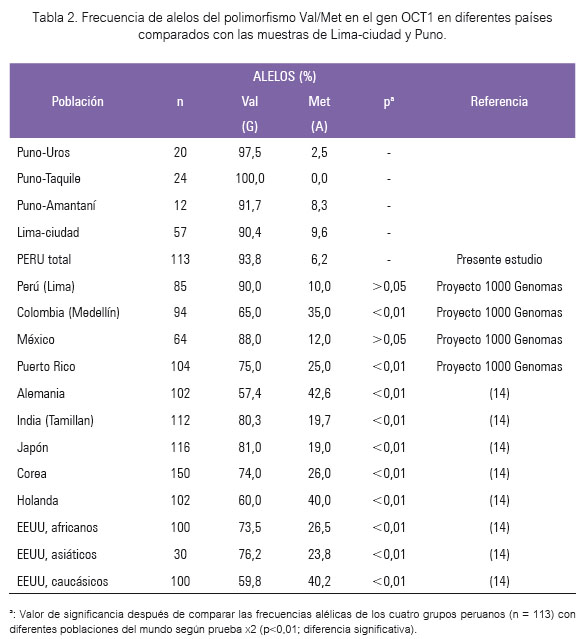
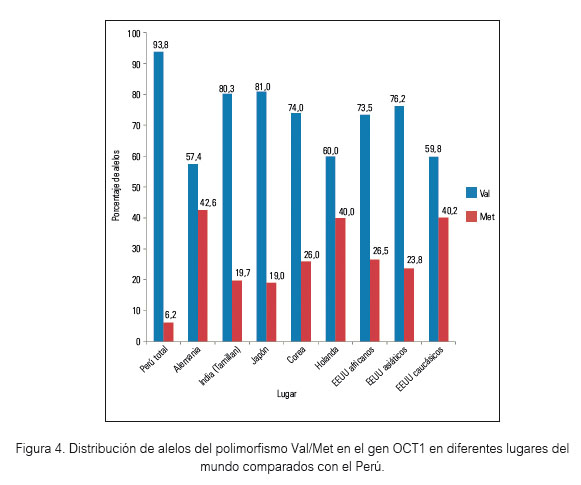
En la tabla 2 se comparan las frecuencias alélicas del gen OCT1 en la población peruana (4 grupos, n= 113) con las encontradas para otras poblaciones del mundo. Existen diferencias significativas (p<0,01) con las poblaciones de Alemania, Holanda, Japón, Corea, India, EEUU (africanos, asiáticos y caucásicos) y, respecto a América Latina, la distribución fue similar a México y significativamente diferente a la de Colombia y Puerto Rico (tabla 2 y figura 4).
DISCUSIÓN
La metformina se difunde a través de la membrana plasmática de los hepatocitos; en el interior se une a la mitocondria, inhibiendo la expresión de genes de la gluconeogénesis mediante la proteína quinasa activada por AMP (AMPK). En paralelo, la activación de LKB1 dependiente de la AMPK activada por depleción de ATP, ejerce un efecto indirecto sobre la sensibilidad a la insulina para controlar la salida de glucosa hepática (18). Existen explicaciones alternativas para la inhibición de la gluconeogénesis por la metformina. Una de ellas es la inhibición leve del complejo I de la cadena respiratoria mitocondrial incrementando el AMP cada vez que cae el ATP, y la otra es otorgar a la metformina una función de regulación alostérica de enzimas, tal como la fructosa-1,6-bisfosfatasa. La metformina es un sensibilizador de la insulina, lo cual se atribuye a sus efectos positivos sobre la expresión del receptor de la insulina y la actividad tirosina quinasa, contribuyendo a la disminución de la resistencia a la insulina que se produce durante la DMT2 (21).
Las investigaciones realizadas sobre la efectividad de la metformina ligada a variaciones genéticas del transportador OCT1 en relación a la presencia de los genotipos Val/Val (GG) o Val/Met (AG) en el exón 7 del gen del transportador OCT1, nos demuestran que los pacientes con genotipo Val/Val tienen el efecto esperado sobre la regulación de glucosa, en comparación con los que presentan el genotipo Val/Met (22,23).
Los resultados de la presente investigación para el gen OCT1, muestran en la totalidad de las muestras (n = 113), que el alelo Val tuvo una prevalencia de 93,8%. Estos datos son similares a los obtenidos por el proyecto 1 000 genomas en una muestra obtenida en la ciudad de Lima (24).
En detalle, al evaluar las subpoblaciones de Lima-ciudad y de Puno por separado (Uros, Taquile y Amantani) se presentan diferencias significativas (p<0,05) entre las frecuencias de genotipos y alelos de Puno-Taquile y Lima-ciudad. Por ello es importante y necesario estudiar subpoblaciones, porque estas diferencias particulares todavía no se reflejan en base de datos internacionales como la del proyecto 1 000 genomas (24). Nuestros estudios previos de ancestralidad en diferentes subpoblaciones demuestran que los peruanos, globalmente, tienen un mayor componente nativo americano (aproximadamente el 70,0%). Por separado se destaca que las muestras de Lima-ciudad tienen mayor grado de mestizaje (componentes caucásico, africano o asiático) respecto a muestras de Puno (componente nativo mayor a 90,0%) (25), lo cual parcialmente se refleja en nuestros resultados al comparar Puno-Taquile y Lima-ciudad.
En la India, en estudios sobre enzimas metabolizadoras y transportadores de fármacos, se muestra la distribución del polimorfismo Val/Met con una frecuencia de 80,3% del alelo Val, mientras que en países europeos como Alemania y Holanda las frecuencias son del 57,4% y 60,0% respectivamente (23).
En general, las poblaciones europeas(23) presentan menor distribución del alelo Val en comparación con las subpoblaciones de Perú, y también en países de América Latina(24), como Colombia y Puerto Rico, se registra esta tendencia, posiblemente por su mayor componente caucásico, con excepción de México cuya distribución alélica es similar a la nuestra. En otros continentes, los estudios poblacionales de farmacogenes son considerados de gran importancia, no solo por su aplicación clínica, sino también para desarrollar la farmacogenómica preventiva y una adecuada evaluación del costo-beneficio farmacológico (26).
Los resultados podrían suponer que la respuesta al tratamiento con metformina para pobladores de Puno-Taquile y Lima-ciudad puede ser diferente, pero debe implicar una evaluación; de esta manera se puede coadyuvar en una estrategia nacional. Para ello sería necesario evaluar la respuesta al tratamiento con metformina en pacientes diabéticos.
Los resultados de la investigación, si bien se ven limitadas por el tamaño de muestra y por el diseño no clínico, coadyuvan al conocimiento para la elección adecuada de un fármaco antidiabético adecuado para los pacientes de nuestro país. Ello significa disminuir los efectos adversos y mejorar la eficacia, con el consiguiente impacto en el tratamiento de la DMT2.
Esta investigación permite conocer la prevalencia de las variantes en el polimorfismo Val/Met en el gen OTC1 que puede ser importante desde una perspectiva genética evolutiva poblacional y también para diseñar estrategias de tratamiento con metformina en los grupos estudiados.
Los resultados muestran una frecuencia mayoritaria del alelo Val (mayor del 93%), la más alta comparada con otras poblaciones del mundo, y cuyas implicancias evolutivas deben ser evaluadas. Para el alelo Met, aun con una baja frecuencia, se notan diferencias entre las subpoblaciones peruanas evaluadas y esto puede tener un impacto farmacogenético.
En conclusión, existen diferencias en las frecuencias genotípicas y alélicas del polimorfismo Val/Met en el gen OCT1 entre las muestras de Puno y Lima. Para el alelo Val se ha encontrado la más alta frecuencia registrada en el mundo. Respecto al alelo Met, aunque es menos frecuente, existen diferencias entre las subpoblaciones peruanas evaluadas, y ese conocimiento puede ayudar en la farmacogenética y la toma de decisiones en el tratamiento con los antidiabéticos orales como la metformina.
AGRADECIMIENTOS
Al Consejo Superior de Investigaciones de la UNMSM por el financiamiento del proyecto CON-CON 2015 de la facultad de Farmacia y Bioquímica (código 150403101), Centro de Investigación de Bioquímica y Nutrición, facultad de Medicina, UNMSM, y al Centro de Genética y Biología Molecular de la facultad de Medicina Humana de la USMP por la financiación y apoyo de la investigación (proyecto E100120015022).
REFERENCIAS BIBLIOGRÁFICAS
1. Ramos E, Doumatey A, Elkahloun A, Shriner D,Huang H, Chen G, et al. Pharmacogenomics, ancestry and clinical decision making for global populations. Pharmacogenomics J. 2014;14:21722. doi: 10.1038/tpj.2013.24. [ Links ]
2. Brunham L, Chan S, Li R, Aminkeng F, Liu X, Saw W, et al. Pharmacogenomic diversity in Singaporean populations and Europeans. Pharmacogenomics J. 2014;14:555-63. doi: 10.1038/tpj.2014.22. [ Links ]
3. Ramírez S, Cabrera C, Huacuja L, Flores L, Pérez G, González J, López A. Implicaciones en la atención primaria en salud de la genética y genómica en la Diabetes mellitus tipo 2. Med Inst Mex SeguroSoc. 2013;51(3):e6-26. [ Links ]
4. Organización Mundial de la Salud. Nota descriptivaN°312 [Internet]. 2013. Disponible en: http://www.who.int/mediacentre/factsheets/fs312/es/ [ Links ]
5. Ministerio de Salud, Perú. Lima [Internet]. 2010. Disponible en: http://www.minsa.gob.pe/portada/ Especiales/2010/diabetes/datos.asp [ Links ]
6. Alwan A. Global status report on noncommunicable diseases. Geneve: World Health Organization [Internet]. 2011. Disponible en: http://www.who.int/nmh/publications/ncd_report_full_en.pdf [ Links ]
7. Ticse R, Peinado A, Baiocchi-Castro L. Características demográficas y epidemiológicas de pacientescon diabetes mellitus tipo 2 hospitalizados porcetoacidosis diabética en un hospital general de Lima-Perú. Rev Med Hered. 2014;25:5-12. doi:10.20453/rmh.v25il.259. [ Links ]
8. Dirección General de Epidemiología. Manual de Procedimientos Estandarizados para la Vigilancia Epidemiológica de la Diabetes Mellitus Tipo 2.México. [Internet]. 2012. Disponible en: http:// www.epidemiologia.salud.gob.mx/doctos/infoepid/vig_epid_manuales/10_2012_Manual_DM2_ vFinal_31OCT12.pdf. [ Links ]
9. Pérez T. Factores determinantes en la adherencia terapéutica en pacientes adultos con diabetes mellitus tipo II. Perú-Trujillo: Oficina de Sistemas e Informática-UNT, 2013. [ Links ]
10. Martínez K, Díaz J. Combinación en dosis fijas: una nueva opción terapéutica para la diabetes mellitustipo 2. Med Int Mex. 2012;28(5):483-90. [ Links ]
11. Becker M, Pearson E, Tkac I. Pharmacogenetics of oral antidiabetic drugs. Int J Endocrinol.2013;2013:686315. doi: 10.1155/2013/686315. [ Links ]
12. Castro M, Castillo V, Ochoa A, Godínez S. La metformina y sus aplicaciones actuales en la clínica. Med Int Méx. 2014;30:562-74. [ Links ]
13. Takane H, Shikata E, Otsubo K, Higuchi S, Ieiri I. Polymorphism in human organic cation transporters and metformin action. Pharmacogenomics.2008;9(4):415-22. doi: 10.2217/14622416.9.4.415. [ Links ]
14. Umamaheswaran G, Praveen R, Arunkumar A,Das A, Shewade D, Adithan C. Genetic analysis Luis Olivares y col. of OCT1 gene polymorphisms in an Indian population. Indian J Hum Genet. 2011;17(3):164-68. doi: 10.4103/0971-6866.92094. [ Links ]
15. Landan G, Cohen N, Mukamel Z, Bar A, Molchadsky A, Brosh R, et al. Epigenetic polymorphism and the stochastic formation of differentially methylated regions in normal and cancerous tissues. Nat Genet. 2012;44(11):1207-14. doi: 10.1038/ng.2442. [ Links ]
16. Perwitasari D, Attobari J. Polymorphism of organic cation transporter 1 (OCT1) in Indonesian cancer patients. Int J Pharm Pharm Sci. 2014;6(5):380-2. [ Links ]
17. Basu N, Goodrich J, Head J. Ecogenetics of mercury: from genetic polymorphisms and epigeneticsto risk assessment and decision-making. EnvironmToxicol Chem. 2014;33(6):1248-58. doi: 10.1002/ etc.2375. [ Links ]
18. Jonker J, Schinkel A. Pharmacological and physiological functions of the polyspecific organic cation transporters: OCT1, 2, and 3 (SLC22A1-3). J Pharmacol Exp Ther. 2004;308(1):2-9. doi: 10.1124/jpet.103.053298. [ Links ]
19. Koepsell H. Lips K, Volk C. Polyspecific organic cation transporters: structure, Function, physiological roles and biopharmaceutical implications. Pharm Res. 2007;24(7):1227-51. doi: 10.1007/ s11095-007-9254-z. [ Links ]
20. SandoVal J, Delgado B, Rivas L, Bonilla B, Nugent D, Fujita R. Variantes del ADNmt en isleños dellago Titicaca: máxima frecuencia del haplotipo B1 y evidencia de efecto fundador. Rev Peru Biol. 2004;24:161-8. doi: 10.15381/rpb.v11i2.2452. [ Links ]
21. Chen L, Shu Y, Liang X, Chen E, Yee S, Zur A, et al. OCT1 is a high-capacity thiamine transporter that regulates hepatic steatosis and is a target of metformin. Proc Natl Acad Sci USA. 2014;11(27):9983-88.doi: 10.1073/pnas.1314939111. [ Links ]
22. Viollet B, Guigas B, Sanz Garcia N, Leclerc J, Foretz M, Andreelli F. Celular and molecular mechanisms of metformin: an overview. Clin Sci (Lond.) 2012;122(6):253-70. doi: 10.1042/CS20110386. [ Links ]
23. Umamaheswaran G, Kumar D, Adithan C. Distribution of genetic polymorphisms of genes encoding drug metabolizing enzymes and drug transporters. A review with Indian perspective. Indian J Med Res.2014;139(1):27-65. [ Links ]
24. 1000 genomes Project. [Internet]. Disponible en: http://www.internationalgenome.org/data [ Links ]
25. Sandoval J, Salazar A, Acosta O, Castillo HW, Fujita R, Pena S et al. Tracing the genomic ancestry of Peruvians reveals a major legacy of pre-Columbianancestors. J Hum Genet. 2013;58(9):627-34. doi: 10.1038/jhg.2013.73. [ Links ]
26. Mizzi C, Dalabira E, Kumuthini J, Dzimiri N,Balogh I, Basak N, et al. A European spectrumof pharmacogenomics biomarkers: Implications for clinical pharmacogenomics. PLoSOne.2016;11(9):e0162866. doi: 10.1371/journal. pone.0162866. [ Links ]
Conflictos de interés entre las instituciones participantes: El estudio fue financiado por el proyecto CON-CON 2015 (código 150403101), CSI, UNMSM y fondos de la facultad de Medicina, USMP (proyecto E10012015022).
Correspondencia:
Oscar Acosta-Conchucos oacostac@unmsm.edu.pe, oacostac@yahoo.com Departamento de Bioquímica. Facultad de Farmacia y Bioquímica, UNMSM. Jr. Puno Nº 100, Lima 1, Perú.
Recibido: 25 enero 2017
Aprobado: 13 marzo 2017