INTRODUCCIÓN
El género Edwardsiella pertenece a la familia Enterobacteriaceae y se caracteriza por ser un bacilo corto, Gram negativo (Ewing et al., 1965). Antes de 2013, este género se encontraba conformado únicamente por tres especies: E. tarda, E. ictaluri y E. hoshinae, las que constituían un grupo heterogéneo de bacterias que afectaban un amplio rango de hospedadores, tales como peces, reptiles, aves y mamíferos (Reichley et al., 2017). Actualmente, el género Edwardsiella ha sido reclasificado en cinco especies, basada en información genómica y filogenética. Dichas especies involucran tres patógenos de peces (E. piscicida, E. anguillarum y E. ictaluri) y dos patógenos que le son inofensivos (E. tarda y E. hoshinae) (Leung et al., 2019).
E. anguillarum es una bacteria facultativa intracelular, la cual ha recibido distintos nombres como E. piscicida-like sp, E. piscicida atípica y E. tarda, siendo originalmente aislada de anguila (Anguilla spp) en China (Shao et al., 2015) y reportada en otros peces como mero blanco (Epinephelus aeneus), ronco (Haemulon sciurus), lubina estriada (Morone saxatilis), pargo japonés (Pagrus major), besugo (Pagellus bogaraveo), dorada (Sparus aurata) y tilapia (Oreochromis sp) (Shao et al., 2015; Buján et al., 2018; Armwood et al., 2019).
La tilapia (Oreochromis spp) es uno de los peces más cultivados a nivel mundial, siendo introducido en el Perú en 1970 (Baltazar, 2009; FAO, 2018). Actualmente, el incremento de la demanda por alimentos de origen acuático trae consigo la intensificación de las densidades en cultivos de esta especie, surgiendo la tecnología biofloc, la cual está constituida por bacterias y otros organismos (fitoplancton, rotíferos, ciliados, flagelados y copépodos) que mejoran la calidad del agua, proveen alimentación suplementaria a los organismos en cultivo y los protegen de enfermedades (McIntosh et al., 2000; Ray et al., 2010).
La rápida expansión e intensificación del cultivo de tilapia ha traído consigo la ocurrencia de enfermedades provocadas por los patógenos bacterianos: E. tarda, Plesiomonas shigelloides y Francisella noatunensis (Soto et al., 2010; Mateo et al., 2011; Sierralta et al., 2016), así como la presencia del virus emergente de la tilapia lacustre (TiLV) (Eyngor et al., 2014). Son escasos los estudios sobre patologías bacterianas en el Perú, por lo cual, en el presente trabajo se planteó como objetivo aislar y caracterizar fenotípica y molecularmente al patógeno E. angui-llarum y analizar las lesiones histopatológicas provocadas en tilapias con signos de enfermedad en una piscigranja de la zona norte de Lima.
MATERIALES Y MÉTODOS
Lugar de Muestreo
El estudio se llevó a cabo en una piscigranja de sistema intensivo con cero recambio de agua y tecnología biofloc, localizada en la provincia de Huaura, departamento de Lima, Perú, durante los meses de marzo de 2014 a octubre de 2015.
Muestras y Aislamiento Bacteriano
Se colectaron 85 tilapias O. niloticus, variedad chitralada, de los estadios de alevinaje, crecimiento y comercial, con signos aparentes de enfermedad, tales como: exoftalmia, melanosis, erosión de las aletas y dificultad para la natación. El número de muestras fue obtenido empleando la metodología descrita por Amos (1985) y de Blas et al. (2006).
Los peces fueron sacrificados mediante la técnica del corte medular (American Fisheries Society, 2011) y se procedió a desinfectar la superficie de la piel con alcohol etílico 70%. Se tomaron diversas muestras para análisis histopatológico, además de un «pool de órganos» (bazo y riñón anterior), que fue homogenizado en solución salina estéril (0.85% Na Cl). El homogenizado fue cultivado en placas con agar tripticasa de soya (TSA) y agar diferencial eosina azul de metileno (EMB) para enterobacterias, las cuales fueron incubadas a 35 ºC durante 2448 horas (Heil, 2009).
Las colonias bacterianas fueron caracterizadas mediante pruebas de tipificación primaria (coloración Gram, citocromo oxidasa y motilidad en gota pendiente). Las colonias presuntivas fueron pasadas a ceparios con agar TSA y mantenidas en estado de pureza en los laboratorios de Ecología Microbiana y Virología Clínica Molecular de la Universidad Nacional Mayor de San Marcos, Lima.
Análisis Histopatológico
De los peces analizados se obtuvieron muestras de branquias y órganos internos como corazón, estómago, hígado, bazo, intestino, ciegos pilóricos, páncreas, riñón anterior, posterior, gónada y músculo esquelético, las cuales fueron fijadas en solución de formaldehído al 10% (v/v) en buffer fosfato (pH 7.0) y procesadas utilizando la metodología propuesta por Luna (1968). Los tejidos fueron cortados a 5 μm de espesor, teñidos con Hematoxilina y Eosina de Harris (HE) y visualizados en un microscopio compuesto (Nikon, modelo Eclipse 90i).
Identificación de E. anguillarum
Se realizaron pruebas para la caracterización fenotípica y bioquímica de E. anguillarum, tales como citocromo oxidasa, fermentación y oxidación de glucosa, Voges Prokauer, citrato, rojo de metilo, indol, producción de sulfuro de hidrógeno, entre otras (Shao et al., 2015). Además, se realizaron pruebas bioquímicas mediante el uso del sistema API 20E para enterobacterias (Biomériux, Francia), siguiendo las indicaciones del fabricante, para posteriormente incubarlas a 35 °C durante 24-48 h.
Identificación Molecular
Las cepas aisladas e identificadas bioquímicamente fueron analizadas molecularmente en la empresa Biodes Laboratorios Soluciones Integrales S.R.L. mediante amplificación del gen 16S ARNr. La extracción de ADN fue realizada por el método estándar CTAB-DTAB (Gustincich et al. 1991), adaptado para células bacte-rianas (Dulanto, 2013). Posteriormente, la región del gen 16S ARNr fue amplificada con los iniciadores universales para bacterias, 8F (5´ AGA GTT TGA TCC TGG CTC AG 3´) y 1510R (5´ GGC TAC CTT GTT ACG A 3´) descritos por Weisburg (1991) y Monsalud et al. (2003) en estudios filogenéticos bacterianos, para la obtención de productos de aproximadamente 1500 pares de bases (pb).
El análisis de PCR fue realizado en 20 μl del volumen final de reacción en microtubos de 0.2 ml. Se preparó una mezcla usando 1.5 mM de MgCl , 2 μl de buffer Taq 10X, 0.2 mM de cada dNTPs (100 mM), 10 pmol de cada cebador, 2 μl de ADN bacteriano extraído y 1 U de Taq ADN polimerasa. El proceso de amplificación se realizó en un termociclador (Blue-Ray Biotech), iniciando con 94 °C durante 5 min, seguida de 30 ciclos de desnaturalización (94 °C durante 1 min), hibridación (55 °C durante 1 min) y elongación (72 °C durante 2 min), concluyendo con una extensión final de 5 min a 72 °C. Los productos de amplificación de la PCR fueron analizados en gel de agarosa al 1% con buffer de migración TAE 1X, coloreados en solución de bromuro de etidio y visualizados empleando un transiluminador UV. Para la secuenciación de ambas cadenas de los productos de amplificación, se remitió 10 ìl de cada muestra a la empresa Macrogen (Corea), incluyendo 5 μl de cada cebador para el gen 16S ARNr.
Análisis Bioinformático
Las secuencias forward y reverse obtenidas de cada producto de la PCR fueron- procesadas mediante el programa SeqMan II v. 5.03 (DNASTAR) para visualizar los cromatogramas y generar las secuencias consenso. Una vez obtenidas dichas secuencias se utilizó la herramienta Nucleotide BLAST (Basic Local Alignment Search Tool) y Ez Biocloud (Yoon et al., 2017) para verificar la identidad de las cepas bacterianas en estudio.
Sensibilidad Antibiótica
Las cepas aisladas fueron sometidas a pruebas de sensibilidad in vitro frente a distintos antibióticos según el método de difusión en agar, utilizando la técnica de KirbyBauer descrita por el National Comitee for Clinical Laboratory Standards (NCCLS, 2015). A partir de colonias bacterianas de 24 h de incubación, se preparó una suspensión bacteriana en suero fisiológico estéril (NaCl 0.85%) ajustada a una concentración equivalente a 0.5 de la escala McFarland (1.5x108 células/ml). Dicha suspensión se sembró por duplicado en placas de agar Müeller Hinton. Posteriormente se aplicaron los sensidiscos con antimicrobianos: florfenicol (30 μg), flumequina (30 μg), gentamicina (10 μg), kanamicina (30 μg), oxitetraciclina (30 μg), sulfatrimetoprim (25 μg), ácido nalidíxico (30 μg) y se incubaron por 24 h a 30 ºC.
RESULTADOS
Signos Clínicos
En 5 de los 85 peces se aisló el patógeno E. anguillarum. Los peces afectados correspondieron a la fase de producción comercial. La mayor frecuencia de signos externos fue la exoftalmia bilateral, eritema en aletas pectorales y alrededor del ano (Cuadro 1). Entre los signos internos se apreciaron nodulaciones blanquecinas en el corazón e hígado, así como congestión en intestino y gónadas.
Hallazgos Histopatológicos
Se apreciaron lesiones asociadas con el aislamiento de E. anguillarum en los peces afectados como necrosis e hiperplasia focal de las lamelas branquiales secundarias; severa pericarditis (Figura 1A), con presencia de células granuloeosinofílicas y reacción inflamatoria a nivel de la aurícula y ventrículo cardiaco. En el hígado se observó degeneración hidrópica y necrosis focal de hepatocitos, así como congestión de capilares sinusoides y reacción inflamatoria perivascular con células granuloeosinofílicas. En el bazo se encontró congestión leve y necrosis focal de tipo licuefactiva en parénquima y cápsula. Igualmente a nivel de tejido pancreático se halló necrosis.
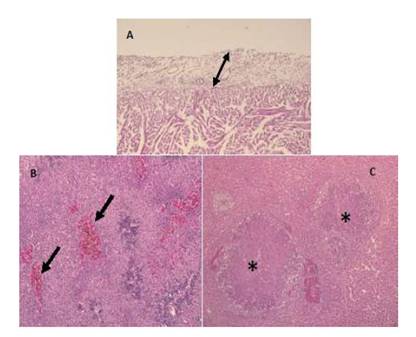
Figura 1 Lesiones histopatológicas producidas por Edwardsiella anguillarum en tilapia (Oreochromis niloticus): A. Marcada pericarditis a nivel del ventrículo cardíaco (flecha) (20X, HE); B. Riñón anterior con congestión focal de vasos sanguíneos (flechas) (20X, HE); C. Tejido hepático con granulomas (asteriscos) (10X, HE)
En el intestino se observó necrosis focal de las vellosidades intestinales y la lámina propia levemente edematosa con presencia de células granuloeosinofílicas y bacterias. En el riñón anterior se halló congestión leve de vasos sanguíneos (Figura 1B) y necrosis focal de tejido hematopoyético, mientras que en el riñón posterior se observó necrosis focal de los túbulos renales.
A nivel de tejido gonadal se apreció severa necrosis y hemorragia. Además, se encontró necrosis de tipo caseosa formando granulomas en el ventrículo cardiaco, tejido hepático (Figura 1C), esplénico, renal y gonadal.
Caracterización de E. anguillarum
Se aislaron cinco cepas bacterianas presuntivas de E. anguillarum a partir de órganos internos. Las colonias presentaron color blanco cremoso, brillosas con bordes regulares y convexas en agar TSA. Asimismo, en agar EMB se visualizaron colonias de color ámbar, brillosas con bordes regulares y convexos. En la observación microscópica, se apreciaron bastones pequeños (2.44-2.57 μm de largo), Gram negativos y mótiles. Las características bioquímicas de las cepas evaluadas mediante métodos convencionales fueron homogéneas, excepto la prueba de inositol (Cuadro 2).
Cuadro 2 Características bioquímicas de E. anguillarum aisladas de cinco tilapias (Oreochromis niloticus) en una piscigranja al norte de Lima, Perú
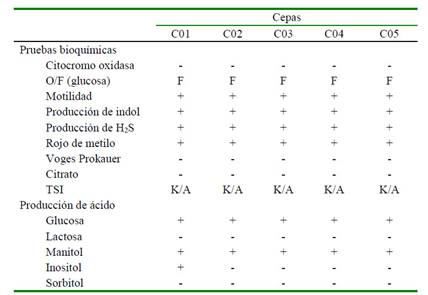
O: oxidativo, F: fermentativo, H2S: sulfuro de hidrógeno, TSI: Agar hierro tres azúcares, K: alcalino, A: ácido
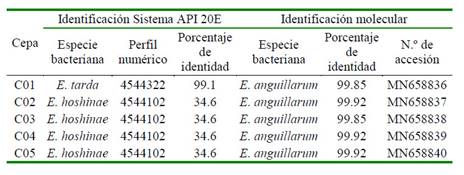
Al emplear el Sistema API 20 E para la identificación de E. anguillarum, se obtuvo un aislado de E. tarda con alto porcentaje de identificación (99.1%) y 4 de E. hoshinae con baja discriminación (34.6%) (Cuadro 3), presentando diferencias bioquímicas en la prueba de inositol.
Identificación Molecular
Utilizando la técnica de PCR con cebadores universales se obtuvieron los productos de amplificación de la región 16S ARNr de las cepas bacterianas de E. anguillarum, las cuales producen amplicones de 1500 pares de bases.
Las secuencias de las bacterias fueron comparadas con la base de datos del Genbank y se obtuvo el consenso de estas con identidades de 99.85 a 99.92% a partir del BLAST, siendo similares con E. anguillarum ET080813 (N.° de accesión en GenBank CP006664; Shao et al., 2015). Las secuencias fueron incorporadas a la base de datos del Genbank con los números de accesión: MN658836, MN658837, MN658838, MN658839 y MN658840 (Cuadro 3).
DISCUSIÓN
La mayoría de las tilapias en las cuales se aisló E. anguillarum mostraron signos clínicos como exoftalmia e internamente se visualizaron nodulaciones blanquecinas en el hígado, así como pericarditis, necrosis intestinal y reacción inflamatoria de tipo granulomatosa en bazo y riñón, guardando semejanza con lo descrito por Armwood et al. (2019). Por otra parte, en el hígado se visualizó degeneración hidrópica y necrosis focal de hepatocitos y en el bazo y riñón se observó depleción focal de esplenocitos y linfocitos, respectivamente, lesiones que no han sido reportadas por otros autores.
Todos los aislados de E. anguillarum produjeron ácido a partir de manitol y arabinosa, lo cual coincide con lo referido por Shao et al. (2015), aunque los resultados referentes a la prueba de inositol difieren de lo reportado por dicho autor. Por otro lado, Reichley et al. (2017) comunicaron la producción de sulfuro de hidrógeno en el medio Hierro Tres Azúcares sin presencia de gas en cepas de este patógeno concordando con los hallazgos del presente estudio.
Al emplear el sistema miniaturizado API 20E se identificó correctamente el taxón Edwardsiella; sin embargo, a nivel de especie se tipificaron cuatro cepas como E. hoshinae con baja discriminación (34.6%) y un aislado como E. tarda (99.1% de confiabilidad), debido a que en la base de datos del sistema no se encuentra la bacteria E. anguillarum. En este sentido, Janda y Abbott (2002) indican que estos sistemas comerciales son relativamente constantes a lo largo del tiempo y a medida que aparecen nuevas especies, las pruebas fenotípicas metabólicas más apropiadas para su discriminación no se encuentran presentes.
La identificación del patógeno mediante el uso de técnicas moleculares concuerda con lo reportado por diversos autores (Reichley et al., 2017; Buján et al., 2018; Armwood et al., 2019) en peces como cherna (Epinephelus aeneus), lubina estriada (Morone saxatilis), ronco catire (Heamulon sciurus), besugo (Pagellus bogaraveo), dorada (Sparus aurata), anguilla moteada gigante (Anguilla marmorata) y tilapia (Oreochromis niloticus).
Cabe destacar que los buenos niveles de sensibilidad alcanzados ante los antimicrobianos utilizados en el antibiograma se deben, probablemente, a que en la piscigranja evaluada no se utilizan antibióticos para tratar enfermedades bacterianas, por lo cual tales patógenos no han adquirido resistencia antibiótica, como ocurre en otras situaciones (Lee et al., 2011; Nadirah et al., 2012).
De acuerdo con lo reportado por Jeney (2017) sobre la adquisición de resistencia bacteriana en peces cultivados con tecnología biofloc, es posible que el sistema de manejo haya favorecido que no se presenten brotes de enfermedad en la piscigranja, pese a que es un sistema intensivo que tiende a generar mayor estrés en los animales y, por ende, a mayor susceptibilidad a las enfermedades.
CONCLUSIONES
Se identificó molecularmente cepas de Edwardsiella anguillarum a partir de bazo y riñón anterior de tilapias (Oreochromis niloticus) con signos clínicos de enfermedad, procedentes de una piscigranja en la zona norte de Lima, Perú.
El daño tisular asociado con E. anguillarum fue la presencia de reacción de tipo granulomatosa en órganos internos.












 uBio
uBio