INTRODUCCIÓN
La leishmaniasis es una enfermedad metaxénica desatendida. En los últimos cinco años, se ha reportado en todo el mundo, aproximadamente, un millón de casos de leishmaniasis cutánea, con un estimado de 220 mil casos por año y más de 399 millo nes de personas en riesgo de infección 1,2. En Perú, la leishmaniasis tegumentaria americana (LTA) es considerada un problema de salud pública por afectar predominantemente a la población más pobre, debido a su amplia distribución geográfica 3 y la diversidad de especies circulantes. Las especies más prevalentes son Leishmania (Viannia) braziliensis y Leishmania (V.) guyanensis, principal mente en las regiones de Madre de Dios, Ucayali y San Martín; Leishmania (V.) peruviana en Lima, Ancash, La Libertad y Cajamarca; mientras que Leishmania (L.) amazonensis ha sido poco reportada, principalmente en las regiones de Junín, Amazonas y Ucayali. Otras especies como Leishmania (V.) lainsoni, Leishmania (V.) shawi, Leishmania (V.) panamensis, Leishmania (V.) colombiensis y la especie híbrida Leishmania braziliensis/peruviana han sido esporádicamente reportadas 4-7.
Entre el 2000 y el 2018, el Ministerio de Salud (MINSA) del Perú reportó 135 233 casos confirmados, con un promedio de 7117 casos anuales, de los cuales aproximadamente el 94% (6685 casos) fueron cutáneos y el 6% (432) fueron de la forma cutáneo-mucosa. Asimismo, en 2018 las regiones de Madre de Dios y Cusco conglomeraron al 25% del total de los casos re portados 3.
La identificación de las especies de Leishmania se realiza mediante el secuenciamiento de múltiples locus (MLST), electro foresis de isoenzimas (MLEE), anticuerpos monoclonales, secuenciamiento de marcadores moleculares específicos 8,9) y por amplificación de múltiples marcadores en la reacción en cadena de la polimerasa (PCR) convencional 10. Por otro lado, el aná lisis de las curvas de disociación o high resolution melting analysis (HRMA) es un método molecular posterior al PCR en tiempo real que permite, mediante el análisis de las curvas de disociación del ADN, la identificación molecular a nivel de especie 11, de poblaciones, así como mutaciones puntuales (SNP) 12.
MENSAJES CLAVE
Motivación para realizar el estudio: Contribuir en la descripción, actualización y distribución geográfica de las especies de Leishmania en el Perú.
Principales hallazgos: La identificación de L. (V.) braziliensis, L. (V.) guyanensis, L. (V.) peruviana, como especies con mayor prevalencia en el país, además de Leishmania (V.) lainsoni y L. (L.) amazonensis, este último identificado en una paciente procedente de Huánuco, área sin antecedente de su circulación.
Implicancias: El análisis de las curvas de disociación por HRMA, permite la identificación rápida y oportuna de la especie de Leishmania in fectante, importante para determinar el pronóstico de la enfermedad, así como para el desarrollo de estudios que permitan conocer las distintas especies de Leishmania que circulan en el Perú.
El Instituto Nacional de Salud (INS) es un organismo público ejecutor del MINSA adonde se refieren pacientes con sos pecha clínica de leishmaniasis, derivados principalmente de hospitales nacionales, Fuerzas Armadas y Sanidad Policial proce dentes de diferentes áreas endémicas del Perú. El INS realiza el diagnóstico confirmatorio de la enfermedad. En casos cutáneos de reciente evolución, el diagnóstico se logra principalmente mediante métodos parasitológicos, como el examen microscópi co directo o frotis, ampliamente usado debido a su bajo costo, y el cultivo in vitro. Mientras que para el diagnóstico inmuno serológico de la forma cutánea crónica y mucosa de la enfermedad se usa la inmunofluo rescencia indirecta (IFI) y la prueba intradermoreacción de Montenegro (IDRM). Por otro lado, a pesar de la eficiencia de los métodos moleculares como el PCR para la detección del parásito, su implementación está restringido a centros especializados con equipamiento, infraestructura y presupuesto adecuado, además de personal especializado que garantice la continuidad del servicio.
El objetivo del presente estudio fue identificar las especies de Leishmania infectante en pacientes derivados al INS en el pe riodo 2006-2011, mediante el análisis de las curvas de disociación del ADN.
EL ESTUDIO
El presente estudio fue de tipo observacional, descriptivo y retrospectivo. La población estuvo conformada por pacientes con sospecha clínica de LTA acudieron al INS entre el 2006 al 2011, derivados de hospitales nacionales por presentar lesiones cutáneas ulcerosas de bordes elevados, nodulares o en placas o a nivel de mucosas, y que fueron confirmados por métodos parasitológicos en el Laboratorio de Referencia Nacional de Leishmaniasis del INS.
La identificación de Leishmania se realizó a partir del ADN genómico extraído de amastigotes obtenidos mediante raspado de la lesión cutánea con lanceta estéril, conservados en alcohol de 70° y de promastigotes a partir del cultivo in vitro. Las mues tras se obtuvieron como parte del desarrollo de la rutina diagnóstica para la confirmación del caso. La extracción del ADN se realizó empleando las recomendaciones del kit comercial PureLink Genomic DNA (Invitrogen, Life-technologies). Las muestras de ADN fueron eluídas en un volumen de 60 µl y conservadas en congelación hasta su posterior uso.
El estudio fue realizado mediante la amplificación de una región del ADN del kinetoplasto (kDNA) en un termociclador Ro torGene Q (Qiagen), en un volumen de reacción de 10 µl; se empleó 2 μl (5 ng/μl) de ADN parasitario, 0,7 μM de los cebadores A1: 5’-CCG CCC CTA TTT TAC ACC AAC CCC-3 y A2: 5’-GGG GAG GGG CGT TCT GCG AA-3’ 13) y 1X de la solución HRM-PCR Master Mix (Kit Tipe-it HRM PCR, Qiagen).
La identificación de las especies de Leishmania se desarrolló mediante el análisis de las curvas de disociación empleando cepas referenciales como muestras control, los códigos OMS se detallan en el Anexo 1. La identificación por HRMA de una muestra problema se logró por comparación del perfil obtenido frente a un control y expresado en porcentaje de similitud o semejanza por el programa Rotor Gene Q Series Software versión 2.3.1.
Asimismo, con la finalidad de confirmar los hallazgos encontrados en la identificación de Leishmania por HRMA, se determinó la concordancia con 14 muestras seleccionadas aleatoriamente por secuenciamiento del gen citocromo B de Leishmania spp, según me todología descrita por Foulet et al. 8 Los cebadores empleados fueron Lei-CytB09: 5’-TTATGGTGTAGGTTTTAGTYTAGGTT-3’ y Lei-CytB12: 5’-TGCTAAAAAACCACTCATAAATATACT-3’. Las secuencias obtenidas fueron analizados y comparados con la base de datos del GenBank (https://www.ncbi.nlm.nih.gov/genbank).
Se determinó la frecuencia de las variables edad, sexo, ocupación, procedencia, tiempo de enfermedad, número de lesiones, tipo de lesiones, localización y superficie total comprometida de la lesión. Además, usando las variables del lugar probable de infección (a nivel regional) y la especie de Leishmania identificada, se elaboró un mapa de distribución con el sistema de infor mación geográfica Quantum GIS versión 3.8.
El estudio contó con la aprobación del Comité de Ética en Investigación del INS. También se solicitó la autorización de uso y análisis de las cepas al Laboratorio de Referencia Nacional de Leishmaniasis.
RESULTADOS
Entre enero del 2006 a diciembre del 2011, 262 pacientes con sospecha clínica de leishmaniasis fueron derivados al INS, sólo 101 pacientes cumplieron los criterios para el diagnóstico parasitológico de la enfermedad, 69 (68,3%) de los cuales fueron confirmados para LTA y 32 (31,7%) fueron negativos en el diagnóstico parasitológico e inmunoserológico, siendo descartados para la enfermedad.
De los pacientes confirmados, mediante HRMA y por comparación con las muestras control, se logró la identificación de Leishmania en 45 pacientes; mientras que, en los 24 restantes no se logró determinar la especie de Leishmania infectante, debi do al valor de similitud encontrado frente a los controles por debajo del valor de corte establecido.
De las 45 muestras de los pacientes confirmados por HRMA, el 40% se identificó como Leishmania (V.) braziliensis, el 28,9% como Leishmania (V.) guyanensis, el 17,8% como Leishmania (V.) peruviana, el 6,7% como Leishmania (L.) amazonensis, y el 6,7% como Leishmania (V.) lainsoni (Figura 1).
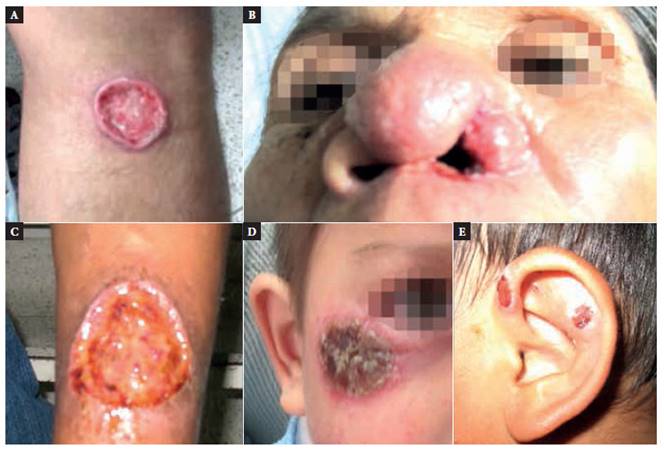
Figura 1 Lesiones causadas por (A) Leishmania (V.) guyanensis, (B) Leishmania (V.) braziliensis, (C) Leishmania (L.) amazonensis, (D) Leishmania (V.) lainsoni, y (E) Leishmania (V.) peruviana
Para la identificación de las especies, se asumió una similitud ≥98% como valor de corte, a excepción de las muestras iden tificadas como Leishmania (V.) lainsoni cuyo valor de corte fue ≥78%. Con este criterio se identificó la especie de Leishmania infectante en un total de 45 pacientes, 42 de los cuales presentaron una similitud ≥98% y los tres restantes una similitud ≥78%.
De los 45 pacientes identificados, 43 (96%) presentaron la forma cutánea, de los cuales 17 (38%) fueron causados por Leishmania (V.) braziliensis procedentes de Amazonas, Cajamarca, Cusco, Huánuco, Loreto, Madre de Dios y San Martín; 12 (27%) por Leishmania (V.) guyanensis procedentes de Amazonas, Cusco, Huánuco, La Libertad, Lima y San Martín 8 (18%) por Lei shmania (V.) peruviana procedentes de Amazonas, Ayacucho, Huánuco, Junín, Lambayeque y Lima; 3 (7%) por Leishmania (V.) lainsoni procedentes de las provincias de La Convención, Satipo y Huaura en las regiones de Cusco, Junín y Lima, res pectivamente; y 3 (7,0%) por Leishmania (L.) amazonensis procedentes de las provincias de La Mar y Huanta en la región de Ayacucho y Churubamba en Huánuco (Anexo 2).
En esta última localidad se logró identificar la especie descrita a partir de una paciente que tenía la forma cutánea diseminada. Por otro lado, el 4% estuvo conformado por dos pacientes procedentes de las provincias de Puerto Inca y Huánuco afectados por la forma cutáneo-mucosa, en quienes se logró identificar Leishmania (V.) braziliensis y Leishmania (V.) guyanensis, respectivamente.
Asimismo, los resultados identificados por HRMA y las 14 muestras secuenciadas aleatoriamente permitieron determinar una concordancia del 100%.
Las características clínico epidemiológicas de los 45 pacientes, en quienes se logró la identificación de la especie de Leish mania infectante se detalla en la Tabla 1.
Tabla 1 Características sociodemográficas y epidemiológicas de casos confirmados de leishmaniasis, 2006-2011 (n=45)

a Comerciante, albañil, ingeniero, profesor, empleado público, obrero; b incluye un caso de Leishmaniasis cutánea diseminada; c pierna, pantorrilla, pie, rodilla, tobillo, muslo, mano, dedo, brazo, antebrazo, codo; d pecho, espalda, cadera; e fosas nasales, labios, orejas, mejillas, frente, mentón, cabeza, cuello
DISCUSIÓN
En la presente investigación, se desarrolló la identificación de Leishmania en 45 pacientes confirmados para LTA, mediante el análisis de las curvas de disociación del kDNA, lográndose la identificación de Leishmania (V.) braziliensis, Leishmania (V.) guyanensis, Leishmania (V.) peruviana, Leishmania (V.) lainsoni y Leishmania (L.) amazonensis, especies reportadas en estudios previos 4,14.
Para la identificación de las especies de Leishmania se estableció un valor de corte relacionado con el grado de divergencia genética, la estrecha relación entre Leishmania (L.) amazonensis y Leishmania (L.) mexicana en el subgénero Leishmania y Leishmania (V.) braziliensis, Leishmania (V.) peruviana, Leishmania (V.) guyanensis y Leishmania (V.) panamensis en el sub género Viannia, ha motivado la elección del ≥98% de similitud como valor de corte, a diferencia del ≥78% de similitud asumido para Leishmania (V.) lainsoni, debido a que esta especie presenta características particulares a nivel de kDNA, que difiere de las demás especies del subgénero Viannia15,16. En nuestro estudio, esta especie fue identificada en muestras de tres pacientes procedentes de Cusco, Junín y Lima, en esta última se logró la confirmación por secuenciamiento del gen citocromo B, mientras que las muestras procedentes de Cusco y Junín fueron confirmadas mediante análisis de fragmentos de restricción de longitud polimórfica (PCR-RFLP) 17. Por el contrario, las muestras de pacientes que no alcanzaron el valor de corte no fueron considera das debido a que la identificación podría no ser correcta.
Asimismo, de las 45 muestras identificadas, el 61% fue aislado de pacientes de procedencia selvática y el 39% de procedencia andina, principalmente de la vertiente occidental de los andes y valles interandinos, lo cual permitió determinar a Leishmania (V.) braziliensis seguido por Leishmania (V.) guyanensis y Leishmania (V.) peruviana como las especies más prevalentes, ampliamente reportadas en nuestro país 7,18. No obstante, la posición ordinal de las mismas puede variar dependiendo de la procedencia de la población, tal como se evidencia en el estudio realizado por Lucas et al. en el cual, a partir de una población selvática se determinó a Leishmania (V.) braziliensis, seguido por Leishmania (V.) peruviana y Leishmania (V.) guyanensis como las más prevalentes.
Del mismo modo, estudios de Arévalo et al. y Kato et al. en poblaciones predominantemente andinas, donde circula principal mente Leishmania (V.) peruviana, determinaron a esta especie como la más prevalente seguido por Leishmania (V.) braziliensis y Leishmania (V.) guyanensis. Otras especies reportadas en la vertiente occidental de los Andes y valles interandinos fueron Leishmania (V.) braziliensis en Ancash y Lima 18, y Leishmania (V.) guyanensis en Lambayeque, Lima (19 y La Libertad 14.
Con respecto a la forma cutáneo-mucosa, a partir de parásitos obtenidos de dos pacientes procedentes de Huánuco, se logró identificar a Leishmania (V.) braziliensis y Leishmania (V.) guyanensis como las especies infectantes, ambas con reconocida capacidad para desarrollar metástasis y evolucionar a formas agresivas de la enfermedad 20,21. Para la forma clínica cutáneo-mucosa se ha repor tado una ocurrencia entre el 5% al 20% del total de casos de leishmaniasis, dependiendo de la región geográfica 21. En Perú, se reporta aproximadamente el 6% de la forma cutáneo-mucosa del total de casos anuales 3. Asimismo, se logró identificar a Leishmania (L.) amazonensis como agente causal de leishmaniasis cutánea diseminada en una paciente proveniente de Huánuco, los casos de leishma niasis con esta forma clínica diseminada, son inusuales y generalmente son comunicados como reportes de casos.
La concordancia de 100% obtenida entre el secuenciamiento por método Sanger y el HRMA, nos permite afianzar la capa cidad de este último, y del kDNA empleado como marcador en la identificación de las especies de Leishmania infectante y, a diferencia del secuenciamiento, con menor consumo de tiempo y recursos económicos.
Las limitaciones del presente estudio están referidas al periodo en que se tomaron las muestras (2006-2011), a la desigual distribución geográfica de los pacientes, predominantemente de la región selva, a la falta de identificación del total de las mues tras confirmadas debido a la baja carga parasitaria lo que no permitió alcanzar el valor de corte establecido para la identi ficación por HRMA, a los altos costos de la identificación por secuenciamiento mediante el método Sanger, por lo que no fue posible confirmar las 31 muestras restantes identificadas.
En conclusión, del análisis de las curvas de disociación del ADN o HRMA de 45 pacientes, se lograron identificar las especies de Leishmania (V.) guyanensis, Leishmania (V.) peruviana, Leishmania (V.) braziliensis, Leishmania (V.) lainsoni y Leishmania (L.) amazonensis, circulantes en 13 regiones de Perú con transmisión autóctona de leishmaniasis. Asimismo, reporta mos por primera vez la circulación de Leishmania (V.) amazonensis en la región Huánuco, identificada a partir de un caso con leishmaniasis cutánea diseminada. La continuación de estudios relacionados a la identificación de especies de Leishmania con un número mayor de muestras, permitirá contar con un mejor conocimiento acerca de la distribución y dispersión de las especies de Leishmania spp, considerando el desplazamiento del vector, las variaciones climatológicas, migraciones, activida des extractivas, entre otros factores.











 texto em
texto em 



