Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.27 no.2 Lima abr./jun. 2016
http://dx.doi.org/10.15381/rivep.v27i2.11661
ARTÍCULOS PRIMARIOS
Variabilidad genética del gato doméstico (Felis catus) en Magangué, Bolívar, Colombia
Genetic variability of the domestic cat (Felis catus) in Magangué, Bolívar, Colombia
Enrique Pardo1,2, Yiris Montes1, Yorlenis Cardales1
1 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Montería, Colombia
2 E-mail: epardop@correo.unicordoba.edu.co
RESUMEN
El objetivo de este trabajo fue determinar la variabilidad genética de las poblaciones de gatos domésticos (Felis catus) utilizando genes que codifican la coloración, el diseño y longitud del pelaje en Magangué, Bolívar, Colombia. Un total de 297 gatos fueron fenotipados mediante observaciones directas realizados en recorridos por las zonas urbanas en Magangué, atendiendo a los marcadores fenotípicos: Orange, Agouti, Tabby, Dilution, Long hair, Spotting white y Dominant white. Se calcularon los parámetros genéticos poblacionales: frecuencia alélica, diversidad genética, flujo génico, equilibrio Hardy-Weinberg y distancia genética y se infirieron las relaciones filogenéticas entre las poblaciones de gatos. Se encontró que el marcador Non-agouti fue el de mayor frecuencia, mientras los genes Tabby blotched y Dominant white presentaron los valores más bajos. La mayor parte de la diversidad genética se encontró dentro de poblaciones y poca entre poblaciones, así como un elevado flujo génico. Se observó un exceso de heterocigotos a nivel poblacional y no hubo equilibrio Hardy-Weinberg. Las poblaciones se encuentran muy relacionadas genéticamente; además se evidenció una posible selección natural y artificial de los marcadores Non-agouti y Spotting white.
Palabras clave: Felis catus, marcadores fenotípicos, diversidad, heterocigosidad, Magangué
ABSTRACT
The aim of this study was to determine the genetic variability of populations of domestic cats (Felis catus) using genes that encode color, design and length of pelage in Magangué, Bolivar, Colombia. A total of 297 cats were phenotyped by direct observations made on tours through urban areas in Magangué, providing the phenotypic markers: Orange, Agouti, Tabby, Dilution, Long hair, Spotting white y Dominant white. Population genetic parameters were calculated: allele frequencies, genetic diversity, gene flow, Hardy-Weinberg equilibrium and genetic distance and phylogenetic relationships among cat populations were inferred. The non-agouti marker was the most frequent, while Tabby blotched and Dominant white gene had the lowest values. Most ofthegeneticdiversitywasfound within populationsand lowamongpopulations as well as high gene flow. An excess of heterozygotes was observed at the population level and there was no Hardy-Weinberg equilibrium. The populations are highly genetically related and was evidenced a possible natural and artificial selection of the Non-agouti and Spotting white markers.
Key words: Felis catus, phenotypic markers, diversity, heterozygosity, Magangué
INTRODUCCIÓN
El gato doméstico (Felis catus) es un mamífero carnívoro de la familia delos félidos, de pequeño tamaño y con peso corporal cercano a los 5 kg, aunque con gran variabilidad entre razas, donde las hembras suelen ser más pequeñas que los machos. Tiene una longitud de unos 50 cm sin incluir la cola. Según Driscoll et al. (2007), los gatos domésticos procederían de cinco líneas maternas de gatos monteses de Oriente Próximo (Felis silvestris lybica) y que habrían sido domesticados a partir de los gatos silvestres (Felis libyca) hace más de 10 000 años, Su áreanatural incluía el norte de África, China, India, sur de Europa, Gran Bretaña e islas del Mediterráneo, y en la actualidad, debido a sus características adaptativas, se encuentran globalmente distribuidos (Ruiz-García et al., 2005).
Los gatos presentan polimorfismos evidentes, coherentes con el color, el patrón y textura de la capa, particularidades factibles de reconocer a simple vista, razón por la cual la recolección de datos sea un procedimiento sencillo. Sumado a esto, los gatos son una especie de elección para este tipo de estudios poblacionales por ser animales cosmopolitas y ser una población panmíctica (Todd 1977).
Los análisis genético poblacionales en gatos son necesarios para revelar la historia de su evolución y para la construcción de hipótesis filogenéticas sobre las relaciones entre los alelos; sin embargo, a pesar la importancia de este tipo de estudios, la información a nivel mundial es escasa y en algunos lugares inexistente.
Magangué puede ser considerada una de las ciudades más importantes del departamento de Bolívar, por ser desde tiempos ancestrales uno de los principales puertos fluviales de Colombia en el rio Magdalena, lo que pudo haber incidido en la diversidad genética de poblaciones de Felis catus. No obstante, no existe información sobre la variabilidad de los marcadores fenotípicos en los genes del pelaje en las poblaciones del gato doméstico en esta ciudad. Por tal motivo, el objetivo de este estudio fue conocer la variabilidad genética de las poblaciones de gatos domésticos (Felis catus) en Magangué mediante marcadores que codifican para el pelaje.
MATERIALES Y MÉTODOS
Recolección de Datos
La recolección de datos se realizó mediante muestreos aleatorios a través de excursiones urbanas y observación directa en 12 barrios urbanos del municipio de Magangué, pertenecientes al departamento de Bolívar, Colombia. Los barrios fueron: Baracoa, Centro, La Candelaria, Córdoba, Simón Bolívar, Villa Juliana, La Florida, Las Delicias, Maracaná, Pueblo Nuevo, San Martín y Versalles.
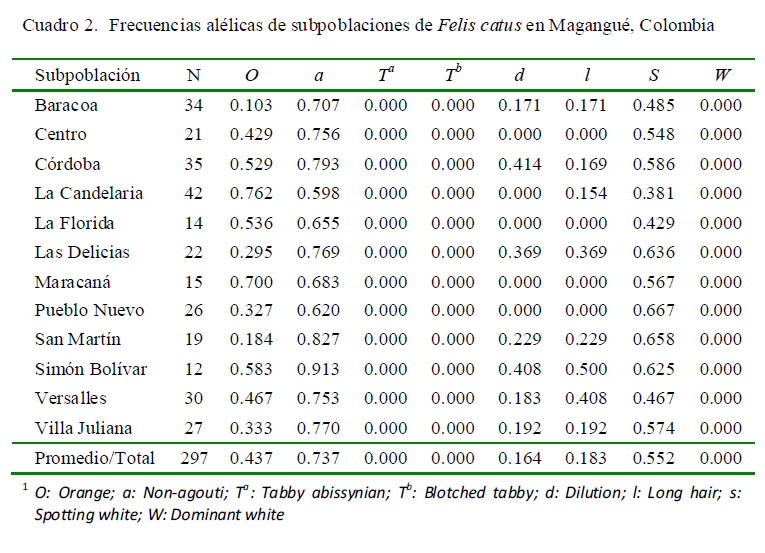
Se realizó una clasificación fenotípica de cada individuo adulto (n=297). Las rutas se utilizaron solo una vez a fin de evitar el remuestreo, atendiendo a la presencia o ausencia de los marcadores autosómicos: Nonagouti(a), Tabby blotched (Tb), Dilution (d), Long hair (l), Spotting white (S)y Dominant white (W), así como el locus ligado al sexo O (Orange) (Cuadro 1). Además, se tomaron registros fotográficos de cada individuo.
Variables de Estudio
Para el estudio de la diversidad genética de las poblaciones de gatos domésticos se tuvieron en cuenta marcadores fenotípicos (Cuadro 1) propuestos por el Committee on Standardized Genetic Nomenclature for Cats (1968): O (Orange. Carácter ligado al sexo) y los loci autosómicos, A (A, a; agouti vs. non-agouti), T (Ta, t+, tb; Abyssinian tabby vs. mackerel o atigrado vs. Tabby blotched), D(D, d; full color vs. Dilution), L (L, l; pelo corto vs. Long hair), S (s+, S; no manchado de blanco vs. Spotting white) y W (w+, W; color normal vs. Dominant white).
Análisis Poblacional
Se estimaron las frecuencias alélicas, la diversidad genética de Nei correspondientes a la heterocigosidad esperada (He) y heterocigosidad de la población total (HT), coeficiente de diferenciación genética (GST), flujo génico (Nm), equilibrio Hardy-Weinberg y la distancia genética entre poblaciones propuestas por Nei (1972) mediante el programa PopGene 1.31 (Yeh et al., 1997).
Para determinar las relaciones genéticas entre las poblaciones analizadas se construyeron árboles filogenéticos que se obtuvieron mediante el algoritmo matemático UPGMA (Unweighted Pair Group Methodwith Arithemetic mean) utilizando datos reportados por Ruiz-García y Álvarez (1999), Causil y Rodríguez (2008), Morales (2008), Peñuela y Cárdenas (2012) y Zambrano y Martínez (2014), y trabajados en el programa MEGA5.2 (Tamura et al., 2011), a partir de la matriz de distancias genéticas.
RESULTADOS
Frecuencias Alélicas
En el análisis de 297 individuos, la frecuencia alélica más alta se presentó en el alelo non-Agouti, especialmente para las subpoblaciones Simón Bolívar (0.913) y San Martín (0.827), mientras que los loci Tabby abissynian, Tabby blotchet y Dominant white no fueron observados en la población estudiada (Cuadro 2).
Diversidad Genética
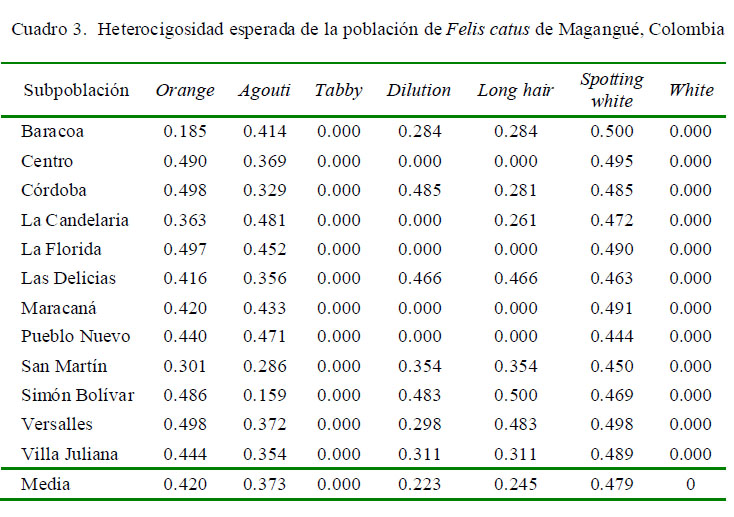
Baracoa fue la subpoblación que presentó el valor más alto de heterocigosidad para el marcador Spotting white, seguido de la subpoblación Simón Bolívar que presentó el valor más alto de heterocigosidad para el marcador Long hair, mientras que los marcadores Whitey Tabby presentaron los valores más bajos de heterocigocidad (Cuadro 3).
El test de equilibrio Hardy-Weinberg (Cuadro 4) a nivel global, mostró ausencia de equilibrio para las poblaciones estudiadas.
Diferenciación Genética y Flujo Génico
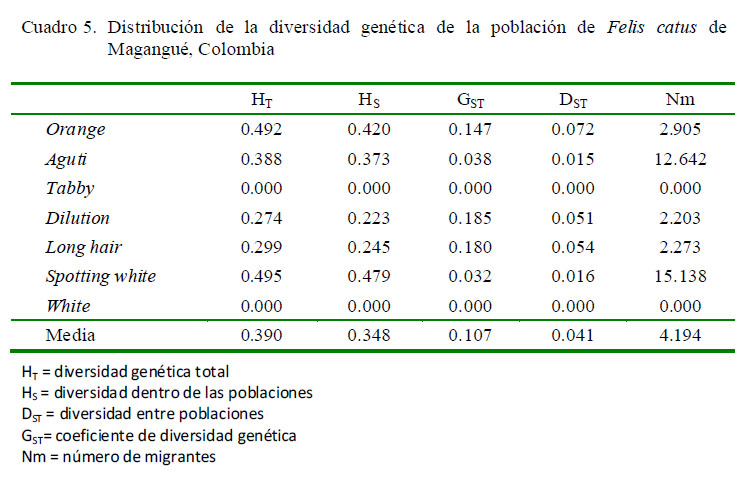
La diversidad genética total (HT) de la población de Magangué es moderada, siendo el promedio igual a 0.390, con un intervalo de 0.274 en el locus Dilution a 0.495 en el locus Spotting white. La mayor parte de la diversidad genética se encuentra dentro de las poblaciones (HS=0.348) y escasa entre las poblaciones (DST=0.041). El coeficiente de diferenciación genética (GST=0.107) indica que el 10.7% de la diversidad genética se encuentra entre poblaciones y el 89.3% dentro de las poblaciones. El valor de Nm mayor a uno indica que existe alto flujo génico entre las subpoblaciones locales estudiadas (Cuadro 5).
Distancia Genética
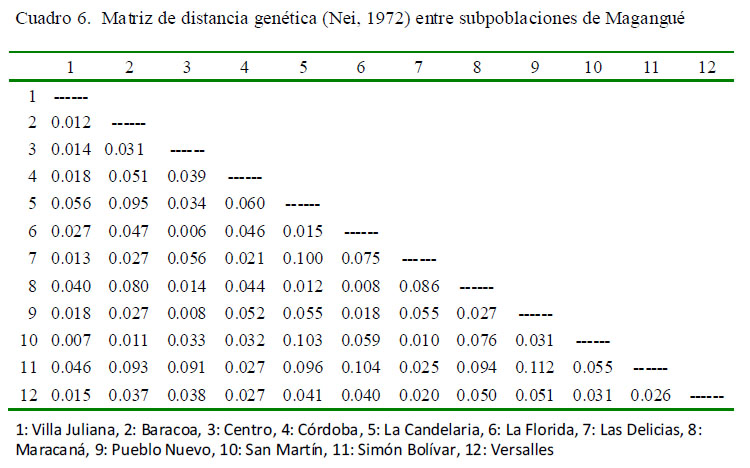
La distancia genética entre las subpoblaciones de Magangué fue baja, siendo San Martín y Villa Juliana las subpoblaciones más cercanas, mientras que Pueblo Nuevo y Simón Bolívar presentaron mayor distancia génica (Cuadro 6).
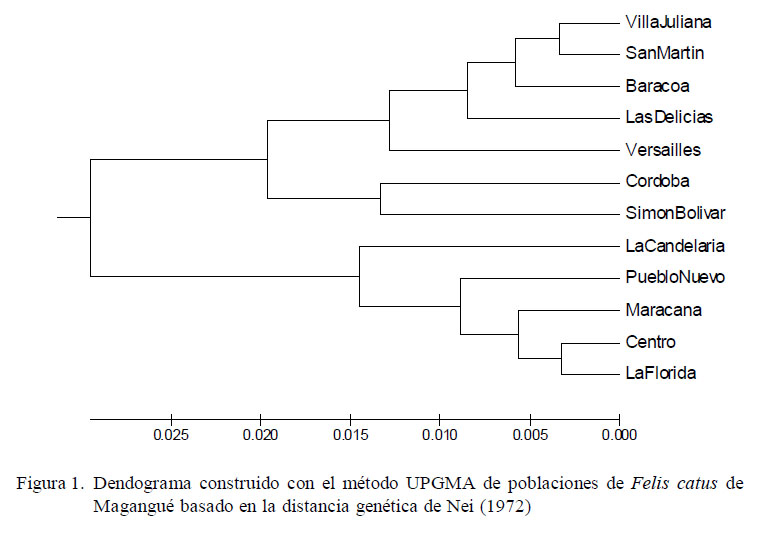
En el dendograma UPGMA elaborado a partir de los valores de distancia genética de Nei (1972) para las doce subpoblaciones de Magangué se puede apreciar claramente la conformación de dos grupos, donde en uno se agrupan las subpoblaciones Villa Juliana, San Martín, Baracoa, Las Delicias, Versalles, Córdoba y Simón Bolívar y el otro conformado por las cinco subpoblaciones restantes (Figura 1).
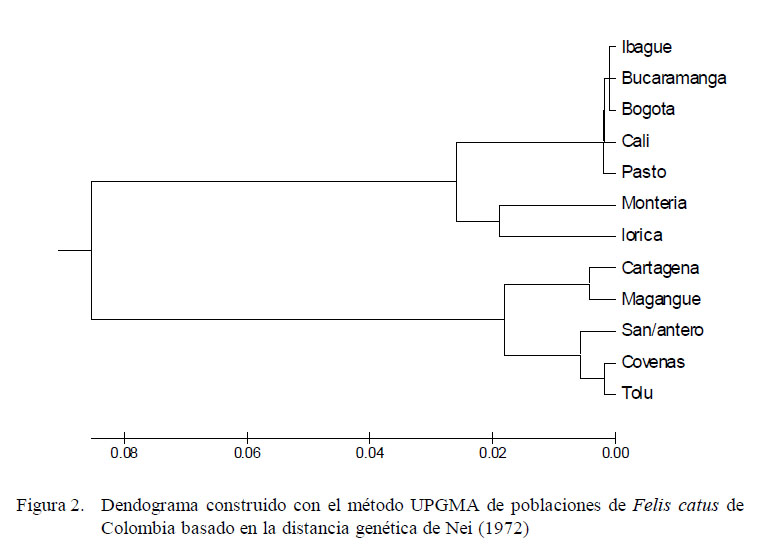
El dendograma UPGMA, elaborado a partir de los valores de distancia genética de Nei (1972) para 12 poblaciones de Felis catus de Colombia, revela la conformación de dos grupos (Figura 2): uno conformado por poblaciones del interior del país como Ibagué, Bucaramanga, Bogotá, Cali y Pasto, a las que se han unido Montería y Lorica, y el segundo grupo formado por poblaciones del Caribe colombiano: Magangué, Cartagena, San Antero, Coveñas y Tolú.
DISCUSIÓN
La elevada frecuencia del alelo nonagouti en Magangué podría estar relacionada con factores ambientales como las elevadas temperaturas, que posiblemente estarían favoreciendo la presencia y aumento de individuos portadores de dicho gen (Ruiz-García et al., 2005, Kaelin et al., 2012), unido a la posible existencia de sucesos selectivos, asociados a una progresiva melanización del pelaje, generando alternativas alélicas que posibilitan que las coloraciones más melánicas se vean favorecidas en el medio urbano (Ruiz-García y Álvarez 2003).
Entre los sucesos selectivos cabe destacar las altas densidades poblacionales y la injerencia humana, debido a que estos individuos son más sociables con otros congéneres, lo que aumenta la posibilidad de co-existir y adaptarse a entornos urbanos (Grahn et al., 2005, Eizirik et al., 2010, Rosenfeld 2010). Estudios realizados en visones (Keeler y Moore 1961; Trapezov et al., 2008) y en zorros (Keeler et al., 1968, Keeler 1975, Borodin 1981) revelan una correlación entre genes de la coloración y ciertos rasgos del comportamiento, resultando los individuos más melánicos menos temerosos, menos agresivos y más resistentes al estrés que produce las zonas urbanas.
El locus Spotting white (s) también presentó altas frecuencias, lo que podría estar relacionado con factores ambientales, ya que este gen se asocia a zonas de altas temperaturas y exposición solar, postulándose como un posible claro ejemplo de selección natural in situ en nuevos hábitats colonizados (Ruiz- Garcia et al., 2005, Kaelin et al., 2012).
La nula frecuencia de los alelos Tabby blotched y Dominant white concuerdan con los resultados de Ruiz-García y Álvarez (2008), quienes afirman que las frecuencias de Tabby blotched en Latinoamérica resultan ser bajas o muy bajas. Por otro lado, la ausencia del alelo Dominant white podría deberse a que este gen se expresa mayormente en lugares donde la temperaturas oscilan entre 28-39 ºC (Morales, 2008), temperaturas bastante menores a las presentadas en Magangué; además, se tiene la particular condición que caracteriza a este gen, pues propicia efectos pleiotrópicos sobre la audición y visión (Geigy et al., 2007), hechos que colocan en desventaja y provocan la reducción sustancial de los individuos que portan este gen.
La presencia de la mayoría de los marcadores estudiados en la población de gatos domésticos de Magangué demuestra la gran variedad de genes disponibles en la zona, situación que posiblemente se puede atribuir a la ubicación geográfica estratégica de dicha población, pues desde hace mucho tiempo es paso obligado de transeúntes, favoreciendo el movimiento de migrantes lo que ha facilitado un considerable flujo génico.
La desviación del equilibrio de Hardy- Weinberg para los alelos O (Orange) y S (Spotting white) puede deberse a diversas causas. El exceso de heterocigotos y déficit de homocigotos encontrado en el estudio podría atribuirse a factores evolutivos como la selección artificial, pues las personas tienen predilección por cierto tipo de carácter en los gatos, lo que ha favorecido algunas característica fenotípica más que a otras. Otro aspecto que podría afectar esta desviación es la cercanía geográfica entre las subpoblaciones estudiadas, lo que ocasiona un aumento sustancial del flujo génico que hay entre las mismas, dado que la existencia de un alto intercambio de genes previene eventos de endogamia al interior de las poblaciones (Cortés, 2008), provocando un aumento de genotipos heterocigotos en la población.
La diversidad genética total (HT) encontrada fue moderada. La mayor parte de la diversidad génica se encuentra dentro de las poblaciones (Hs) y poca entre las poblaciones (DST), lo cual indica que las subpoblaciones locales comparten una gran proporción de la diversidad total. El flujo de genes Nm fue de 4.194 y, en general, se considera que valores cercanos o mayores de 5 son considerados suficientes para mantener una relativa homogeneidad del pool de genes (Slatkin, 1987). Los resultados indican que aparentemente no hay diferenciación entre las subpoblaciones muestreadas. La falta de diferenciación genética entre poblaciones es generalmente considerada como el resultado de un suficiente flujo de genes, típicamente dado por migraciones, ocurriendo en todas las poblaciones para contrarrestar los efectos de la selección o la deriva genética. Según Lowe et al. (2004), un Nm mayor a 1 permite que las poblaciones conserven conectividad genética, ya que el flujo genético sobrepasa los efectos de la deriva e impide la diferenciación local.
El elevado grado de flujo génico permite inferir que las poblaciones se encuentran muy relacionadas genéticamente y se comportan como una metapoblación, situación a la cual se atribuye la aproximación de todas las poblaciones desde el punto de vista estructural (Ruiz-García et al., 2005).
El dendograma UPGMA elaborado a partir de los valores de distancia genética de Nei (1972) en Magangué, muestra como los resultados obtenidos responden a la antigüedad de los barrios (Peña-Cruz et al., 2015).
En el dendograma UPGMA elaborado a partir de los valores de distancia genética de Nei (1972) indicó que Magangué se comporta genéticamente como una sola población, y al compararla con otras ciudades colombianas se encontró similitud con ellas, siendo mayor dicha relación entre Magangué con Cartagena, San Antero, Coveñas y Tolú. La homogeneidad genética hallada en estas ciudades del Caribe colombiano puede estar relacionada con el vertiginoso desplazamiento de los conquistadores españoles por el río Magdalena, donde establecieron muchas ciudades en poco tiempo (Ruiz-García et al., 2005, Peña-Cruz et al., 2015). Además, este gran parecido genético entre las poblaciones puede corresponder a un evento fundador común, las cuales se originaron a partir de las poblaciones españolas (Ruiz-García et al., 2005), al ser una especie ausente en América hasta la inmigración de los europeos (Ruiz- García y Álvarez, 1999). Sumado a esto, cabe resaltar la similitud de las condiciones ambientales de estas ciudades.
CONCLUSIONES
Las frecuencias alélicas más elevadas correspondieron a los alelos Non-agouti y Spotting White, mientras que los alelos Tabby bloched y Dominant white presentaron las menores frecuencias.
Al no encontrarse los loci Orange y Spotting white en equilibrio de Hardy- Weinberg, se presume que en las poblaciones analizadas puede estar ocurriendo selección sobredominante, mutación, deriva génica o la ocurrencia de migraciones.
Se evidenció una posible selección natural y artificial de los marcadores Nonagouti y Spotting white.
LITERATURA CITADA
1. Borodin PM. 1981. Phenotype and gene frequencies in red fox populations of Russian America in 1803-1832. J Hered 72: 343-346. [ Links ]
2. Causil L, Rodríguez A. 2008. Análisis genético poblacional de gatos domésticos (Felis catus) mediante genes del pelaje en el municipio de Santa Cruz de Lorica. Tesis de pregrado. Montería: Universidad de Córdoba. 75 p. [ Links ]
3. Commitee on Standardized Genetic Nomenclature for Cats. 1968. Standardized genetic nomenclature for the domestic cat. J Hered 59: 39-40. [ Links ]
4. Cortés O. 2008. Análisis de la variabilidad genética en la raza bovina de lidia utilizando información molecular. Tesis doctoral. Madrid: Universidad Complutense de Madrid. 244 p. [ Links ]
5. Driscoll CA, Menotti-Raymond M, Roca AL, Hupe K, Johnson WE, Geffen E, Harley EH, et al. 2007. The Near Eastern origin of cat domestication. Science 317: 519-523. doi: 10.1126/ science.1139518 [ Links ]
6. Eizirik E, David VA, Buckley-Beason V, Roelke ME, Schäffer AA, Hannah SS, Narfström K, et al. 2010. Defining and mapping mammalian coat pattern genes: multiple genomic regions implicated in domestic cat stripes and spots. Genetics 184: 267-275. doi: 10.1534/genetics.109.109629 [ Links ]
7. Geigy CA, Heid S, Steffen F, Danielson K, Jaggy A, Gaillard C, et al. 2007. Does a pleiotropic gene explain deafness and blue irises in white cats? Vet J 173: 548-553. doi: 10.1016/ j.tvjl.2006.07.021 [ Links ]
8. Grahn RA, Lemesch BM, Millon LV, Matise T, Rogers QR, Morris JG, Fretwell N, et al. 2005. Localizing the X-linked orange colour phenotype using feline resource families. Anim Genet 36: 67-70. doi: 10.1111/j.1365-2052.2005. 01239.x [ Links ]
9. Kaelin CB, Xu X, Hong LZ, David VA, McGowan KA, Schmidt-Küntzel A, Roelke ME, et al. 2012. Specifying and sustaining pigmentation patterns in domestic and wild cats. Science 337: 1536-1541. doi: 10.1126/science.1220893 [ Links ]
10. Keeler C, Moore L. 1961. Psychosomatic synthesis of behaviour trends in the taming of mink. Bul Georgia Acad Sci 19: 66-74. [ Links ]
11. Keeler C, Ridgway S, Lipscomb L, Fromm E. 1968. The genetics of adrenal size and tameness in colour phase foxes. J Hered 59: 82-94. [ Links ]
12. Keeler C. 1975. Genetics of behaviour variations in color phases of the red fox. In: Fox MW (ed). The wild canids: their systematics, behavioral ecology, and evolution. New York: Dogwise. p 399- 417. [ Links ]
13. Lowe A, Harris S, Ashton P. 2004. Ecological genetics: design, analysis, and application. UK: Blakwell Publishing. 344 p. [ Links ]
14. Morales J. 2008. Estudio de la estructura genética existente en las poblaciones de gatos domésticos (Felis catus) en el municipio de Montería - Córdoba. Tesis de pregrado. Montería: Universidad de Córdoba, Colombia. 71 p. [ Links ]
15. Nei M. 1972. Genetic distance between populations. Am Nat 106: 283-292. [ Links ]
16. Peña-Cruz AF, Sandoval S, Patiño A, Bedoya M, Rodríguez A, Orjuela J, Ortega A, et al. 2015. Análisis genético de la población de gatos del norte y sur de Cali, Colombia. Acta Biol Colomb 20: 109 116. doi: 10.15446/abc.v20n1.41610 [ Links ]
17. Peñuela M, Cárdenas H. 2012. Marcadores genéticos del pelaje en gatos domésticos de Capurganá-Colombia. Momentos de Ciencia 9(1): 28-33. [ Links ]
18. Rosenfeld CS. 2010. Animal models to study environmental epigenetics. Biol Reprod 82: 473-488. doi: 10.1095/ biolreprod.109.080952 [ Links ]
19. Ruiz-García M. 1994. Genetic profiles from coat genes of natural Balearic cat populations: an eastern Mediterranean and North-African origin. Genet Sel Evol 26: 39-64. doi: 10.1186/1297-9686-26-1-39 [ Links ]
20. Ruiz-García M, Álvarez D. 1999. Análisis filogenético de 21 poblaciones latinoamericanas de gatos mediante 10 loci morfológicos utilizando métodos de matrices de distancias genéticas y de máxima parsimonia. Bol. Real Soc Espan Hist Nat 95: 139-164. [ Links ]
21. Ruiz-García M, Álvarez D. 2003. Posible origen europeo de seis poblaciones latinoamericanas de gatos y no existencia de paralelismo con el modelo colonizador británico al utilizar genes del pelaje y microsatélites. Acta Zool Mex 89: 261-286. [ Links ]
22. Ruiz-Garcia M, Alvarez D, Shostell JM. 2005. Population genetic analysis of cat populations from Mexico, Colombia, Bolivia, and the Dominican Republic: identification of different gene pools in Latin America. J Genet 84: 147-171. doi: 10.1007/BF02715841 [ Links ]
23. Ruiz-García M, Álvarez D. 2008. Biogeographical population genetics perspective of the colonization of cats in Latin America and temporal genetic changes in Brazilian cat populations. Genet Mol Biol 31: 772-782. doi: 10.1590/ S1415-47572008000400026
24. Slatkin M. 1987. Gene flow and the geographic structure of natural populations. Science 236: 787-792. doi: 10.1126/science.3576198 [ Links ]
25. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol 28: 2731-2739. doi: 10.1093/molbev/msr121 [ Links ]
26. Todd NB. 1977. Cats and Commerce. Sci Am 237: 100-107. doi: 10.1038/ scientificamerican1177-100 [ Links ]
27. Trapezov O, Trapezova L, Sergeev E. 2008. Effect of coat color mutations on behavioral polymorphism in farm populations of American minks (Mustela vison Schreber, 1777) and sables (Martes zibellina Linnaeus, 1758). Genetika 44: 516-523. [ Links ]
28. Yeh, FC, Yang RC, Boyle TB, Ye ZH, Mao JX. 1997. POPGENE, the userfriendly shareware for population genetic analysis. Canada: Molecular Biology and Biotechnology Centre, University of Alberta. 10 p. [ Links ]
29. Zambrano JD, Martínez MT. 2014. Estudio de la diversidad genética en poblaciones de gato doméstico (Felis catus) mediante genes de pelaje en los municipios de San Antero, Córdoba, Coveñas y Tolú, Sucre. Tesis de pregrado. Montería: Univ de Córdoba, Colombia. 82 p. [ Links ]
Recibido: 22 de octubre de 2015
Aceptado para publicación: 4 de febrero de 2016