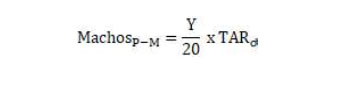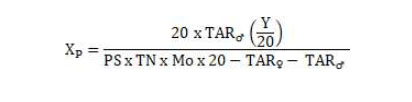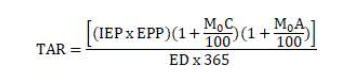Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.29 no.3 Lima jul./set. 2018
http://dx.doi.org/10.15381/rivep.v29i3.14840
ARTÍCULOS PRIMARIOS
Esquema de reproductores macho de referencia para un núcleo genético disperso de alpacas (Vicugna pacos) Huacaya en la región Pasco, Perú
Sire Referencing Scheme for A Dispersed Genetic Nucleus ff Alpacas (Vicugna Pacos) Huacaya in the Pasco Region, Peru
Eliet Amanca H.1,4, Jorge Calderón V.1, Vanerlei Roso M.2, Jorge Mendoza D.3, Gustavo Gutiérrez R.1
1Departamento Académico de Producción Animal, Universidad Nacional Agraria la Molina (UNALM), Lima, Perú
2GenSys Consultorios Asociados, Porto Alegre, Brasil
3Facultades y Programas de Investigación, Universidad Nacional Agraria la Molina, Lima, Perú
4E-mail: amancah.08@gmail.com
RESUMEN
Los rebaños de alpacas en la Región Pasco del Perú presentan escasos vínculos genéticos conocidos, de allí que el mérito genético de los reproductores de los rebaños no pueda ser comparado, ya que los efectos ambientales pueden enmascarar la diferencia genética entre ellos. El propósito del presente estudio fue evaluar un esquema de reproductores macho de referencia para un núcleo genético disperso de alpacas Huacaya en la región Pasco. Se recopiló información para evaluar la capacidad organizacional de los productores y caracterizar la distribución poblacional de seis rebaños en el año 2016. Se calculó el tamaño del núcleo en base a la necesidad de reemplazo anual de reproductores machos en la majada. Se simularon genealogías para tres esquemas de reproductores macho de referencia (EMR) con 3, 6 y 9 machos, que empadraron al 10, 20 y 30% de hembras en el núcleo respectivo en cada generación. Los grupos contemporáneos (GC) fueron definidos por rebaño-año durante tres generaciones. Finalmente, se calculó el grado del vínculo genético utilizando el método de número total de lazos genéticos directos (LGD) entre GC. Se encontró que los rebaños evaluados poseen una estructura genética central y abierta, que mantienen un bajo flujo de animales entre plantel y majada.
El tamaño mínimo del núcleo genético disperso fue de 747 hembras y 41 machos. Se reportó en los tres escenarios de EMR simulados en el núcleo genético disperso un alto grado de vínculo genético en tres generaciones, que varió entre 98.20 y 100% de LGD.
Palabras clave: alpaca; macho referencia; núcleo disperso; vínculo genético
ABSTRACT
The alpaca herds in Pasco region, Peru, have few known genetic links, so the breeding value of sires from different herds cannot be compared since environmental effects can mask the genetic differences among them. The aim of the study was to evaluate a sire referencing scheme for a dispersed genetic nucleus of Huacaya alpacas in Pasco. Data was collected to evaluate the organizational capacity of the breeders and characterize the population structure of six herds belonging to the nucleus in 2016. The size of the nucleus was calculated based on the need for annual replacement of sires. The genealogy was simulated for three scenarios of sire reference schemes (SRS) with 3, 6 and 9 males mating 10, 20 and 30% of dams in the nucleus per generation respectively. The contemporary groups (CG) were determined by herd-year for three generations. Finally, the degree of the genetic link was calculated using the method of total number of direct genetic links (TDGL) among CG. Results showed that the evaluated herds have a central and open genetic structure, with a low flow of animals between the nucleus and the based group. The minimum size of the dispersed genetic nucleus was 747 dams and 41 males. The three SRS scenarios in the dispersed genetic nucleus showed a high degree of genetic link in three generations, ranged from 98.20 to 100% of TDGL.
Key words: alpaca; sire reference; dispersed nucleus; genetic link
INTRODUCCIÓN
Los sistemas de crianza de alpaca se caracterizan por poseer rebaños con pocos animales y con modalidades de crianza independiente de otros rebaños, lo cual causa una rápida disminución de su variabilidad genética, si no se recurre a la adquisición de machos fuera del rebaño. Sin embargo, el productor se enfrenta a la incertidumbre sobre el mérito genético de dichos animales, de sus características productivas de importancia, y de no poder comparar con los animales de su propio rebaño, debido a que se encuentran genéticamente desvinculados o mantienen un débil vínculo, donde los efectos ambientales pueden enmascarar la diferencia genética de los animales (Lewis y Simm, 2000; Kuehn, et al. 2007; Haile et al., 2011).
Establecer el vínculo genético entre rebaños permite realizar comparaciones entre los animales que lo conforman (Lewis y Simm, 2000). Sin embargo, la exactitud de las comparaciones depende del grado de vínculo genético entre los grupos en evaluación (Roso et al., 2004). Por esta razón, una metodología para crear vínculo genético en rebaño de ovinos bastante utilizada es el esquema reproductor macho de referencia, que consiste en el uso compartido de ciertos machos (Roden, 1996). El esquema de reproductores macho de referencia, con el uso amplio de inseminación artificial podría crear altos grados de vínculos entre los rebaños (Simm et al., 2001).
El objetivo del presente este estudio fue estructurar un esquema de machos de referencia para un núcleo genético disperso de alpacas Huacaya, en seis rebaños de sistemas semi-tecnificados y con monta natural en la región Pasco, Perú. Los objetivos específicos fueron; i) describir la estructura genética de los rebaños; ii) calcular el tamaño del núcleo; y iii) simular el esquema de reproductores machos de referencia para obtener un alto grado de vínculo genético en el núcleo disperso.
MATERIALES Y MÉTODOS
Localidad y Animales
Se evaluaron seis rebaños de alpacas pertenecientes a la Cooperativa Comunal de San Pedro de Racco (R1), Empresa Ganadería y Comercio del Centro SRL (R2), Granja Comunal de Huayllay (R3), Cooperativa Comunal Yurajhuanca (R4), Asociación Pecuaria Janampa Sanjo (R5) y Asociación de Ganaderos Hermanos Tufino Cachipampa (R6), ubicados en la región Pasco, Perú entre 4000 y 5000 msnm.
Se utilizó información proveniente de registros de 7038 hembras y 1815 machos alpacas Huacaya, según inventario y planilla de octubre de 2016. Los rebaños evaluados forman parte del Proyecto ‘Mejorando los sistemas de producción de alpacas en pastizales en la sierra central del Perú’, ejecutado por la Universidad Nacional Agraria la Molina en cooperación con el Concejo de Universidades Flamencas (VLIR) desde 2011. El trabajo consistió en tres etapas: descripción de la estructura genética de los rebaños evaluados, cálculo del tamaño de núcleo para la estructura por núcleo genético disperso y simulación estocástica de genealogía para escenarios de esquema reproductores macho de referencia y, finalmente, el cálculo del grado de vínculo genético generado en dichos escenarios.
Estructura Genética de los Rebaños
Se realizaron tres visitas a cada establecimiento para realizar las entrevistas al encargado del rebaño, observar el manejo del sistema y conversar con los representantes del proyecto VLIR. La estructura organizacional de los rebaños fue descrita según los siguientes criterios: capacidad organizacional, toma de decisiones, recursos económicos, poder adquisitivo, infraestructura y manejo. Para el caso de este último se consideró la alimentación, empadre controlado, destete, selección y esquila, los cuales fueron calificados de malos a muy buenos. Los criterios considerados para la calificación se muestran en el Cuadro 1 y fueron derivados de estudios realizados por Barrantes (2012) y Ruiz et al. (2015) para la caracterización de sistemas de producción pecuaria en la región Pasco.
La población del estrato superior, denominado plantel, fue clasificada según edad en DL (<1 año), 2D (2-3 años), 4D (3-4 años) y BLL (>4 años), sexo y clase. Para las clases se tuvieron los siguientes grupos:
-Clase S: Clase Súper. Libres de defectos congénitos, poseen excelente conformación y uniformidad de rizos en los puntos de visualización paleta, costillar medio y muslo, densa, excelente finura, sin canas, color blanco entero;
-Clase A: Libre de defecto congénito, poseen buena conformación, pero menor que la clase S, buena uniformidad en los puntos de visualización paleta, costillar medio y muslo, buena densidad, buena finura, sin canas, color blanco entero;
-Clase B: Libre de defectos congénitos, tienen una conformación regular menor que la A, menor densidad, son de color blanco entero, poseen canas casi invisibles, uniformidad del vellón, rizos y diámetro de fibra menos uniformes;
-Clase C: Libre de defectos congénitos, color blanco entero, mayor diámetro de fibra que las clases anteriores, menor densidad al tacto. En el caso de hembras son colocadas en el estrato de núcleo, pero en el caso de machos son llevados a la majada;
-Clase R: Animales de rechazo, con defectos congénitos, vellón manchado, mala conformación, fibra demasiado gruesa y canosa;
-Clase RV: Animales de rechazo por vejez.
La población de la majada fue descrita según edad y sexo, considerándose el total de animales reproductores registrados en las planillas mensuales menos los animales inventariados en el plantel. El flujo de animales se obtuvo de los registros de cada rebaño, de los traslados de reproductores entre estratos en cada rebaño y los registrados del ingreso de otros rebaños.
Cálculo del Tamaño de Núcleo
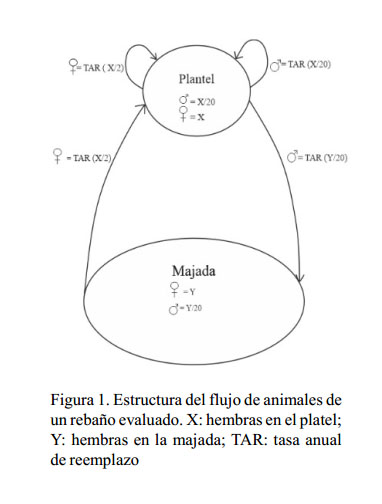
Con los datos previos se realizó el cálculo del número de reproductoras hembras en el plantel (X) con base al número de hembras en la majada (Y) (Figura 1). Se hicieron los siguientes pasos:
Paso 1: Y, indica el número de hembras en la majada (la relación macho-hembra en este estrato se mantiene en 1/20), y TAR corresponde a la tasa anual de reemplazo. Los machos mejorados de la majada son provenientes del plantel y se calcula por:
Paso 2: El valor de X en cada rebaño se halló según a la necesidad de reemplazo de hembras y machos en el mismo plantel y machos en la majada. La proporción de selección (PS) fue considerada entre 70 y 90%. Además, se consideró la tasa de natalidad (TN) y de mortalidad (Mo). Este cálculo se realizó con el uso del Excel, modificado de Haile et al (2011) y Mueller (2013).
Paso 3: El número total de hembras en el núcleo, se obtuvo de la suma de los valores X calculados de los seis rebaños. Se incluyó en la fórmula de reemplazo de animales machos y hembras para obtener la cantidad de animales requeridos para el tamaño de majada.
El porcentaje de natalidad bruta (TN) de52% para R1,R2,R3 y R4 y de37% para R5 y R6, así como el 5% de mortalidad (Mo) fueron obtenidos de Ruiz et al. (2015), mientras que 37% en hembras y 44% en machos de tasa anual de reemplazo (TAR) fue calculado siguiendo la metodología planteada por Gonzáles et al. (2002).
donde IEP: Intervalo entre partos (se consideró 365); EPP: Edad al primer parto (3 años en machos, ya que en los machos inician la actividad reproductiva a los 2 años, previa revisión de la liberación pene-prepucio, de modo que al siguiente año ya se cuenta con sus progenies y 2 años en hembras para R1, R2, R3 y R4, y 3 años en hembras para R5 y R6); M0C y M0A: Mortalidad de crías y mortalidad de adultos (se consideró 5%); ED: Edad al descarte (10 años en machos y 8 años en hembras para R1,R2,R3 y R4,y 9 años para R5 y R6, dado que ingresaron en forma tardía a la edad reproductiva).
Esquema de Reproductor Machos de Referencia
El tamaño de núcleo obtenido sirvió de referencia para la simulación estocástica de datos de genealogía por tres generaciones de alpacas con el uso compartido de machos en común por los seis rebaños evaluados. Se realizó con tres escenarios de esquemas de reproductores macho de referencia (EMR), para lo cual se usó el lenguaje computacional FORTRAN 95.
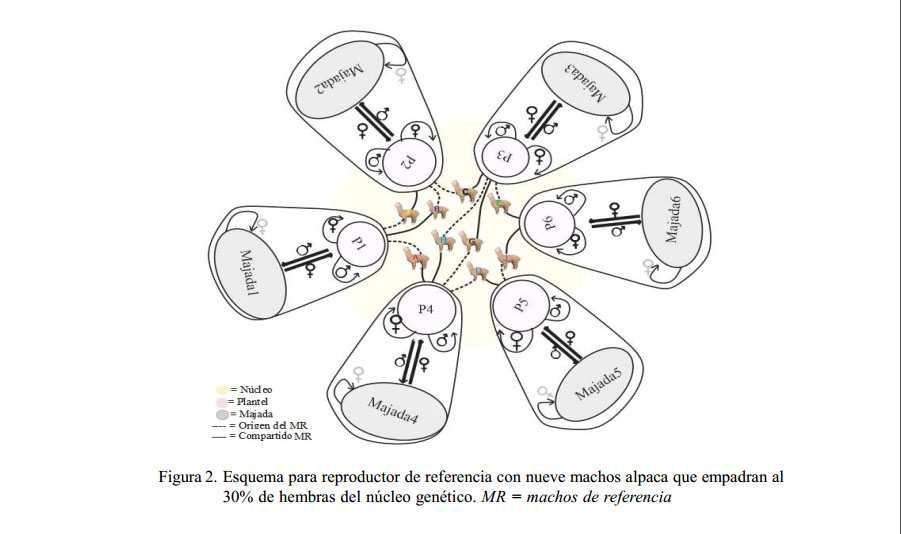
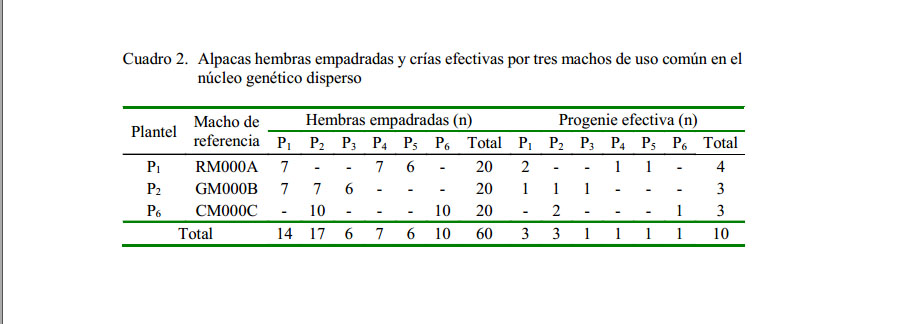
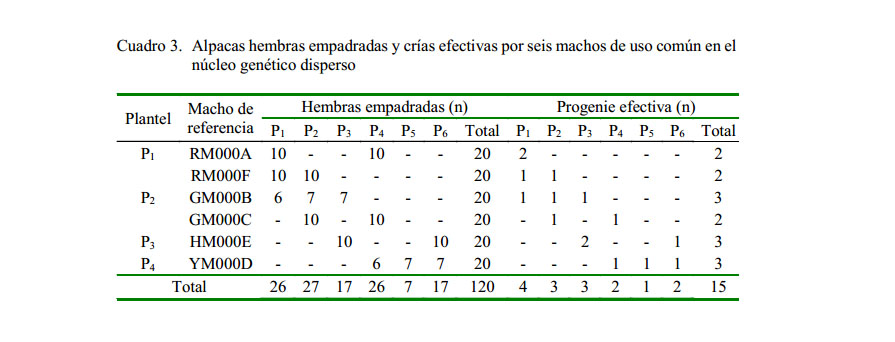
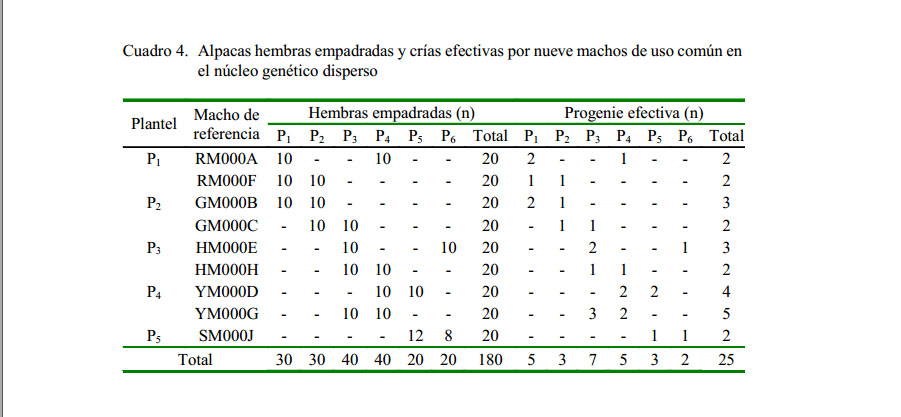
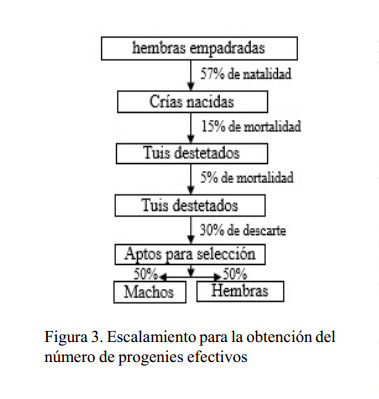
Los escenarios fueron de 3, 6 y 9 reproductores machos de referencia que empadraron al 10, 20 y 30%, respectivamente, de hembras del núcleo genético disperso (Figura 2). La relación macho hembra fue 1/20. La cantidad de progenies efectivas se presenta en los cuadros 2, 3 y 4. Los machos de referencia que llegaron a la selección fueron asumidos asumió según los parámetros mostrados en la Figura 3 (Quispe, 2010).
Por lo tanto, cada escenario de EMR fue simulado según a la cantidad de hembras y progenies mostrados en los cuadros 2 al 4. Los reproductores machos de referencia poseen un código asignado como CM0000A: C (Rebaño), M (Sexo), 0 (Año); 00A (número del animal, alfanumérico).
Finalmente, se calculó el grado de vínculo genético en los tres escenarios de EMR. Para un tema de comparación se identificaron los grupos contemporáneos (GC) agrupados por rebaño-año en los datos simulados. Los 18 GC eran animales sometidos a las mismas condiciones de medio ambiente, y se utilizaron para registros de genealogía por las tres generaciones
El análisis del grado del vínculo genético entre GC se realizó por el método de número total de lazos genéticos directos (LGD) entre grupos contemporáneos. Se utilizó el software AMC-A Computer Program to Assess the Degree of Connectedness Among Contemporary Groups (Roso y Schenkel, 2006).
RESULTADOS
Estructura Genética de los Rebaños
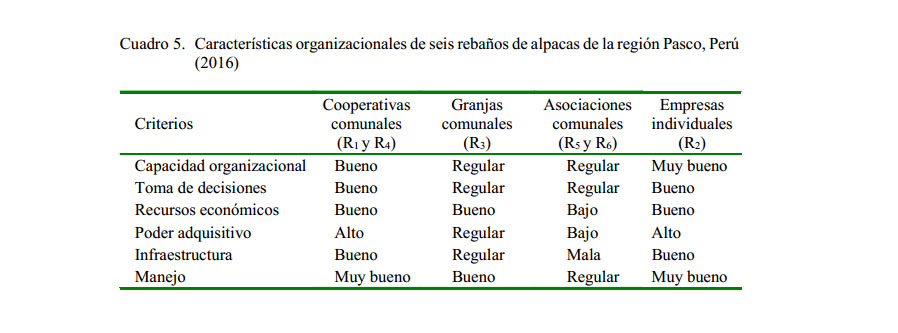
El Cuadro 5 muestra las características distintivas de la estructura organizacional de los propietarios de los seis rebaños de alpacas. La empresa individual y las cooperativas comunales presentaron las calificaciones superiores. Además, se resalta los criterios más importantes para la planificación de la estructura genética en los programas de mejoramiento genético, que son la capacidad organizacional y el manejo de la crianza, ya que en todos los rebaños fueron calificadas de regular a muy buena.
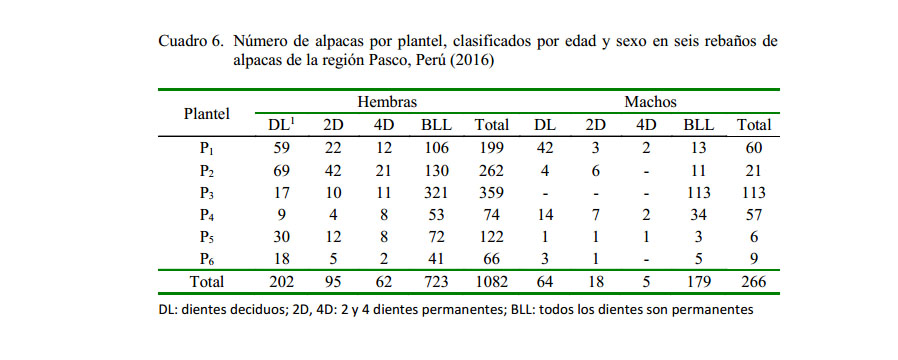
Los rebaños evaluados están formados por dos estratos: un estrato plantel con animales de calidad superior y otra denominada majada. El estrato plantel de todos los rebaños está compuesto por alpacas hembras y machos con edades de dientes deciduos (DL), 2D, 4D y boca llena (BLL), que a su vez hacen el núcleo de la estructura genético disperso. La distribución de las alpacas dentro de los planteles, por sexo y por edad, se presenta en el Cuadro 6, donde se observa la gran proporción de hembras y machos con edad de BLL.
Los planteles están conformados por alpacas clasificadas, según evaluación visual en: Clase S, Clase A, Clase B y Clase C, lo cual está relacionado a la finura de fibra y peso de vellón, así como a la apariencia general del animal.
El número de alpacas en los planteles, según el inventario, fueron de 720 hembras seleccionadas para reproducción y 120 machos distribuidas en las clases S, A, B y C. No fueron inventariados como reproductores las alpacas de la clase R (rechazo) por no cumplir con los estándares mínimos para pertenecer al plantel; así mismo, los de la clase RV son alpacas que ya cumplieron su vida reproductiva.
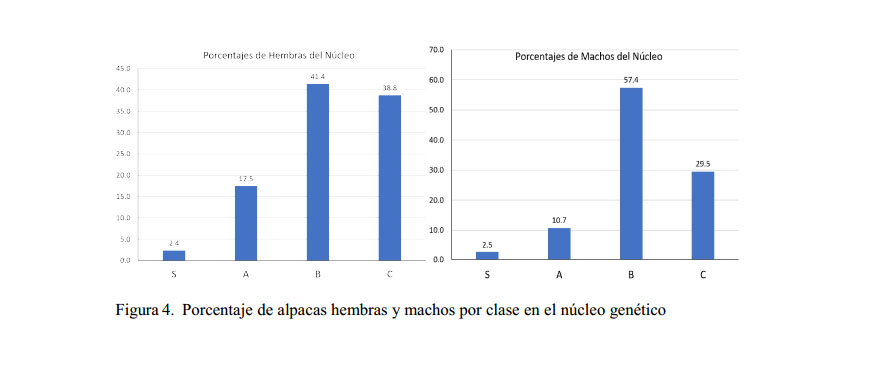
En la Figura 4 se presentan los porcentajes de alpacas existentes en el núcleo por clase, donde el 2.4% de hembras y el 2.5% de los machos pertenecen a la clase S, y el 17.5% de hembras y 10.7% de machos son de la clase A. Además, se muestra que la clase B tiene la mayor población de los dos sexos, seguido de la clase C.
La majada está compuesta por alpacas hembras pertenecientes a las clases C y R (n= 5956) y por machos de las clases B y C. Con relación al flujo de animales registrados en 2016, hembras de plantel fueron transferidas a la majada de los rebaños R1, R2, R5 y R6. Así mismo, solo el R2 trasladó un macho reproductor del plantel a la majada. Por otro lado, machos reproductores fueron trasladados de las majadas al plantel en los rebaños R1, R2 y R6. Además, solo tres rebaños compartieron machos (Cuadro 7).
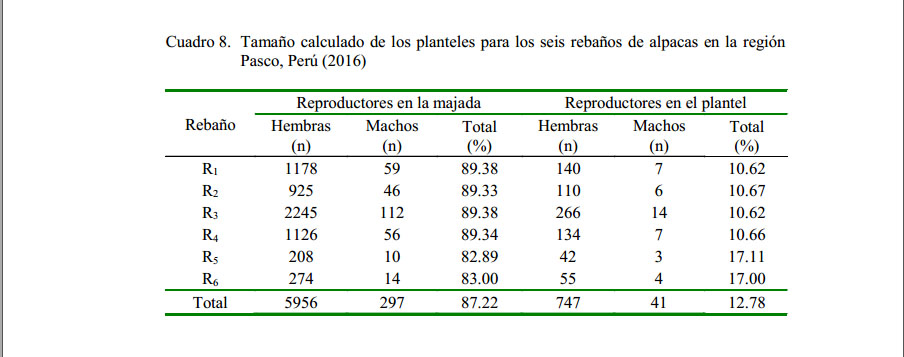
Tamaño de Núcleo para el Núcleo Genético Disperso
El número de alpacas calculadas para el núcleo se basó en las consideraciones de manejo de los rebaños miembros del núcleo genético disperso. Se tuvo 41 machos y 747 hembras reproductores en el plantel ( Cuadro 8), los cuales generan 107 hembras y 16 machos para reemplazo propio, y136 machos para reemplazo de la majada. Según al resultado de la descripción, se dice que por ser un núcleo abierto, el 50% del reemplazo de hembras del núcleo son aportados por la majada, entonces 107 hembras de la majada fueron al núcleo.
Grado de Vínculo Genético del Esquema de Reproductor Macho de Referencia
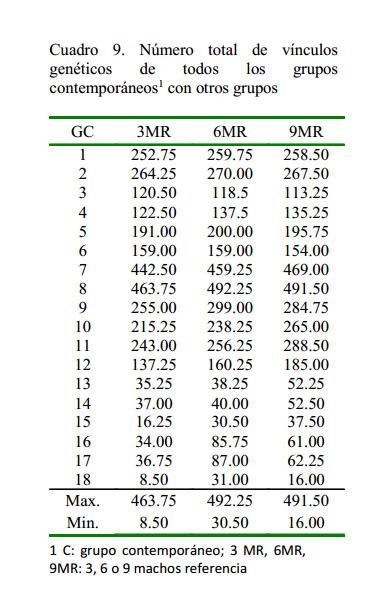
En el primer escenario de tres EMR se obtuvo el mayor valor para el GC-8 con 463.75 de total de lazos genéticos (TLG). Así mismo, se observa que el GC-18 registró 8.5 TLG, lo cual significa que este no mantiene un vínculo genético con el grupo principal (Cuadro 9). Sin embargo, en vista que su valor es superior a cero, el método de TLG permite afirmar que al menos mantiene vínculo con los demás grupos secundarios; por lo tanto, se considera que el GC-18 generó un vínculo genético indirecto con el grupo principal.
En el escenario de seis EMR se obtuvo el mayor valor para el GC-8 con 492.25 TLG, por lo que se le considera como el grupo principal. El menor valor fue de 30.5 TLG para el GC-15; sin embargo, es superior al límite inferior para mantener el vínculo con el grupo principal (>10), por lo que se considera que este grupo aún mantiene un vínculo directo con el grupo principal. No se encontraron grupos secundarios ni desvinculados (Cuadro 9).
El tercer escenario de nueve EMR tuvo como grupo principal al GC-8 con 491.5 TLG. Los grupos secundarios fueron los 17 restantes. El menor valor encontrado fue para GC18 con 16 TLG. No se presentaron GC con vínculos genéticos indirectos ni grupos desvinculados ( Cuadro 9).
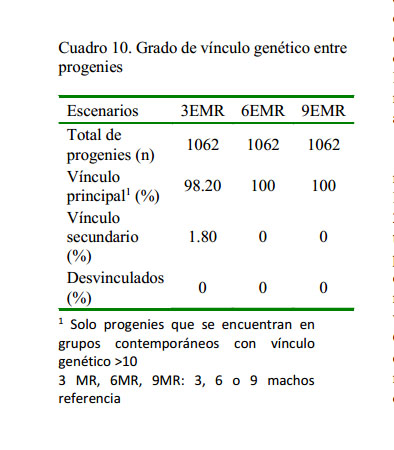
Los resultados del porcentaje de vínculos generados por las progenies muestran que en el escenario de tres EMR, el grado de vínculo principal fue de 98.2%, el vínculo secundario de 1.8% y no existieron animales desvinculados. Por otro lado, las progenies de los escenarios para seis EMR y nueve EMR establecieron el 100% de vínculo genético principal ( Cuadro 10).
DISCUSIÓN
El éxito de las estructuras genéticas planteadas para pequeños rumiantes es la activa participación de todos los involucrados (Haile et al., 2011). Así mismo, Mueller (2017) menciona que los programas de mejoramiento genético en pequeños rumiantes deben basarse en la organización de los productores e instituciones por tener participación clave para su sostenibilidad en sistemas extensivos y semi-intensivos. La adecuada organización y planificación de actividades de los productores permite el mejor control en el manejo, sobre todo en el apareamiento de los animales del plantel (Gizaw et al., 2014a).
La estratificación del rebaño en sistemas semi-intensivos se inicia básicamente con la evaluación visual de los animales (Mueller, 2013), a pesar de que Corredor (2015) mostró una coincidencia con las altas respuestas productivas de las alpacas en los rebaños evaluados en el presente trabajo para las características de diámetro de fibra y peso de vellón con las clases S y A. Sin embargo, Gizaw et al. (2014b) mencionan la importancia de los registros para la evaluación genética, motivo por el cual se realiza la estratificación de los animales para un adecuado manejo de genealogía. En efecto, se desea que el núcleo esté compuesto por los mejores machos y las mejores hembras, dado que ambos aportan a la progenie la mitad de sus genes (Mueller, 2001; Ahuya et al., 2005). La finalidad del sistema de núcleos es concentrar la ganancia genética (Haile et al., 2011).
La distribución por edad de animales en el núcleo en el presente trabajo difiere a lo reportado por Canazas (2001), quien encontró mayor frecuencia en animales de 4D (39.5%) y menor proporción de BLL (13.1%), y a lo encontrado por Cáceres y Díaz (2007), que registraron mayor cantidad de alpacas de 2D (33.9%).
La principal finalidad del plantel es la de proporcionar machos mejoradores a la majada (Haile et al., 2011; Iñiguez et al., 2013); sin embargo, los rebaños evaluados en 2016 registraron bajos niveles de flujo de animales entre sus estratos plantel y majada. Esta práctica realizada en la presente descripción del flujo genético no coincide con lo señalado por otros autores (Haile et al., 2011; Mueller, 2013; Gizaw et al., 2014a), quienes mencionan que el núcleo genético abierto se caracteriza por permitir el ingreso de las mejores hembras de estratos inferiores hasta en un 50% del reemplazo del plantel.
Por otro lado, el tamaño de núcleo calculado para la estructura genética de núcleo disperso fue similar a los reportado por Mueller et al. (2015) con el 16.8% de plantel para una población de cabras en un sistema de empadre natural. Así mismo, Haile et al. (2011) indican que entre 5 y 15% de la población de ovinos representa un plantel genético abierto. Resultados distintos son reportados por Gicheha et al. (2006) para ovejas en un sistema de empadre natural, donde el tamaño del plantel fue el 5% de la población total para proporcionar el 30% de la población base en un núcleo cerrado.
Con respecto a los escenarios de machos de referencia planteados en la estructura de núcleo disperso, los resultados obtenidos del grado de vínculo genético entre grupos de comparación son inferiores a lo reportados por Roso et al. (2004) de 707 TLG, lo cual podría ser explicado porque en dicho estudio se trabajó con poblaciones de vacunos bajo inseminación artificial donde el reproductor macho de referencia podía compartir progenies en varios rebaños. En el presente estudio, sin embargo, los escenarios simulados fueron para reproducción de alpacas por monta natural.
Como era de esperarse, los escenarios con mayor número de progenies de los machos de referencia generaron mayores TLG entre grupos. Similares resultados fueron reportados por Kuehn et al. (2007) evaluando escenarios de machos de referencia para bovinos, donde registraron 20 progenies del macho reproductor común para que el esquema tenga claras ventajas en la disminución de sesgos de 50 a 33% de la evaluación genética.
Los resultados de vínculos genéticos también coinciden con Tarrés et al. (2010), quienes mencionan que la calidad de diseño del EMR, a través de la proporción de progenies de machos comunes dentro de un rebaño, permite mejorar el sesgo en la comparación de valores genéticos entre rebaño. Mathur (2005) señala, asimismo, que alrededor del 15% de las progenies de reproductores macho de referencia en un rebaño son suficientes para alcanzar un buen nivel de vínculo genético en porcinos. Por otro lado, Hanocq et al. (1999) mencionan que solo con realizar un adecuado vínculo se incrementa la tendencia genética (+20%) en cada ciclo de selección. Kennedy y Trus (1993) mostraron que el vínculo aumenta con la relación entre los grupos, mientras que disminuye cuando aumenta la relación dentro del grupo.
En la revisión de otras investigaciones, se observa que Kuehn et al. (2008) reportaron 20% de grado de vínculo genético en 15 rebaños de ovinos con modelo macho, y que el empleo de 15 GC con machos de referencia permite minimizar el riesgo de comparar valor de cría del animal. Por su parte Tarrés et al. (2010) registró un vínculo genético alto de 70.7% para una evaluación genética con modelo animal en 18 rebaños de bovinos. Esto indica que los porcentajes de vínculo de este estudio son altos, y similares a lo reportado por Roso et al. (2004) de 94.5% de TLG para rebaños de bovinos de carne.
CONCLUSIONES
-
Los rebaños de alpacas en el año 2016 estaban establecidos por un núcleo genético central y abierto, con bajo flujo de animales entre plantel y majada.
-
Bajo los parámetros zootécnicos del estudio y una población base de 5956 hembras, el tamaño mínimo del núcleo de alpacas Huacaya es de 747 hembras y 41 machos reproductores.
-
El esquema de reproductores macho de referencia para el núcleo genético disperso permitirá establecer vínculos genéticos directos que fluctúan de 98.2 a 100%.
LITERATURA CITADA
1. Ahuya CO, Okeyo AM, Mwangi-Njuru, Peacock C. 2005. Development challenges and opportunities in the goat industry: the Kenya experience. Small Ruminant Res 60: 197-206. doi: 10.1016/ j.smallrumres.2005.06.013 [ Links ]
2. Barrantes C. 2012. Caracterización de planteles en los sistemas de producción alpaquera de la Sierra Central. Tesis de Maestría. Lima, Perú: Univ. Nacional Agraria La Molina. 97 p. [ Links ]
3. Cáceres M, Díaz G. 2007. Estructura poblacional y variabilidad fenotípica de alpacas (Vicugna pacos) en el distrito de Paratia, provincia de Lampa-Puno. Arch Latinoam Prod Anim 15(Supl 1): 480-481. [ Links ]
4. Canazas F. 2001. Estructura poblacional y variabilidad fenotípica de alpacas en tres anexos del distrito San Juan de Tarucani en la Reserva Nacional de Salinas y Aguada Blanca. Tesis de Médico Veterinario Zootecnista. Arequipa, Perú: Univ. Católica de Santa María. 78 p. [ Links ]
5. Corredor A. 2015. Relación entre las clases de evaluación visual y el peso de vellón, peso vivo y finura en alpacas Huacaya de Pasco. Tesis de Maestría. Lima, Perú: Univ. Nacional Agraria La Molina. 69 p. [ Links ]
6. Gicheha GM, Kosgey IS, Bebe BO, Kahi AK. 2006. Evaluation of the efficiency of alternative two tier nucleus breeding systems designed to improve meat sheep in Kenya. J Anim Breed Genet 123: 247-257. doi: 10.1111/j.14390388.2006.00598.x [ Links ]
7. Gizaw S, Goshme S, Getachew T, Haile A, Rischkowsky B, van Arendonk J, Valle-Zárate A, et al. 2014a. Feasibility of pedigree recording and genetic selection in village sheep flocks of smallholder farmers. TropAnim Health Pro 46: 809-814. doi: 10.1007/s11250-014-0569-6 [ Links ]
8. Gizaw S, van Arendonk JA, Valle Zárate A, Haile A, Rischkowsky B, Dessie T, Mwai AO. 2014b. Breeding programs for smallholder sheep farming systems: II. Optimization of cooperative village breeding schemes. J Anim Breed Genet 131: 350-357. doi: 10.1111/jbg.12102 [ Links ]
9. Gonzáles C, Fajardo M, Corredor A, Dallos. 2002. Protocolo de análisis de información de finca ganaderas. Fondo Nacional del Ganado. Bogotá, Colombia. [Internet]. Disponible en: http://www.compuagro.net/Tour/Protocolo%20de%20analisis.pdf [ Links ]
10. Haile A, Wurzinger M, Mueller J, Mirkena T, Duguma G, Okeyo A, Sölkner J, et al. 2011. Guidelines for setting up community-based sheep breeding programs in Ethiopia: lessons andexperiences for sheep breedinginlowinput systems. Syria: ICARDA. 37 p. [ Links ]
11. Hanocq E, Tiphine L, Bibé B. 1999. Le point sur la notion de connexion en génétique animale. Inra Prod Anim 12: 101-111. [ Links ]
12. Iñiguez L, Mueller J, Faco O, Wurzinger M, Sölkner J, Rodríguez T, Salinas H. 2013. Limitaciones y sostenibilidad del mejoramiento genético comunitario para pequeños productores en las zonas áridas de Latinoamérica. En: Iñiguez LR (ed). La producción de rumiantes menores en las zonas áridas de Latinoamérica. Brasilia, Brasil: EMBRAPA. p 516-538. [ Links ]
13. Kennedy BW, Trus D. 1993. Considerations on genetic connectedness between management units under an animal model. JAnim Sci 71: 2341-2352. doi: 10.2527/1993.7192341x [ Links ]
14. Kuehn LA, Lewis RM, Notter DR. 2007. Managing the risk of comparing estimated breeding values across flocks or herds through connectedness: a review and application. Genet Sel Evol 39: 225-247. doi: 10.1186/1297-9686-39-3-225 [ Links ]
15. Kuehn LA, Notter DR, Nieuwhof GJ, Lewis RM. 2008. Changes in connectedness over time in alternative sheep sire referencing schemes. J Anim Sci 86: 536-544. doi: 10.2527/jas.20070256 [ Links ]
16. Lewis R, Simm G. 2000. Selection strategies in sire referencing schemes in sheep. Livest Prod Sci 67: 129-141. doi: 10.1016/S0301-6226(00)00182-2 [ Links ]
17. Mathur PK. 2005. Importance of connectedness between herds for effective across herd genetic evaluation. J S China Agric Univ 26: 61-68. [ Links ]
18. Mueller J. 2001. Mejoramiento genético de las majadas patagónicas. En: Borreli P, Oliva G (eds). Ganadería sustentable en la Patagonia Austral. Ed INTA Región Patagonia Sur 10: 209-222. [ Links ]
19. Mueller J. 2013. Experiencias con estructuras genéticas para el mejoramiento de rumiantes menores en las zonas áridas. En: Iñiguez LR (ed). La producción de rumiantes menores en las zonas áridas de Latinoamérica. Brasilia: Ed EMPRADA. p 497-513. [ Links ]
20. Mueller J. 2017. Programas de mejora genética de rumiantes menores basados en comunidades. Arch Latinoam Prod Anim 25: 59-73. [ Links ]
21. Mueller JP, Ansari-Renani HR, Momen SM, Ehsani M, Alipour O, Rischkowsky B. 2015. Implementation of a Cashmere goat breeding program amongst nomads in Southern Iran. Small Ruminant Res 129: 69-76. doi: 10.1016/ j.smallrumres.2015.05.011 [ Links ]
22. Quispe E. 2010. Estimación del progreso genético de seis esquemas de selección en alpacas (Vicugna pacos) Huacaya con tres modelos de evaluación en la región altoandina de Huancavelica. Tesis Doctoral. Lima, Perú: Univ. NacionalAgraria La Molina. 114 p. [ Links ]
23. Roden JA. 1996. A comparison of alternative nucleus breeding systems and a sire referencing scheme for sheep improvement.Anim Sci 62: 265-270. doi: 10.1017/S1357729800014569 [ Links ]
24. Roso V, Schenkel F. 2006. AMC-A computer program to assess the degree of connectedness among contemporary groups. In: Proc 8th World Congress on Genetics Applied to Livestock Production. Belo Horizonte, Minas Gerais, Brazil. [ Links ]
25. Roso VM,Schenkel FS, Miller SP. 2004. Degree of connectedness among groups of centrally tested beef bulls. Can J Anim Sci 84: 37-47. doi: 10.4141/A02-094 [ Links ]
26. Ruiz J, Gutiérrez G, Flores E. 2015. Índices pecuarios de tres unidades de producción de alpacas en Pasco. VII World Congress on South American Camelids. Puno, Perú [ Links ].
27. Simm G, Lewis RM, Collins JE, Nieuwhof GJ. 2001. Use of sire referencing schemes to select for improved carcass composition in sheep. JAnim Sci 79: E255-E259. doi: 10.2527/jas2001.79E-SupplE255x [ Links ]
28. Tarrés J, Fina M, Piedrafita J. 2010. Connectedness amongherds of beef cattle bred under natural service. Genet Sel Evol 42: 1-9. doi: 0.1186/1297-9686-42-6 [ Links ]
Recibido: 1 de febrero de 2018
Aceptado para publicación: 30 de junio de 2018
Fuente financiera: CONCYTEC y Proyecto VLIR -UNALM «Mejorando los sistemas de producción de alpacas en pastizales en la sierra central del Perú»