INTRODUCCIÓN
La Salmonella spp es una de las principales causas de gastroenteritis en humanos y animales a nivel mundial. Estas infecciones se reconocen como zoonosis y son transmitidas por los alimentos (ETA). Existen diferentes reportes en animales como fuente de contagio, pero no hay suficientes estudios acerca de las posibles infecciones producidas (Woodward, 1997; Zamudio et al., 2011).
La Salmonella se elimina por las heces y se disemina en el medio ambiente, donde puede sobrevivir durante un tiempo variable, según las condiciones de temperatura, pH y humedad. Esta bacteria provoca la salmonelosis, la cual puede afectar a todas las especies de animales domésticos. También ataca a los animales silvestres, principalmente aves y reptiles, siendo más susceptibles los jóvenes y las hembras gestantes (Zamudio et al., 2011; OIE, 2018). En especies de quelonios se ha demostrado que forma parte de la flora normal, siendo por este motivo catalogados como portadores asintomáticos. Esta bacteria, al tener un comportamiento de parásito intracelular, es oportunista y puede llegar a generar altos niveles de inmunosupresión en animales bajo condiciones de cautiverio, como en zoológicos y zoocriaderos, o en aquellos mantenidos como mascotas o como parte de tráfico ilegal (Fowler y Miller, 2003).
La tortuga taricaya (Podocnemis unifilis) es criada como fuente de proteína animal, como mascota y para su exhibición en parques naturales y zoológicos (Fachín y Matheus, 2003; Sánchez et al., 2006). Esta tortuga es comercializada y criada en diferentes regiones del país de manera ilegal, sin tomar en cuenta el riesgo zoonótico que representa y la posible diseminación de cepas multidrogorresistentes durante su manipulación, manejo y cría. El objetivo de este estudio fue serotipificar y determinar el perfil de sensibilidad frente a antibacterianos de Salmonella spp en tortugas taricaya, mediante hisopados cloacales de la población total del Parque Zoológico Huachipa y del Parque Natural de Pucallpa.
MATERIALES Y MÉTODOS
Se analizaron 73 muestras de hisopados cloacales de la población total del Parque Zoológico Huachipa (n = 30), Lima, y del Parque Natural de Pucallpa (n = 43), Ucayali, Perú. Los hisopos fueron colocados en tubos Falcon con medio de transporte bacteriano Stuart (Merck) y llevados al Laboratorio de Microbiología de la Universidad Científica del Sur, Lima.
Las muestras se inocularon en caldo tetrationato (Merck) e incubadas a 42 °C por 24 horas. De allí fueron sembradas por agotamiento en medios selectivos: Agar xilosalisina-desoxicolato (XLD) y SalmonellaShigella (SS) e incubados a 37 °C por 24 horas, para proceder a la identificación de las colonias «sospechosas» de Samonella spp utilizando pruebas bioquímicas estándar: fermentación de glucosa, lactosa, producción de ácido de la glucosa (rojo de metilo), producción de ureasa, Vorges Proskauer, fenilalanina, movilidad, malonato y citrato de Simmons, descarboxilación de arginina, lisina y ornitina, oxidación del ácido glucónico y producción de sulfhídrico, siguiendo las recomendaciones descritas en el formato de instrucciones de la técnica de identificación bioquímica bacteriana del Instituto Nacional de Salud (INS, 2013).
La tipificación serológica de las cepas se hizo en el laboratorio de Salud Pública de la Facultad de Medicina de la Universidad Autónoma de México, utilizando el esquema de Kauffmann-White con sueros específicos (DIFCO), basada en la identificación de los antígenos somáticos (O) y flagelares (H) (Popoff et al., 2000). La determinación del patrón de susceptibilidad antibacteriana de Salmonella enterica se hizo según el método de Kirby Bauer, utilizando discos de cloranfenicol (30 µg), enrofloxacina (5 µg), oxitetraciclina (30 µg), gentamicina (10 µg), trimetroprim sulfatrimetropim (25 µg), estreptomicina (10 µg), fosfomicina (5 µg) y furazolidona (100 µg).
RESULTADOS
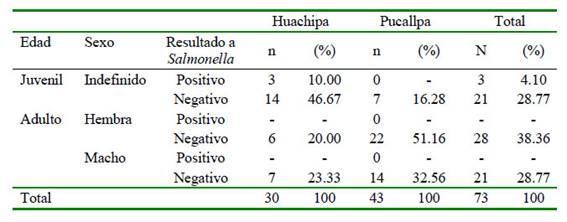
Se aislaron 68 cepas bacterianas de las 73 taricayas muestreadas, resultando tres compatibles a Salmonella por identificación bioquímica, siguiendo la metodología recomendada por el INS. Sin embargo, en 32.87% (24/73) de las taricayas no se pudo determinar el sexo, debido a su inmadurez reproductiva (Cuadro 1).
Cuadro 1 Aislamiento de Salmonella spp de hisopados cloacales en tortugas taricaya del Parque Zoológico Huachipa (n=30), Lima, y del Parque Natural de Pucallpa (n=43), Ucayali, Perú
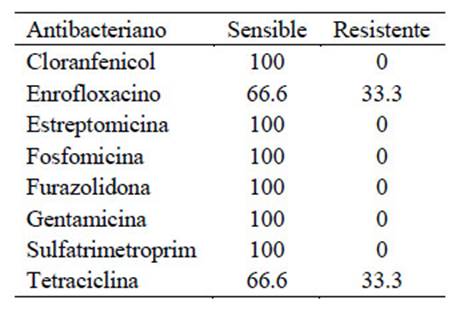
Los resultados del antibiograma realizados con las cepas aisladas de Salmonella spp se presentan en el Cuadro 2, observándose una alta sensibilidad frente a la mayoría de los antibacterianos evaluados. Entre las cepas identificadas se encontró 1.47% de Salmonella spp y 2.94% de Salmonella javiana O9,12:I,Z28:H5 (Cuadro 3), todos ellos de un solo centro de cautiverio y de animales juveniles.
Cuadro 2 Resultado (%) del antibiograma de tres cepas de Salmonella spp aisladas de hisopados cloacales de tortugas taricaya de zoológicos (Perú)
DISCUSIÓN
En estudios previos de prevalencia de infección por Salmonella spp en tortugas taricayas (Podocnemis unifilis) se han reportado diferentes valores entre 4.4 y 25% (Jackson y Jackson, 1971; Duque y Giraldo, 2008; Pachón, 2009). Jackson y Jackson (1971) muestrearon varias especies de tortugas terrestres y acuáticas mantenidas en cautiverio en varios zoológicos de EEUU, entre ellas taricayas, encontrando 12.1% (15/124) de positividad a Salmonella spp. Pachón (2009) trabajando con taricayas en cautiverio en la estación de Biología Tropical de la Universidad Nacional de Colombia (Villavicencio) encontró 4.4% (12/27) de positivos a Salmonella spp, mientras que Toledo (2009) tuvo 13% (3/23) de positivos en tortugas de oreja roja (Trachemys scripta) provenientes de una tienda de mascotas en la ciudad de Valdivia, Chile, lo cual demuestra una variable distribución y frecuencia de presentación, poniendo en evidencia el riesgo para la salud pública.
Se logró aislar tres cepas bacterianas compatibles con Salmonella spp por identificación bioquímica (Cuadro 1), que representan el 4.1% (3/73) de la población en estudio. Este bajo porcentaje de distribución de Salmonella spp puede deberse a la característica de infección y colonización de la bacteria, sumada a la característica intracelular facultativa y eliminación intermitente por las heces que presenta la bacteria (Chiodini, 1982). En este sentido, Burnham et al. (1998) observaron en 10 cultivos de heces de un mismo ejemplar de Iguana iguana, que solo uno de ellos dio positivo para Salmonella spp. En algunos casos se ha requerido un múltiple muestreo para una mejor detección de Salmonella, más aún cuando se decide utilizar la prueba por cultivo bacteriano como método de diagnóstico (Saelinger et al., 2006).
Se ha comprobado que los reptiles que se encuentran en condiciones de cautiverio tienen más posibilidad de eliminación y aislamiento de Salmonella spp que reptiles en estado libre, porque esta bacteria está asociada a factores estresantes (Pachón et al., 2009). Los animales que no están sometidos a estos factores predisponentes albergan cantidades menores de Salmonella que dificultan el aislamiento microbiológico (Burham et al., 1998; Pasmans, 2003; Saelinger et al., 2006). Los tres casos del presente estudio dentro de una población de 73 tortugas muestreadas ocurrieron en solo uno de los dos centros de cautiverio, el cual se caracteriza por presentar ambientes pequeños con alta densidad animal; por otro lado, Duque y Giraldo (2008) encontró la presencia de Salmonella spp en tortugas Podocnemis unifilis que fueron llevadas al centro donde las albergan para ser mantienen en cautiverio, donde el factor estrés pudo afectar la respuesta inmune. Este mismo efecto se observa en el estudio de Toledo (2009), quien reportó Salmonella spp en tortugas acuáticas Trachemys scripta elegans criadas en peceras provenientes del comercio como mascotas o casas donde las mantenían como mascotas.
Los aislamientos de Salmonella spp provinieron de animales juveniles de sexo indefinido (Cuadro 1). La madurez la alcanzan a los 4-5 años, edad en que con base a peso, tamaño, forma de la entrada del caparazón con la cloaca, dirección y tamaño de cola, permite diferenciar los sexos (Sánchez et al., 2006). Esto podría estar relacionado a una mayor oportunidad de colonización de la bacteria y una pobre respuesta inmunológica (Duque y Giraldo, 2008).
En la serotipificación desarrollada por el método de Kauffman-White se identificó en dos de los tres aislados de Salmonella a la cepa Salmonella javiana, con la fórmula antigénica I,Z28:H5, la cual pertenece a la especie Salmonella enterica subespecie enterica serovar javiana (S. javiana) con el serotipo Javiana. Esta posee un antígeno somático O que es un lipopolisacárido (LPS), el cual es un determinante importante de la virulencia de la bacteria (Popoff et al., 2000; Grimont y Weill, 2007). El LPS está compuesto de un lípido interno de una región integrada en la membrana, mientras que la otra región del núcleo O protege al microorganismo mediante el aumento de la distancia con la membrana celular (Brenner et al., 2000). S. javiana ha sido vinculada con alimentos comerciales contaminados y está geográficamente reportada en ciertas áreas de los Estados Unidos desde hace décadas; por ejemplo Alley y Pijoan (1942) reportaron un brote de Salmonella Javiana en 15 pacientes por consumo de queso cottage en Nuevo México; Hedberg et al. (1992), reportaron un brote múltiple de 136 casos de S. Javiana asociados al consumo de queso mozarella; Burnham et al. (1998) aislaron 66 cepas de S. Javiana de alimentos manipulados en un brote de gastroenteritis en Massachusetts; Srikantiah et al. (2001) analizaron 55 casos de S. Javiana, donde 30 tuvieron contactos con anfibios; e Iveson y Bradshaw (1973) encontraron S. Javiana en las muestras fecales de un bebé de 14 meses que estuvo estrechamente asociado con un quokka (Setonix brachyurus) y con una serpiente. Por otro lado, el CDC (2013) realizó un cuadro de los 10 serotipos más importantes respecto a las incidencias de enfermedades transmitidas por alimentos contaminados con Salmonella reportadas por FoodNet (Foodborne Diseases Active Surveillance Network), en el cual S. Javiana se ubicó en un cuarto lugar, con 739 casos durante el 2011 en EEUU. Tomando en cuenta estos estudios y reportes, se considera que la S. Javiana es un riesgo de infección por el consumo de alimentos contaminados o el contacto directo con animales infectados.
El antibiograma arrojó que Salmonella Javiana mostró mayor sensibilidad a fosfomicina y la sulfa-trimetroprim (Cuadro 2), mientras que la tetraciclina y la enrofloxacina resultaron resistentes. Dicha resistencia fue encontrada también por Pachón (2009) quien aisló Salmonella spp (perteneciente a los serogrupos B, C1, C2, D1 y otros atípicos) que resultaron resistentes a tetraciclina. Singh et al. (2011) reportaron cepas de Salmonella Javiana resistentes a la penicilina y la estreptomicina. Así mismo, Chen et al. (2011) realizaron un estudio en reptiles identificando 44 serotipos de Salmonella que resultaron resistentes a ampicilina, cloranfenicol, gentamicina, estreptomicina, sulfa-trimetroprim y tetraciclina; sin embargo no existe un reporte específico de la sensibilidad de la Salmonella Javiana a antibacterianos en tortugas taricayas. Esta variada presentación de resistencia a los diferentes antibióticos es una evidencia de que este proceso es propio de una evolución de las cepas bacterianas en relación con su entorno, y no una respuesta de especies o géneros bacterianos.
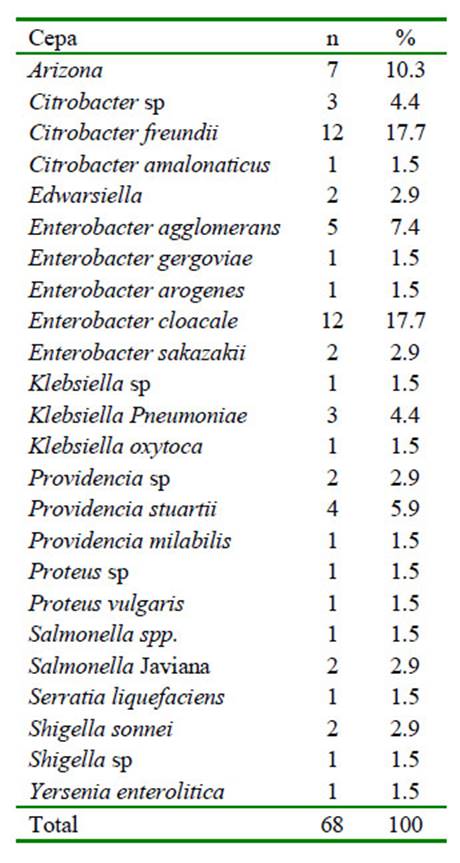
Las tortugas poseen diversidad de enterobacterias en su flora intestinal normal. En el presente estudio, de las 73 muestras se identificaron 22 especies en 68 de ellas (Cuadro 3). Duque y Giraldo (2008), en Medellín Colombia, identificó en tortugas taricayas, aparte de Salmonella spp, Serratia odorífera, Serratia liquefaciens, Providencia rettgeri, Raoutella ornithinolytica, Enterobacter sakazaki, Citrobacter braakii, Kluyvera sp y Enterobacter cloacal. De estas, solo E. cloacae coincide con los resultados obtenidos en el presente estudio, posiblemente por la diferente ubicación de los centros de cautiverio. Por otro lado, Santoro et al. (2006), en el Pacífico Norte de Costa Rica, muestrearon 45 tortugas golfinas (Lepidochelys olivacea) mediante hisopados cloacales y obtuvieron 35 aislados bacterianos, entre ellos Aeromonas spp (13/45), Citrobacter freundi (6/45), Salmonella spp (3/45), Acinetobacter spp (3/45), Alcaligenes fecalis (1/45), Enterobacter faecalis (2/45), Escherichia coli (3/45), Proteus mirabilis (1/45) y Pseudomonas aeruginosa (3/45).












 uBio
uBio