Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. v.19 n.1 Lima abr. 2012
TRABAJOS ORIGINALES
Degradación de tiocianato por hongos aislados de ambientes mineros y evaluación de su capacidad degradativa
The thiocyanate degradation by fungi isolated from mining environments and evaluation of degradative capacity
Susan Medina1, Marisela Torres1, Yerson Durán1, Rina Ramírez2, Juan Herrera1 y Pablo Ramírez1*
1 Laboratorio de Microbiología Molecular y Biotecnología,
2 Laboratorio de Sistemática Molecular y Filogeografía. Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Avenida Venezuela s/n, Lima 1, Perú.
Resumen
El Perú es uno de los principales países en la extracción de minerales como el oro, zinc, plomo y estaño. En la producción de oro se utiliza cianuro, el cual es tóxico para el medio ambiente, y que por reacción natural con el azufre se convierte en tiocianato (-SCN). En el presente trabajo se aislan hongos con capacidad de degradar tiocianato procedentes de aguas y suelos de zonas mineras de Junín y Tumbes. Estas cepas se sometieron a ensayos con concentraciones de tiocianato entre 5 y 600 mM. Asimismo, se evaluó la cinética de degradación de tiocianato en medio Kwon partiendo de 1,2 g.L-1 de KSCN y luego se realizó la identificación morfológica. De un total de 58 mohos aislados, obtuvimos 4 mohos con excelentes capacidades de degradar tiocianato y que pueden ser utilizados en biorremediación. La capacidad degradativa de estas cepas fue en promedio 10,05 mg.L-1 de -SCN con una velocidad de 28,77 mg.L-1.h-1. Dos cepas fueron identificadas fenotípica y molecularmente como Fusarium trincictum usando el marcador ITS del gen rDNA.
Palabras claves: tiocianato, hongos, Fusarium trincictum, degradación, mineria.
Abstract
Peru is one of the principal countries in mining extraction of minerals like gold, zinc, plumb and tin. Cyanide is used in gold production this compound is very toxic especially for environment, by nature reaction with sulfur it converts into thiocyanate (-SCN). In this paper, we isolate -SCN degrading fungus from Junin and Tumbes mining zones. Strains were assayed on 5 to 600 mM -SCN concentration, at least thiocyanate degrading kinetic was assayed at 1.2 g.L-1 of KSCN initial concentration, then the best strains had been morphological, biochemical and molecular identified using ITS RNA molecular marker. From 58 isolated fungus we found 4 unparfait funguses with a great thiocyanate degrading capacity those strains could be used for bioremediation processes. Thiocyanate degrading capacity of these strains was 50 g.L-1 on plate into 72 hours and their average capacity was 10.05 mg.L-1 -SCN with an standard velocity of -SCN degradation of 28.77 mg.L-1.h-1. Two strains were molecular identified as Fusarium trincictum by using ITS rDNA gene.
Keywords: thiocyanate, fungus, Fusarium trincictum, degradation, mining.
Introducción
El tiocianato (-SCN) es un intermediario de la conversión del cianuro por una reacción de adición de azufre (Sorokin et al. 2004). En dosis bajas el tiocianato es menos tóxico que el cianuro, pero es altamente tóxico cuando la dosis exceden los 0.3 g.L-1 (Boucabeille 1994). En peces se ha probado que este compuesto causa alteración en el equilibrio, la presión osmótica y la respiración, los individuos sometidos a concentraciones mayores a 10 mM de tiocianato presentan mareos, alteraciones en las comunicaciones cerebrales, pérdida de equilibrio y orientación. Al igual que el cianuro, el tiocianato es biodegradable por microorganismos.
La existencia de microorganismos con actividad de degradar tiocianato ha sido descrita, entre ellos tenemos a bacterias como Thiobacillus thioparus (Katayama et al. 1992), Pseudomonas y Acinetobacter (Dubey & Holmes 1995) y hongos como Acremonium (Kwon et al. 2002), Trichoderma harzianum (Faull et al. 1994), Fusarium solani, F. oxysporum, Trichoderma polysporum, Scytalidiym thermophilum y Penicilium miczynski (Barclay et al. 1998) que pueden degradar productos derivados del cianuro, entre ellos los tiocianatos. Estos microorganismos han desarrollado mecanismos metabólicos para la degradación de estos compuestos utilizando enzimas como las cianasas que producen como productos finales amonio y dioxido de carbono (Dorr & Knowles 1989), cianuro hidratasas (Clunnes et al. 1993) que conjuntamente con las amidasas convierten el cianuro hasta formato (Dumestre et al. 1997), otros microorganismos utilizan las rodanasas (Ezzi & Lynch 2002; Ramírez et al. 2002), esta reacción puede ser relevante en la desintoxicación del cianuro en los organismos vivos (Ramírez et al. 2002; Bordo & Bork 2002). El tiocianato se halla en los desechos industriales del procesamiento del carbón y de la extracción del oro y la plata conjuntamente con otros compuestos tóxicos como el cianuro libre y cianuro acomplejado a metales (Boucabeille et al. 1994). Pocas investigaciones se han realizado sobre la degradación del -SCN por cultivos puros y en consecuencia, las vías metabólicas no se conocen aun en su totalidad.
Entre los hongos tenemos a Acremonium strictum que degradó 7,4 g.L-1 de -SCN y fue aislado de aguas servidas de la industria del carbón. Los productos finales fueron amonio y sulfato en una relación estequiométrica 1:1 (Kwon et al. 2002). Actualmente se encuentran algunos reportes del uso de microorganismos para el tratamiento de efluentes mineros que contienen cianuro o tiocianatos como en Brasil a nivel de birreactores (Souza-Fagundes et al. 2004). Los cultivos mixtos o consorcios removieron 5 mM de -SCN en 36 horas. En este sentido, el conocimiento de los procesos de destoxificación del -SCN y cianuro resultarán de mucha importancia para mejorar la conservación del ambiente y a su vez la extracción de metales a partir de los minerales que los contienen.
Por otro lado, se están aplicando métodos moleculares en la identificación de los hongos. Éstos se basan en la comparación de las secuencias de nucleótidos de genes conservados, o de regiones conservadas de estos genes en una base de datos, la cual nos permite determinar la especie. En estos eucariontes usualmente se amplifica el gen 28S rRNA LSU que presenta tres regiones altamente polimórficas y también se amplifica las regiones espaciadoras o ITS (Martin et al. 2000; Trout et al. 1997). El presente trabajo tuvo como finalidad identificar hongos aislados de aguas y suelos contaminados con elevadas capacidades de degradación de tiocianato, con potenciales aplicaciones en procesos de biorremediación. Se evaluó la velocidad de degradación del -SCN de 58 mohos y obtuvimos cuatro con excelentes capacidades de degradar -SCN. Asimismo, se ha determinado su relación filogenética mediante el análisis del ITS del gen rDNA.
Material y métodos
Áreas de estudio: se colectó muestras de agua y suelo de zonas influenciadas con desechos mineros ubicadas en dos regiones del Perú. Una de éstas fue en los alrededores del Lago Chinchaycocha, ubicado al Nor-Oeste del Departamento de Junín en la sierra central a 4115 m de altitud. La otra zona fue el río Tumbes, ubicado al nor-oeste del departamento de Tumbes en la costa norte del país a 243 m de altitud.
Aislamiento de cepas nativas degradadoras de tiocianato: Las muestras de agua y suelo se sembraron en los medios líquidos de Kwon (Kwon et al. 2002) con 123 mM de -SCN y M9 con 5 mM de -SCN en agitación rotatoria de 200 rpm. Luego, todas las muestras enriquecidas se sometieron al ensayo de mínima concentración inhibitoria de tiocianato de potasio (MIC) entre 100 a 600 mM, los consorcios se sembraron en medio M9 por 2 días a cada concentración, considerando como positivo aquel consorcio que presente crecimiento superior a la escala 0,5 de McFarland. En el caso del medio Kwon se cultivó por 5 días y se seleccionó las cepas con el mismo criterio. Luego que crecieron a concentraciones de 500 y 600 mM de -SCN se sembraron en agar Kwon y M9 respectivamente. Las colonias seleccionadas fueron repicadas a Medio Saboraud para su mantenimiento.
Evaluación de la capacidad degradativa del -SCN: A las mejores cepas de mohos que crecieron en altas concentraciones de -SCN (diámetro de la colonia >2 cm) se les sometió a los ensayos de cinética de degradación de -SCN vs Tiempo. Este ensayo se realizó en medio líquido Kwon con 1,2 mM de -SCN en agitación constante a 200 rpm y temperatura ambiente durante 90 horas. Se usó un inóculo inicial de 100 mg de peso húmedo de cada cepa. Se tomaron alícuotas de 2 mL del cultivo cada doce horas hasta las 90 horas, estas alícuotas fueron centrifugadas a 13000 x g por tres minutos y en el sobrenadante se cuantificó el -SCN residual. Asimismo, se determinó por cepa, el peso seco a 50 °C durante tres horas para construir una curva de crecimiento vs tiempo.
La cuantificación de la cantidad de -SCN residual en los cultivos se realizó mediante análisis espectrofotométrico con el reactivo de nitrato férrico a DO460 nm (Kelly & Wood 1998). Se preparó un volumen de reacción de 5,5 mL con 50 µL de muestra, este ensayo se realizó con cuatro réplicas para cada tiempo por cepa comenzando de 1,2 g.L-1 de -SCN en el medio (Kwon et al. 2002).
Identificación de cepas: Los mohos aislados fueron identificados mediante ensayos microbiológicos estándares y aplicando el sistema Sacardo (Barnett & Hunter 1998) para hongos imperfectos. Posteriormente, en algunos casos, se usó métodos moleculares.
Para la identificación molecular se extrajo DNA cromosómico, las cepas se cultivaron en medio líquido Saboraud con 123 mM de -SCN a 200 rpm de agitación rotatoria a 28 °C durante dos dias y se utilizó el Wizard genomic DNA Purification kit (Promega®) (Orbegozo et al. 2008) utilizando 200 U de la enzima Liticasa para la ruptura de la pared celular de los hongos.
El DNA genómico fue usado para amplificar por PCR la región ITS del gen 28S rDNA (Orbegozo et al. 2008; Sonnenberg et al. 2007). Los iniciadores usados fueron el ITS1 (5´-TCCGTAGGTGAACCTGCGG-3´) y ITS4 (5´-TCCTCCGCTTATTGATATGC-3´) (White et al. 1990). El programa de PCR fue: desnaturalización a 95 °C durante 3 minutos (1 ciclo), seguido de 30 ciclos a 95 °C por 30 segundos, hibridación a 58 °C por 30 segundos, síntesis a 72 °C por 2 minutos y extensión final a 72 ºC por 5 minutos. La mezcla de reacción fue: 0,5 µL de cada iniciador, 0,5
μL de dNTPs, 5 μL de buffer 5X, 1 μL de MgCl2, 0,125 µL de Taq, 14,875 µL NFW y 2,5 µL de DNA molde. Para visualizar el DNA cromosomal y los productos de PCR se utilizaron geles de agarosa 1% en TAE 0,5X. Las muestras se mezclaron con solución de carga (0,25% azul de bromofenol, 40% sacarosa). Como marcador de tamaño molecular se utilizó 1 kb plus DNA Ladder (Promega®). La tinción se realizó con bromuro de etidio, bandas se visualizaron en un transiluminador de luz UV y la secuenciación se realizó bajo las condiciones de BigDyeTM terminator cycling y la corrida utilizando Automatic Sequencer 3730xl en MACROGEN USA (http://www.macrogenusa.com).Filogenia molecular: Se realizó comparando las secuencias de la región ITS del gen rDNA 28S de los hongos nativos con aquellas disponibles en las base de datos del NCBI Genbank/EMBL/DDBJ y usando el programa de alineamiento local de secuencias BlastN versión 2.0 (Altschul et al. 1997), y Cap3 (Huang & Madan 1999) para obtener la secuencia consenso. Se alinearon las secuencias de nuestras cepas con las secuencias obtenidas del NCBI con el programa Clustal X (Larkin et al. 2007) y se trabajó el alineamiento con el BioEdit 7.0.9 (Hall 2009), y se convirtió a formato Mega en el programa MEGA 4.1 (Kumar et al. 2008). Se construyó la filogenia con el algoritmo Neighboard Joining con boostrap de 1000 replicas, considerando el modelo de sustitución nucleotídica de Kimura 2-p.
Resultados
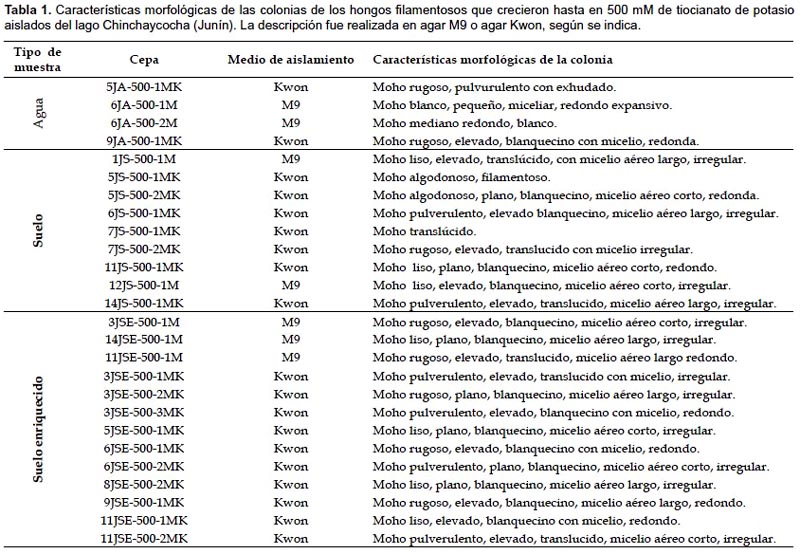
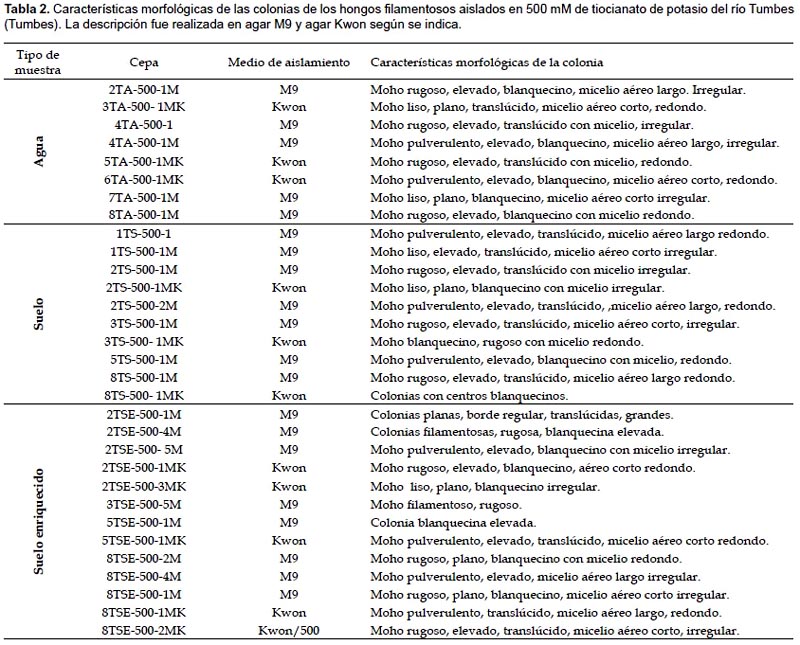
Aislamiento de hongos degradadores de -SCN: En total se procesaron 46 muestras de suelos y aguas que fueron enriquecidas en medio M9 y Kwon (Kwon et al. 2002) en presencia de -SCN. En total se aislaron 58 cepas de mohos, de las cuales 26 fueron de Junín (Tabla 1; 5 de agua, 8 de suelo y 13 de suelo enriquecido) y 32 de Tumbes (Tabla 2; 9 de agua, 9 de suelo y 14 de suelo enriquecido), sólo 12 cepas crecieron a 500 mM de -SCN en placa, éstas cepas fueron de Junín (3 de suelo y 9 de suelo enriquecido). En las Tablas 1 y 2 se describen las características morfológicas de las colonias de las cepas de mohos en medio M9 o Kwon, aislados de Junin y Tumbes según la muestra procesada (agua, suelo o suelo enriquecido).
Evaluación de la capacidad degradativa del -SCN: Se realizó el ensayo de MIC a 58 cepas de mohos. Del total de éstos, sólo 12 crecieron a 500 mM de -SCN en placa: Fusarium sp.1JS-500-1M, Fusarium sp. 1JSE-500-1M, Fusarium sp. 11JSE-500-1MK, Fusarium sp. 14JSE-500-1M, Fusarium sp. 9JSE-500-1MK, Fusarium sp. 11JSE-500-1M, Paecilomyces sp. 5JS-500-2MK, Bispora sp. 7JS-500-1MK, Cladosporium sp. 3JSE-500-3MK, Monillia sp. 11JSE-500-2MK, 9JSE-500-1M, 3JSE-500-1M, 3JSE-500-1MK (Tabla 3). La capacidad de degradativa de -SCN a éstas se les evaluó y cuantificó mediante ensayo espectofotométrico. Las 12 cepas fueron aisladas de muestras procedentes de Junín (3 de suelo y 9 de suelo enriquecido).
Evaluación cuantitativa de la degradación del -SCN. Se cuantificó la cantidad de -SCN residual en los cultivos de 13 cepas que degradan altos niveles de -SCN (500 mM) mediante ensayo espectrofotométrico, los resultados se muestran en la Tabla 4. Las cepas que presentaron los mayores valores de velocidades de degradación de -SCN y un rendimiento mayor a 50% respecto al consumo de -SCN fueron: Paecilomyces sp. 5JS-500-2MK, Fusarium sp. 14JSE-500-1M, y Fusarium sp. 9JSE-500-1MK.
Asimismo, se realizó la cinética de crecimiento de las 13 mejores cepas obteniendo los resultados que se presentan en la Tabla 5. Se consideró como las mejores cepas aquellas que mostraron una producción de biomasa como peso seco mayor a 0,090 g.L-1.h-1,en presencia de -SCN. Entre éstas tuvimos a Fusarium sp. 14JSE-500-1M, Fusarium sp. 1JS-500-1M, Fusarium sp. 9JSE-500-1MK, Paecilomyces sp. 5JS-500-2MK, 1TS-500-2K y 3JSE-500-1MK.
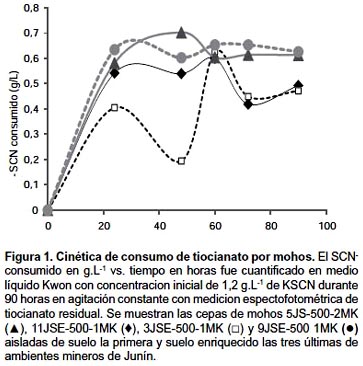
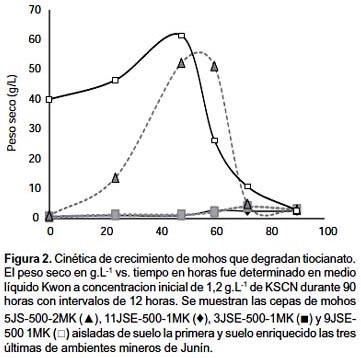
Solamente las cepas que mostraron los mejores rendimientos fueron evaluadas en consumo y crecimiento en -SCN vs tiempo (Fig. 1 y 2). Entre éstas tuvimos a Paecilomyces sp. 5JS-500-2MK y los Fusarium sp. 11JSE-500-1MK, 9JSE-500 1MK y 3JSE-500-1MK. La cepa de Paecilomyces sp. fue aislada de suelo y los Fusarium sp. de suelo enriquecido de ambientes mineros de Junín.
Identificación de cepas nativas degradadoras de -SCN: Se identificaron 9 mohos de acuerdo a sus estructuras reproductivas y vegetativas con la clave Saccardo (Barnett 1998), de las cuales cinco cepas resultaron Fusarium sp., una Paecilomyces sp., dos Cladosporium sp. y Bispora sp. También se identificaron 5 mohos a partir de las estructuras en microcultivo: cuatro Aspergillus sp. y un Trichophyton sp.
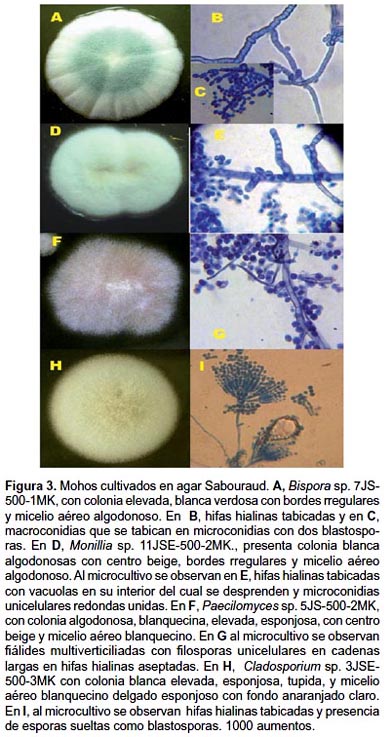
En la Figura 3A se muestra la colonia de Bispora sp. 7JS-500-1MK, elevada, blanca verdosa con bordes irregulares y micelio aéreo algodonoso. Al microcultivo se observan hifas hialinas tabicadas (Fig. 3B) y macroconidias que se tabican en microconidias con dos blastosporas en su interior (Fig. 3C). En tanto que, Monillia sp. 11JSE-500-2MK presentó colonia blanca algodonosa con centro beige, bordes rregulares y micelio aéreo algodonoso (Fig. 3D). Al microcultivo se observan en hifas hialinas tabicadas con vacuolas en su interior del cual se desprenden y microconidias unicelulares redondas unidas (Fig. 3E). Entre tanto, Paecilomyces sp. 5JS-500-2MK, (Fig. 3F) presentó colonias algodonosas, blanquecina, elevada, esponjosa, con centro beige y micelio aéreo blanquecino, que al microcultivo se observa fiálides multiverticiliadas con filosporas unicelulares en cadenas largas en hifas hialinas aseptadas (Fig. 3G). Por otro lado, Cladosporium sp. 3JSE-500-3MK con colonia blanca elevada, esponjosa, tupida, y micelio aéreo blanquecino delgado esponjoso con fondo anaranjado claro (Fig. 3H) con hifas hialinas tabicadas y presencia de esporas sueltas como blastosporas (Fig. 3I).
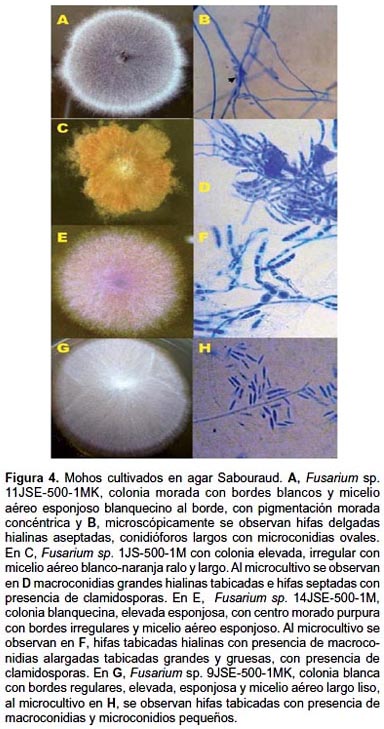
Asimismo, se identificó a la cepa 11JSE-500-1MK como Fusarium sp. (Fig. 4A), ésta presentó una colonia morada con bordes blancos y micelio aéreo esponjoso blanquecino al borde, con pigmentación morada concéntrica, que microscópicamente se observan hifas delgadas hialinas aseptadas, conidióforos largos con microconidias ovales. La flecha indica microconidias pequeñas de bordes romos (Fig. 4B). Además, se identificó un Fusarium sp. 1JS-500-1M con colonia elevada, irregular con micelio aéreo blanco-naranja ralo y largo (Fig. 4C), que al microcultivo mostró macroconidias grandes hialinas tabicadas e hifas septadas con presencia de clamidosporas (Fig. 4D). Además, identificamos a Fusarium sp. 14JSE-500-1M con colonia blanquecina, elevada esponjosa, con centro morado purpura con bordes irregulares y micelio aéreo esponjoso (Fig. 4E). Al microcultivo se observan hifas tabicadas hialinas con presencia de macroconidias alargadas tabicadas grandes y gruesas con presencia de clamidosporas (Fig. 4F). También a Fusarium sp. 9JSE-500-1MK con colonia blanca de bordes regulares, elevada, esponjosa y micelio aéreo largo liso (Fig. 5G), al microcultivo se observan hifas tabicadas con presencia de macroconidias y microconidios pequeños (Fig. 4H).
Filogenia molecular: La región ITS de las cepas 1JS-500-1M y 3JSE-500-1MK fueron sometidas al alineamiento mediante el programa BlastN. Asimismo, se obtuvo el porcentaje de identidad de cada una de ellas con las secuencias existentes en la base de datos. Las cepas 1JS-500-1M y 3JSE-500-1MK presentaron un 100% de identidad con F. trincictum. Se seleccionaron secuencias de la base de datos del NCBI relacionadas a las nuestras y se construyó el árbol filogenético con el marcador ITS de los mohos filamentosos 1JS-500-1M y 3JSE-500-1MK y se ubicaron en el clado robusto de F. trincictum (Fig. 5).

Discusión
Aislamiento de cepas nativas degradadoras de -SCN: Las cepas de mohos aislados de Junín fueron mejores degradadoras de -SCN que las de Tumbes. En comparación con lo propuesto por Baxter y Cumming (2006), Yamasaki et al. (2002) y Patil (2008), el enriquecimiento con -SCN incrementó la actividad degradativa de las cepas, lo que corrobora los buenos resultados al aislar las cepas en medios mínimos enriquecidos con -SCN especialmente al usar el medio M9 a pH neutro utilizado por Patil (2008) para el aislamiento de comunidades bacterianas. En nuestro caso el enriquecimiento fue exitoso ya que el número de mohos aislados a partir de este medio fue similar al obtenido en medio Kwon.
Evaluación de la capacidad degradativa del -SCN: al evaluar la velocidad de degradación de -SCN, la velocidad de crecimiento, el total de -SCN degradado por hora, la cantidad de biomasa producida y la tendencia de su curva de crecimiento, consideramos que las mejores cepas fueron: Paecilomyces sp. 5JS-500-2MK, Fusarium sp. 9JSE-500-1Mk y F. trincictum 3JSE-500-1MK. Estas presentaron velocidades de degradación superiores a 20 mg.L-1.h-1 y un rendimiento superior al 50% respecto al consumo de –SCN (Figs. 1 y 2). En este sentido, éstas cepas podrían ser usadas en procesos de biorremediación ex situ. Por otro lado, el Fusarium sp. 11JSE-500-1MK degrada alrededor del 50% de -SCN con baja producción de biomasa, por lo que podría ser un buen candidato para aplicarla en procesos in situ. ya que en éstos hay que controlar la producción de biomasa.
Asimismo, las cepas de F. trincictum 11JSE-500-1MK y 1JS-500-1M crecieron en medio sólido Kwon con 500 mM de -SCN, esta concentración de -SCN es mayor a las anteriormente reportadas por Souza-Fagundes et al. (2004) de 5 mM y por Kwon et al. (2002) de 123 mM. Nuestros aislados tuvieron capacidad para degradar -SCN muy superior a Thiobacillus thioparus, ésta degrada solo 5 mM de KSCN en 40 horas (Yamasaki et al. 2002), en comparación a 50 g.L-1 en placa en 72 horas y en promedio degradan 10,05 mg.L-1.h-1 con una velocidad promedio 28,77 mg.L-1.h-1 en 60 horas partiendo de una concentración inicial de 1,2 g.L-1, en comparación con las bacterias Pseudomonas sp. y Bacillus sp. que degradan 0,65 g.L-1 en placa en 48 horas a partir de una concentración de 5 mM -SCN (Boucabeille et al. 1994), y con Acremonium strictum (Kwon et al. 2002) que degrada 7,4 g -SCN en 85 horas.
Identificación de cepas nativas degradadoras de tiocianato: En reportes previos, A. strictum (Kwon et al. 2002) tolera altas concentraciones de -SCN, similares resultados obtuvimos con nuestros aislados de Fusarium sp. 11JSE-500-1M, 11JSE-500-1MK, 1JS-500-1M, 14JSE-500-1M, 9JSE-500-1MK, Paecilomyces sp. 5JS-500-2MK, Cladosporium sp. 3JSE-500-3MK. y Bispora sp. 7JS-500-1MK. Algunas de éstas cepas fueron identificadas molecularmente con el marcador ITS como F. trincitum: 1JS-500-1M y 3JSE-500-1MK. Estas presentaron porcentaje de identidad y cobertura de 100% con un E-value de 0,0, confirmando la identificación genérica obtenida por las bases de datos MycoBand (MICOBAND 2008) y DrFungus (DrFungus 2008), entre otras existentes en el NCBI y la reportada por Arias y Piñeros (2008).
Existen pocos casos de reportes de identificación molecular de las especies de Fusarium de ambientes contaminados con cianuro o sus derivados, un Fusarium fue identificado molecularmente en muestras de paramo (Arias & Piñeros 2008). Resulta interesante mencionar que los dos aislados nuestros se encuentran en el clado correspondiente a Fusarium trincictum que indica que la ITS nos permite separar esta especie de otras, sin embargo observamos incluidas otras especies como F. oxysporium, por lo que sería necesario usar otro marcador más polimórfico, como el fragmento del gen del factor de elongación de la traducción (TEF-1
α) reportado por Nietschke et al. (2009).En conclusión se aislaron 58 mohos degradadores de -SCN de muestras de suelos y aguas de los cuales Fusarium sp. 14JSE-500-1M, Fusarium sp. 9JSE-500-1Mk y Paecilomyces sp. 5JS-500-2MK presentaron elevadas capacidades degradativas de -SCN. Se identificaron molecularmente con el marcador ITS a Fusarium trincictum 3JSE-500-1MK y 1JS-500-1M . En general la capacidad degradativa del -SCN de los hongos aislados de aguas y suelos contaminados de Junín y Tumbes fueron de 50 g.L-1 y en promedio 10,05 mg.L-1.h-1 con una velocidad promedio 28,77 mg.L-1.h-1 en 60 h partiendo de una concentración inicial de 1,2 g.L-1, estas cepas consumieron hasta un 52,3% del -SCN inicial.
Agradecimientos
Al Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica (CONCYTEC) por el financiamiento al proyecto mediante contrato No 223-2008-CONCYTEC-OAJ. Esta publicación fue parte de la tesis de título profesional de Bióloga Microbióloga-Parasitóloga de Susan Medina.
Literatura citada
Altschul S.F., T.L. Madden, A.A. Schäffer, J. Zhang, Z. Zhang, W. Miller & D.L. Lipman.
1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucl. Acids Res. 25:3389-3402.Arias E. & P. Piñeros. 2008. Aislamiento e identificación de hongos filamentosos de muestras de suelo de los Paramos de Huasca y Cruz Verde.Tesis para optar el Grado-Pontificia Universidad Javeriana-Facultad de Ciencias. Bogotá-Colombia. 45-57.
Barclay M., A. Hart, C.J. Knowles, J.C.L. Meeussen & V.A. Tett. 1998.
Biodegradation of metal cyanides by mixed and pure cultures of fungi. Enz. Microb. Technol. 22: 223-231.Barnett H. L. & B. B. Hunter. 1998. Illustrated Genera of Imperfect fungi. The american Phytopathological Society 2nd. Ed.
Baxter J. & S.P. Cummings. 2006. The Current and Future Applications of Microorganism in the Bioremediation of Cyanide Contamination. Antonie van Leeuwenhoek, 90: 1-17.
Boucabeille C., A. Bories & P. Ollivier. 1994. Degradation of thiocyanate by a bacterial coculture. Biotechnology Letters 16:425-430.
Bordo D. & P. Bork. 2002. The rhodanese/Cdc25 phosphatase superfamily Sequence–structure–function relations. EMBO reports 3(8):741–746
Cluness M. J., P. D. Turner, E. Clements, D. T. Brown & C. Reilly. 1993. Purification and properties of cyanide hydratase from Fusarium lateritium and analysis od the correspondyng Chy1 gene. J. Gen. Microbiol. 139: 1807-1815
Dorr P. K. & C. J. Knowles. 1989. Cyanide oxygenase and cyanase activities of Pseudomonas fluorecens NCIMB 11746. FEMS Microbiol. Lett. 50: 289-294.
DrFungus. 2008. Mycological database - Image bank. http://www.doctorfungus.org/imageban/.2008.
Dubey S.K. & D.S. Holmes. 1995. Biological cyanide destruction mediated by microorganisms. World J. Microb. Biot. 11:257-265.
Dumestre A., T. Chone, J.M. Portal, M. Gerard & J. Berthelin. 1997. Cyanide degradation under alkaline conditions by a strain of Fusarium solani isolated from contaminated soils. Appl. Environ. Microbiol. 63: 2729-2734.
Ezzi M. & J.M. Lynch. 2002. Cyanide catabolizing enzymes in trichoderma spp. Enz. Microb. Technol. 31:1042-1047.
Faull J. L., K. A. Graeme-Cook & B. L. Pilkington. 1994. Production of an isonitrile antibiotic by an UV-induced mutant of Trichoderma harziamu. Phytochemistry 36: 1273-1276.
Hall T. 2009. BioEdit 7.0.9 Ibis BioSciences, Carlsbad.
Huang X. & A. Madan. 1999. CAP3: A DNA sequence assembly program. Genome Res., 9: 868-877
Katayama Y., Y. Narahara, Y.Inoue, F. Amano, T. Kanagawa & H. Kuraishi. 1992. A thiocyanate hydrolase of Thiobacillus thioparus. A novel enzyme catalyzing the formation of carbonyl sulfide from thiocyanate. J. Biol. Chem. 267:9170-5.
Kelly D.P. & A.P. Wood. 1998. Microbes of the sulfur cycle. En R.S. Burlage, R. Atlas, D. Stahl, G. Geesey y G. Sayler (eds), Techniques in microbial ecology p:31-57. Oxford University Press. USA.v
Kumar S., J. Dudle, M. Nei & K. Tamura. 2008. MEGA: A biologist-centric software for evolutionary analysis of DNA and protein sequences. Briefings in Bioinformatics 9: 299-306.
Kwon H. K., S. H. Woo & J. M. Park. 2002. Thiocyanate degradation by Acremonium strictum and inhibition by secondary toxicants. Biotechnology Letters 24:1347-1351.
Larkin M.A., G. Blackshields, N.P. Brown, R. Chenna, P.A. McGettigan, H. McWilliam, F. Valentin, I.M. Wallace, A. Wilm, R. Lopez, J.D.Thompson, T.J.Gibson & D.G.Higgins. 2007. Clustal W and Clustal X version 2.0. Bioinformatics, 23, 2947-2948.
Martin R.R., Delano J.Y. & C.A. Levesque. 2000. Impacts of molecular diagnostic technologies on plant disease management. Annu. Rev. Phytopathol. 38:207-239
MICOBAND. 2008. Fungal Databases Nomenclature and Special Taxonomic Novelties Submission. Administered by internationl association. http://www.mycobank.org
Nietschke E., M. Nihlgard & M. Varrelmam. 2009. Differentiation of eleven Fusarium spp. Isolated from sugar beet using restriction fragment analysis of a polymerase chain reaction-amplified translation elongation factor 1α gene fragment. Phytopathotogy 99:921-929
Orbegozo J., M. Abanto, R. Garcia & P. Ramirez. 2008. Identificacion molecular de Pichia guillermondi aislada de aguas ácidas de minas del Perú y su resistencia a metales pesados. Rev. Peru. Biol. 15(1): 91-96
Patil Y. 2008 Degradation of thiocyanate from aqueous waste by a mixed Bacterial community. Res. J. chem. Environ. 12(1):69-75
Ramirez P., H. Toledo, N. Guilliani & C.Jerez. 2002. An exported rodhanase-like protein is induced during growth of Thiobacillus ferroxidans in metal sulfides and different sulfur compounds. Appl. Environ. Microbiol.68: 1837-1845
Sonnenberg R., A.W. Nolte & D. Tautz. 2007. An evaluation of LSU rDNA D1-D2 sequences for their use species identification. Front. Zool. 4:6.
Souza-Fagundes E., L. Rosa, N. Gomes, M. Santos, & P. Pimentel. 2004. Thiocyanate degradation by pure and mixed cultures of microorganisms. Brazilian Journal of Microbiology 35:333-336.
Sorokin D.Y., T.P. Tourova, A.M. Lysenko & J.G. Kuenen. 2001. Microbial thiocyanate utilization under highly alkaline conditions. Appl Environ Microbiol 67:528-538.
Yamasaki M., Y. Matsushita, M. Namura, H. Nyunoya & Y. Katayama. 2002. Genetic and immunochemical characterization of thiocyanate-degrading bacteria in lake water. Appl. Environ. Microbiol. 68:942-6.
White, T. J., T. Bruns, S. Lee & J.W. Taylor. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. Pp. 315-322 In: PCR Protocols: A Guide to Methods and Applications, eds. Innis, M. A., D. H. Gelfand, J. J. Sninsky, and T. J. White. Academic Press, Inc., New York.
Autor para correspondencia:
Pablo Ramírez,
E-mail: pramirezr@unmsm.edu.pe
Presentado: 05/01/2012
Aceptado: 05/08/2012
Publicado online: 01/10/2012













