Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de la Sociedad Química del Perú
versión impresa ISSN 1810-634X
Rev. Soc. Quím. Perú vol.79 no.3 Lima jul./set. 2013
TRABAJOS ORIGINALES
Purificación de ADN genómico a partir de hojas, raíces y neumatóforos de Mauritia flexuosa "aguaje"
Genomic DNA purification from leaves, roots and pneumatophores of Mauritia flexuosa "aguaje"
Juan Carlos Castro Gómeza*, Gino Andersson Navarro Jiméneza, Luis Alexander Cerdeira Gutiérreza, Marianela Cobos Ruiza
a* Unidad Especializada de Biotecnología, Centro de Investigación de Recursos Naturales (CIRNA), Universidad Nacional de la Amazonía Peruana (UNAP), Psje. Los Paujiles S/N AAHH Nuevo San Lorenzo, San Juan Bautista, Iquitos, Perú. juanccgomez@yahoo.es
RESUMEN
Mauritia flexuosa es una palmera amazónica muy importante porque brinda servicios ecosistémicos y múltiples beneficios. Sin embargo, los estudios moleculares de la especie son escasos, debido en parte a la falta de un protocolo simple, económico y reproducible para purificar ADN de hojas y otros órganos. Por tal razón, el objetivo fue estandarizar un protocolo para purificar ADN genómico a partir de hojas, raíces y neumatóforos de M. flexuosa. El ADN genómico se purificó con un protocolo modificado (sin el empleo de nitrógeno líquido). La calidad y cantidad del ADN se analizó por métodos estándares. La alta calidad del ADN obtenido de los tres tipos de órganos fue demostrada por espectrofotometría (A260/A230 > 2 y A260 /A280 de 1,77 a 1,81), por hidrólisis del ADN con la enzima Hind III, por los amplicones RAPD sintetizados y la integridad de las bandas de ADN en el análisis electroforético. En conclusión, el protocolo descrito es simple, económico y reproducible y nos permite obtener ADN genómico de alta pureza tanto de las hojas como de las raíces y de los neumatóforos. Por tanto, tal como ha sido demostrado experimentalmente, el ADN purificado es apropiado para realizar estudios moleculares de M. flexuosa.
Palabras clave: Diversidad genética, estudios moleculares, palmera amazónica.
ABSTRACT
Mauritia flexuosa is a very important amazonian palm tree because it offers ecosystemic services and multiple benefits. However, molecular studies of the species are few, partly due to the lack of a simple, economic and reproducible protocol to purify DNA of leaves and other organs. For this reason, the objective was to standardize a protocol to purify genomic DNA from leaves, roots and pneumatophores of M. flexuosa. Genomic DNA was purified with a modified protocol (without the use of liquid nitrogen). DNA quality and quantity were analyzed by standard methods. The high quality of the DNA obtained from the three types of organs was demonstrated by spectrophotometry (A260/A230 > 2 and A260/A280 of 1.77 to 1.81), DNA hydrolysis with Hind III, synthesized RAPD amplicons, and DNA integrity by electrophoresis analysis. In conclusion, the described protocol is simple, economic and reproducible, and it allows us to obtain genomic DNA of high purity from leaves, roots, and pneumatophores. Consequently, these results demonstrated experimentally that purified DNA is appropriate for molecular studies of M. flexuosa.
Key words: genetic diversity, molecular studies, amazonian palm tree.
INTRODUCCIÓN
La amazonía peruana se caracteriza por su gran biodiversidad de recursos fitogenéticos promisorios. Entre éstos, la palmera Mauritia flexuosa L.f. "aguaje" que frecuentemente forma los "aguajales" tiene una gran importancia socio-cultural , nos brinda servicios ambientales2 y sus frutos tienen buena aceptación y una gran demanda por el alto valor nutricional, medicinal e industrial.3-5 Pero, esta creciente demanda está promoviendo la tala selectiva de plantas hembras para la cosecha de los frutos, lo cual podría causar su erosión genética y sobreexplotación. Estos impactos negativos pueden ser minimizados con métodos alternativos de cosecha y la inmediata implementación de programas de reforestación, de domesticación y de mejoramiento genético. Para dar soporte científico a estos programas, es necesario evaluar la diversidad genética, genotipificar ecotipos importantes, identificar y clonar marcadores gene-específicos asociados con el sexo y con resistencia a factores bióticos y abióticos adversos y realizar estudios del genoma.
Sin embargo, para realizar los estudios de genética molecular mencionados, debemos disponer de protocolos rápidos, simples, económicos y reproducibles para purificar el ADN genómico a partir de diferentes tipos de tejidos. Como no existe un protocolo universal para purificar el ADN de las plantas con las características mencionadas, se ha reportado varios protocolos de purificación de ADN especie-específicos a partir de hojas de varias especies vegetales.6-10 Pero estos protocolos no siempre son útiles para purificar ADN de alta calidad de hojas o de distintos tejidos de otras especies vegetales, debido a la diversidad de metabolitos que interfieren con el proceso de purificación y en los análisis moleculares posteriores.11-13
Probablemente, por estas dificultades y debido a la rápida oxidación de los tejidos, no hay protocolos publicados sobre la purificación del ADN genómico de M. flexuosa. Aunque hay reportes sobre estudios moleculares de la especie en base al ADN obtenido de las hojas, pero no dan detalles metodológicos ni muestran evidencias de la cantidad ni calidad del material genético obtenido.14,15 Debido a esta falta de información científica y a las dificultades de colectar hojas de plantas adultas, por su gran altura (15 a 30 m), pero siendo factible purificar el ADN de otros tejidos accesibles como las raíces y los neumatóforos, se ha realizado esta investigación cuyo objetivo principal fue estandarizar un protocolo adecuado.
PARTE EXPERIMENTAL
Colecta de material botánico
Las hojas, raíces y neumatóforos fueron colectadas en el fundo "Leonardo", ubicado en las coordenadas 03°46?50,5" LS y 73°18?25,3"LE a la altura del km 1,5 de la carretera a Santa Clara del Distrito de San Juan Bautista-Iquitos. Se seleccionó aleatoriamente 12 plantas: 4 plantas de 6 meses a 2 años, 4 plantas >2 hasta 4 años y 4 plantas >4 años. De cada planta, se obtuvo segmentos de 3 cm de hojas, raíces y neumatóforos (figura 1). Éstos se lavaron con agua destilada estéril y solución antioxidante (ácido L-ascórbico 150 µg/mL y ácido cítrico 100 µg/mL). Las muestras fueron transportadas en solución antioxidante a 4°C y protegidas de la luz a la Unidad Especializada de Biotecnología-CIRNA. En el laboratorio, de las hojas se obtuvo segmentos de ~25 mm2; de las raíces y de los neumatóforos se extrajo porciones del ápice de ~15 mg y ~4 mg, respectivamente. Todo este proceso se realizó con el material botánico embebido en solución antioxidante fría (~4°C).

Purificación del ADN genómico
Se realizó en base a Porebski et al.11 con modificaciones. Estas modificaciones fueron: a) exclusión de la etapa de pulverización del material vegetal con nitrógeno líquido, porque este insumo no está disponible en la amazonía, b) uso de arena estéril para triturar las muestras, c) empleo de alta proporción del tampón de extracción: muestra (10:1) y d) reducción del tiempo de purificación a sólo una hora. Para estandarizar el protocolo se realizó diversos experimentos con la cantidad de muestra botánica, la cantidad de arena, el tiempo de trituración, la proporción tampón de extracción:muestra y el volumen de cloroformo:alcohol isoamílico. Finalmente, el protocolo estandarizado consta de los siguientes pasos:
-
Poner en un mortero 10 a 90 mg de la muestra, 100 mg de arena estéril y 10 volúmenes del tampón de extracción (Tris-HCl 300 mM pH 8,0; NaCl 2,0 M, EDTA25 mM, CTAB 2%, polivinilpirrolidona 40000 2%, albúmina de suero bovino 0,3%) y β-mercaptoetanol 2%, triturar por 1 a 2 minutos.
-
Transferir el homogenizado a microtubos estériles de 1,5 mL e incubar a 75°C por 10 minutos, homogenizar por inversión cada 2 minutos.
-
Añadir igual volumen de cloroformo:alcohol isoamílico, homogenizar en el vortex por 10 segundos y centrifugar a 14000 x g a 4°C por 5 minutos.
-
Transferir el sobrenadante a un microtubo, repetir el paso 3) y transferir el sobrenadante a un nuevo microtubo.
-
Añadir igual volumen de alcohol isopropílico helado, homogenizar suavemente por inversión e incubar a -20°C por 10 minutos.
-
Centrifugar a 10000 x g a 4°C por 5 minutos, descartar el sobrenadante; y el ADN precipitado lavar dos veces con 250 µLde etanol de 70°C frío.
-
Centrifugar a 5000 x g a 4°C por 5 minutos, descartar el sobrenadante, secar a 40°C por 10 minutos y resuspender con 100 µL de tampón TE (Tris-HCl 10 mM pH 8,0; EDTA1 mM) y almacenar a -20°C.
Análisis espectrofotométrico
Para el análisis espectrofotométrico en las tres etapas de purificación (E) se tomó alícuotas de 50 µL después de los pasos 1 y 2 (E1), pasos 3 y 4 (E2) y después de los pasos 5, 6 y 7 (E3) del proceso de purificación. De estas muestras se midió su absorbancia desde 190 hasta 1100 nm. Para determinar la calidad y cantidad del ADN purificado se midió absorbancias de las muestras a 230, 260 y 280 nm, con los que se determinó los ratios de calidad (A260/A230 y A260/A280). La cantidad de ADN se determinó de acuerdo a Sambrook et al.16
Análisis electroforético
Una mezcla de ADN genómico purificado de los tres grupos etáreos de hojas, raíces o neumatóforos se sembraron en gel de agarosa al 1% y se realizó la electroforesis con TBE 0,5X (Tris-borato 45 mM pH 8,0; EDTA 1 mM) a 80 V por una hora. El gel se tiñó con bromuro de etidio (0,1 mg/mL) y las bandas de ADN se observaron en un transiluminador UV.
Hidrólisis del ADN con endonucleasa
Una mezcla de 20 µg de ADN genómico obtenido de los tres grupos etáreos de hojas, raíces o neumatóforos se incubó con 50 U de la enzima de restricción Hind III a 37ºC por 24 horas. Los fragmentos de ADN fueron resueltos por electroforesis en gel de agarosa al 1% a 80 V por una hora. Las bandas de ADN se observaron con luz UV después de tratar el gel con bromuro de etidio (0,1 mg/mL).
Amplificación con cebadores aleatorios
Los componentes para la reacción en cadena de la polimerasa fueron: tampón 1X, MgCl2 1,87 mM, dNTPs 0,5 mM, cebadores aleatorios A (5'-GGGTAACGCC-3'), B (5'-CCGCAGCCAA-3') ó C (5'-GCGATCCCCA-3') 0,25 µM, Amplitaq Gold 0,025 U/µL, ADN genómico 40 ng y agua ultrapura para un volumen final de 20 µL. Las amplificaciones se realizaron en un termociclador Eppendorf ep gradient con las siguientes condiciones: un ciclo de 95°C por 4 minutos, 40 ciclos de 95°C por 1 minuto, 40°C por 1 minuto y 72°C por 1 minuto y una extensión final a 72°C por 10 minutos. Los amplicones se resolvieron por electroforesis a 80 V por 3 h en gel de poliacrilamida al 8%. Las bandas de ADN se revelaron por tinción argéntica según Bassam y Greshold17.
Análisis estadístico
Se determinó el promedio y desviación estándar de la cantidad de ADN aislado a partir de los tres tipos de tejidos y de diferentes cantidades de muestras (10 a 90 mg). También, para determinar diferencias en la cantidad promedio de ADN genómico obtenido se realizó el ANVA con test HSD de Tukey con un nivel de confianza del 95% y considerando diferencias estadísticas significativas cuando p < 0,05. El coeficiente de correlación de Pearson se empleó para analizar este parámetro entre la cantidad de muestra botánica empleada y la cantidad de ADN genómico obtenido. Todos los análisis se hicieron con el programa IBM SPSS Statistics 21.
RESULTADOS Y DISCUSIÓN
Se ha purificado ADN genómico a partir de hojas, raíces y neumatóforos de M. flexuosa, el cual se caracteriza por su alta calidad y cantidad apropiadas para estudios moleculares de esta especie. La alta calidad se evidencia por los resultados de los análisis espectrofotométricos durante la purificación, que muestran desaparición gradual de los picos de absorbancia de sustancias contaminantes. Así, en la última etapa de purificación (E3) se observó un único pico de máxima absorbancia a 260 nm, el cual corresponde al ADN genómico puro (figura 2). Asimismo, en la tabla 1 se muestra que los ratios A260/A230 son mayores de 2, lo cual nos indica que el ADN purificado no está contaminado con polisacáridos ni polifenoles, porque ratios bajos sugieren contaminación con estos compuestos.11,18 También, los ratios A260/A280 adecuados nos indican que el ADN no está contaminado con proteínas ni está degradado.10,16

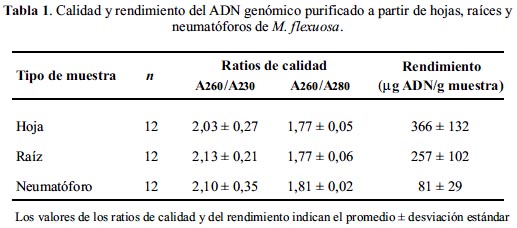
Adicionalmente, otras evidencias experimentales nos indican la alta pureza del ADN genómico obtenido (figura 3). Primero, al hidrolizar el ADN genómico con la enzima Hind III, nos revela ausencia de inhibidores para este tipo de enzimas (figura 3,1). Segundo, al poder sintetizar múltiples amplicones con los tres cebadores (figura 3,2), evidenciados por la alta intensidad de las bandas de ADN, nos muestra que no contiene inhibidores para la ADN polimerasa taq. Porque varias sustancias que coprecipitan con el ADN durante la purificación interfieren con los análisis moleculares mencionados.11-13
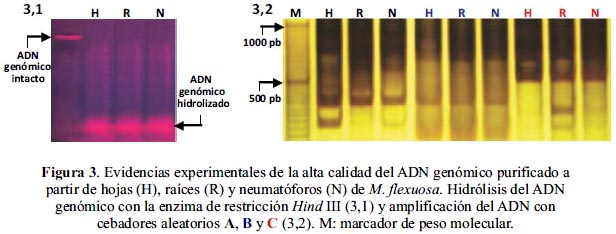
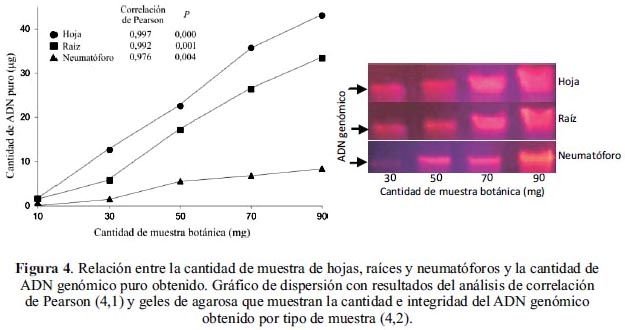
En cuanto a la cantidad de ADN genómico obtenido los resultados indican que dependió del tipo (tabla 1) y cantidad de muestra botánica (figura 4). De tal forma, que de acuerdo al rendimiento promedio obtenido (µg ADN/g tejido) observamos que hoja > raíz > neumatóforo. Así, de las hojas se obtuvo 1,4 veces más ADN que de las raíces y 4,5 veces más que de los neumatóforos. También, de las raíces se obtuvo 3,2 veces más ADN que de los neumatóforos, siendo estas diferencias estadísticamente significativas (F = 25,7, gl = 2, p < 0,05). Adicionalmente, la reproducibilidad del protocolo se muestra con la alta correlación de Pearson (> 0,97) entre la cantidad de tejido utilizado (hoja, raíz o neumatóforo) y la cantidad de ADN puro obtenido (figura 4).
Las diferencias en la cantidad de ADN genómico obtenido por tipo de órgano puede deberse a sus diferencias histológicas e histoquímicas. Así, el ADN obtenido de las hojas procede de diferentes células, porque las hojas de palmeras tienen células que forman la epidermis, la hipodermis, el mesófilo y los tejidos del floema y xilema. Todas las células que constituyen estos tejidos son nucleadas y tienen paredes celulares finas (excepto las células del xilema).19
Además, las células del mesófilo presentan cloroplastos y al ser lisadas liberan las clorofilas. Estos compuestos fueron detectados en la primera etapa de purificación del ADN de hojas, pero no de los demás órganos (figura 2). En segundo lugar, el ADN obtenido de las raíces deriva principalmente de los meristemos, los cuales se encuentran en la parte interna del ápice, ligeramente por encima de la cofia.19 Finalmente, el ADN genómico obtenido de los neumatóforos también provienen principalmente de los meristemos apicales. Estos órganos están muy desarrollados en suelos húmedos y gran parte de la estructura está formada por células que constituyen el aerénquima, caracterizados por grandes espacios intercelulares para facilitar el intercambio de gases;19 consecuentemente, en estos órganos la densidad celular por área o cantidad de material botánico es mucho menor que en las hojas o en las raíces. Lo que nos permite explicar la menor cantidad de ADN obtenido de estos órganos. Por tanto, las particulares características histológicas e histoquímicas de los tres tipos de órganos empleados explica las diferencias en la cantidad de ADN genómico purificado.
Al comparar el rendimiento de nuestro protocolo con los reportes de purificación de ADN a partir de hojas de otras palmeras encontramos algunas diferencias. En este estudio obtuvimos en promedio > 350 µg ADN/g de hoja (tabla 1). Mientras, que de Cocos nucifera obtienen 300 µg de ADN/g de hoja20 y de Phoenix dactilyfera sólo de 30 a 35 µg de ADN/g de hoja.21
Estas variaciones se deben a diferencias en varios aspectos: a) edad, y composición química de los tejidos foliares; b) la anatomía e histología de las hojas y c) diferencias en los protocolos empleados, particularmente en los tipos y concentraciones de los componentes del tampón de extracción, la proporción tampón: muestra, entre otros.
La investigación realizada podría tener algunas limitaciones. Porque no se ha evaluado la hidrólisis del ADN con otras enzimas de restricción y no se ha obtenido el ADN de plantas procedentes de distintas zonas geográficas, dado que es posible que diversos metabolitos inhibitorios pueden ser sintetizados en respuesta a factores ambientales variables. Finalmente, no se ha probado si el protocolo descrito es apropiado para purificar ADN de otros órganos de M. flexuosa.
Con este trabajo se ha superado una primera brecha, que es el de contar con un protocolo simple, económico y reproducible para purificar ADN de M. flexuosa. Aún queda por hacer mucha investigación a nivel molecular de esta especie. Porque hasta la fecha no se ha identificado marcadores moleculares asociados con el sexo. Si esto se logra, se sentarán las bases para que los programas de cultivo y repoblamiento de la especie sean exitosos. Asimismo, falta identificar marcadores genéticos asociados con resistencia al estrés causado por factores abióticos y bióticos y aquellos marcadores genéticos que determinan el tamaño, forma, color y composición nutricional de los frutos de M. flexuosa. También es necesario realizar estudios de diversidad genética de la especie e iniciar proyectos de secuenciamiento del genoma y transcriptoma. Si logramos estos avances, daremos soporte científico al establecimiento de programas de mejoramiento genético asistido por marcadores moleculares, que aceleraría significativamente la obtención de variedades mejoradas de la especie.
CONCLUSIONES
El protocolo descrito es simple, económico y reproducible y nos permite obtener ADN genómico de alta pureza tanto de las hojas como de las raíces y de los neumatóforos. Por tanto, tal como ha sido demostrado experimentalmente, el ADN purificado es apropiado para realizar estudios moleculares de M. flexuosa.
AGRADECIMIENTOS
Alos Dres. Jorge L. Marapara y Lastenia Ruiz Mesía por brindarnos las facilidades de acceso a las instalaciones y uso de equipos de la Unidad Especializada de Biotecnología del Centro de Investigaciones de la Amazonía-UNAP. Un especial agradecimiento al Dr. J. Dylan Maddox por la revisión crítica del Abstract.
BIBLIOGRAFÍA
1. Gilmore MP, Endress BA, Horn CM. The socio-cultural importance of Mauritia flexuosa palm swamps (aguajales) and implications for multi-use management in two Maijuna communities of the Peruvian Amazon. J Ethnobiol Ethnomed 2013;9:29.
2. Freitas L, Otárola E, del Castillo D, Linares C, Martínez P, Malca GA. Servicios ambientales de almacenamiento y secuestro de carbono del ecosistema aguajal en la Reserva Nacional Pacaya Samiria, Loreto-Perú. IIAP. Documento Técnico N° 29. 2006; p. 1-62.
3. Villachica H. Frutales y Hortalizas Promisorias de la Amazonía Peruana. En Tratado de Cooperación Amazónica. Iquitos-Perú. Secretaria Pro tempore. 1996; p.76-83.
4. Rojas R, Salazar C, Llerena C, Rengifo C, Ojanama J, Muñoz V, et al. Industrialización primaria del aguaje (Mauritia flexuosa L.f.) en Iquitos (Perú). Folia Amazónica 2001;12(1-2):107-121.
5. Ferreira BS, de Almeida CG, Faza LP, de Almeida A, Diniz CG, da Silva VL, et al. Comparative properties of Amazonian oils obtained by different extraction methods. Molecules. 2011;16(7):5875-1585.
6. Doyle JJ, and Doyle JL. Arapid DNAisolation procedure for small quantities of fresh leaf tissue. Phytochem. Bull. 1987;19:11-15.
7. Wang X, Wang Z, and Zou Y. An improved procedure for the isolation of nuclear DNA from leaves of wild grapevine dried with silica gel. Plant Mol. Biol. Rep. 1996;14(4):1-5.
8. Zhang J, and Stewart J. Economical and rapid method for extracting cotton genomic DNA. The J. Cotton Sci. 2000;4:193-201.
9. Sharma AD, Gill PK, and Singh P. DNA isolation from dry and fresh samples of polysaccharide-rich plants. Plant Mol. Biol. Rep. 2002;20:415a-415f.
10. Castro JC, Cobos M, Ramírez R, Imán SA. Aislamiento de ADN genómico de Myrciaria dubia (HBK) "camu camu" apropiado para análisis moleculares. Ciencia Amazónica 2012;2(1):7-15.
11. Porebski S, Bailey L, and Baum B. Modification of a CTAB DNAextraction protocol for plants containing high polysaccharide and polyphenol components. Plant Mol. Biol. Rep. 1997;15: 8-15.
12. Merlo DJ, and Kemp D. Effect of polysaccharides on kinetics of DNA. Plant Physiol. 1976;58:1522-1526.
13. Pandey R, Adams R, and Flournoy L. Inhibition of random amplified polymorphic DNAs (RAPDs) by plant polysaccharides. Plant Mol. Biol. Rep. 1996;14(1):17–22.
14. Picanço LR, Gomes MT, da Silva JL, Silva W, Costa P and Serrão LA. Genetic diversity in natural populations of Buriti (Mauritia flexuosa L. f.). CBAB, Crop Breed. Appl. Biotechnol. 2011;11: 216-223.
15. Menezes EV, Souto WF, Ciampi AY, Azevedo VC, Valério HM, Pimenta MA. Development and characterization of DNA microsatellite primers for buriti (Mauritia flexuosa L.f.). Genet Mol Res. 2012;11(4):4058-4062.
16. Sambrook J, Fritsch E, and Maniatis T. Molecular Cloning: A Laboratory Manual. First Edition. Ed. Coll Spring Harbor Laboratory publications. New York. USA. 1989; p. 545.
17. Bassam BJ, Gresshoff PM. Silver staining DNA in polyacrylamide gels. Nat. Protoc. 2007;2(11):2649-2654.
18. Loomis MD. Overcoming problems of phenolics and quinones in the isolation of plant enzymes and organelles. Methods Enzymol 1974;31:528–544.
19. Tomlinson PB. The Structural Biology of Palms. Oxford Science Publication. New York. U.S.A. 1990.
20. Upadhyay A, Jose J, Manimekalai R, and Parthasarathy V. Molecular analysis of phylogenetic relationship among coconut accessions. Man. Plant Gen. Div. 2002;1:1-6.
21. Quenzar B, Hartmann C, Rode A, and Benslimane A. Date palm DNA mini-preparation without liquid nitrogen. Plant Mol. Biol. Rep. 1998;16(3):263-269.
Recibido el 16-07-2013
Aprobado el 24-07-2013













