INTRODUCCIÓN
La resistencia bacteriana a los antimicrobianos tiene una base genética que puede ser intrínseca y/o extrínseca del microorganismo. En los últimos años, ha tomado importancia por el aumento de la mortalidad intrahospitalaria, en gran medida, debido al uso irracional de los antimicrobianos, por lo que ha sido declarada una de las 10 principales amenazas en contra de la salud pública 1. También, se suman costos en salud, debido a la utilización de antibióticos caros, mayor estancia hospitalaria y una repercusión social con respecto al impacto salarial y productivo del paciente afectado 2.
Se conoce que el espectro de resistencia antimicrobiana es variable, por lo cual se define como microorganismo con multirresistencia a los medicamentos (MDR) cuando no presenta sensibilidad al menos a un agente en tres o más categorías antimicrobianas; microorganismo extremadamente resistente a los medicamentos (XDR), cuando no presenta sensibilidad a al menos un agente en todas las familias con excepción de una o dos de ellas; es decir, los aislamientos bacterianos siguen siendo sensibles solo a una o dos familias; microorganismo pandroga-resistente (PDR), cuando no presenta sensibilidad a todos los agentes en todas las categorías de antimicrobianos; es decir, ningún agente probado como sensible para ese organismo 3.
El mapa microbiológico es un documento que proporciona información acerca de los microorganismos que circulan en los diferentes servicios de un hospital, evalúa la sensibilidad de estos microorganismo frente a los antimicrobianos en uso, contribuye, de esta manera, al inicio de un efectivo y oportuno tratamiento empírico en los pacientes que presentan infecciones, a la disminución de la estancia hospitalaria y también a la reducción de los costos que implica la atención médica 4.
En algunos hospitales del Perú: En Lima, Arequipa o Trujillo se han venido elaborando mapas microbiológicos, pero en muchas otras regiones del país, no se cuenta con dicha información. Es propicio mencionar que luego de la pandemia por la COVID-19, se desarrolló, en gran medida, un uso irracional de antimicrobianos para el tratamiento de neumonía por SARS-CoV-2, lo que favoreció, significativamente, al avance de la resistencia antimicrobiana 2,5. Debido a la falta un mapa microbiológico local y al elevado reporte de infecciones por patógenos multirresistentes, el presente estudio tiene por objetivo identificar los microorganismos y su perfil de resistencia antimicrobiana en las infecciones desarrolladas en los servicios clínicos y quirúrgicos.
METODOLOGÍA
Estudio descriptivo transversal, el cual evaluó los resultados de cultivos y antibiogramas procedente muestras clínicas de pacientes con sospecha de infección internados entre enero y diciembre de 2021 en un hospital de la selva peruana, el cual es uno de los principales nosocomios en la región Loreto y cuenta con 191 camas hospitalarias, 56 del servicio de Emergencia: Adultos 42, pediátricas 10, gineco-obstétricas 4; además, cuenta con 21 camas de la Unidad De Cuidados Intensivos (UCI): 11 adulto, 4 neonatal y 6 pediátricas; asimismo, tiene 14 camas en el servicio de Pediatría; 26 camas, en Cirugía; 19, en Gineco-obstetricia; 14, en el servicio de Traumatología; 29, en Medicina Interna y 12 camas en Infectología; la población asignada es de 240 mil personas. Para la identificación microbiológica, se usó VITEK ® 2 (bioMérieux), un sistema automatizado de evaluación espectrofotométrica del crecimiento de los microorganismos (bacterias y hongos) y susceptibilidad a los antimicrobianos, según los puntos de corte actualizados de las guías del Instituto de Estándares Clínicos y de Laboratorio (CLSI). Se identificó el tipo de microorganismo, sensibilidad antimicrobiana, fuente de aislamiento, edad del paciente y servicio de hospitalización. El estudio fue aprobado por el Comité de Ética hospitalario. Los datos fueron recolectados en una hoja de cálculo Excel v.2016, codificados, tabulados y analizados con SPSS Statistics 25.00; se realizó un análisis estadístico descriptivo, medidas de tendencia central y distribución de frecuencias.
RESULTADOS
Se identificaron 477 cultivos positivos correspondientes a 453 pacientes; 262 (54.9%) cultivos procedieron de muestras de secreción bronquial, 168 (35.2 %) hemocultivos, 31 (6.5 %) urocultivos y cinco o menos en muestras de secreción de herida, dren laminar, punta de catéter venoso central, secreción traqueal, líquido cefalorraquídeo, líquido pleural y secreción peritoneal. La edad de los pacientes varió entre un día de nacido y 88 años (mediana de 56 años). Los microorganismos aislados con más regularidad fueron Acinetobacter baumannii complex y Klebsiella pneumoniae ssp., fueron encontrados con mayor frecuencia en cultivo de secreción bronquial, en los servicios de emergencias y las unidades de cuidados intensivos (Figura 1).
En el grupo de bacterias gramnegativas Acinetobacter baumannii complex, presentó sensibilidad a tigeciclina y colistina en un 53 % y 97 %, así como resistencia a meropenem e imipenem en un 97 % y 98 % respectivamente, siendo XDR en un 88.5%. Con respecto a Klebsiella pneumoniae ssp., el 58 % fue BLEE (+), con sensibilidad a ceftazidima en 10 % y resistencia en un 100 % a ceftriaxona, además de resistencia a meropenem e imipenem en un 44 % y 34 %, siendo MDR en un 56.3 %. En el caso de Pseudomonas aeruginosa, presentó sensibilidad a colistina y amikacina en un 96 % y 70 %, así como resistencia a meropenem e imipenem en un 85 % y 82 %, siendo XDR en 54.7 %. Con respecto a Escherichia coli, el 89% fue BLEE (+) con sensibilidad a ceftazidima y ceftriaxona en 6 % y 13 % respectivamente (Tabla 1).
Con respecto a las bacterias grampositivas, todos procedieron de hemocultivos; Staphylococcus epidermidis fue el más frecuente, seguido por Staphylococcus haemolyticus, siendo meticilino-resistentes en un 88 % y 100 % y no se reportó resistencia alguna a vancomicina para ambos microorganismos. Con respecto a Staphylococcus aureus, fueron meticilino-resistentes en un 67 % no reportando resistencia a vancomicina y con respecto a Entereococcus faecium, la única cepa aislada fue resistente a ampicilina, pero sensible a vancomicina (Tabla 2).
Con relación a hongos, fueron aislados en un total de 43 muestras, de los cuales sólo se realizaron en 6 de estos cultivos el perfil de sensibilidad antimicótica respectiva; se encontró en el hemocultivo de un paciente de 66 años, la presencia de Cándida parapsilosis resistente a fluconazol y voriconazol (Tabla 3).
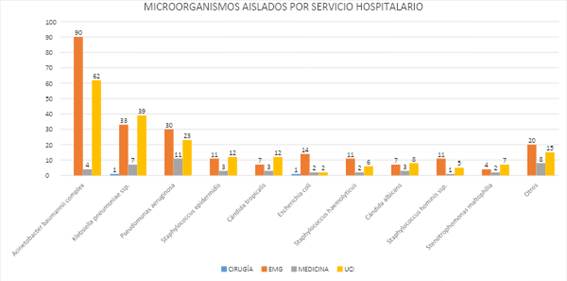
Figura 1 Microorganismo aislado según servicio hospitalario en un nosocomio general de la selva peruana 2021
Tabla 1 Sensibilidad antimicrobiana de bacterias gramnegativas aisladas en un hospital general de la selva peruana 2021
| BACTERIAS GRAMNEGATIVAS | AISLADOS | SENSIBILIDAD ANTIMICROBIANA | |||||||||||||||||||||||||
| BLEE (+) | AMPICILINA | AMOXICILINA/ ÁCIDO CLAVULÁNICO | AMPICILINA/ SULBACTAM | PIPERACILINA/ TAZOBACTAM | CEFALOTINA | CEFAZOLINA | CEFUROXIMA | CEFOTAXIMA | CEFTAZIDIMA | CEFTRIAXONA | CEFEPIMA | ERTAPENEM | IMIPENEM | MEROPENEM | AMIKACINA | GENTAMICINA | TOBRAMICINA | CIPROFLOXACINO | LEVOFLOXACINO | NORFLOXACINO | FOSFOMICINA | TIGECICLINA | COLISTINA | NITROFURANTOÍNA | TRIMETROPIM/ SULFAMETOXAZOL | ||
| Acinetobacter baumannii complex | 156 | N | N | N | 2% | N | N | N | N | N | N | N | 1% | N | 2% | 3% | 36% | 9% | 0% | 1% | 0% | N | N | 53% | 97% | N | 1% |
| Klebsiella pneumoniae ssp. | 80 | 58% | N | 8% | 6% | 34% | N | 1% | 11% | 13% | 10% | 0% | 11% | 67% | 66% | 56% | 99% | 35% | 7% | 14% | 0% | N | N | 75% | 87% | 6% | 18% |
| Pseudomonas aeruginosa | 64 | N | N | N | N | 22% | N | N | N | N | 48% | N | 51% | N | 18% | 15% | 70% | 50% | 33% | 25% | 25% | 0% | N | N | 96% | N | N |
| Escherichia coli | 19 | 89% | 0% | 27% | 24% | 76% | 0% | 5% | 0% | 0% | 6% | 13% | 5% | 95% | 94% | 100% | 89% | 47% | 43% | 5% | 0% | 0% | 100% | 100% | 100% | 88% | 33% |
| Stenotrophomonas maltophilia | 13 | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | 100% | N | N | N | N | N | 92% |
| Serratia marcescens | 7 | N | N | N | 0% | 43% | N | N | N | 14% | 14% | N | 29% | 14% | 14% | 29% | 71% | 57% | N | 14% | N | N | N | 43% | N | N | 14% |
| Enterobacter aerogenes | 4 | N | N | N | 0% | 0% | N | N | N | 0% | 25% | 0% | 25% | 25% | 50% | 33% | 100% | 50% | 0% | 25% | 0% | N | N | 100% | 100% | 0% | 50% |
| Sphingomonas paucimobilis | 4 | N | N | N | N | 25% | N | 25% | N | 50% | 25% | 0% | 25% | N | 25% | 50% | 25% | 25% | 0% | 50% | 0 | N | N | 100% | N | N | 50% |
| Enterobacter cloacae complex | 2 | N | N | N | 0% | 100% | N | N | N | 50% | 100% | N | 100% | 100% | 100% | 100% | 100% | 50% | N | 50% | N | N | N | 100% | 100% | N | 100% |
| Proteus mirabilis | 2 | N | N | 0% | 0% | 100% | N | N | N | 0% | 0% | N | 100% | 0% | N | 0% | 100% | 0% | N | 0% | N | N | N | N | N | N | 0% |
| Citrobacter freundii | 1 | N | N | N | 0% | 100% | N | N | N | N | 100% | 0% | 100% | 100% | 100% | N | 100% | 100% | 100% | 0% | 0% | N | N | N | N | 100% | 0% |
| Morganella morganii ssp. | 1 | N | N | N | 0% | 100% | N | 0% | N | N | 100% | 100% | 100% | 100% | 0% | N | 100% | 100% | 100% | 0% | 0% | N | N | N | N | N | 0% |
| Proteus hauseri | 1 | N | N | 100% | 100% | 100% | N | 0% | 0% | 100% | 100% | N | 100% | 100% | 0% | 100% | 100% | 100% | N | 100% | N | N | N | N | N | N | 0% |
| Providencia stuartii | 1 | N | N | N | 0% | 100% | N | N | N | 0% | 0% | N | 100% | 0% | 0% | 0% | 100% | N | N | 0% | N | N | N | N | N | N | 0% |
| Pseudomonas stutzeri | 1 | N | N | N | N | 100% | N | N | N | 100% | 100% | N | 100% | N | 100% | 100% | 100% | 100% | N | 100% | N | N | N | 100% | N | N | 100% |
N: No se realizó sensibilidad para este antibiótico
Tabla 2 Sensibilidad antimicrobiana de bacterias grampositivas aisladas en un hospital general de la selva peruana 2021
| BACTERIAS GRAM POSITIVAS | AISLADOS | SENSIBILIDAD ANTIMICROBIANA | ||||||||||||||||||||
| BETA-LACTAMASA (+) | DETECCIÓN DE CEFOXITINA (+) | BENCILPENICILINA | AMPICILINA | OXACILINA | GENTAMICINA | ESTREPTOMICINA | CIPROFLOXACINO | LEVOFLOXACINO | MOXIFLOXACINO | RESISTENCIA IND A CLINDAMICINA (+) | ERITROMICINA | CLINDAMICINA | QUINUPRISTINA/ DALFOPRISTINA | LINEZOLID | VANCOMICINA | TETRACICLINA | TIGECICLINA | NITROFURANTOÍNA | RIFAMPICINA | TRIMETROPIM/ SULFAMETOXAZOL | ||
| Staphylococcus epidermidis | 26 | 100% | 92% | 0% | N | 12% | 50% | 0% | 31% | 44% | 35% | 0% | 12% | 19% | 100% | 100% | 100% | 85% | 100% | 100% | 35% | 54% |
| Staphylococcus haemolyticus | 19 | 100% | 100% | 0% | N | 0% | 11% | 0% | 0% | 5% | 5% | 0% | 0% | 0% | 95% | 95% | 100% | 79% | 95% | 100% | 18% | 37% |
| Staphylococcus hominis ssp. | 17 | 100% | 71% | 0% | N | 35% | 65% | 0% | 65% | 65% | 65% | 0% | 18% | 41% | 100% | 100% | 100% | 59% | 100% | 100% | 82% | 59% |
| Enterococcus faecalis | 5 | 40% | N | 80% | 80% | N | 67% | 40% | 80% | 80% | N | 0% | 25% | N | 0% | 100% | 100% | 60% | 100% | 100% | N | 100% |
| Staphylococcus aureus | 3 | 100% | 67% | 0% | N | 33% | 67% | 0% | 67% | 67% | 67% | 0% | 67% | 67% | 100% | 100% | 100% | 67% | 100% | 100% | 67% | 67% |
| Staphylococcus capitis | 2 | 100% | 0% | 0% | N | 50% | 100% | 0% | 100% | 100% | 100% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 50% | 100% | 100% | 100% |
| Staphylococcus saprophyticus | 2 | 100% | 100% | 0% | N | 0% | 50% | 0% | 50% | 50% | 50% | 0% | 0% | 50% | 100% | 100% | 100% | 100% | 100% | 100% | 50% | 0% |
| Enterococcus faecium | 1 | 0% | N | 0% | 0% | N | 100% | 0% | 100% | 0% | N | 0% | 0% | N | 100% | 100% | 100% | 0% | 100% | 0% | N | N |
| Kocuria kristinae | 1 | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N |
| Staphylococcus lugdunensis | 1 | 100% | 100% | 0% | N | 0% | 100% | 0% | 0% | 0% | 0% | 100% | 0% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 100% | 100% |
| Staphylococcus warneri | 1 | 100% | 0% | 0% | N | 100% | 100% | 0% | 100% | 100% | 100% | 0% | 0% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 100% | 100% |
N: No se realizó sensibilidad para este antibiótico
Tabla 3 Sensibilidad antimicrobiana de hongos aislados en un hospital general de la selva peruana 2021
| BACTERIAS GRAM POSITIVAS | AISLADOS | SENSIBILIDAD ANTIMICROBIANA | ||||||||||||||||||||
| BETA-LACTAMASA (+) | DETECCIÓN DE CEFOXITINA (+) | BENCILPENICILINA | AMPICILINA | OXACILINA | GENTAMICINA | ESTREPTOMICINA | CIPROFLOXACINO | LEVOFLOXACINO | MOXIFLOXACINO | RESISTENCIA IND A CLINDAMICINA (+) | ERITROMICINA | CLINDAMICINA | QUINUPRISTINA/ DALFOPRISTINA | LINEZOLID | VANCOMICINA | TETRACICLINA | TIGECICLINA | NITROFURANTOÍNA | RIFAMPICINA | TRIMETROPIM/ SULFAMETOXAZOL | ||
| Staphylococcus epidermidis | 26 | 100% | 92% | 0% | N | 12% | 50% | 0% | 31% | 44% | 35% | 0% | 12% | 19% | 100% | 100% | 100% | 85% | 100% | 100% | 35% | 54% |
| Staphylococcus haemolyticus | 19 | 100% | 100% | 0% | N | 0% | 11% | 0% | 0% | 5% | 5% | 0% | 0% | 0% | 95% | 95% | 100% | 79% | 95% | 100% | 18% | 37% |
| Staphylococcus hominis ssp. | 17 | 100% | 71% | 0% | N | 35% | 65% | 0% | 65% | 65% | 65% | 0% | 18% | 41% | 100% | 100% | 100% | 59% | 100% | 100% | 82% | 59% |
| Enterococcus faecalis | 5 | 40% | N | 80% | 80% | N | 67% | 40% | 80% | 80% | N | 0% | 25% | N | 0% | 100% | 100% | 60% | 100% | 100% | N | 100% |
| Staphylococcus aureus | 3 | 100% | 67% | 0% | N | 33% | 67% | 0% | 67% | 67% | 67% | 0% | 67% | 67% | 100% | 100% | 100% | 67% | 100% | 100% | 67% | 67% |
| Staphylococcus capitis | 2 | 100% | 0% | 0% | N | 50% | 100% | 0% | 100% | 100% | 100% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 50% | 100% | 100% | 100% |
| Staphylococcus saprophyticus | 2 | 100% | 100% | 0% | N | 0% | 50% | 0% | 50% | 50% | 50% | 0% | 0% | 50% | 100% | 100% | 100% | 100% | 100% | 100% | 50% | 0% |
| Enterococcus faecium | 1 | 0% | N | 0% | 0% | N | 100% | 0% | 100% | 0% | N | 0% | 0% | N | 100% | 100% | 100% | 0% | 100% | 0% | N | N |
| Kocuria kristinae | 1 | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N | N |
| Staphylococcus lugdunensis | 1 | 100% | 100% | 0% | N | 0% | 100% | 0% | 0% | 0% | 0% | 100% | 0% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 100% | 100% |
| Staphylococcus warneri | 1 | 100% | 0% | 0% | N | 100% | 100% | 0% | 100% | 100% | 100% | 0% | 0% | 0% | 100% | 100% | 100% | 100% | 100% | 100% | 100% | 100% |
N: No se realizó sensibilidad para este antibiótico.
DISCUSIÓN
En nuestra Institución, se encontró que el 74.6 % de cultivos positivos correspondieron a bacterias gramnegativas, seguidas por grampositivas en 16.4 % y hongos el 9 % restante. Hubo una alta frecuencia de cultivos de secreción bronquial de pacientes hospitalizados en los servicios de Emergencia y Cuidados Intensivos. Hallazgo semejante se ha descrito en los mapas microbiológicos en otros hospitales de América Latina y de nuestro país 6-8, relacionados principalmente a infecciones asociadas a la atención de la salud (IAAS). Se destaca una muy elevada tasa de resistencia y multidrogoresistencia en bacterias gramnegativas y grampositivas.
Las IAAS son más frecuentes a nivel de vías respiratorias, torrente sanguíneo, sitio quirúrgico, piel-mucosas y tracto urinario. Durante la pandemia del SARS-CoV-2, se pudo observar un incremento de IAAS, principalmente en pacientes internados en áreas críticas como el reporte que se presenta, por lo que fue oportuna la realización de cultivos antimicrobianos para determinar de manera específica los patógenos asociados 2,9-11.
La situación de pandemia permitió mayor acceso a cultivos y confirmar el incremento del perfil de resistencia antimicrobiana, similar a lo reportado en una institución de salud en Colombia 12. Las áreas de UCI y emergencia son las que reportan el mayor aislamiento de microorganismos en el presente estudio, asociados a mayor frecuencia de técnicas invasivas en los pacientes que facilitan el ingreso y, concomitantemente, el desarrollo de la infección por estos microorganismos. La baja frecuencia de cultivos positivos en otros servicios hospitalarios, probablemente, se deba a la falta de recursos para toma de muestras, pues se priorizan áreas críticas o la falta de conocimiento por parte del personal en la necesidad de confirmar infecciones (especialmente IAAS) en áreas no críticas del hospital.
Dentro del grupo de las bacterias gramnegativas, está el Acinetobacter baumannii complex como la más frecuentemente aislada con un 32.7 %, que se evidencia en una escasa sensibilidad a imipenem y meropenem: 2 % y 3 %, respectivamente, lo cual resulta alarmante, ya que, al ser estos fármacos de reserva, se suelen utilizar en infecciones complicadas de manera empírica y queda en evidencia la poca efectividad con los resultados mostrados 13.
En los últimos años, Acinetobacter baumannii complex ha aumentado significativamente su prevalencia y, con ella, los mecanismos de resistencia 14,15; en un estudio realizado en los años 2012-2016 sobre el perfil microbiológico y de sensibilidad a los antibióticos en dos hospitales de alta complejidad del seguro social de salud del Perú, mostró a E. coli como el principal organismo gramnegativo aislado 16; en contraste con nuestro estudio, queda claro que es necesario continuar con la realización de estudios de sensibilidad antimicrobiana que permita evidenciar un panorama actualizado.
Además, se observa una resistencia a tigeciclina en un 47 %; si bien la primera aparición notificada de resistencia a la tigeciclina se dio en 2007, un estudio reciente sobre la prevalencia de Acinetobacter baumannii complex mostró que la resistencia a la tigeciclina era inferior al 5,5 % en Corea, India y China 17. Se evidenció una preocupación en la utilidad de este fármaco en infecciones asociadas a Acinetobacter baumannii complex en nuestro medio.
La presencia de Acinetobacter baumannii complex, en frecuencias tan elevadas, sugiere que no hay medidas de aislamiento de contacto adecuadas, por lo que es importante el cumplimiento estricto de la higiene de manos por el personal de salud como parte fundamental para controlar una situación de endemia por este patógeno 18.
Teniendo una alta frecuencia de microorganismos resistentes en el lugar de estudio: Acinetobacter baumannii complex, Klebsiella pneumoniae ssp. y Pseudomonas aeruginosa), las terapias empíricas a emplear deberían incluir combinaciones de agentes antimicrobianos de reserva como colistina, ampicilina/sulbactam, tigeciclina, carbapenems, ceftazidima/avibactam, aztreonam o aminoglucósidos. La principal debería ser aislar y caracterizar adecuadamente el microorganismo y más aún implementar medidas preventivas adecuadas.
Con respecto a las bacterias grampositivas, Shtaphylococcus aureus presentó una baja frecuencia en comparación a otros hospitales 19, debido, probablemente, a que no se realizaron muchos aislamientos de lesiones cutáneas en el presente estudio.
Los Staphylococcus coagulasa negativa (SCN) suelen ser considerados como los microorganismos más frecuentemente aislados en los laboratorios de microbiología 20. En nuestro estudio, significó el 87 % de todos los microorganismos grampositivos, pero no se pudo determinar la implicancia clínica de dichas bacterias, por lo que pudieron ser considerados desde contaminantes y/o microorganismos patógenos. Aun así, se sugiere evaluar los protocolos para la correcta toma de hemocultivos e incidir en la capacitación del personal para evitar confusión en el manejo clínico del paciente.
La prevalencia micótica en hemocultivos fue baja con mayor presencia en secreción bronquial. El tipo de hongo más frecuentemente con aislamiento fue Candida albicans (42.9 %) al igual que lo encontrado en un hospital nacional 21, y su tasa de resistencia aún es baja.
En relación con el uso de antimicrobianos, es importante optimizar dicha terapia en los hospitales. Al respecto, es importante mencionar que, en algunas instituciones del país, aún mantienen una elevada frecuencia de prácticas injustificada relacionadas a estos fármacos, que contribuyen así en un aumento de resistencia antimicrobiana 22,23. El empleo de antimicrobianos como aminopenicilinas asociadas a un inhibidor de betalactamasas (IBL), carbapenémicos, glicilciclinas (tigeciclina) y glicopéptidos deben estar vigiladas por el Comité de Optimización de Antimicrobianos y la Unidad del Programa de Optimización Antimicrobiana, entidades hospitalarias que ayudan a optimizar adecuadamente el uso de antimicrobianos, promueven el cambio positivo en la prescripción y la utilización de tratamientos costo-efectivos, contribuyen en la mejora de resultados clínicos de pacientes con infecciones a su vez que reducen la selección de microorganismos resistentes y los riesgos para el paciente en la utilización de antimicrobianos, ya que, como se observó, existe una alta tasa de resistencia en bacterias gramnegativas y positivas, por lo que es necesario mantener una adecuada vigilancia epidemiológica 24,25.
El presente mapa microbiológico elaborado consiste en un estudio pasivo a partir de las muestras microbiológicas; al respecto, no se puede determinar con exactitud cuántos y cuáles de los microorganismos descritos han sido colonizantes y/o contaminantes. A su vez, no se pudo establecer cuáles han sido los patógenos causales de IAAS ni la tasa de positividad de cultivos por no contar con el número total de muestras de cultivos tomadas ni los genes implicados en la resistencia; sin embargo, a pesar de estas limitaciones, el presente estudio constituye uno de los primeros reportes locales y, a pesar de incluirse un solo hospital, es importante para conocer la situación de nuestro medio para tener evidencia y continuar con el desarrollo del mapa microbiológico regional que brinde información indispensable para optimizar la terapia antimicrobiana en el manejo de las infecciones.
Se concluye que las bacterias gramnegativas son las más prevalentes en el hospital de estudio y los servicios hospitalarios más afectados son emergencia y las unidades de cuidados intensivos. Las muestras de los cultivos más involucradas fueron secreción bronquial y sangre, con un elevado porcentaje de resistencia antimicrobiana principalmente en Acinetobacter baumannii complex (XDR en 88.5 %), Klebsiella pneumoniae ssp. (MDR en 56. 3%), Pseudomonas aeruginosa (XDR en 54.7 %), Staphylococcus epidermidis (MDR en 92.3 %) y Staphylococcus haemolyticus (MDR en 100 %).











 texto en
texto en 


