Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de Neuro-Psiquiatría
Print version ISSN 0034-8597
Rev Neuropsiquiatr vol.82 no.2 Lima Apr. 2019
http://dx.doi.org/https://doi.org/10.20453/rnp.v82i2.3539
ARTÍCULO ORIGINAL
Diagnóstico molecular de pérdida de heterocigosidad para 1p/19q en tumores oligodendrogliales por PCR multiplex en el Instituto Nacional de Enfermedades Neoplásicas, Lima- Perú.
Molecular diagnosis of heterozygosity loss for 1p/19q in oligodendroglial tumors by multiplex PCR at the National Institute of Neoplastic Diseases, Lima-Peru.
Jimena Murguía-Cuadros 1,a, Ruddy Liendo-Picoaga 2,b, Karina Cancino-Maldonado 2,a, Dany Cordova- Mamani 2,c, Tatiana Vidaurre-Rojas 3,d,Pamela García-Corrochano 4,e, Enrique Orrego-Puelles 4,e,f, Sandro A. Casavilca-Zambrano 5,g
1 Servicio de Genética del Departamento de Patología, Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.
2 Banco de Tejidos Tumorales, Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.
3 Departamento de Medicina Oncológica, Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.
4 Departamento de Neurocirugía, Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.
5 Departamento de Patología, Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.
a Bióloga; b Biólogo Molecular, MSc ; c Tecnólogo Médico ; d Médico-Oncólogo, Mg; e Médico-Neurocirujano; f Doctor; g Médico Patólogo Oncológo, Mg.
RESUMEN
La pérdida de heterocigosidad 1p/19q tiene valor pronóstico clínico y está fuertemente asociada con características histológicas clásicas de oligodendroglioma. Objetivos: El presente artículo, propone un método molecular para determinar la pérdida de heterocigosidad (LOH por sus siglas en inglés) para 1p/19q y permitir la clasificación de tumores oligodendrogliales. Material y Métodos: Se utilizaron muestras en fresco del Banco de Tejidos Tumorales del Instituto Nacional de Enfermedades Neioplásicas (INEN) y biopsias de tejido embebido en parafina de tumores oligodendrogliales, con diagnóstico patológico de oligodendroglioma y oligoastrocitoma. Los métodos propuestos son PCR Multiplex y amplificación de fragmentos por electroforesis capilar de los productos de PCR, y fueron aplicados a un total de 39 casos que presentaban grado histológico II y III. Resultados: Los resultados obtenidos permiten una adecuada clasificación molecular de los tumores oligodendrogliales.
PALABRAS CLAVE: Oligodendroglioma, PCR Muliplex, pérdida de heterocigosidad.
SUMMARY
A heterozygosity loss of 1p/19q has clinical prognostic value and is strongly associated with classical histologic features of oligodendroglioma. Objectives: The present article proposes a molecular method to determine the loss of heterozygosity (LOH) for 1p/19q and to allow the classification of oligodendroglial tumors. Material and Methods: Fresh samples from the National Institute of Neoplastic Diseases Tumor BioBank and paraffin-embedded tissue biopsies of oligodendroglial tumors with pathological diagnosis of oligodendroglioma and oligoastrocytoma were used. The proposed methods are Multiplex PCR and amplification of fragments by capillary electrophoresis of PCR products, and were applied to a total of 39 cases which presented histological grade II and III. Results: The results obtained allow an adequate molecular classification of oligodendroglial tumors.
KEYWORDS: Oligodendroglioma, Multiplex PCR, loss of heterozygosity.
INTRODUCCIóN
Según el Sistema de Vigilancia Epidemiológica del Ministerio de Salud (MINSA), para los años 2006 al 2011, los tumores del Sistema Nervioso Central (SNC) corresponden al 1.8% de las neoplasias en el Perú. El Instituto Nacional de Enfermedades Neoplásicas (INEN), atendió para el periodo 2005-2014 un total de 1541 casos de tumores del SNC de los cuales el 10% correspondieron a oligodendroglioma y 6% a oligoastrocitoma, grado II y III (datos estadísticos no publicados). La reciente incorporación de patrones genéticos en la clasificación molecular de tumores del SNC de la Organización Mundial de la Salud (OMS) crea la necesidad de implementar herramientas validadas, que faciliten su diagnóstico (1, 2, 3). La actualización de la Clasificación de tumores del sistema nervioso central (SNC) de la OMS para el año 2016, se basa en el Consenso de Harlem (2014) (1) en donde se recomienda que el diagnóstico debe ser determinado en base a la clasificación histopatológica (i.e. inmunohistoquímica) (4), grado OMS e información molecular (i.e. biomarcadores); como un diagnostico integrado, anteriormente, esta información molecular se proporcionaba principalmente como información complementaria dentro de categorías histológicas ya definidas (5). Este enfoque contribuye a la reproducibilidad inter observador, uno de los problemas más frecuentes en las prácticas de neuropatología quirúrgica y particularmente en el diagnóstico de neoplasias oligodendrogliales (2).
En los últimos años, se ha desarrollado el concepto de marcadores moleculares, el más utilizado en neoplasias oligodendrogliales es la codeleción del brazo corto del cromosoma 1 y el brazo largo del cromosoma 19 (1p/19q) (6). Por otro lado, en gliomas difusos de la línea astrocítica y oligodendroglial, grado II y III. La clasificación de los gliomas difusos de tipo adulto sigue los criterios estándares actuales de la OMS para astrocitomas y oligodendrogliomas, con excepción de algunas circunstancias como cuando las características de la muestra dificultan su graduación. Este tipo de excepciones es más relevante en el glioma difuso de morfología ambigua, que no es claramente de subtipo oligodendroglial puro o astrocítico puro, ya sea en el contexto de una pequeña biopsia en la que el muestreo selectivo puede dificultar el diagnóstico morfológico o debido a la falta de estudios moleculares (1,7). Esto toma particular relevancia en la toma de decisiones terapéuticas, debido a las diferencias en el manejo entre gliomas difusos de la línea astrocítica u oligodendroglial, siendo estos últimos de mejor pronóstico y sensibles a la quimioterapia con agentes alquilantes como Temozolamida.
Siendo el INEN el centro de referencia para diagnóstico y tratamiento de neoplasias del SNC se hace necesario implementar la determinación de la heterocigocidad (LOH) 1p/19q mediante pruebas moleculares, que asociada a pruebas de inmunohistoquímica, permitirán el desarrollo de un método sensible y específico para el diagnóstico preciso de los tumores del Oligodendrogliales.
El presente trabajo tuvo como objetivo proponer un método molecular para determinar la pérdida de heterocigosidad (LOH por sus siglas en inglés) para 1p/19q y permitir la clasificación de tumores oligodendrogliales.
MATERIAL Y MéTODOS
Consideraciones éticas
Las muestras pertenecen al Banco de Tejidos Tumorales del Instituto Nacional de enfermedades Neoplásicas (INEN) y son donadas por los pacientes, mediante un consentimiento informado, aprobado por el Comité de ética Institucional del INEN. La información brindada y las muestras colectadas podrán ser utilizadas en el estudio.
Identificación de la región tumoral y desparafinización del tejido
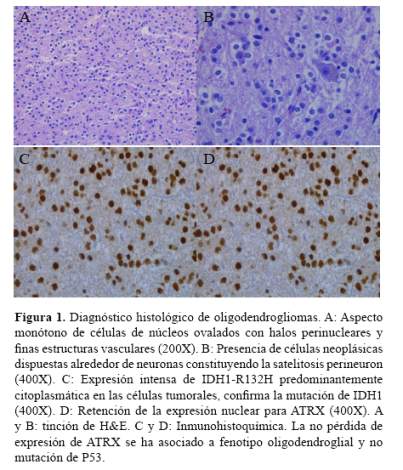
Se analizó ADN tumoral a partir de biopsias o tejido fijado en parafina con no menos del 90% de células tumorales de 39 casos del INEN (figura 1). Se realizaron 10 a 15 cortes histológicos de 20 µm de espesor utilizando un micrótomo y se colocaron en un tubo de 1.5 mL.
En una cabina para extracción de gases, se desparafinaron las muestras en 1 mL de Xilol, con la ayuda de un vortex por 10 segundos y se centrifugaron a 14000 rpm por 2 minutos. Se eliminó el sobrenadante y se realizó un lavado con 1 mL de etanol absoluto, se centrifugó a 14000 rpm por 2 min. Se eliminó el sobrenadante y se dejó secar el tubo a 57°C por 10 min.
Extracción de ADN y cuantificación
Cada muestra fue tratada con el kit QIAamp® DNA FFPE Tissue siguiendo las instrucciones del fabricante. Brevemente, se resuspendió el precipitado en 180 µL de buffer ATL (animal tissue lyss buffer) y 20 µL de proteinasa K (20mg/mL), se incubó a 56°C por 1 hora hasta que el tejido estuviese completamente lisado. Inmediatamente después se incubó la mezcla a 90°C por 1 hora. Luego de un breve spin, se le agregó 200 µL de buffer AL y 200 µL de etanol absoluto, y se mezcló por vortex. Se transfirió cada muestra a las columnas QIAampMinElute y se centrifugó a 8000 rpm por 1 min. Luego se realizaron lavados sucesivos con las soluciones del lavado Wash Buffer 1 y Wash Buffer 2 del kit, se centrifugó a 14000 rpm por 3 min. El ADN fue eluído con 30 µL de buffer ATE del kit, se centrifugó a 14000 rpm por 1 min. La cuantificación de ADN se llevó a cabo con equipo Qubit®.
Reacción en cadena de la polmerasa Múltiple (Multiplex PCR)
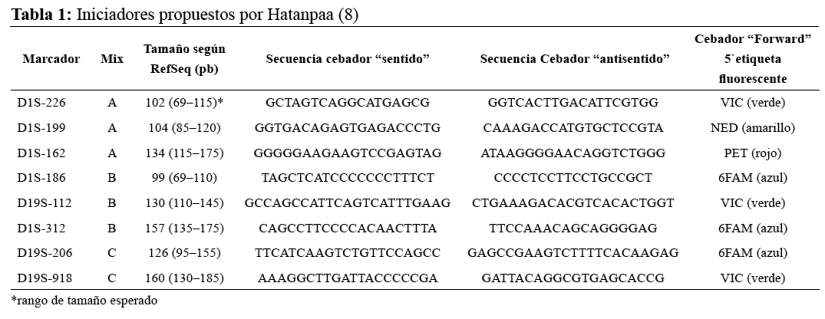
El volumen final de cada reacción fue de 10 µL que contiene 0,4 unidades de Taq Thermo Scientific Maxima Hot Start Taq DNA Polymerase, 0.3 mM de dNTPs, 1.5 mM de ión Mg++ y 1-2 ng de ADN, los cebadores utilizados para el Mix A son: D1S162F, D1S162R, D1S199F, D1S199R, D1S226F, D1S226R, para el Mix B son: D1S186F, D1S186R, D1S312F, D1S312R, D19S112F, D19S112R, para el Mix C son: D19S918F, D19S918R, D19S206F, D19S206R.
Las condiciones de ciclos de PCR para el Mix A y el Mix B: 95°C por 4 minutos, luego 40 ciclos de 95°C por 45 segundos seguidos de 55°C por 45 segundos y 72°C por 1 minuto, seguido por una etapa de extensión final a 72°C por 20 min. El Mix C amplificó con el siguiente protocolo de TouchDown PCR: 95°C por 4 minutos, luego 23 ciclos de 95°C por 45 segundos y 61°C (AutoDelta A -0.2°C) por 45 segundos, seguidos de 10 ciclos de 95°C por 45 segundos y 55°C por 45 segundos; finalmente 72°C por 1 minuto, seguido por una etapa de extensión final a 72°C por 20 min. Los cebadores utilizados fueron propuestos por Hatanppa (8) (tabla 1).
Los fragmentos amplificados fueron denaturados según el protocolo indicado en la tabla 2.
Análisis de fragmentos amplificados en analizador ABI 3500
Los amplicones generados fueron separados por electroforesis en capilar usando el analizador genético ABI 3500 de Applied Biosystems, se utilizó el polímero POP 4, se utilizó la configuración y parámetros estándares del analizador ABI 3500.
Los electroferogramas generados se analizaron con la herramienta bioinformática Genemapper. Esta metodología permitió separar los amplicones generados por tamaño y color para analizarlos independientemente.
Análisis de perfiles de cada alelo obtenido
Se utilizó el software Genemapper, se identificó cada alelo por su tamaño en pares de bases y se comparó los perfiles alélicos normal y tumoral en búsqueda de indicios de pérdidas alélicas. Se consideró la morfología típica de los microsatélites de repeticiones dinucleotidicas para poder identificar adecuadamente los diferentes patrones de heterocigocidad existentes.
RESULTADOS
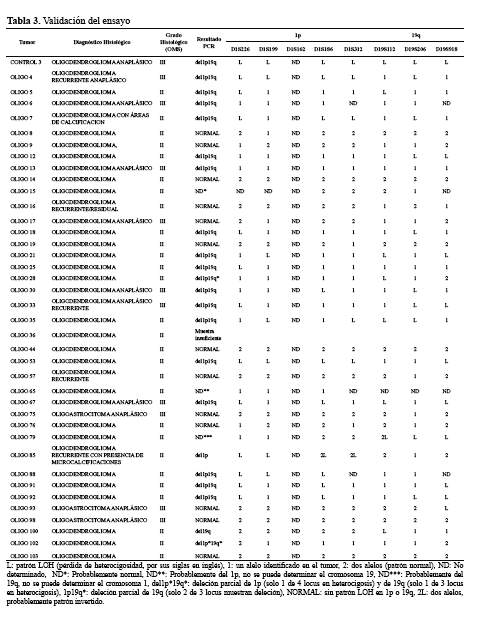
Las técnicas utilizadas fueron Multiplex PCR y el análisis de fragmentos amplificados por electroforesis capilar como se indica en la metodología del estudio. Se propuso determinar la pérdida de heterocigosidad 1p y 19q en 39 tumores con diagnóstico de Oligodendrogliomas y Oligoastrocitomas, grado II y III (OMS) de pacientes del Instituto Nacional de Enfermedades Neoplásicas (INEN), identificando diferentes patrones en las muestras analizadas. Del total de tumores en estudio (39 casos de tumores oligodendrogliales) el 71.8% corresponden a grado II, 28.2% corresponden a grado III (tabla 3).
Del total de muestras con Grado Histológico II (OMS), 46.4% presentan deleción 1p/19q, 3.6% con deleción 19q y 32.1% presentan patrones normales (1 ó 2 alelos identificados en el tumor). En un tumor con grado histológico II no se obtuvo suficiente muestra para realizar las pruebas moleculares propuestas.
Del total de muestras con Grado Histológico III (OMS), 63.6% presentan deleción 1p/19q y 36.4% presentan patrones normales (1 ó 2 alelos identificados en el tumor).
De los cinco microsatélites reportados en la bibliografía para el cromosoma 1 brazo q, no se pudo determinar el estado de heterocigocidad para D1S162 en todas las muestras de tumor en estudio.
El tumor con código OLIGO100, con grado Histológico II (OMS), muestra un patrón LOH solo en 19q.
De los 21 tumores con LOH en 19q, hubo 2 tumores que mostraron solo el patrón de alelo único en los 3 loci de 19q (códigos OLIGO13 y OLIGO25).
DISCUSIóN
El oligodendroglioma es la tercera neoplasia glial más común y se presenta más frecuentemente en el lóbulo frontal y en el sexo masculino. La manifestación máxima es entre los 40 y 50 años, afectando a la población económicamente activa y la presentación clínica es a menudo prolongada presentando convulsiones la mayoría de los pacientes, (1,2).
Diversos estudios previos han demostrado la importancia de introducir el Diagnóstico Molecular para un tratamiento adecuado, el desarrollo de la aplicación de tecnologías y del uso de herramientas moleculares que mejoren el diagnóstico de tumores oligodendrogliales.
Está ampliamente reportado que la codeleción 1p/19q se detecta en hasta el 80% de los tumores oligodendrogliales y 60% de oligodendrogliomas anaplásicos y 40% de oligoastrocitomas. Por otro lado solo está presente en el 40% de los oligoastrocitomas,30% de los oligoastrocitomas anaplásicos, y en el 10% de los gliomas astrocíticos difusos, incluyendo el glioblastoma multiforme (9,10), éste último considerado como el tipo de glioma más agresivo (11). Es importante conocer el patrón de cada una de las muestras para el microsatétile D1S162, para evaluar la probabilidad de que existen tumores con patrón de alélo único en los cinco loci de microsatélites en 1p.
La inmunohistoquímica desempeña un rol importante en el suministro de información genética para tumores (figura 1) (12). Los tipos de alteraciones moleculares en tumores son detectables potencialmente por ésta técnica e incluyen traslocaciones cromosómicas específicas, mutaciones específicas, deleción o pérdida genética, amplificación genica y virus asociados a tumores (8), sin embargo el diagnóstico histológico puede ser variable entre observadores, incluso entre neuropatólogos altamente experimentados (13), lo que dificulta los estudios clínicos de gliomas (14).
En la actualidad, ningún enfoque puede describir todo el espectro de gliomas en adultos, es importante incorporar y combinar nuevas técnicas como análisis de mutaciones, secuenciamiento de ARN, genómica y metilación del ADN que nos permitan el refinamiento de la clasificación de los gliomas en adultos (15).
En el Perú, aún no se ha publicado una técnica validada para el diagnóstico molecular de Oligodendrogliomas, siendo un factor importante para el diagnóstico la determinación de la codeleción 1p/19q. Es por ello que se propone aplicar la técnica de Reacción en Cadena de la Polimerasa Múltiple (multiplex PCR) para la determinación de la pérdida de la heterocigocidad para la codeleción 1p/19q e incorporar esta metodología, como potencial herramienta de diagnóstico de tumores oligodendrogliales en el INEN.
REFERENCIAS BIBLIOGRáFICAS
1. Koeller KK, Rushing EJ. From the archives of the AFIP: Oligodendroglioma and its variants: radiologic- pathologic correlation. Radiographics. 2005;25(6):1669-88. DOI: 10.1148/rg.256055137 [ Links ]
2. Dehghani F, Schachenmayr W, Laun A, Korf HW. Prognostic implication of histopathological, immunohistochemical and clinical features of oligodendrogliomas: a study of 89 cases. Acta neuropathologica. 1998;95(5):493-504. [ Links ]
3. Louis DN, Ohgaki H, Wiestler OD, Cavenee WK, editors. WHO classification of tumours of the central nervous system. Lyon, Francia: International Agencyfor Research on Cancer; 2016. [ Links ]
4. Las Heras F, Diocares G. Neuropatología: diagnóstico con biología molecular. Revista Médica Clínica Las Condes. 2017; 28(3): 352-359. [ Links ]
5. Wesseling P, Capper D. WHO 2016 classification of gliomas. Neuropathology and applied neurobiology. 2018; 44(2): 139-150. [ Links ]
6. Hinrichs BH, Newman S, Appin C L, et al. Farewell to GBM-O: Genomic and transcriptomic profiling of glioblastoma with oligodendroglioma component reveals distinct molecular subgroups. Acta neuropathologica communications. 2016; 4(1): 4. doi: 10.1186/s40478-015-0270-7 [ Links ]
7. Louis DN, Perry A, Reifenberger G, et al. The 2016 World Health Organization classification of tumors of the central nervous system: a summary. Acta neuropathologica. 2016; 131(6): 803-820. doi:10.1007/s00401-016-1545-1 [ Links ]
8. Hatanpaa KJ, Burger PC, Eshleman JR, Murphy KM, Berg KD. Molecular diagnosis of oligodendroglioma in paraffin sections. Laboratory investigation. 2003; 83(3): 419. [ Links ]
9. Pekmezci M, Perry A. Practical molecular pathologic diagnosis of infiltrating gliomas. Surgical Pathology Clinics. 2015;8(1):49–61. [ Links ]
10. Horbinski C. Something old and something new about molecular diagnostics in gliomas. Surgical Pathology Clinics. 2012;5(4):919–39. [ Links ]
11. Popova SN, Bergqvist M, Dimberg A, et al. Subtyping of gliomas of various WHO grades by the application of immunohistochemistry. Histopathology. 2014; 64(3): 365-379. [ Links ]
12. Martínez-Ricarte F, Mayor R, Martínez-Sáez E, et al. Molecular diagnosis of diffuse gliomas through sequencing of cell-free circulating tumor DNA from cerebrospinal fluid. Clinical Cancer Research. 2018;24(12): 2812-2819. [ Links ]
13. Smith JS, Perry A, Borell TJ, et al. Alterations of chromosome arms 1p and 19q as predictors of survival in oligodendrogliomas, astrocytomas, and mixed oligoastrocytomas. Journal of Clinical Oncology. 2000; 18(3): 636-636. [ Links ]
14. Ueki K, Nishikawa R, Nakazato Y, et al. Correlation of histology and molecular genetic analysis of 1p,19q, 10q, TP53, EGFR, CDK4, and CDKN2A in 91 astrocytic and oligodendroglial tumors. Clinical Cancer Research. 2002; 8(1): 196-201. [ Links ]
15. Barritault M, Meyronet D, Ducray F. Molecular classification of adult gliomas: recent advances and future perspectives. Curr Opin Oncol. 2018; 30(6):375-382. doi: 10.1097/CCO.0000000000000482 [ Links ]
Fuentes de financiamiento: Autofinanciado
Declaración de conflictos de interés: No existe conflicto de interés para los autores.
Correspondencia:
Sandro A. Casavilca-Zambrano
Calle Las Lilas 105 Dpto. 302, Urbanización Valle Hermoso, Monterrico, Surco, Perú.
Teléfono: (51) 996499092
Correo electrónico: scasavilca@inen.sld.pe
Recibido: 06/05/2019
Aceptado: 21/06/2019