Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Medica Herediana
versión impresa ISSN 1018-130Xversión On-line ISSN 1729-214X
Rev Med Hered v.21 n.1 Lima ene. 2010
Staphylococcus aureus
resistente a meticilina adquirido en la comunidad aislados en tres hospitales de Lima-Perú.Methicillin resistant Staphylococcus aureus isolates acquired in the community in three hospitals in Lima, Peru.
Jesús Tamariz 1, Juan Agapito 1,2, Gertrudis Horna 1,2, Elena Tapia1,3, William Vicente4, María Silva4,Rito Zerpa5, Humberto Guerra1,2.
1
Universidad Peruana Cayetano Heredia. Lima, Perú.2
Instituto de Medicina Tropical Alexander von Humboldt. Lima, Perú.3
Hospital Nacional Cayetano Heredia. Lima, Perú.4
Instituto Nacional de Enfermedades Neoplásicas. Lima, Perú.5
Instituto Nacional de Salud del Niño. Lima, Perú.
RESUMEN
Staphylococcus aureus es un importante patógeno involucrado en una serie de infecciones e intoxicaciones, presenta múltiples factores de virulencia y su impacto se incrementa por su notable resistencia a los antimicrobianos. Objetivo: Determinar la frecuencia de Staphylococcus aureus meticilino resistente adquiridos en la comunidad, en hospitales de Lima- Perú. Material y métodos: Se realizó un estudio descriptivo multicéntrico. La resistencia a meticilina se determinó por el método Oxacillin Agar Screen. El origen de la cepa fue determinado mediante los criterios de los CDC; la Leucocidina de Panton Valentine fue identificada por métodos moleculares. Resultados: Se aislaron 276 cepas de Staphylococcus aureus, 160 fueron resistentes a meticilina (58%), 9 de ellas fueron identificadas como adquiridas en la comunidad (5,6%). La PVL fue identificada en 25 cepas (9,1%), 14 fueron MSSA y 11 MRSA, de éstas últimas solo 4 fueron MRSAcom, 7 fueron MRSAhosp (p<0,001). Conclusiones: El estudio revela niveles elevados de resistencia a meticilina, pero niveles bajos de MRSAcom. En nuestro medio la presencia de PVL no constituiría un marcador para la identificación de los MRSAcom. (Rev Med Hered 2010;21:4-10).
PALABRAS CLAVE: Staphylococcus aureus, resistente, meticilina, comunidad.
SUMMARY
Background : Staphylococcus aureus is an important pathogen involved in a series of infections and toxin mediated syndromes, has many virulence factors and its impact increases its resistance to antimicrobial agents. Objectives: To determine the frequency of community acquired methicillin resistant Staphylococcus aureus among hospitals in Lima -Peru. Material and Methods: We performed a prospective multicenter study. The resistance to methicillin was determined by the Oxacillin Agar Screen method. The origin of the strain was determined using CDC criteria, the Panton Valentine Leucocidin was identified by molecular methods. Results: We isolated 276 strains of Staphylococcus aureus, 160 were resistant to methicillin (58%). 9 strains were identified as community acquired MRSA (5.6%). The Panton Valentine Leucocidin was identified in 25 strains (9.1%), 14 were MSSA and 11 MRSA, only 4 of the latter were CA MRSA, 7 were HA MRSA (p <0.001). Conclusions: The study showed frequencys of methicillin resistance, but low CA MRSA. In our environment the presence of PVL would not be a marker for the identification of CA MRSA. (Rev Med Hered 2010;21:4-10).
KEYWORDS: Staphylococcus aureus, methicillin-resistant, community.
INTRODUCCIÓN
Staphylococcus aureus, uno de los patógenos humanos mas importantes, se encuentra involucrado en una diversidad de infecciones e intoxicaciones, pudiendo comprometer cualquier órgano del sistema (1-4). El tratamiento de estas infecciones se ve dificultado por su elevada capacidad de generar resistencia a los antimicrobianos.
En 1946, se reportó un 60% de resistencia a penicilinas naturales, en 1960 ésta se incrementó a 80%, obligando a su abandono. Hacia 1961 se reportó en Europa la aparición de Staphylococcus aureus resistente a meticilina (MRSA), extendiéndose posteriormente a diversas partes del mundo (5,6), constituyendo en la actualidad un serio problema de salud pública (7,8,9).
Estudios en nuestro país muestran una elevada prevalencia de MRSA, sobre todo a nivel hospitalario (10,11). En un estudio multicentrico en 6 hospitales de Lima Metropolitana (1996) encontramos 63,3% de cepas MRSA (11).
El mecanismo molecular de resistencia a la meticilina consiste en la producción, por el gen mec A, de una proteína de unión a penicilina adicional (PBP2a) que no presenta afinidad por los betalactámicos, originando resistencia a penicilinas semi-sintéticas (meticilina y oxacilina), cefalosporinas de primera a cuarta generación y a los carbapenems; extendiéndose a otras familias antibióticas como las quinolonas y lincosamidas (5,6). El gen mec A está localizado en una isla genómica móvil conocida como Staphylococcal Cassette Chromosome mec (SCCmec). En la actualidad se han identificado siete tipos de SCCmec (I al VII), sus tamaños varían de 20,9 a 66,9 kb. El SCCmec tipo I (34,3 kb), IV (20,9–24,3 kb), V (28 kb), VI (20,9 Kb), y VII (35,9 kb) codifican solo resistencia al grupo de betalactámicos, mientras que los SCCmec tipo II (53,0 kb) y III(66,9 kb) generan resistencia a múltiples grupos de antibióticos (12).
En 1993 emergen las cepas de Staphylococcus aureus resistentes a meticilina adquiridos en la comunidad (MRSAcom), cepas de elevada virulencia implicadas principalmente en infecciones severas de piel y tejidos blandos, neumonía necrotizante graves y fulminantes (13-16).
MRSAcom es diferente del Staphylococcus aureus meticilino resistente de origen hospitalario (MRSAhosp) conocido hasta entonces. The Centers for Disease Control and Prevention - CDC la define como cepas de MRSA aisladas de pacientes no hospitalizados o dentro de las primeras 48 horas de hospitalización, sin antecedentes de infección o colonización con MRSA, sin hospitalización en el año anterior al aislamiento, ni empleo de dispositivos protésicos permanentes (13).
Genéticamente los MRSA-com poseen el SCCmec de los tipos tipo IV,V y VII a diferencia de los MRSAhos, que poseen los tipos I, II, III y VI (12,14-16). Los MRSA-com poseen factores de virulencia específicos como una exotoxina: Leucocidina de Panton Valentine (PVL), elemento no solo relacionado con su elevada patogenicidad, sino también empleado como marcador molecular para su identificación. Se ha reportado además que estas cepas tienen mayor rapidez de duplicación y elevada capacidad de diseminación.
Recientemente se ha informado un nuevo factor de virulencia los péptidos modulina soluble en fenol (PSM), con capacidad para aglutinar, activar y lisar neutrófilos humanos, bloqueando de esta manera la primera línea de respuesta inmune celular contra estas infecciones (9,13-20).
Los MRSAcom presentan un perfil de susceptibilidad a antimicrobianos particular: son resistentes a betalactámicos (penicilinas, cefalosporinas, aminopenicilinas asociadas a inhibidores de betalactamasas, carbapenems) así como a los macrólidos-azálidos (eritromicina, claritromicina, azitromicina). Sin embargo a diferencia de los MRSAhos, son susceptibles a sulfametoxazole/trimetoprim, clindamicina, gentamicina, rifampicina, entre otros (15,16).
La identificación de las cepas MRSAcom es de suma importancia para el abordaje terapéutico adecuado de las infecciones y disminuir la elevada mortalidad, así mismo para la implementación de medidas de control que impidan su propagación. Sin embargo, en nuestro medio no se han realizado estudios orientados a su identificación.
El estudio tuvo como objetivo determinar la frecuencia de Staphylococcus aureus resistente a meticilina adquirido en la comunidad en hospitales de Lima Metropolitana.
MATERIAL Y MÉTODOS
Estudio de tipo descriptivo, transversal. Entre mayo 2005 y septiembre 2006, se recolectaron 276 cepas de Staphylococcus aureus de los laboratorios de 3 hospitales de Lima: Hospital Nacional Cayetano Heredia (HNCH), Instituto Especializado de Salud del Niño (INSN), Instituto Nacional de Enfermedades Neoplásicas (INEN). Las cepas fueron verificadas mediante pruebas de coagulasa, DNAsa y almacenadas hasta su uso.
Determinación de la Resistencia a Meticilina
Se empleó el método Oxacillin Agar Screen propuesto por Clinical and Laboratory Standards Institute (CLSI). En placas de agar Mueller Hinton suplementado con oxacilina (6 µg/mL) y NaCl (4%), se inocularon 104 UFC. Las placas fueron incubadas entre 33-35°C durante 24h. La presencia de una o más colonias sobre el inóculo fue indicador de resistencia a meticilina.
Determinación de cepas MRSAcom
Las cepas MRSA-com fueron catalogadas en base a los datos epidemiológicos obtenidos. Las cepas de MRSA aisladas de pacientes no hospitalizados o en las primeras 48 horas de internamiento, sin antecedentes de uso de prótesis o uso de antimicrobianos prolongado, fueron considerados MRSAcom.
Extracción del DNA
La extracción del ADN se realizó a partir de un cultivo de la cepa, resuspendiendo 2 a 3 colonias de la bacteria en 200 µL de solución tampón TE (1X), conteniendo 50 µL de Lisozima y 25 µL de Lisostafina, incubándose a 37 ºC por 20 minutos. Se agregó SDS/Proteinasa K (75/5 µL) y se incubó por 1 hora a 37° C; posteriormente se añadieron 84 µL de NaCl 5M y 60 µL de CTAB incubándose a 65º C por 20 minutos. Se adicionaron 750 µL de una solución de Cloroformo/Alcohol Isoamílico (24:1), se centrifugó por 15 minutos a 12000 g y el sobrenadante fue transferido a un nuevo tubo. El DNA fue precipitado con 400 µL de Isopropanol, e incubado a - 20ºC por 30 minutos. La muestra fue centrifugada por 15 minutos a 12000 g., lavada con 1 mL de etanol a 70%, centrifugada por 15 minutos a 12000 g y finalmente secada y resuspendida en 100 µL de solución TE. El DNA fue guardado a 4° C hasta su posterior uso.
Detección de la Leucocidina de Panton Valentine - PVL
La PVL fue identificada mediante PCR, los "cebadores" empleados fueron los diseñados por Lina et al (34) de acuerdo a la secuencia de genes de PVL (Gen Bank N° X72700 y AB006796). Para luk PV-1, 5´-ATCATTAGGTAAAATGTCTGGACATGATCCA-3´ y para luk-PV-2, 5-GCATCAASTGTATTGGATAGCAA
AAGC-3´. La amplificación se realizó en un termociclador Perkin Elmer 9700 (Applied Biosystems), según el siguiente esquema: un ciclo inicial de 94° C por 5min; seguido de 30 ciclos de 94° C por 30 seg, 55° C por 30 seg, 72° C por 1 min; extensión final 72° C por 1 min. Los productos de PCR fueron separados por electroforesis en gel de azarosa al 1,5% conteniendo bromuro de etidio para su posterior visualización con luz UV. Las condiciones de corrida fueron: 60V durante 60 min.
Análisis estadístico
Los datos fueron analizados mediante el programa SPSS versión 15,0, se realizaron tablas de frecuencia y contingencia, se utilizó la prueba de chi cuadrado para el análisis de los datos, se consideró como significativo un valor de p< 0,05.
RESULTADOS
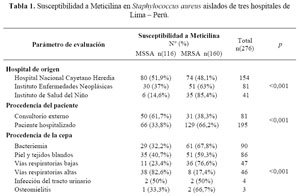
De las 276 cepas de Staphylococcus aureus, 81 provenían de consultorios externos y 195 de pacientes hospitalizados, la mayoría provenían de hemocultivos, heridas de piel y tejidos blandos, con 90 y 86 cepas respectivamente (Tabla 1).
Resistencia a Meticilina
Ciento sesenta cepas (58%) fueron identificadas como MRSA, la distribución de las cepas MRSA y MSSA fue significativamente diferente según el hospital de origen, procedencia del paciente, y procedencia de la cepa (p<0,001). Los MRSA fueron más frecuentes en pacientes hospitalizados (66,2%) en las cepas aisladas de las bacteriemias y de las infecciones de piel y tejidos blandos. Fueron menos frecuentes en las cepas provenientes de vías respiratorias altas (Tabla 1).
Origen de las cepas MRSA
De las cepas MRSA aisladas, 9 (5,6%) fueron catalogadas como adquiridas en la comunidad (MRSAcom), 129 (80,6%) fueron adquiridas en el hospital (MRSAhos) y 22 (13,8%) resultaron indeterminadas al no cumplir con los criterios establecidos por CDC o carecer de información concluyente. De las cepas MRSAcom, 5 fueron aisladas de infecciones en piel y tejidos blandos, 3 de infecciones de vías respiratorias bajas y una cepa de hemocultivo. Se encontró diferencia estadísticamente significativa entre el origen de las cepas y el lugar anatómico de su aislamiento (p<0,001). Seis cepas fueron aisladas en el HNCH y 3 cepas en el INSN.
Presencia de PVL
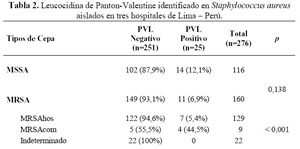
La Leucocidina de Panton Valentine fue identificada en 25 cepas (9,1%); 11 cepas MRSA (6,9%) y 14 cepas de MSSA (12,1%). La mayor proporción de PVL fue identificada en cepas MSSA frente a las cepas MRSA (12,1% vs 6,9%).
En las cepas MRSAhos, 7 cepas (5,4%) resultaron positivas para la PVL, en las cepas MRSAcom 4 (44,5%) fueron positivas para PVL (Tabla 2).
El análisis estadístico mostró diferencia significativa en el origen de las cepas respecto a la presencia de la PVL (p<0,001) y muestra independencia en la susceptibilidad a meticilina respecto a la presencia de PVL (p=0,138) (Tabla 2).
DISCUSIÓN
En la actualidad Staphylococcus aureus se presenta como un patógeno cada vez más importante, ello debido al arsenal de factores de virulencia que presenta el microorganismo sumada a su elevada capacidad de generar resistencia a los antimicrobianos (1-4).
Nuestro estudio muestra niveles elevados de resistencia a meticilina, el 58% de cepas aisladas resultaron MRSA, los reportes en nuestro medio muestran valores de frecuencia variable entre 50 – 90%, pero empleando metodología y poblaciones diversas.
Es importante destacar que las cepas MRSA manifiestan fenotípicamente homoresistencia o heteroresistencia, siendo éste último el más frecuente y en el que solo una mínima cantidad de bacterias en una población (1 en 100 000), expresan el fenotipo resistente, dificultando su identificación in vitro, lo que obliga al empleo de métodos especiales para su identificación. En nuestro estudio se empleó el "Oxacillin Agar Screen", método recomendado por CLSI y que detecta tanto cepas homoresistentes como heteroresistentes, siendo considerado el método fenotípico de referencia. Con ésta misma metodología, en 1996, en un estudio en 6 hospitales de Lima, encontramos 63,3% de MRSA, resultados similares a los actuales, considerando además que ambos fueron estudios multicéntricos (11).
El incremento en la prevalencia de infecciones por MRSA, está conduciendo a un mayor uso de los glucopéptidos, lo que a su vez origina un incremento notable de los costos de tratamiento de estas infecciones, pero más grave aún es la presión selectiva antibiótica que conduce a la generación de resistencia a estas drogas, que se evidencia en la actualidad por la aparición de las cepas con resistencia intermedia a Vancomicina – VISA y las cepas con resistencia absoluta – VRSA, como lo muestran diversos reportes (21-24).
Tradicionalmente, el Staphylococcus aureus meticilino resistente (MRSA) ha sido considerado un patógeno nosocomial, con características propias y factores predisponentes bien determinados. Sin embargo en la última década aparecen infecciones por cepas MRSA con características fenotípicas y genotípicas diferentes, denominándoseles como adquiridos en la comunidad (MRSAcom). Este nuevo patógeno constituye una mayor preocupación que las cepas convencionales de MRSA (9).
Nuestro estudio identificó, en base a los criterios de CDC, 5,6% de cepas MRSAcom, ninguno de los pacientes presentaba antecedentes relevantes de contacto directo o indirecto con ambientes hospitalarios, permanencia en instituciones de cuidados crónicos, ni de uso prolongado de antibióticos, que hicieran sospechar la infección previa por MRSA. Las cepas aisladas provenían de infecciones en piel y partes blandas, neumonías y septicemia. Estos datos coinciden plenamente con las características de los MRSAcom cuyo espectro de infecciones va desde los cuadros cutáneos locales (forúnculos, abscesos, celulitis) hasta enfermedad sistémica severa incluyendo la neumonía necrotizante, bacteriemia y abscesos profundos (13-16).
Este estudio constituye el primer reporte de MRSAcom en nuestro país, su frecuencia es aún baja si la comparamos a reportes en el resto del mundo, incluso países de Sudamérica en los que se han reportado niveles crecientes, llevando a calificarlo como un problema emergente, obligando a implementar diversas medidas para su control (25-27). Sin embargo, el patógeno está presente en nuestro medio y amerita programas de vigilancia epidemiológica constante y la implementación de medidas que permitan el control del problema, más aún si existe el riesgo que estas cepas se diseminen de la comunidad al ambiente hospitalario, con niveles insospechados de mortalidad y consecuencias mucho más severas, dado el mayor compromiso de los pacientes hospitalizados, como ya han sido descritos en Alemania, Israel, Reino Unido y EUA (28).
Diversos reportes asignan a los MRSAcom propiedades particulares como su mayor velocidad de replicación, mayor patogenicidad como consecuencia de factores de virulencia adicionales como la Leucocidina de Panton-Valentine, citotoxina formadora de poros, que produce destrucción de los leucocitos mononucleares y polimorfonucleares, causando la muerte celular por necrosis o apoptosis, la necrosis tisular en la neumonía necrotizante y las infecciones severas de piel, partes blandas, siendo propuesta como marcador característico de las cepas MRSAcom (15-20). Por esta razón se incorporó en el estudio la identificación de esta proteína.
Nuestro estudio muestra que la PVL, no es exclusividad de las MRSAcom, el factor de virulencia fue identificado tanto en cepas resistentes como susceptibles a meticilina. Así mismo fue encontrada en cepas MRSA de origen hospitalario como adquiridas en la comunidad. Recientes estudios han puesto en discusión la utilidad de la PVL como marcador de las cepas MRSAcom, Rossney et al. (29), en un estudio realizado en Irlanda, encontraron que solo 6,7% de los MRSAcom aislados resultaron positivos para PVL, concluyendo que esta proteína no deberìa ser usado como un único marcador para MRSAcom, siendo necesaria la combinación de distintas características fenotípicas y que se requieren posteriores estudios que definan un marcador confiable o una combinación de marcadores que faciliten su reconocimiento (30).
Los resultados del estudio nos permiten concluir que en nuestro medio están presentes cepas de Staphylococcus aureus de elevada virulencia, aun en baja frecuencia, pero que amerita la implementación de medidas de control oportunas y efectivas. Así mismo que la PVL no constituye un marcador suficiente para la identificación de cepas de MRSAcom.
REFERENCIAS BIBLIOGRAFICAS
1.Archer GL. Staphylococcus aureus: a well-armed pathogen. Clin Infect Dis 1998;26:1179-81. [ Links ]
2.Murray P. Manual of clinical microbiology. 9th edition. Washington D.C: American Society for Microbiology; 2007. [ Links ]
3.Isenberg H. Clinical microbiology procedures handbook. 2nd Ed. Washington D.C.:American Society for Microbiology; 2004. [ Links ]
4.Honeyman A, Friedman H, Bendinelli M.Staphylococcus aureus infection and disease.New York:Kluwer Academic Publishers; 2002. [ Links ]
5.Chambers H. Methicillin-Resistant Staphylococci. Clin Microbiol Reviews 1988; 1(2): 173-186. [ Links ]
6.Chambers H. Methicillin Resistance in Staphylococci: Molecular and biochemical basis and clinical implications. Clin Microbiol Reviews 1997; 10(4): 781–791. [ Links ]
7.Cosgrov SE, Sakoulas G, Perencevich EN, Schwaber MJ, Karchmer AW, Carmeli Y. Comparison of mortality associated with methicillin-resistant and methicillin-susceptible Staphylococcus aureus bacteremia: a metaanalysis. Clin Infect Dis 2003;36:53-9. [ Links ]
8.Engemann JJ, Carmeli Y, Cosgrove SE, et al. Adverse clinical and economic outcomes attributable to methicillin resistance among patients with Staphylococcus aureus surgical site infection. Clin Infect Dis 2003;36:592-8. [ Links ]
9.Flynn N, Cohen S. The Continuing Saga of MRSA. J Infectious Diseases 2008; 197:1217-9. [ Links ]
10.Echevarria J, Iglesias D. Estafilococo Meticilino resistente, un problema actual en la emergencia de resistencia entre los Gram positivos. Rev Med Hered 2003; 14:195-203. [ Links ]
11.Echevarria J, Ore L, Zerpa R, et al. Prevalence of Methilillin Resistant Staphylococcus strains, in Hospitalized patients and susceptibility to Teicoplanin in Lima Peru. Sidney – Australia: 20th International Congress of Chemotherapy. International Society of Chemotherapy; Junio 1997. [ Links ]
12.Deurenberg R, Stobberingh E. The evolution of Staphylococcus aureus. Infection, genetics and evolution 2008;8(6):747-763. [ Links ]
13.Saïd-Salim B, Mathema B, Kreiswirth BN. Community-acquired methicillin-resistant Staphylococcus aureus: an emerging pathogen. Infect Control Hosp Epidemiol 2003; 24:451-5. [ Links ]
14.Buck JM, Como-Sabetti K, Harriman KH, et al. Community-associated methicillin-resistant Staphylococcus aureus, Minnesota, 2000–2003. Emerg Infect Dis 2005; 11(10):1532–8. [ Links ]
15.Tenover F, McDougal L, Goering R, et al. Characterization of a Strain of Community-Associated Methicillin-Resistant Staphylococcus aureus Widely Disseminated in the United States. J Clin Microbiol 2006; 44:108-118. [ Links ]
16.Huang H, Flynn N,. King J, Monchaud C, Morita M, Cohen S. Comparisons of Community-Associated Methicillin-Resistant Staphylococcus aureus (MRSA) and Hospital-Associated MSRA Infections in Sacramento,California. J Clin Microbiology 2006; 44: 2423–2427. [ Links ]
17.Boubaker K, Diebold P, Blan DS, et al. Panton–Valentine leucocidin and staphyloccoccal skin infections in schoolchildren. Emerg Infect Dis 2004; 10(1): 121-124. [ Links ]
18.Diep B, Sensabaugh G, Somboona N, Carleton H, Perdreau Remington F. Widespread skin and soft-tissue infections due to two Methicillin- Resitant Staphylococcus aureus Strains Harboring the Genes for Panton – Valentine Leucocidin. J Clin Microbiology 2004; 42: 2080-2084. [ Links ]
19.Yves G, Bertrand I. Association between Staphylococcus aureus strains carrying gene for Panton – Valentine Leucocidin and hightly lethal necroting pneumonia in young immunocomponent patients. Lancet 2002; 359:753-59. [ Links ]
20.Wannet W, Spalburg E, Heck M, et al. Emergence of Virulent Methicillin-Resistant Staphylococcus aureus Strains Carrying Panton-Valentine Leucocidin Genes in The Netherlands. J Clin Microbiology 2006; 43: 3341– 3345. [ Links ]
21.Hiramatsu K. Reduced Susceptibility of Staphylococcus aureus to Vancomycin Japan, 1996. MMWR 1997; 46: 624-626. [ Links ]
22.Hiramatsu K, Aritaka N, Hanaki H, Kawasaki S, Hosoda Y, Hori S. Vancomycin-resistant Staphylococcus aureus: dissemination of heterogeneously resistant strains in Japanese hospital. Lancet 1997; 350: 1670-1673. [ Links ]
23.Kantzanou M, Tassios PT, Tseleni-Kotsovili A, Legakis NJ, Vatopoulos AC. Reduced susceptibility to vancomycin of nosocomial isolates of methicillin-resistant Staphylococcus aureus. J Antimicrob Chemother 1999; 43: 729-731. [ Links ]
24.Rotun SS, McMath V, Schoonmaker J, et al. Staphylococcus aureus with reduced susceptibility to vancomycin isolated from a patient with fatal bacteremia. Emerg Infec Dis 1999; 5; 147-149. [ Links ]
25.Paganini H, Della Latta M, Muller Opet B, et al. Estudio multicéntrico sobre las infecciones pediátricas por Staphylococcus aureus meticilino-resistente provenientes de la comunidad en la Argentina. Arch Argent Pediatr 2008; 106(5):397-403. [ Links ]
26.Ribeiro A, Dias C, Silva-Carvalho MC, et al. First report of infection with community-acquired methicillin-resistant Staphylococcus aureus in South America. J Clin Microbiol 2005; 43:1985-8. [ Links ]
27.Pereira W, Galiana A. Una nueva clona de Staphylococcus aureus con resistencia a Meticilina y alta virulencia emergente en la comunidad y en los grandes hospitales en Uruguay. Montevideo, Uruguay: Instituto de Higiene- Universidad de la República; 2002. [ Links ]
28.Strommenger B, Braulke C, Pasemann B, Schmidt C, Witte W. Multiplex PCR for Rapid Detection of Staphylococcus aureus Isolates Suspected to Represent Community-Acquired Strains. J Clin Microbiology 2008; 46: 582–587. [ Links ]
29.Rossney A, Shore A, Morgan P, Fitzgibbon M, OConnell B, Coleman D. The Emergence and Importation of Diverse Genotypes of Methicillin-Resistant Staphylococcus aureus (MRSA) Harboring the Panton-Valentine Leukocidin Gene (pvl) Reveal that pvl Is a Poor Marker for Community-Acquired MRSA Strains in Ireland. J Clin Microbiology 2007; 45: 2554–2563. [ Links ]
30.Takizawa Y, Taneike I, Nakagawa S, et al. A Panton- Valentine Leucocidin (PVL)-Positive Community- Acquired Methicillin-Resistant Staphylococcus aureus (MRSA) Strain, Another Such Strain Carrying a Multiple-Drug Resistance Plasmid, and Other More-Typical PVL-Negative MRSA Strains Found in Japan. J Clin Microbiology 2006;43: 3356–3363. [ Links ]
Correspondencia:
Jesús Humberto Tamariz Ortiz
Facultad de Medicina Alberto Hurtado
Universidad Peruana Cayetano Heredia
Av. Honorio Delgado Nº 430 – San Martin de Porres
Lima Peru.
Correo electrónico: jesus.tamariz@hotmail.com