Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de Gastroenterología del Perú
Print version ISSN 1022-5129
Rev. gastroenterol. Perú vol.29 no.3 Lima July/set. 2009
ARTÍCULO DE REVISIÓN
Métodos de investigación molecular en la detección de mutaciones germinativas en cáncer colorrectal hereditário
Molecular research methods in the detection of germinal mutations in hereditary colorectal cancer
MV Dominguez1,2 ; EP Bastos1 ; SD Silva3 ; BM Rossi2
1 Laboratorório de Genómica y Biologia Molecular del Centro de Investigaciones del Hospital AC Camargo, São Paulo, Brazil.
2 Departmento de Cirugia Pélvica del Hospital AC Camargo, São Paulo, Brazil.
3 Departmento de Cabeza y Cuello y Otorrinolaringologia del Hospital AC Camargo, São Paulo, Brazil.
RESUMEN
El cáncer colorrectal (CCR) es una de las principales causas de muerte por neoplasia en países sudamericanos. Las formas hereditarias de CCR son, poliposis adenomatosa familiar (FAP) y cáncer colorrectal hereditario sin poliposis (HNPCC) o Síndrome de Lynch (SL), que es la forma más común. La detección de mutaciones en los genes de reparo de ADN (MMR) y en el gen APC permite el desarrollo de estrategias de prevención. Algunas de estas metodologías de diagnóstico molecular son aplicadas para la investigación y detección de mutaciones en estos genes, tales como el Test de la Proteína Truncada (PTT), Análisis de polimorfismos de conformación de cadena simple (SSCP), Cromatografia liquida desnaturante de alta performance (DHPLC) y Reacción en Cadena de la Polimerasa (PCR) en tiempo real (qPCR).
PALABRAS CLAVES: mutaciones germinativas, SL, FAP, PTT, SSCP, DHPLC, qPCR.
SUMMARY
Colorectal cancer (CRC) is one of the main causes of death in South American countries. The hereditary forms of CRC are, familial adenomatous (FAP) and hereditary nonpolyposis colorectal cancer (HNPCC) or Lynch Syndrome (LS), which is the most common form. The detection of mutations in the DNA repair genes (MMR) and in the APC genes enables the development of prevention strategies. Some of these methods for molecular diagnosis are applied in research and the detection of mutations of these genes, such as the partial thromboplastin time test (PTT), the single strand conformational polymorphism test (SSCP), the Denaturing High Performance Liquid Chromatography test (DHPLC) and the Polymerase Chain Reaction (PCR) in real time (qPCR).
KEY WORDS: Germinal mutations, LS, FAP, PTT, SSCP, DHPLC, qPCR.
INTRODUCCIÓN
Las mutaciones constitucionales deletéreas en genes involucrados en el reparo del ADN o control de la proliferación celular están frecuentemente asociadas a un alto riesgo de cáncer hereditario (1,2). La poliposis adenomatosa familiar (FAP) y el cáncer colorrectal hereditario sin poliposis (HNPCC) o Síndrome de Lynch (SL) son síndromes de cáncer colorrectal (CCR) asociados a defectos hereditarios en genes que regulan la proliferación celular y adhesión, APC, MUTYH, o genes de reparo de ADN (MMR), MLH1, MSH2, MSH6, PMS2 y PMS1 (3, 4, 5).
En familias FAP, la mutación en el gen APC está asociada a numerosos pólipos adenomatosos, con un riesgo de aproximadamente 100% de desarrollar CCR durante la vida y con una edad media de 40 años. El fenotipo atenuado de FAP se presenta en un inicio tardío, con pocos pólipos, presencia de tumores en el lado derecho y está asociado a mutaciones en el exón 9 y terminaciones 5´o 3´ del gen APC, o también con alguna mutación bialélica del gen MUTYH. Los portadores de mutación en el gen APC presentan un riesgo incrementado de desarrollar otros tumores, principalmente tumores desmoides benignos (5).
Mutaciones en los genes MMR ligados a SL, están asociados con el desarrollo temprano de CCR (<50 años) e instabilidad de microsatélites (IMS) en tejidos malignos, como consecuencia de la pérdida del alelo de tipo salvaje durante la oncogénesis. Un amplio espectro tumoral es frecuentemente observado en el contexto de las mutaciones de los genes MLH1 y MSH2, principalmente cáncer de estómago, endometrio, pelvis renal, intestino delgado, entre otros. El gen MSH2 representa aproximadamente 40% de las mutaciones de los genes MMR asociados a CCR, entre mutaciones puntuales y grandes deleciones genómicas. Raros homocigotos de inicio temprano de CCR han sido observados para los genes MLH1, MSH2 y PMS2. Estos homocigotos podrían presentar fenotipos adicionales, entre malignidades hematológicas y neurológicas en la infancia, y señales intermediarias de neurofi bromatosis en homocigotos del gen MLH1. Dobles heterocigotos también han sido observado en diferentes combinaciones de genes, como: APC más MSH6, MSH6 más MUTHY, y MSH6 más PMS2, y un caso conocido de APC más MSH2. En estos casos, el diagnóstico de cáncer de colon se debe realizar en una edad más temprana (3).
La identificación de mutaciones germinativas mediante la técnica de secuenciación directa consume mucho tiempo y no es aplicable para protocolos o estudios clínicos a gran escala. Por tanto, estrategias de investigación molecular, comprendiendo el test de la proteína truncada (PTT), análisis de polimorfismo de conformación de cadena simple (SSCP), cromatografía liquida desnaturante de alta performance (DHPLC) y Reacción en cadena de la Polimerasa (PCR) en tiempo real (qPCR) han sido descritos para facilitar la detección de mutaciones. Idealmente, estos métodos son usados para el análisis de mutaciones de un gran número de fragmentos de ADN y son caracterizados por presentar una alta sensibilidad, especificidad, y ser considerados como métodos inofensivos, relativamente baratos y totales o semi-automatizados para minimizar costos y tiempo (6).
En el presente estudio, nosotros revisamos 4 métodos de investigación para mutaciones germinativas en SL y FAP, tales como PTT, SSCP DHPLC, y qPCR.
MÉTODOS DE SCREENING
Test de la proteína truncada (PTT)
El PTT está basado en la transcripción y traducción in vitro de una región amplificada de un gen o mRNA alvo mediante PCR de transcripción reversa (RT-PCR), seguido de un análisis para fragmentos de polipeptideos menores a partir del produto de PCR (7). Las proteínas de bajo peso molecular surgen como consecuencia de un truncamiento (framsehift) o por mutaciones de parada (stop códon) en el gen analizado, las cuales difi eren de las proteínas de tamaño completo esperado. El PTT está compuesto por 4 pasos: el primero, la separación de ácidos nucleicos: genómico, ARN total o poly (A)+ ARN Segundo, amplificación de una región especifica del gen de interés. Tercero, transcripción y traducción in vitro del producto de reacción de amplificación; y finalmente el cuarto paso, detección de los productos traducidos. Los productos proteicos truncados (alelos mutantes) son fácilmente distinguidos del producto proteico de tamaño completo (alelo normal) (Figura 1) (8). Adicionalmente, PTT facilitó la detección de nuevos tipos de mutación, debido a que un cambio en la secuencia genera una región altamente mutable y detectable en la superficie del ARN (9).
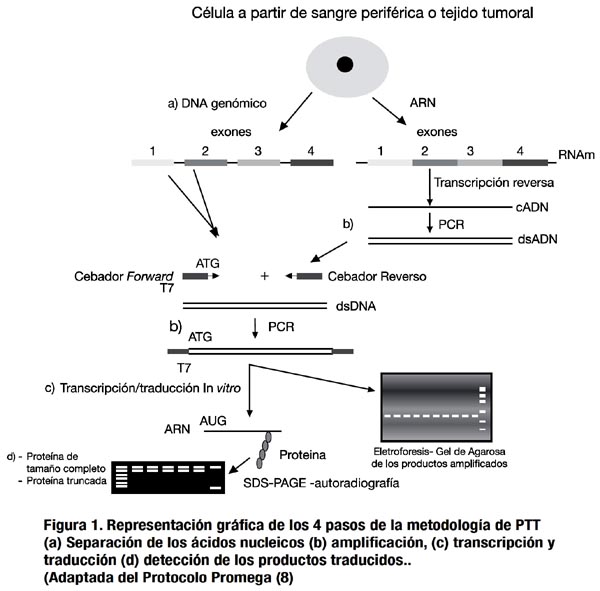
Las primeras mutaciones publicadas en los genes PMS1 y PMS2 en pacientes HNPCC fueron originalmente detectadas por PTT. Tao y colaboradores, (2001) utilizaron la técnica de detección basada en RT-PCR acoplada a PTT como punto de partida. Rastrearon 84 familias no relacionadas para el análisis de mutaciones germinativas en los genes PMS1 y PMS2. No fueron encontradas mutaciones en el gen PMS2. Cinco bandas aberrantes fueron identificadas para el gen PMS1 en siete familias (seis de Finlandia y una de Italia). La secuenciación de cDNA reveló una deleción en todas esas familias (10). Recientemente, Hes y colaboradores (2008) utilizaron la metodología de PTT para elucidar la magnitud de la mutación en el gen APC en familias FAP. Mediante el PTT, fue encontrado 5% de casos nuevos de FAP identificados como mosaicos, probablemente debido al número significativo de mutaciones en el gen APC. Estos estudios demuestran que PTT es una importante herramienta para rastrear mutaciones de sospecha de neoplasia (11,12,13).
Análisis de polimorfismo de conformación de cadena simple (SSCP)
El principio de SSCP se basa en la conformación secundaria a partir de moléculas de ADN de cadena simple basadas en secuencias específicas sobre condiciones no desnaturantes. Las moléculas de tamaño normal pueden diferenciarse de las pequeñas con una simple substitución de base, formando diferente conformación y migración en un gel de poliacrilamida no desnaturante (Figura 2) (14).
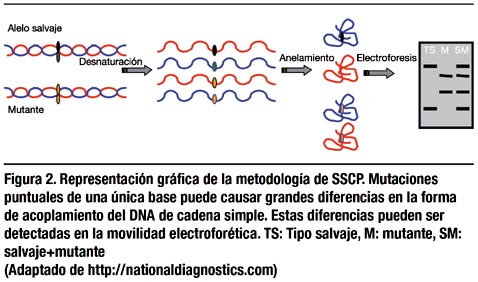
La sensibilidad de SSCP varía dramáticamente de acuerdo al tamaño del fragmento de ADN. Por lo tanto, el producto de PCR tiene que ser preferentemente pequeño (aproximadamente 150pb) para aplicar satisfactoriamente esta metodología en la detección de mutaciones. Otro requisito importante, es la selección de cebadores de PCR (fl anquean la región de interés) o la digestión de productos grandes de PCR en sitios de internos de restricción. Cuando la investigación para polimorfismos no es crítica, el análisis de fragmentos tamaño grande puede ser realizado. La sensibilidad de SSCP es de aproximadamente 80 a 90%, siempre y cuando se cumplan con todas las condiciones (15).
Isidro y colaboradores (2003) detectaron mediante SSCP y DHPLC, 4 mutaciones nuevas en el gen MLH1 y un nuevo polimorfismo silencioso en el gen MSH2 en familias portuguesas de Lynch (16). De la Fuente y colaboradores (2007) detectaron 87% de mutaciones en el gen APC por SSCP; de las cuales, más del 50% no habían sido descritas. El alto porcentaje de mutaciones nuevas encontradas puede ser por la composición genética de la población chilena, la cual, es una mistura de amerindios y españoles (17).
En la población española, la mutación en el codón 1061 del gen APC es una de las más comunes en las Islas Baleáricas. Aunque este codón sea un hot-spot, el análisis de haplotipo de estas familias es consistente con la presencia del efecto fundador de la deleción de 5pb en el codón 1061 en familias FAP de las Islas Baleáricas españolas (18). La relevancia de estos estudios es tener un mayor conocimiento de la susceptibilidad al cáncer hereditario, y la posibilidad de identificar mediante el diagnóstico molecular a los familiares de alto riesgo (19).
El número de pólipos rectales y las regiones de las mutaciones en el gen APC están siendo utilizados para direccionar el tratamiento quirúrgico en pacientes con FAP familiar. Por lo tanto, la identificación de mutaciones germinativas en los genes APC o MYH puede direccionar la selección del tratamiento quirúrgico (20).
Cromatografia Líquida Desnaturante de Alta Performance (DHPLC)
En sus inicios, esta metodología presentó una efectiva aplicación para la separación de oligonucleotidos, fragmentos de PCR y análisis de productos formados a partir de RT-PCR para determinar los niveles de expresión génica.
Esta técnica está basada en la detección automatizada de segmentos de DNA mediante una cromatografía liquida de alta performance de fase íonica-pareada reversa. El principio de DHPLC se basa en la desnaturación y reanelamiento de amplicones de tipo salvaje y mutado y la comparación entre sus perfiles (21).
Para alcanzar resultados más confiables, se sugiere un análisis preliminar de la cualidad de los amplicones a temperaturas no desnaturantes de 50ºC. En estas condiciones, la presencia de mutaciones de pequeñas repeticiones también puede ser detectadas (Figura 3).
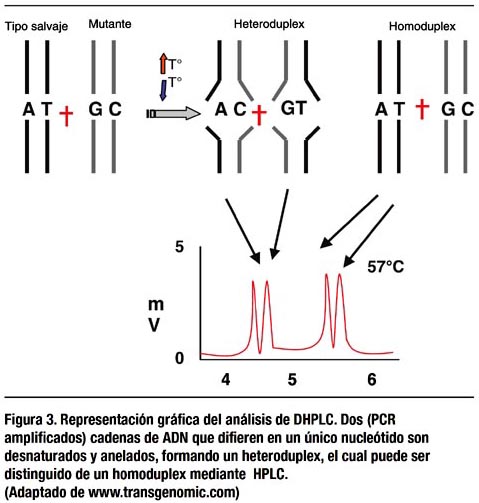
Los productos de PCR no necesitan ser purificados para el análisis por DHPLC. El analizador de DHPLC (Transgenomic Wave) presenta una manipulación automatizada de 96 muestras simultáneamente, lo que disminuye la mano de obra del operador. Aproximadamente, 10ul (50-200ng) de producto de PCR son utilizados para el análisis mediante DHPLC y consecuentemente, el producto remanente de PCR (20-50ul) puede ser usado para purificación, secuenciación u otro procedimiento experimental.
En conclusión, esta metodología parece ser adecuada para el análisis de mutaciones como rutina de laboratorio (22,23).
El análisis mediante DHPLC también puede ser usado satisfactoriamente para la detección de mutaciones y polimorfismos en el cromosoma Y, factor IX, genes de la neurofibromatosis tipo 1, regulador de conductancia transmembrana de la fibrosis quística (CFTR), homólogos de la fosfatasa y tensina en el cromosoma 10 (PTEN) y en los genes BRCA1 y BRCA2 (24,25,26).
PCR en tiempo real (qPCR)
La metodología de PCR en tiempo real (qPCR) direcciona un análisis de datos cuantitativos de expresión génica en estudios biológicos, de medicina, biotecnología, microbiología y diagnóstico de enfermedades (27). qPCR utiliza colorantes reporter fluorescentes para combinar la amplificación y los pasos de detección de la reacción de PCR en un único paso (28,29). El ensayo mide el incremento de la señal fluorescente, la que es proporcional a la cantidad de ADN procesado durante cada ciclo de PCR (27) (Figura 4).
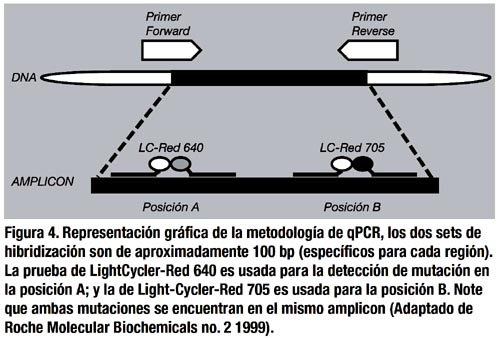
Los instrumentos requeridos en un ensayo de qPCR consisten de un termociclador y un fluorimetro de microvolumen que permite la rápida detección de mutaciones con una alta acurácia. Las alteraciones de múltiples bases son discriminadas mediante el uso de pruebas de hibridización y curvas de melting fluorescentes. A través del análisis de estas curvas, este sistema proporciona una única e innovada aproximación para detectar polimorfi smos de una única base (SNPs). El análisis de la curva de melting monitora la hibridización, que se trata de la temperatura-dependiente de una única cadena. Las pruebas de hibridización se ligan perfectamente al ADN alvo marcado, requiriendo una alta temperatura de melting (Tm) para separar las pruebas de hibridización unidas al ADN que contiene errores de desestabilización, es esta diferencia la base de la detección de mutaciones. La prueba de hibridización, comprendiendo la región de una mutación potencial (mutation probe), tiene un bajo Tm que la prueba adyacente (anchor probe), asegurando el señal fluorescente generado durante la curva de melting, ( que es determinado sólo por la prueba de mutación). Los híbridos Probe/ADN contienen el melt de error a bajas temperaturas que los híbridos marcados perfectamente. Esto refleja las diferentes Tms durante el análisis de la curva de melting. Por lo tanto, el tipo salvaje, mutante, la mezcla de ellos (por ejemplo, genotipo heterocigoto) pueden ser distinguidos mediante las diferencias que presentan en sus respectivas temperaturas de melting. El análisis de la curva de melting es realizada inmediatamente después del paso de amplificación (30).
Por lo tanto, qRT_PCR es aplicado para una rápida detección de genes involucrados en neoplasias humanas y enfermedades infecciosas. En la investigación de la selección de de métodos, este es el principal método para una cuantifi cación de la expresión génica. La disponibilidad práctica y el análisis son simples y puede ser fácilmente extendible al ambiente clínico a diferencia de aquellas técnicas de PCR tradicionales (Figura 5).
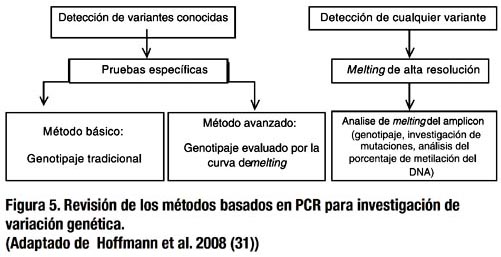
En CCR, la mutación V600E del gen BRAF, codificador de quinasas pertenecientes a la vía de señalización RAS/ RAF/MAPK, sugiere un origen esporádico de esta enfermedad, excluyendo los criterios para cáncer colorrectal hereditario (tipo FAP y SL).
La combinación de las pruebas de TaqMan MGB con las condiciones de un ensayo universal permite un eficiente flujo de trabajo para el análisis de grandes números de diferentes SNPs en laboratorios clínicos (32).
DISCUSIÓN Y CONCLUSIÓN
El análisis de mutaciones en pacientes con padrón hereditario de CCR, generalmente se basa en el análisis de la secuencia genómica de ADN, las que pueden ser rastreadas por diferentes metodologías. La identificación, acurácia e interpretación de mutaciones de los portadores son esenciales para el manejo clínico de los pacientes con CCR, y para investigaciones científicas (2).
Una de las primeras metodologías desarrolladas en esta área es PTT, la cual presenta varias características importantes como una alta sensibilidad y un bajo porcentaje de falsos positivos. El principal problema técnico de esta metodología está relacionado con el hecho que, generalmente PTT usa un alvo de ARN, presentando dificultades en la potencial expresión génica diferencial y estabilidad de los transcritos resultantes de los alelos estudiados. Esta desventaja puede ser como consecuencia de las modificaciones en el tipo captura del gen así como el uso de PTT digital, que disminuye el límite de detección de mutaciones y permite el uso de PTT en la detección de mutaciones de fluidos corporales (9). Otra desventaja de PTT convencional es el uso de marcadores radioactivos para la detección de proteínas. Recientemente, modificaciones en el tipo marcadores fluorescentes o el uso de epitopes alvos fueron establecidos, lo que permite la detección del producto traducido no radioactivo. Cuando varios epitopes son usados en diferentes campos de lectura, el espectro de detección de mutación puede ser expandido para todas las posibles mutaciones del tipo frameshift (10). Estas modificaciones permiten que PTT sea una poderosa técnica no radioactiva que permite detectar mutaciones con una alta sensibilidad.
Clínicamente, utilizar técnicas de rastreamiento tal como SSCP y DHPLC es más aceptable en el análisis genético de familias FAP y HNPCC (16). La mayor ventaja de DHPLC es la facilidad de rastrear un gran número de muestras evitando el uso de electroforesis, preparación y análisis de gel. Este sistema es idealmente utilizado para detección de los genes MLH1 y MSH2 en la rutina laboratorial, debido a que (a) es rápido, tomando aproximadamente de 5-7 minutos por muestra, (b) presenta una alta acurácia, (c) es económico, costando aproximadamente $0,7 por muestra y (d) es menos laborioso, DHPLC presenta la ventaja de genotipar una gran cantidad de muestras sin supervisión humana y puede funcionar durante la noche, generando resultados automáticamente.
Si las condiciones de PCR son cuidadosamente optimizadas y la inyección del amplicón es única y pura, el padrón de análisis muestra un único y reconocible pico, previniendo así la necesidad de ser analizados por dos observadores independientes (21). Comparando DHPLC y otras metodologías para el mismo propósito, DHPLC tiene la ventaja de presentar una facilidad en el manejo de las muestras y tiene problemas significantemente mínimos de interpretación de datos. Además de sus aplicaciones para la detección de mutaciones en los genes MLH1 y MSH2, DHPLC es una herramienta poderosa para análisis clínicos y poblacionales de MMR en general (25,26).
Otra metodología recientemente descrita para el rastreamiento de mutaciones, es qPCR. Desde su introducción, qPCR revolucionó el campo del diagnóstico molecular y está siendo aplicada en un número cada vez mayor de experimentos clínicos. Las ventajas de este procedimiento en comparación a los métodos convencionales incluyen, su alta sensibilidad, rango dinámicamente grande, y su acurácia en la cuantificación, lo que incrementa su sensibilidad (27,28,29). Esta metodología es simple, rápida y reproducible para uso en aplicaciones clínicas, laboratoriales. La alta tecnología de qPCR ha permitido el rastreamiento de mutaciones no conocidas o modificaciones de ADN (31,32). Actualmente, la detección de mutaciones es realizada mediante el análisis de tamaño de fragmento de restricción (33), PCR de alelo específico (35), análisis de SSCP (34), y secuenciación automatizada directa. Estos últimos métodos son ampliamente aceptados como estándar, a pesar de consumir bastante tiempo.
La metodología de qPCR presenta ventajas en costo y labor, y es una alternativa de la técnica de secuenciación directa automatizada, debido a sus medidas seriales, pesar de la secuenciación directa sea más eficiente, es más laboriosa, más cara en términos de reactivos y análisis de datos y no es 100% eficiente.
REFERENCIAS
1. BRIEGER A, TROJAN J, RAEDLE J, ROTH WK, ZEUZEM S. Identification of germline mutations in hereditary nonpolyposis colorectal cancer using base excision sequence scanning analysis. Clin Chem. 1999; 45(9):1564-7.
2. HAMPEL H, FRANKEL WL, MARTIN E, ARNOLD M, KHANDUJA K, KUEBLE P, NAKAGAWA H, SOTAMAA K, PRIOR TW, WESTMAN J, PANESCU J, FIX D, LOCKMAN J, COMERAS I, DE LA CHAPELLE A. Screening for the Lynch syndrome (hereditary nonpolyposis colorectal cancer). N Engl J Med. 2005; 352(18):1851-60.
3. UHRHAMMER N, BIGNON YJ. Report of a family segregating mutations in both the APC and MSH2 genes: juvenile onset of colorectal cancer in a double heterozygote. Int J Colorectal Dis. 2008;23(11):1131-5.
4. NICOLAIDES, N. C., PAPADOPOULOS, N., LIU, B., WEI, Y. F., CARTER, K. C., RUBEN, S. M., ROSEN, C. A., HASELTINE, W. A., FLEISCHMANN, R. D., FRASER, C. M., et al. Mutations of two PMS homologues in hereditary nonpolyposis colon cancer. Nature . 1994; 371: 75–80,.
5. LEACH, F. S., NICOLAIDES, N. C., PAPADOPOULOS, N., LIU, B., JEN, J., PARSONS, R., PELTOMAKI, P., SISTONEN, P., AALTONEN, L. A., NYSTROM-LAHTI, M., et al. Mutations of a mutS homolog in hereditary nonpolyposis colorectal cancer. Cell.1993;75: 1215–1225.
6. RANGEL MC, DA SILVA SD, PEREIRA NC, DOMINGUEZ MV. Essentials of Molecular Biology in Cancer Research. Applied Can Res. 2008; 28(1):2-10.
7. OLIVER HAUSS, OLIVER MULLER, Protein Truncation Test in Mutation Detection and Molecular Diagnosis. Springer Protocols. 2007; 375: 151-164,.
8. Promega Protocol. The Protein Truncation Test chapter Four, www.promega.com/
9. SADANAND GITE, MARK LIM, RICK CARLSON, JERZY OLEJNIK, BARBARA ZEHNBAUER, KENNETH ROTHSCHILD. A high-throughput nonisotopic protein truncation test. Nature Biotechnology. 2003; 21: 194 – 197,
10. TAO LIU, HAI YAN, SHANNON KUISMANEN, ANTONIO PERCESEPE, MARIE-LOUISE BISGAARD, MONICA PEDRONI, PIERO BENATTI, KENNETH W. KINZLER, BERT VOGELSTEIN, MAURIZIO PONZ DE LEON, PA¨IVI PELTOMAKI, AND ANNIKA LINDBLOM. The Role of hPMS1 and hPMS2 in Predisposing to Colorectal Cancer. Cancer Reasearch. 2001; 61: 7798–7802,.
11. HES FJ, NIELSEN M, BIK EC, KONVALINKA D, WIJNEN JT, BAKKER E, VASEN HF, BREUNING. Somatic APC mosaicism: an underestimated cause of polyposis coli. Gut. 2008;57(1):71-6.
12. TRAVERSO G, B.A., ANTHONY SHUBER, M.S., BERNARD LEVIN, M.D., CONSTANCE JOHNSON, R.N., M.S., LOUISE OLSSON, M.D., DAVID J. SCHOETZ, JR., M.D., STANLEY R. HAMILTON, M.D., KEVIN BOYNTON, B.S., KENNETH W. KINZLER, PH.D., AND BERT VOGELSTEIN, M.D. Detection of APC mutation in fecal DNA from patients with Colorectal tumor. N Engl J Med. 2002; 346(5):302-4.
13. JOHAN T. DEN DUNNEN *, GERT-JAN B. VAN OMMEN The protein truncation test: A review. Van Ommen Human Mutation 1999;14(I2):95 – 102,
14. ATHA DH, KASPRZAK W, O'CONNELL CD, SHAPIRO BA. Prediction of DNA single-strand conformation polymorphism: analysis by capillary electrophoresis and computerized DNA modeling. Nucleic Acids Res. 2001;29(22):4643-53.
15. SHEFFIELD VC, BECK JS, KWITEK AE, SANDSTROM DW, STONE EM. The sensitivity of single-strand conformation polymorphism analysis for the detection of single base substitutions. Genomics. 1993;16(2):325-32.
16. ISIDRO G, MATOS S, GONÇALVES V, CAVALEIRO C, ANTUNES O, MARINHO C, SOARES J,BOAVIDA MG. NOVEL MLH1 mutations and a novel MSH2 polymorphism identified by SSCP and DHPLC in Portuguese HNPCC families. Hum Mutat. 2003;22(5):419-20.
17. DE LA FUENTE MK, ALVAREZ KP, LETELIER AJ, BELLOLIO F, ACUÑA ML, LEÓN FS, PINTO E, CARVALLO P, LÓPEZ-KÖSTNER F. Mutational screening of the APC gene in Chilean families with familial adenomatous polyposis: nine novel truncating mutations. Dis Colon Rectum. 2007;50(12):2142-8.
18. GONZÁLEZ S, BLANCO I, CAMPOS O, JULIÀ M, REYES J, LLOMPART A, CABEZA E, GERMÀ JR, Obrador A, Capellá G. Founder mutation in familial adenomatous polyposis (FAP) in the Balearic Islands. Cancer Genet Cytogenet. 2005;158(1):70-4.
19. BELLOLIO R F, ALVAREZ V K, DE LA FUENTE L M, LEÓN G F, FULLERTON M DA, SOTO D G, CARVALLO DE S Q P, LÓPEZ-KÖSTNER F. Hereditary colorectal cancer: Molecular analysis of APC and MLH1 genes. Rev Med Chil. 2006;134(7):841-8.
20. CASEY G, LINDOR NM, PAPADOPOULOS N, THIBODEAU SN, MOSKOW J, STEELMAN S, BUZIN CH, SOMMER SS, COLLINS CE, BUTZ M, ARONSON M, GALLINGER S, BARKER MA, YOUNG JP, JASS JR, HOPPER JL, DIEP A, BAPAT B, SALEM M, SEMINARA D, HAILE R; Colon Cancer Family Registry. Conversion analysis for mutation detection in MLH1 and MSH2 in patients with colorectal cancer. JAMA. 2005;293(7):799-809.
21. XIAO W, OEFNER PJ. Denaturing high-performance liquid chromatography: a review. Hum Mutation 2001;17:439-74.
22. HOLINSKI-FEDER E, MÜLLER-KOCH Y, FRIEDL W, MOESLEIN G, KELLER G, PLASCHKE J, BALLHAUSEN W, GROSS M, BALDWIN-JEDELE K, JUNGCK M, MANGOLD E, VOGELSANG H, SCHACKERT HK, LOHSEA P, MURKEN J, MEITINGER T. DHPLC mutation analysis of the hereditary nonpolyposis colon cancer (HNPCC) genes hMLH1 and hMSH2. J Biochem Biophys Methods. 2001;47(1-2):21-32.
23. COHEN V, AGULNIK JS, JARRY J, BATIST G, SMALL D, KREISMAN H, TEJADA NA, MILLER WH JR, CHONG G. Evaluation of denaturing highperformance liquid chromatography as a rapid detection method for identification of epidermal growth factor receptor mutations in non small-cell lung cancer. Cancer. 2006;107(12):2858-65.
24. KOYAMA RG, CASTRO RM, DE MELLO MT, TUFIK S, PEDRAZZOLI M. Simple detection of large InDeLS by DHPLC: the ACE gene as a model. J Biomed Biotechnol. 2008;562183.
25. COBB CJ, SCOTT G, SWINGLER RJ, WILSON S, ELLIS J, MACEWEN CJ, MCLEAN WH. Rapid mutation detection by the transgenomic wave analyser DHPLC identifies MYOC mutations in patients with ocular hypertension and/or open angle glaucoma. Br J Ophthalmol. 2002; 86(2):191-5.
26. DOMINGUEZ MV, BASTOS EP, SANTOS EM, OLIVEIRA LP, FERREIRA FO, CARRARO DM,ROSSI BM. Two new MLH1 germline mutations in Brazilian lynch syndrome families. Int J Colorectal Dis. 2008 Jul 2.
27. NOLAN T, HANDS RE, Bustin SA. Quantification of mRNA using real-time RT-PCR. Nature Protocols. 2006;1: 1559-82.
28. BJARNADOTTIR H, JONSSON JJ. A rapid realtime qRT-PCR assay for ovine beta-actin mRNA. J Biotechnol. 2005;117 (2): 173-82.
29. WONG ML, MEDRANO JF. Real-time PCR for mRNA quantitation. Biotechniques. 2005;39 (1): 75-85.
30. BUSTIN SA. Real-time, fluorescence-based quantitative PCR: a snapshot of current procedures and preferences. Expert Rev Mol Diagn. 2005;5 (4): 493-8
31. HOFFMANN M, GEULEN O, WEILKE C. The LightCycler® 480 real-time PCR system: a versatile platform for genetic variation research. Nature Methods. 2008; 5.
32. BENLLOCH S, PAYÁ A, ALENDA C, BESSA X, ANDREU M, JOVER R, CASTELLS A, LLOR X, ARANDA FI, MASSUTÍ B. Detection of BRAF V600E mutation in colorectal cancer: comparison of automatic sequencing and real-time chemistry methodology. J Mol Diagn. 2006;8(5):540-3.
33. MCGIVERN A, WYNTER CV, WHITEHALL VL, KAMBARA T, SPRING KJ, WALSH MD, BARKER MA, ARNOLD S, SIMMS LA, LEGGETT BA, YOUNG J, JASS JR: Promoter hypermethylation frequency and BRAF mutations distinguish hereditary non-polyposis colon cancer from sporadic MSI-H colon cancer. Fam Cancer 2004, 3:101-107
34. IKEHARA N, SEMBA S, SAKASHITA M, AOYAMA N, KASUGA M, YOKOZAKI H: BRAF mutation associated with dysregulation of apoptosis in human colorectal neoplasms. Int J Cancer 2005, 115:943-950
35. DOMINGO E, NIESSEN RC, OLIVEIRA C, ALHOPURO P, MOUTINHO C, ESPIN E, ARMENGOL M, SIJMONS RH, KLEIBEUKER JH, SERUCA R, AALTONEN LA, IMAI K, YAMAMOTO H, SCHWARTZ S, JR, HOFSTRA RM: BRAF-V600E is not involved in the colorectal tumorigenesis of HNPCC in patients with functional MLH1 and MSH2 genes. Oncogene 2005, 24:3995- 3998
36. HUGGETT J, DHEDA K, BUSTIN S, ZUMLA A. Real-time RT-PCR normalisation; strategies and considerations. Genes Immun. 2005; 6 (4): 279-84.
37. FRONHOFFS S, TOTZKE G, STIER S, WERNERT N, ROTHE M, BRÜNING T, KOCH B, SACHINIDIS A, VETTER H, Ko Y. A method for the rapid construction of cRNA standard curves in quantitative real-time reverse transcription polymerase chain reaction. Mol Cell Probes. 2002; 16 (2): 99-110.
38. SKRZYPSKI M. Quantitative reverse transcriptase real-time polymerase chain reaction (qRT-PCR) in translational oncology: Lung cancer perspective. Lung Cancer. 2008; 59 (2): 147-54.
39. LAY MJ, WITTWER CT. Real-time fluorescence genotyping of factor V Leiden during rapid-cycle PCR. Clin. Chem. 1997; 43: 2262-2267
40. OSGOOD-MCWEENEY D, GALLUZZI JR, Ordovas JM. Allelic discrimination for single nucleotide polymorphisms in the human scavenger receptor class B type 1 gene locus using fluorescent probes. Clin Chem. 2000; 46(1):118-9.
41. AOSHIMA T, SEKIDO Y, MIYAZAKI T, KAJITA M, MIMURA S, WATANABE K, SHIMOKATA K, NIWA T. Rapid detection of deletion mutations in inherited metabolic diseases by melting curve analysis with LightCycler. Clin Chem. 2000 Jan;46(1):119-22.
42. VALANZANO R, FICARI F, CURIA MC, ACETO G, VESCHI S, CAMA A, BATTISTA P, TONELLI F. Balance between endoscopic and genetic information in the choice of ileorectal anastomosis for familial adenomatous polyposis. J Surg Oncol. 2007;95(1):28-33.
43. YOUNG J, BARKER MA, SIMMS LA, WALSH MD, BIDEN KG, BUCH ANAN D, BUTTENSHAW R, WHITEHALL VL, ARNOLD S, JACKSON L, KAMBARA T, SPRING KJ, JENKINS MA, WALKER GJ, HOPPER JL, LEGGETT BA, JASS JR: Evidence for BRAF mutation and variable levels of microsatellite instability in a syndrome of familial colorectal cancer. Clin Gastroenterol Hepatol 2005, 3:254-263.
Correspondencia:
Mev Dominguez Valentin,
Departamento de Cirugia Pélvica del Hospital AC Camargo,
Rua Professor Antônio Prudente, 211, Liberdade,
São Paulo 01509-010, São Paulo, Brazil.
Tel.: +55-11-2189-5000 ext.1131
Email: mev_dv@yahoo.com













