Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista de Gastroenterología del Perú
versión impresa ISSN 1022-5129
Rev. gastroenterol. Perú vol.36 no.1 Lima ene./mar. 2016
ARTÍCULOS ORIGINALES
Inestabilidad de microsatélites en pacientes con diagnóstico de cáncer colorrectal
Microsatellite instability in patients with diagnostic of colorectal cancer
César Ortiz1,a, Kenny Dongo-Pflucker1,b, Luis Martín-Cruz1,b, Claudia Barletta Carrillo1,c, Pamela Mora-Alferez1,d, Abelardo Arias1,e
1 Laboratorio de Biología Molecular, Instituto Nacional de Enfermedades Neoplásicas (INEN). Lima, Perú.
a Biólogo Genetista Biotecnólogo, b Biólogo con mención en Biología Celular y Genética, c Biólogo. Magister en Biología Molecular,
d Médico Genetista, e Médico Patólogo Genetista
RESUMEN
Objetivo: Determinar la presencia de inestabilidad de microsatélites en pacientes con cáncer colorrectal usando el panel molecular Bethesda y discutir su importancia en pacientes con sospecha de cáncer colorrectal hereditario no polipósico (HNPCC) o con sospecha de síndrome de Lynch. Materiales y métodos: Se trabajó con muestras de sangre periférica y tejido tumoral de 28 pacientes con diagnóstico de cáncer colorrectal remitidos al laboratorio de Biología Molecular del Instituto Nacional de Enfermedades Neoplásicas (INEN) de Lima, bajo sospecha de Síndrome de Lynch. El ADN fue extraído utilizando kits de extracción de ácidos nucleicos para sangre periférica y tejido tumoral embebido en parafina. Se amplificaron los cinco marcadores microsatélites del panel Bethesda: BAT25, BAT26, D2S123, D5S346 y D17S250, por reacción en cadena de la polimerasa. El análisis de IMS fue realizado mediante electroforesis en chip en el bioanalizador Agilent 2100. Resultados: Del total de pacientes estudiados 11 tuvieron IMS alta (IMS-H) y uno no pudo ser totalmente clasificado quedando como IMS-H/IMS-L. En todos los casos de IMS-H tanto BAT25 como BAT26 resultaron inestables. La IMS-H en estos pacientes indica mayor probabilidad de HNPCC o síndrome de Lynch, lo cual debe ser contrastado con el análisis genético de los genes MMR. Conclusión: La técnica permitió determinar cuáles son los pacientes que deben continuar con el estudio de los genes del sistema de reparación de mal apareamiento del ADN, para establecer si estamos frente a casos de HNPCC o ante casos de cáncer colorrectal esporádicos.
Palabras clave: Cáncer colorrectal; Inestabilidad de microsatélites; Síndrome de Lynch; Reacción en cadena de la polimerasa (fuente: DeCS BIREME).
ABSTRACT
Objective: To determine the presence of microsatellite instability in patients with colorectal cancer using the molecular panel Bethesda and discuss its significance in patients with suspected hereditary nonpolyposis colorectal cancer (HNPCC) or Lynch Syndrome. Materials and methods: We worked with samples of peripheral blood and tumor tissue of 28 patients diagnosed with colorectal cancer referred to the Laboratory of Molecular Biology of the Instituto Nacional de Enfermedades Neoplasicas (INEN), Lima, with suspected of Lynch syndrome. DNA was extracted using kits of nucleic acid extraction of peripheral blood and paraffin-embedded tumor tissue. Five microsatellite markers of Bethesda panel were amplified: BAT25, BAT26, D2S123, D5S346 and D17S250, by polymerase chain reaction. IMS analysis was performed by electrophoresis on chip in the Bioanalyzer Agilent 2100. Results: Of the patients studied, 11 had high IMS (IMS-H) and one could not be fully ranked, staying as MSI-H / IMS-L. In all cases of IMS-H both BAT26 and BAT25 were unstable. The IMS-H in these patients indicates high probability of HNPCC or Lynch syndrome; it must be contrasted with the genetic analysis of MMR genes. Conclusion: The technique allowed determine which patients have to continue with the study of system mismatch repair genes, for establish whether we facing to HNPCC or sporadic colorectal cancer.
Key words: Colorectal cancer; Microsatellite instability; Lynch syndrome; Polymerase chain reaction (source: MeSH NLM).
INTRODUCCIÓN
La inestabilidad de microsatélites (IMS) corresponde a una vía molecular patogénica presente en alrededor del 15% de los casos de cáncer colorrectal (CRC) (1). Se origina cuando el sistema de reparación de mal apareamiento del ADN, conocido como sistema MMR, es disfuncional (2), lo cual conlleva a la acumulación de mutaciones originadas por la ADN polimerasa, como mutaciones puntuales, deleciones e inserciones (3). Las dos últimas se dan principalmente en las secuencias microsatélites, distribuídas ampliamente en nuestro genoma, y genera una disminución o aumento del tamaño de los microsatélites en las células cancerosas (3). A este cambio en la longitud de los microsatélites se le denomina inestabilidad de microsatélites (IMS). Así, la IMS es una huella genética de la disfunción del sistema MMR.
La IMS está presente en cáncer hereditario y esporádico, teniendo en ambos casos implicancias clínicas para el paciente. En cáncer de tipo hereditario se presenta en pacientes con síndrome de Lynch cuya etiología se debe a mutaciones germinales en los genes del sistema MMR (4). Este síndrome fue descrito por primera vez por Warthin AS en 1913 como cáncer de colon hereditario no polipósico (HNPCC), reportando una predisposición hereditaria a desarrollar cáncer de colon, estómago y endometrio (3). Es así que la detección de IMS es útil como herramienta de apoyo al diagnóstico en pacientes con sospecha clínica de síndrome de Lynch.
La IMS tiene además valor predictivo de resistencia a terapia adyuvante basada en 5-fluorouracilo (5,6), lo cual ha sido estudiado principalmente en pacientes con CRC esporádico, existiendo pocos datos para los casos hereditarios (3,5). Aún así, en ambos casos la IMS presenta la misma biología, por lo que el beneficio de conocer el estatus de IMS colaboraría en el tratamiento del paciente con CRC, tanto en casos esporádicos como hereditarios. La IMS también ha sido asociada con pronóstico favorable, relacionado con la elevada infiltración de linfocitos observada en estos tumores, que indica un grado de respuesta inmunológica contra el tumor. Al respecto, se ha reportado que la células tumorales con IMS presentan péptidos inmunogénicos de cambio del marco de lectura (FSP, frameshift peptides), originados por deleciones e inserciones en microsatélites que se encuentran dentro de genes, como TGFRβII y BAX (5), alterando el marco de lectura y produciendo péptidos cuya secuencia y estructura son reconocidas como ajenas por el sistema inmunológico. El valor pronóstico también se ha asociado al bajo porcentaje de metástasis a órganos distantes que hacen las células tumorales con IMS (3).
Con la finalidad de analizar la IMS, Boland et al. (7) recomendaron el uso del panel Bethesda, el cual consiste en el análisis de cinco marcadores microsatélites, tres de repeticiones dinucleotídicas: D2S123, D5S346 y D17S250; y dos de repeticiones mononucleotídicas: BAT25 y BAT26. Se estableció que el 40% o más de los marcadores probados (≥2) deben encontrarse inestables para clasificar a un tumor con inestabilidad de microsatélites alta (IMS-H), el cual tiene aplicación clínica demostrada. Si solo un marcador es inestable se clasifica como inestabilidad de microsatélites baja o IMS-L, cuyo significado biológico y clínico aún no ha sido dilucidado (8).
En el presente trabajo realizamos un estudio en pacientes con diagnóstico de cáncer colorrectal y sospecha clínica de cáncer hereditario, para determinar inestabilidad de microsatélites utilizando el panel Bethesda. Se discute la implementación de la prueba molecular como análisis rutinario y se hace énfasis en el significado clínico de la prueba en pacientes con sospecha de síndrome de Lynch.
MATERIALES Y MÉTODOS
Muestras analizadas
Se estudiaron 28 pacientes con diagnóstico de cáncer colorrectal evaluados inicialmente en el consultorio de Genética Médica del Instituto Nacional de Enfermedades Neoplásicas (INEN), bajo criterios clínicos de sospecha de predisposición genética a cáncer colorrectal (CRC). Las muestras de los pacientes seleccionados fueron remitidas al Laboratorio de Biología Molecular del INEN entre junio del 2013 a mayo del 2014. Cada paciente fue consultado bajo consentimiento informado respecto a su participación en el presente estudio. Se utilizaron muestras de sangre periférica, como tejido normal, y muestras de tumor embebido en parafina con un mínimo de 30% de tejido tumoral.
Extracción y cuantificación de ADN
La extracción de ADN a partir de sangre periférica se realizó empleando el kit PureLinkTM Genomincs DNA (InvitrogenTM) según protocolo modificado en nuestro laboratorio. El ADN de las muestras de tejido tumoral embebido en parafina fue extraído a partir de cuatro cortes de 5 µm de espesor, los cuales fueron desparafinados con xilol-alcohol y sometidas al kit QIAamp® DNA FFPE Tissue (Qiagen). El ADN fue cuantificado empleando la plataforma de cuantificación QubitTM con 1 µl de cada muestra, según indicaciones de manufactura.
Reacción en cadena de la polimerasa (PCR)
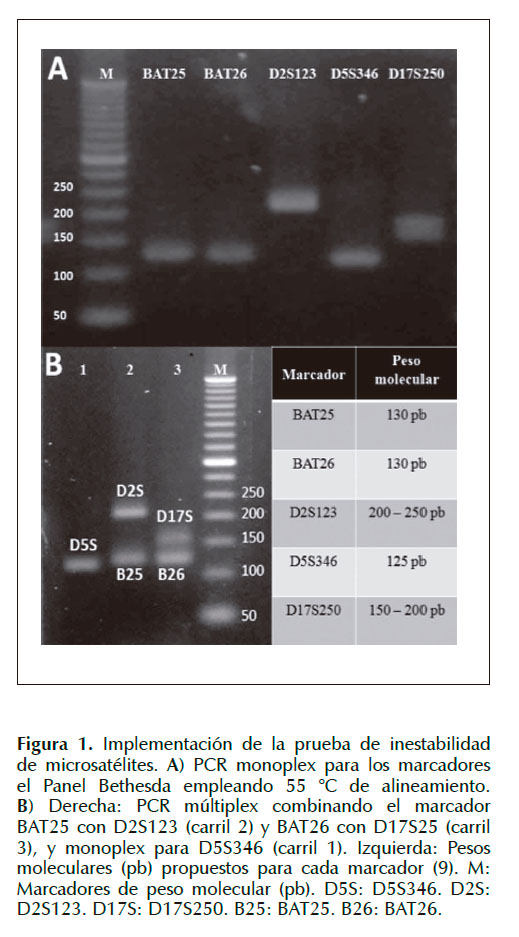
Se amplificaron por PCR los marcadores microsatélites BAT25, BAT26, D2S123, D5S346 y D17S250 propuestos en el panel Bethesda(9). Para la implementación de la prueba se realizó una reacción de PCR para cada marcador, probando temperaturas de hibridización entre 50 a 58°C, concentraciones de cebadores entre 0,2 a 0,8 µM, y de ión Mg+2 entre 2 a 3 mM. Luego se ensayaron dos PCR múltiplex, la primera con los marcadores BAT25 y D2S123, y la segunda con BAT26 y D17S250. En todos los casos se usó buffer 1X, 300 µM de dNTPs, 0,6 unidades de Taq polimerasa de alta fidelidad Platinum® (InvitrogenTM) y 50 ng de ADN. Las condiciones de PCR fueron: 94 °C por 2 minutos; 40 ciclos de 94 °C por 35 segundos, 52-58 °C por 40 segundos, y 68 °C por 35 segundos; y una extensión final a 68 °C por 5 minutos. La estandarización fue realizada usando ADN extraído de sangre periférica y tejido tumoral parafinado.
Electroforesis en gel de agarosa
Los productos de PCR fueron analizados en gel de agarosa al 3% en buffer TAE 1X. La electroforesis se realizó en una cámara de electroforesis horizontal Fisher Biotecha 100 V por 60 minutos. El gel fue coloreado en SYBR® Safe por 20 minutos y observado en un transiluminador UV.
Electroforesis en chip en Bioanalizador y clasificación de inestabilidad
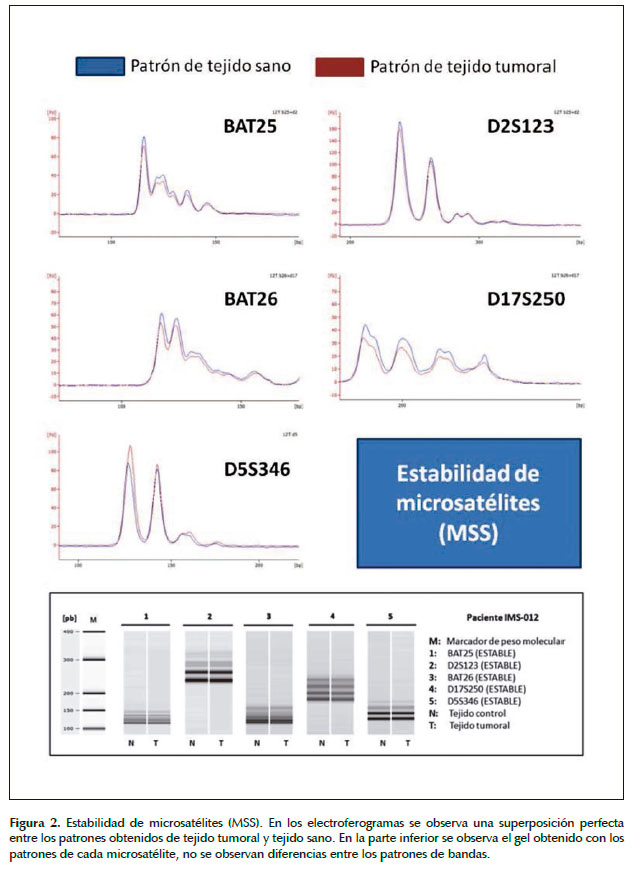
La determinación de IMS se realizó en el Bioanalizador Agilent 2100 (Agilent Technologies, Inc.) empleando el kit DNA 1000 LabChip y el software Agilent’s 2100 expert según protocolo modificado en nuestro laboratorio. Se compararon los patrones de cada marcador microsatélite entre tejido normal y tumoral. Se interpretó como marcador microsatélite estable (MSS) cuando el patrón obtenido fue el mismo en ambos tejidos; por otro lado, si se observaron diferencias se clasificó como microsatélite inestable (IMS). Se consideró que un tumor presenta inestabilidad de microsatélites alta (IMS-H) si al menos dos marcadores, de los cinco estudiados, resultaron inestables.
RESULTADOS
Se estableció la concentración de ión Mg++en 2,5 mM, de cebadores en 0,4 µM excepto para D5S346 que fue de 0,2 µM. Los cinco marcadores analizados amplificaron de forma específica en todo el rango de temperaturas de hibridización probadas, estableciéndose la temperatura a 55 °C para cada marcador (Figura 1A). La PCR múltiplex fue óptima mostrando la misma eficiencia de amplificación en cada marcador (Figura 1B) e indistinto si el ADN fue extraído sangre periférica o de tejido tumoral parafinado.
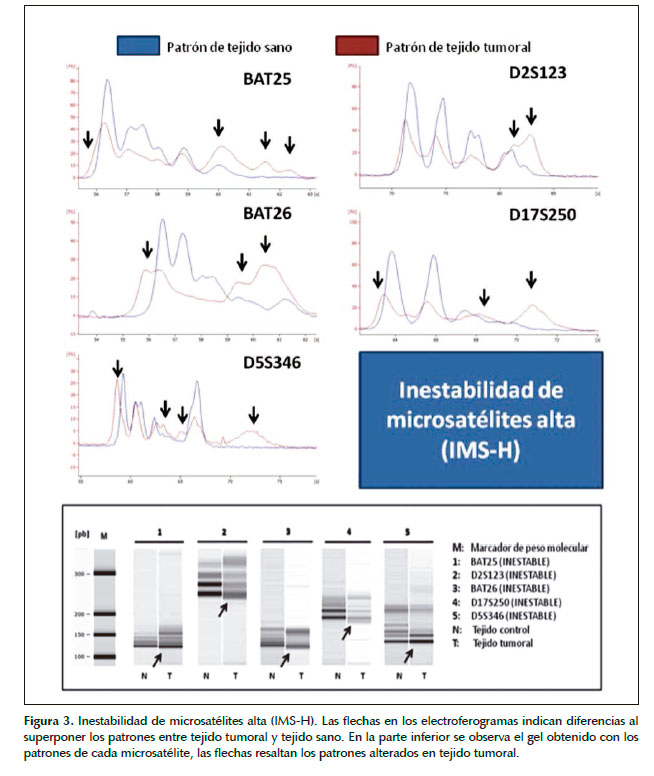
El grupo de pacientes evaluados estuvo constituido por individuos entre 25 y 65 años de edad, de ambos sexos. El análisis de IMS se realizó en un Bioanalizador Agilent 2100 comparando los patrones obtenidos entre tejido normal y tumoral (Figuras 2 y 3). Se detectó IMS-H en 11 de los 28 casos, todos menores de 50 años y enviados por el consultorio de Genética del
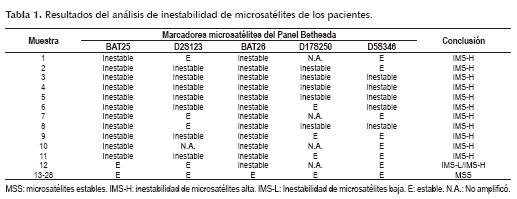
INEN bajo sospecha de síndrome de Lynch/cáncer colorrectal no polipósico hereditario. BAT26 y BAT25 fueron inestables en todos los casos positivos, siendo estos los marcadores más sensibles. En cuatro casos los marcadores D2S123 y/o D17S250 no pudieron ser amplificados (Tabla 1); sin embargo, el análisis del resto de marcadores fue suficiente para clasificar a estos tumores, excepto en un paciente en el cual no se pudo establecer IMS-H o IMS-L.
DISCUSIÓN
El cáncer colorrectal, es el tercer cáncer más frecuente y cuarto cáncer más letal a nivel mundial (10). En el Perú corresponde al 3,3% de los casos de cáncer registrados entre el 2006 y 2011 (11). Tiene su inicio en lesiones en la mucosa epitelial del colon en donde se producen los primeros cambios genéticos que conducen al desarrollo de las células cancerosas (12). Se han propuesto tres vías patogénicas de desarrollo de estos tumores: inestabilidad cromosómica (INC), fenotipo metilador en las islas CpG (CIMP) e inestabilidad de microsatélites (IMS) (13). Por lo general, una de las tres vías predomina, por lo que raras veces se traslapan. La INC está asociada con la desregulación secuencial de genes supresores de tumores y oncogenes, tales como APC, KRAS, DCC/ SMAD4 y TP53. El CIMP consiste en una hipermetilación de las islas CpG en varios loci. Por su parte, la IMS se presenta en los casos con síndrome de Lynch y en tumores esporádicos, y es causada principalmente por la inactivación del sistema reparador del ADN por mal apareamiento o sistema MMR.
La IMS representa 15% de los casos de CRC, aunque ello depende de la población en estudio, siendo del 15% en Estados Unidos, 20 al 45% en afroamericanos y de hasta 37% en población egipcia (13-15). Del total de casos de IMS un 20% corresponden a síndrome de Lynch, mientras que el 80% restante forman parte de un CRC esporádico (13). En general las características de los tumores con IMS suelen presentarse en el colon proximal, ser diagnosticado en estadios patológicos bajos (13) y caracterizarse histológicamente por abundante infiltración de linfocitos al tumor, pobre diferenciación, células en anillo de sello y fenotipo mucinoso (16).
La IMS representa la disfuncionalidad del sistema MMR, y de sus principales genes componentes: MLH1, PMS2, MSH2 y MSH6 (2,3). El fallo del sistema MMR lleva a la célula a un estado de inestabilidad genética en el cual se produce acumulación de mutaciones durante la replicación del ADN. La acumulación es especialmente evidente en las secuencias repetitivas cortas o microsatélites, debido a la poca eficacia que tiene la ADN polimerasa de unirse a secuencias repetitivas durante la replicación (2). Es así que una célula con el sistema MMR deficiente es incapaz de reparar los errores ocurridos en estas regiones produciendo un cambio en la longitud del microsatélite, situación conocida como inestabilidad de microsatélites.
La aparición temprana de CRC con IMS está relacionada con la inactivación de los genes MMR producida por mutaciones germinales. Por otro lado, la aparición tardía suele desarrollarse en un contexto de hipermetilación que lleva al silenciamiento del gen MLH1 (4,13). Así, en un paciente menor a 60 años con CRC e IMS la probabilidad de cáncer de tipo hereditario (síndrome de Lynch) es mayor, siendo importante establecer el estatus de IMS como estrategia inicial de estudio en estos pacientes. La prueba que permite confirmar el síndrome de Lynch es el secuenciamiento de los genes MMR, proceso bastante complejo si consideramos el número y tamaño de los genes implicados, además de la complejidad de las mutaciones que se pueden encontrar. En ese contexto, se deben realizar dos análisis complementarios antes del secuenciamiento: expresión de proteínas MMR por inmunohistoquímica (IHC) y el análisis de la mutación V600E del gen BRAF. La primera indicará qué genes del sistema MMR estarían disfuncionales, y la segunda, si fuera positiva a la mutación, determinará en el paciente CRC esporádico (17,18). Ambas pruebas aumentan la efectividad del tamizaje disminuyendo el número de casos que pasarían a ser analizados por secuenciamiento de los genes MMR.
Respecto a la prueba de inmunohistoquímica (IHC) para detección de proteínas MMR, ha mostrado elevada sensibilidad y especificidad para la identificación de pacientes con los genes MMR disfuncionales. Debido al bajo costo, fácil implementación y sensibilidad de la metodología, algunos autores la prefieren sobre la prueba de IMS. Sin embargo, la IHC tiene la desventaja de que la tinción puede ser heterogénea en todo el tumor, afectando la sensibilidad del test (19). Esta dificultad se ha descrito en la detección de la expresión proteica del gen MLH1, cuya mutación abarca hasta el 50% de todos los casos de síndrome de Lynch (2,19,20). Además, en los tumores puede ocurrir pérdida parcial o incompleta de la expresión proteica, dificultando la interpretación del resultado. Por lo indicado, se recomienda realizar tanto la IHC como el análisis de IMS para concluir sobre un caso en particular (21).
La IMS se determina usando cinco marcadores microsatélites: BAT25, BAT26, D2S123, D5S346 y D17S250; que en conjunto constituyen el panel Bethesda (7). Respecto al estatus de IMS se ha establecido tres categorías: estabilidad de microsatélites (MSS), inestabilidad de microsatélites baja (IMS-L) e inestabilidad de microsatélites alta (IMS-H), siendo este último el que posee las aplicaciones clínicas descritas (8). Para la clasificación de IMS-H se debe considerar que al menos dos de los cinco marcadores sean inestables, es decir, el tamaño de dichos microsatélites es distinto entre tejido tumoral y tejido sano. Sin embargo, la observación de dicho evento depende de la metodología utilizada: electroforesis en geles de poliacrilamida, electroforesis capilar o electroforesis en chip. En el presente trabajo se utilizó electroforesis en chip, metodología para realizar electroforesis de alta resolución, que identifica un microsatélite inestable cuando de la comparación entre tejido sano y tumoral, resultan dos patrones distintos, no observándose necesariamente diferencias en la longitud del marcador. Si bien los otros dos tipos de electroforesis son los más utilizados debido a que la lectura es mucho más sencilla, la electroforesis en chip es una metodología rápida y poco compleja, con una sensibilidad comparable a las otras metodologías (22,23).
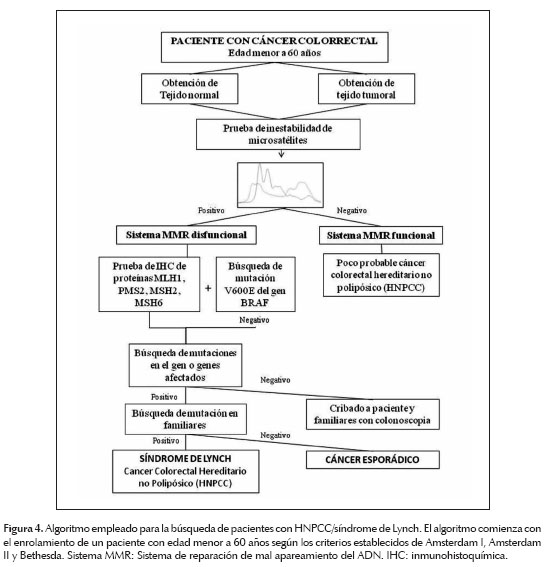
En el presente trabajo se reporta la estandarización de la metodología para el análisis de los cinco marcadores microsatélites del panel Bethesda: BAT25, BAT26, D2S123, D5S346 y D17S250, el cual culminó con el establecimiento de un protocolo de PCR múltiplex para su análisisenunbioanalizadormedianteelectroforesisenchip. Se encontraron 11 pacientes con IMS-H. Considerando que estos pacientes fueron enviados por el consultorio de Genética del INEN por sospecha de síndrome de Lynch, los siguientes pasos serían complementar el estudio evaluando la inmunohistoquímica (IHC) de expresión de proteínas MMR, buscar la mutación V600E del gen BRAFy el subsecuente análisis de mutaciones en los genes MMR de los casos negativos para V600E del BRAF. Los genes a evaluar serán determinados por la inmunohistoquímica (IHC). Este procedimiento forma parte de un algoritmo establecido para la búsqueda de casos de Síndrome de Lynch/HNPCC (Figura 4).
En conclusión, la inestabilidad de microsatélites es una vía molecular del cáncer colorrectal cuya detección constituye un tamizaje molecular de pacientes con sospecha de síndrome de Lynch. Posee además valor predictivo de resistencia a tratamiento con 5-fluorouracilo. La implementación de la prueba de IMS en el laboratorio de Biología Molecular del INEN permitió definir a los pacientes que entrarían en los subsecuentes estudios, tales como detección de la mutación V600E del gen BRAF y el secuenciamiento de los genes del sistema MMR, con miras a determinar un diagnóstico genético definitivo.
Conflicto de interés: Los autores declaran no tener conflictos de interés en la concepción, desarrollo y publicación del presente trabajo.
BIBLIOGRAFÍA
1. Pancione M, Remo A, Colantuoni V. Genetic and epigenetic events generate multiple pathways in colorectal cancer progression. Pathology Research International. 2012;2012(1):1-11. [ Links ]
2. Vilar E, Gruber S. Microsatellite instability in colorectal cancerthe stable evidence. Nat Rev Clin Oncol. 2010;7(3):153-62. [ Links ]
3. Kloor M, Staffa L, Ahadova A, von Knebel Doeberitz M. Clinical significance of microsatellite instability in colorectal cancer. Langenbecks Arch Surg. 2014;399(1):23-31. [ Links ]
4. Aaltonen L, Jonhs L, Jarvinen H, Mecklin J-P, Houlston R. Explaining the familial colorectal cáncer risk associated with mismatch repair (MMR)-deficient and MMR-stable tumors. Clin Cancer Res. 2007;13(1):356-61. [ Links ]
5. Saeterdal I, Bjørheim J, Lislerud K, Gjertsen MK, Bukholm IK, Olsen OC, et al. Frameshift-mutation-derived peptides as tumor-specificantigens in inherited and spontaneous colorectal cancer. Proc Natl Acad Sci U S A. 2001;98(23):13255-60. [ Links ]
6. Ribic CM, Sargent DJ, Moore MJ, Thibodeau SN, French AJ, Goldberg RM, et al. Tumor microsatellite-instability status as a predictor of benefit from fluorouracil-based adjuvant chemotherapy for colon cancer. N Engl J Med. 2003;349(3):247-57. [ Links ]
7. Boland CR, Thibodeau SN, Hamilton SR, Sidransky D, Eshleman JR, Burt RW, et al. A National Center Institute Workshop on Microsatellite Instability for cancer detection and familial predisposition: development of international criteria for the determination of microsatellite instability in colorectal cancer. Cancer Res. 1998;58:5248-57. [ Links ]
8. De la Chapelle A, Hampel H. clinical relevance of microsatellite instability in colorectal cancer. J Clin Oncol. 2010;28(20):3380-7. [ Links ]
9. Umetani N, Sasaki S, Watanabe T, Ishigami H, Ueda E, Nagawa H. Diagnostic primer sets for microsatellite instability optimized for a minimal amount of damaged DNA from colorectal tissue samples. Ann Surg Oncol. 2000;7(4):276-80. [ Links ]
10. Wilmink ABM. Overview of the epidemiology of colorectal cancer. Dis Colon Rectum. 1997;40(4):483-93. [ Links ]
11. Perú, Ministerio de Salud, Dirección General de Epidemiología. Análisis de la situación del cáncer en el Perú 2013. Lima: MINSA; 2013. [ Links ]
12. Gryfe R, Kim H, Hsieh ET, Aronson MD, Holowaty EJ, Bull SB, et al. Tumor microsatellite instability and clinical outcome in young patients with colorectal cancer. N Engl J Med. 2000;342(2):69-77. [ Links ]
13. Boland CR, Goel A. Microsatellite instability in colorectal cancer. Gastroenterology. 2010;138(6):2073-2087.e3. [ Links ]
14. Duval A, Hamelin R. Mutations at coding repeat sequences in mismatch repair‑deficient human cancers: toward a new concept of target genes for instability. Cancer Res. 2002;62(9):2447-54. [ Links ]
15. Rajagopalan H, Bardelli A, Lengauer C, Kinzler KW, Vogelstein B, Velculescu VE. Tumorigenesis: RAF/RAS oncogenes and mismatch‑repair status. Nature. 2002;418(6901):934. [ Links ]
16. Hyde A, Fontaine D, Stuckless S, Green R, Pollett A, Simms M, et al. A histology-based model for predicting microsatellite instability in colorectal cancers. Am J Surg Pathol. 2010;34(12):1820-9. [ Links ]
17. Samowitz WS, Sweeney C, Herrick J, Albertsen H, Levin TR, Murtaugh MA, et al. Poor survivalassociatedwiththe BRAF V600E mutation in microsatellite-stable colon cancers. Cancer Res. 2005;65(14):6063-9. [ Links ]
18. Weisenberger DJ, Siegmund KD, Campan M, Young J, Long TI, Faasse MA, et al. CpG island methylator phenotype underlies sporadic microsatellite instability and is tightly associated with BRAF mutation in colorectal cancer. Nat Genet. 2006;38(7):787-93. [ Links ]
19. Shia J. Immunohistochemistry versus microsatellite instability testing for screening colorectal cancer patients at risk for hereditary nonpolyposis colorectal cancer syndrome. Part I. The utility of immunohistochemistry. J Mol Diagn. 2008;10(4):293-300. [ Links ]
20. Mangold E, Pagenstecher C, Friedl W, Fischer HP, Merkelbach- Bruse S, Ohlendorf M, et al. Tumours from MSH2 mutation carriers show loss of MSH2 expression but many tumours from MLH1 mutation carriers exhibit weak positive MLH1 staining. J Pathol. 2005;207(4):385-95. [ Links ]
21. Iacopetta B, Grieu F, Amanuel B. Microsatellite instability in colorectal cancer. Asia Pac J Clin Oncol. 2010;6(4):260-9. [ Links ]
22. Banerjea A, Phillips SM, Dorudi S, Bustin SA. Colorectal cancers with mononucleotide microsatellite instability can be identified using microfabricated chip technology. Anal Biochem. 2003;322(1):130-3. [ Links ]
23. Baldus S, Barta N, Lammers S, Dienes H, Salowsky R, Odenthal M. Label-free analysis of microsatellite instability in colorectal carcinoma by on-chip electrophoresis. Palo Alto, CA: Agilent Technologies; 2005. [ Links ]
Correspondencia: César Alexander Ortiz Rojas
Francisco de Toledo, 159. Urb. Cercado. Santiago de Surco. Lima, Perú.
E-mail: cesar.alexander04@gmail.com
Recibido: 24-06-2015
Aprobado: 10-08-2015