Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Anales de la Facultad de Medicina
Print version ISSN 1025-5583
An. Fac. med. vol.66 no.1 Lima ene./Mar. 2005
Presencia de b-lactamasas de espectro extendido
en dos hospitales de Lima, Perú
José-Luis Morales 1, Karina Reyes 1, Mario Monteghirfo 2, Mirtha Roque 1, José Irey 1
1 Facultad de Farmacia y Bioquímica, UNMSM. Lima, Perú.
2 Centro de Investigación de Bioquímica y Nutrición. Facultad de Medicina, UNMSM. Lima, Perú.
Resumen
Objetivo: Estudiar la presencia de b-lactamasas de espectro extendido (BLEE) e identificar los tipos Temoniera (TEM) y sulfidrilo variable (SHV) producidas por cepas de Escherichia coli y Klebsiella pneumoniae en dos hospitales de Lima. Material y Métodos: La selección y confirmación de cepas productoras de BLEE se realizó mediante pruebas de susceptibilidad antimicrobiana, utilizando los criterios de la National Committee for Clinical Laboratory Standards (NCCLS); la identificación de los genes blaTEM y blaSHV se realizó mediante el análisis de la reacción en cadena de la polimerasa (PCR) para su posterior secuenciamiento genético. Resultados: Se recolectó consecutivamente entre julio y septiembre de 2000, 137 cepas de Escherichia coli y 18 cepas de Klebsiella pneumoniae. La mayoría mostró alta resistencia a las cefalosporinas de tercera generación y aztreonam; 2,9% del total de E.coli y 44,4% del total de K.pneumoniae aisladas fueron confirmadas como productoras de BLEE. Todas las cepas productoras de BLEE fueron multirresistentes y la mayoría presentó co-resistencia a sulfametoxazol/trimetoprim, amikacina, gentamicina y ciprofoxacina. Se identificó la presencia del gen blaTEM en 4 cepas (3 K. pneumoniae y 1 E. coli) y el gen blaSHV en 6 cepas (3 K. pneumoniae y 3 E. coli). El secuenciamiento de los correspondientes genes confirmó las BLEEs TEM-10 y SHV-5. Conclusión: Se demostró la presencia de BLEE tipo TEM y SHV, asociado a multirresistencia antibiótica.
Palabras clave: Beta-lactamasas; Escherichia coli; Klebsiella pneumoniae; resistencia microbiana a las drogas; resistencia a múltiples drogas.
Presence of extended-spectrum b-lactamases in two hospitals of Lima, Perú
Abstract
Objective: To study the presence in two hospitals of Lima of extended spectrum b-lactamases (BLEE) and to identify TEM and SHV types produced by Escherichia coli and Klebsiella pneumoniae strains. Material and Methods: The selection and confirmation of BLEE producing strains was done by antimicrobial susceptibility tests. The identification of bla TEM and bla SHV genes was achieved by polymerase chain reaction (PCR) and DNA sequencing. Results: One hundred and thirty seven Escherichia coli and 18 Klebsiella pneumoniae strains were isolated from hospitalized patients between July and September 2000. Most showed high resistance to third-generation cephalosporins and aztreonam; 2,9% of all E.coli and 44,4% of all K.pneumoniae isolates were confirmed as BLEE producer strains. All BLEE producer strains were multidrug-resistant and most presented co-resistance to trimethoprim/sulfamethoxazole, amikacin, gentamicin and ciprofloxacin. The blaTEM gene was found in 4 strains (3 K. pneumoniae y 1 E. coli) and blaSHV gene in 6 strains (3 K. pneumoniae y 3 E. coli). Sequencing of the corresponding genes confirmed TEM-10 and SHV-5 BLEEs. Conclusion: We determined the presence of TEM and SHV BLEE types, associated to antibiotic multiresistance.
Key words: Beta-lactamases;Eescherichia coli; Klebsiella pneumoniae; drug resistance, microbial; drug resistance, multiple.
INTRODUCCIÓN
La introducción de cefalosporinas de espectro extendido facilitó un seguro y efectivo tratamiento de infecciones moderadas a severas en bacilos gram negativos. Desafortunadamente, el uso indiscriminado de estos agentes está estrechamente relacionado con cifras elevadas de la resistencia (1). Esta resistencia puede ser mediada por disminución de la permeabilidad de la membrana, la hiperproducción cromosomal de b-lactamasas, producción de b-lactamasas de espectro extendido, o combinación de estos mecanismos (2).
Las b-lactamasas de espectro extendido (BLEE) son principalmente producidas por cepas de Escherichia coli y Klebsiella pneumoniae y generan resistencia a cefalosporinas de tercera generación y aztreonam (3-5). Son de rápida propagación y se las ha hallado en diferentes partes del mundo, usualmente en hospitales; la prevalencia varía de país en país (6,7).
A nivel nosocomial, las BLEE son consideradas como causas importantes del incremento en la morbilidad y mortalidad de pacientes hospitalizados, de la prolongada estancia hospitalaria y del aumento de los costos globales de salud. Asimismo, las pruebas rutinarias de la susceptibilidad antimicrobiana no son suficientes para determinar la presencia de cepas productoras de BLEE, por lo que el Comité Nacional para la Normalización de los Laboratorios Clínicos (NCCLS) ha diseñado una metodología de laboratorio práctica para su identificación rápida y a bajo costo (8). Las principales BLEE derivan de los tipos TEM y SHV debido a mutaciones puntuales en la estructura del gen que las codifica (9-11). Los genes que codifican a estas enzimas han podido ser identificadas por técnicas moleculares como la reacción en cadena de la polimerasa (PCR, polymerase chain reaction) y la secuenciación de los genes que las codifican (12).
Los efectos de no identificar los tipos de BLEE producidas por cepas de Escherichia coli y Klebsiella pneumoniae pueden resultar en un fracaso del tratamiento, debido a una dosificación inapropiada de los antibióticos de espectro extendido (13-15), seguido de un aumento de la tasa de morbimortalidad y de los costos que implica el tratamiento y la estancia hospitalaria.
En el presente estudio realizado con cepas de E. coli y K. pneumoniae recolectadas de los Hospitales Nacionales Guillermo Almenara Irigoyen y Edgardo Rebagliati Martins, se ha detectado la presencia de BLEE mediante el uso de técnicas microbiológicas e identificado los tipos TEM y SVH mediante PCR y secuenciación del ADN.
MATERIAL Y MÉTODOS
Entre julio y setiembre de 2000, un total de 137 cepas de E. coli y 18 cepas de K. pneumoniae fueron recolectadas de los Hospitales Nacionales Guillermo Almenara Irigoyen y Edgardo Rebagliati Martins.
La detección de cepas productoras de BLEE y pruebas de susceptibilidad, se realizó mediante la prueba de sinergia de doble disco y las pruebas de susceptibilidad recomendadas por el Comité Nacional para la Normalización de los Laboratorios Clínicos (NCCLS).
La obtención del ADN se realizó mediante la técnica de Pitout (1). Los cebadores usados para la obtención de los genes blaTEM fueron: T1 (5-ATA AAA TTC TTG AAG ACG AAA-3) y T2 (5-GAC AGT TAC CAA TGC TTA ATC-3) correspondientes a las posiciones –5 a 16 y 1073 a 1053, respectivamente. Los cebadores usados para la obtención de los genes blaSHV fueron: S1 (5-TGG TTA TGC GTT ATA TTC GCC-3) y S2 (5-GGT TAG CGT TGC CAG TGC T-3) correspondientes a las posiciones 120 a 140 y 990 a 972, respectivamente. Para la amplificación de los genes blaTEM se utilizó la técnica descrita por Mabilat (2) y para los genes blaSHV se usó la técnica descrita por Kim (3).
Los productos de PCR fueron liofilizados y enviados para su purificación y secuenciación a AFIGEN S.L. (Aplicaciones Funcionales de Ingeniería Genética S.L.). Se secuenció ambas cadenas de los productos de PCR, utilizando los mismos cebadores de la amplificación. El análisis de las secuencias nucleotídicas se realizó mediante el software BLAST.
RESULTADOS
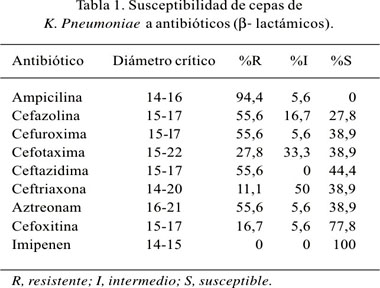
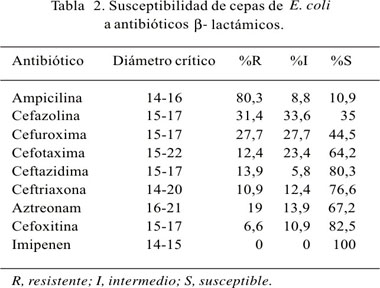
Se recolectó 137 cepas clínicas de Escherichia coli y 18 de Klebsiella pneumoniae. En ambas bacterias se observó una elevada resistencia a la ampicilina, cefazolina y cefuroxima y total susceptibilidad a imipenem. Los valores de susceptibilidad en K. pneumoniae fueron de 38,9%, 44,4%, 38,9% y 38,9% para cefotaxima, ceftazidima, ceftriaxona y aztreonam, respectivamente; mientras que para E. coli fueron 64,2%, 80,3%, 76,6% y 67,2%.
Doce cepas fueron positivas a la prueba de confirmación fenotípica de BLEE (10,2%). Se obtuvo 4 (2,9%) de un total de 137 cepas de E. coli y 8 (44,4%) de un total de 18 cepas de K. pneumoniae, como cepas productoras de BLEE. Estas cepas fueron designadas como C1 a C12, siendo C1-C4, C8 y C11 cepas de K. pneumoniae y C5-C7 y C9 cepas de E. coli.
Todas las cepas fueron resistentes a la ampicilina y cefazolina. Las cepas de K. pneumoniae fueron más resistentes a cefalosporinas de tercera generación y aztreonam que las cepas de E. coli. 10 cepas fueron intermedios o resistentes con diámetro de inhibición para: cefotaxima (12 a 19 mm), ceftazidima (6 a 9 mm), ceftriaxona (10 a 16 mm) y aztreonam (6 a 12 mm). Dos cepas de E. coli (C7 y C9) mostraron susceptibilidad a cefalosporinas de tercera generación y aztreonam. Todas fueron susceptibles a imipenem. La mayoría se presentó resistente a amoxicilina/ácido clavulánico y solamente una cepa (C11) se mostró resistente a cefoxitina (Tabla 1).
La mayoría de las cepas productoras de BLEE fueron resistentes a las 3 clases de antibióticos (sulfonamidas, aminoglucósidos y fluoroquinolonas). La co-resistencia a sulfametoxazol/trimetoprim fue común a todas las cepas, seguido de ciprofloxacina, gentamicina y amikacina. Todas estas cepas fueron halladas en diferentes muestras biológicas y procedían de diferentes salas de hospitalización (Tabla 2).
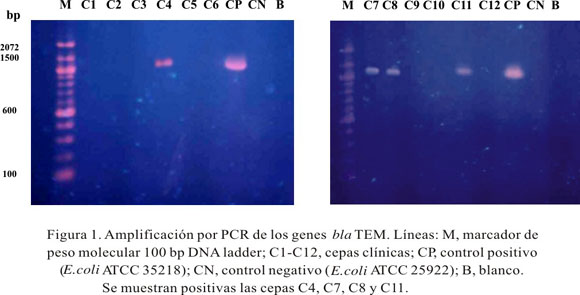
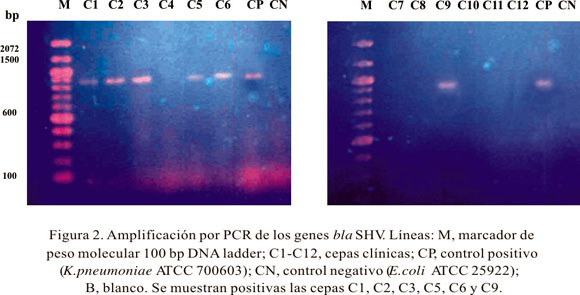
Se investigó sobre el tipo de BLEE de las cepas confirmadas fenotípicamente como productoras de BLEE, mediante amplificación de ADN por PCR. Los productos de PCR (amplicones) fueron del tamaño esperado, de aproximadamente 1078 pb para los genes blaTEM y 870 pb para blaSHV, representando la parte de los genes en donde ocurren las mutaciones características de los tipos de b-lactamasas respectivos. Este análisis de PCR reveló que 4 cepas, 1 E. coli (C7) y 3 K. pneumoniae (C4, C8 y C11), contenían el gen de b-lactamasa blaTEM; 6 cepas, 3 E. coli (C5, C6 y C9) y 3 K. pneumoniae (C1,C2 y C3) contenían el gen de b-lactamasa blaSHV y 2 cepas de K. pneumoniae (C10 y C12) no contenían los genes blaTEM o blaSHV. Estos resultados se muestran en las Figuras 1 y 2.
En la secuenciación del ADN de los 2 productos de PCR que fueron positivos a blaTEM y 3 productos de PCR que fueron positivos a blaSHV se obtuvo como resultado que los genes secuenciados codifican para las BLEE TEM-10 y SHV-5, que presentan sustituciones aminoacídicas para TEM en las posiciones 164 y 240, respectivamente, con respecto a TEM-1; y para SHV en las posiciones 238 y 240, con respecto a SHV-1 (numeración de las posiciones de acuerdo a Ambler (16)). Figuras 3 y 4.
DISCUSIÓN
El presente estudio se inició realizando un reconocimiento de los patrones de susceptibilidad a antibióticos b-lactámicos de 137 cepas clínicas de E. coli y 18 de K. pneumoniae procedentes de los hospitales de cuarto nivel de Lima Guillermo Almenara Irigoyen y Edgardo Rebagliati Martins. De acuerdo a los resultados obtenidos, estos patrones eran indicativos de la presencia de cepas de BLEE. Al compararse los resultados con los obtenidos por un estudio realizado por el SENTRY (4) en Latinoamérica (Argentina, Brasil, Colombia, Chile, México y Venezuela), se encontró en ambos una elevada resistencia a ampicilina y cefazolina. Pero, lo diferente y lo más alarmante fue encontrar en nuestro estudio valores menores de susceptibilidad a cefalosporinas de tercera generación y aztreonam, tal como se evidenció en K. pneumoniae, cuyos porcentajes de susceptibilidad fueron 44,4%, 38,9% y 38,9% para ceftazidima, ceftriaxona y aztreonam, respectivamente, mientras que para el SENTRY fueron 79,2%, 81,3%, 79,2%; de manera similar ocurrió con E. coli.
Del total de cepas presuntas productoras de BLEE, 12 cepas fueron confirmadas como productoras de BLEE, lo cual indica que el número de cepas que no fueron productoras de BLEE involucran otros mecanismos de resistencia, tales como b–lactamasas AmpC cromosómicas o mediadas por plásmidos y/o reducción de permeabilidad de la membrana externa (5).
Un estudio de vigilancia en Latinoamérica reportó que la frecuencia de BLEE obtenidas de las cepas de Argentina, Brasil, Chile, Colombia, México y Uruguay fue de 41,8% (136/325) en K. pneumoniae y de 10,3% (64/620) en E. coli (4). Estos resultados son bastante similares a los encontrados en nuestro estudio, principalmente para K. pneumoniae, cuyo porcentaje de cepas productoras de BLEE fue 44,4% (8/88), mientras que para E. coli fue 2,9% (4/137). Como se puede apreciar, las BLEE están presentes en nuestro medio y confirman que este mecanismo de resistencia constituye un problema creciente en instituciones medicas de Latinoamérica.
En pruebas de susceptibilidad frente a los antibióticos b-lactámicos, las 12 cepas confirmadas fenotípicamente como productoras de BLEE fueron intermedias o resistentes a cefalosporinas de tercera generación (ceftazidima, cefotaxima y ceftriaxona) y a aztreonam, y susceptibles a imipenem. Particularmente, C7 y C9 no mostraron un perfil de resistencia compatible con una BLEE, debido posiblemente a una baja o relativamente baja expresión de la producción de BLEE, las cuales siendo productoras de BLEE pudieron haber sido calificadas erróneamente como sensibles a cefalosporinas de tercera generación y aztreonam en un antibiograma de rutina.
En cuanto a la susceptibilidad a amoxicilina/ácido clavulánico, se observó que 10 de las 12 cepas productoras de BLEE fueron resistentes al inhibidor de b-lactamasa (ácido clavulánico), lo cual podría deberse a diversos mecanismos. Esta resistencia puede deberse probablemente a alteraciones de la permeabilidad de la membrana externa de la bacteria por modificación de las porinas (proteínas que actúan como canales), o a la producción de b-lactamasa AmpC (6) o por la hiperproducción de BLEE (7).
En general, las BLEE presentan susceptibilidad a cefoxitina, pero las 12 cepas productoras de BLEE, 4 cepas (C2, C5, C6 y C11) no fueron susceptibles, posiblemente debido a la producción de AmpC o pérdida de porinas de la membrana externa (8).
La multirresistencia determinada en las 12 cepas productoras de BLEE se debería a mutaciones cromosómicas (9-11) y extra cromosómicas (14-15) en los genes que dan origen a los blancos terapéuticos de estos antibióticos (aminoglicósidos, sulfametoxazol/ trimetroprim, ciprofloxacina). Por tanto, las opciones terapéuticas para el tratamiento de infecciones causadas por BLEE en nuestro medio estarían limitadas al uso de carbapenems.
En el presente estudio, se describe las cepas de E. coli y K. pneumoniae productoras de BLEE, principalmente de los tipos TEM y SHV, 4 para TEM y 6 para SHV, determinadas por PCR utilizando cebadores específicos y controles (positivo y negativo). Sin embargo, en dos cepas productoras de BLEE no se obtuvo producto de amplificación para ninguno de los genes estudiados (blaTEM y blaSHV) con los cebadores utilizados. Esto indicaría que las dos cepas podrían contener genes que codifican otras BLEE como las del tipo CTX-M descritas en Argentina (17) y Brasil (18).
La secuencia de ADN reveló la presencia de dos tipos de BLEE; TEM-10, que ha sido hallada principalmente en EU de A (19) y con menor frecuencia en Europa (20); la otra SHV-5 ha sido hallada en muchos países (21-24). El presente trabajo es el primer reporte de TEM-10 y SHV-5 hallados en nuestro país y es el primer reporte de TEM-10 en Latinoamérica.
Las BLEE halladas son de particular importancia, ya que en comparación con otras están más frecuentemente asociadas con la multirresistencia, hecho demostrado en este estudio y por otros autores (14,25) y que pueden mantenerse por prolongados periodos de tiempo y causar brotes en los hospitales (26). La alternativa terapéutica más recomendable para el tratamiento de bacterias productoras de TEM-10 y SVH-5 sería el imipenem, pero el uso indiscriminado de esta droga podría generar el incremento de la frecuencia de P. aeruginosa y Acinobacter spp resistente al imipenem (27,28). Por ello, seria necesario hacer una revisión más minuciosa y continua de la literatura sobre los tratamientos contra estos tipos de BLEE, sobre todo por parte de los prescriptores.
Tanto TEM-10 y SVH-5 fueron hallados en ambos hospitales y probablemente se encuentre en otros hospitales de Lima y provincias. Los factores que podrían haber facilitado el desarrollo de tal situación epidemiológica incluyen principalmente el largo uso de cefalosporinas de espectro ampliado, en especial ceftazidima, la transferencia de pacientes entre salas y hospitales y la falta de una adecuada monitorización y control de infecciones, causados por organismos productores de BLEE.
AGRADECIMIENTOS
A la Dra. Sara Palomino Berríos y al Dr. Eduardo Céliz Gutiérrez por habernos facilitado la recolección de cepas clínicas de los Hospitales Edgardo Rebagliati Martins y Guillermo Almenara Irigoyen.
REFERENCIAS BIBLIOGRÁFICAS
1. Pitout JDD, Thomson KS, Hanson ND, Ehrhardt AF, Moland ES, Sanders CC. b-lactamases responsible for resistance to expanded-spectrum cephalosporins in Klebsiella pneumoniae, Escherichia coli and Proteus mirabilis isolates recovered in South Africa. Antimicrob Agents and Chemother. 1999;42(6):1350-4. [ Links ]
2. Mabilat C, Goussard S. PCR detection and identification of genes for extended-spectrum b-lactamases. En: Persing DH, Smith TF, Tenover FC, White TJ (ed.). Diagnostic molecular microbiology: principles and applications. Washington, D.C.: American Society for Microbiology; 1993. p. 553-9. [ Links ]
3. Kim J, Lee H. Rapid discriminatory detection of genes coding for SHV b-lactamases by ligase chain reaction. Antimicrob Agents Chemother. 2000;44(7):1860-4. [ Links ]
4. Gales C, Jones R, Gordon K, Sader H, Wilke W, Beach M. Activity and spectrum of antimicrobial agents tested again urinary tract infection pathogens in hospitalized patients in Latin America: report from the second year of the SENTRY antimicrobial surveillance program. J Antimicrob Chemother. 1998;45:295-303. [ Links ]
5. Philippon A, Arlet G, Jacoby GA. Plasmid-determined AmpC-type b-lactamases. Antimicrob Agents Chemother. 2002;46(1):1-11. [ Links ]
6. Stapleton P, Pei-Jun W, King, A, Shannon K, French G, Phillips I. Incidence and mechanisms of resistance to the combination of amoxicillin and acid clavulanic in Escherichia coli. Antimicrob Agents Chemother. 1995;39:2478-83. [ Links ]
7. French GL, Shannon KP, Simmons, M. Hospital outbreak of Klebsiella neumoniae resistant to broad spectrum cephalosporins and betalactam-betalactamases inhibitor combinations by hyperproduction of SHV-5 betalactamase. J Clin Microbiol. 1996;34:358-63. [ Links ]
8. Martins M, Aguiar B, Lorenzato V, Rocha MR, DosSantos G, Dutra M. Molecular epidemiology of extended-spectrum b-lactamase-producing Klebsiella pneumoniae isolated from neonatal intensive care unit patients involved in hospital infection cases in Rio de Janeiro, Brazil. Rev Latin Microbiol. 2001;43(2):88-95. [ Links ]
9. Paterson DL, Mulazimoglu L, Casellas JM, Ko WC, Goossens H, Gottberg AV. Epidemiology of ciprofloxacin resistance and its relationship to extended spectrum b-lactamase production in Klebsiella pneumoniae isolates causing bacteremia. Clin Infect Dis. 2000;30:473-8. [ Links ]
10. Huovinen P, Sundstrom L, Swedberg G, Skold O. Trimethoprim and sulfonamide resistance. Antimicrob Agents Chemother. 1995;(2):279-89. [ Links ]
11. Minueto-Leclerco, M, Glupcsynskl Y, Tulkens PM. Aminoglycosides: activity and resistance. Antimicrob Agents Chemother. 1999;(4):727-37. [ Links ]
12. Bradford P. Extended –spectrum b-lactamase in the 21st Century: Characterization, epidemiology and detection of this important resistant threat. Clin Microbiol Rev. 2001;14(4):933-51. [ Links ]
13. Arlet G, Rouveau M, Casin I, Bouvet PJ, Lagrange PH, Philippon A. Molecular epidemiology of Klebsiella pneumoniae strains that produce SHV-4 b-lactamase that produce SHV-4 and which were isolated in 14 French hospitals. J Clin Microbiol. 1994;32:2553-8. [ Links ]
14. Sekowska, A, Janicka G, Klyszejko C, Wojda M, Wróblewski M, Szymankiewicz M. Resistance of Klebsiella pneumoniae strains producing and not producing ESBL (extended-spectrum betalactamase) type enzymes to selected non-beta-lactam Antibiotics. Med Sci Monit. 2002;8(3):BR100-4. [ Links ]
15. Lautenbach E, Patel JB, Bilker WB, Edelstein PH. Firshman NO. Extended-spectrum b-lactamase-producing Escherichia coli and Klebsiella pneumoniae: Risk factors for infection and impact of resistance on outcomes. Clin Infect Dis. 2001;32:1162-71. [ Links ]
16. Ambler RP, Coulson AF, Frère JM, Ghuysen JM, Joris B, Forsman M. A standard numbering scheme for the class A b-lactamases. Biochem J. 1991;276:269-70. [ Links ]
17. Guzmán-Blanco M, Casellas JM, Silva H. Bacterial resistance to antimicrobial agents in Latin America the giant is awakening. Infectious Disease Clinics of North America. 2000;14(1):67-81. [ Links ]
18. Bonnet R, Dutour C, Sampaio J, Chanal C, Sirot D, Labia R. Novel cefotaximase (CTX-M-16) with increased catalytic efficiency due to substitution Asp-240-Gly. Antimicrob Agents Chemother. 2001;45(8):2269-75. [ Links ]
19. Yang Y, Bhanchech N, Bradford PA, Jett BD, Sahm DF, Bush B. Ceftazidime-resistant Klebsiella pneumoniae and Escherichia coli isolates producing TEM-10 and TEM-43 b-lactamases from St. Louis, Missouri. Antimicrob Agents Chemother. 1998;42(7):1671-6. [ Links ]
20. Barroso H, Freitas-Vieira A, Lito l.M, Melo J, Salgado M J, Ferreira H. Survey of Klebsiella pneumoniae producing extended-spectrum b-lactamases at a Portuguese hospital: TEM-10 as the endemic enzyme. J Antimicrob Chemother. 2000;45:611-6. [ Links ]
21. Gniadkowski M, Schneider I, Jungwirth R, Hryniewicz W, Bauernfeind A. Ceftazidime-reistant Enterobacteriaceae isolates from three Polish hospitals: Identification of three novel TEM- and SHV-5-type extended-spectrum b-lactamases. Antimicrob Agents Chemother. 1998;42(3):514-20. [ Links ]
22. Siu LK, Lu P, Hsueh PR, Lin FM, Chang SC, Luh KT. Bacteremia due to extended-spectrum b-lactamase-producing Escherichia coli and Klebsiella pneumoniae in a pediatric oncology ward: Clinical features and identification of different plasmids carrying both SHV-5 and TEM-1 genes. J Clin Microbiol. 1999;37(12):4020-7. [ Links ]
23. Szabo D, Filetoth Z, Szentandrassy J, Nemedi M, Toth E, Jeney C. Molecular epidemiology of a cluster of cases due to Klebsiella pneumoniae producing SHV-5 extended-spectrum b-lactamase in the premature intensive care unit of a Hungarian hospital. J Clin Microbiol. 1999;37(12):4167-9. [ Links ]
24. Prodinger WM, Fille M, Bauernfeind A, Stemplinger I, Amann S, Pfausler B. Molecular epidemiology of Klebsiella pneumoniae producing SHV-5 b-lactamase: Parallel outbreaks due to multiple plasmid transfer. J Clin Microbiol. 1996;34(3):564-8. [ Links ]
25. Fridkin SK, Gaynes RP. Antimicrobial resistance in intensive care units. Clinics in Chest Medicine. 1999;20(2):303-16. [ Links ]
26. Rice LB, Eckstein EC, De Vente J, Sales DM. Ceftazidime-resistant Klebsiella pneumoniae isolates recovered at the Cleveland Department of Veterans Affairs Medical Center. Clin Infect Dis. 1996;23:118-24. [ Links ]
27. Nathisuwan S, Burgess D, Lewis J. Extended-spectrum b-lactamases: Epidemiology, detection, and treatment. Pharmacotherapy. 2001;21(8):920-8. [ Links ]
28. Wong A. Therapeutic challenges associated with extended-spectrum b-lactamase-producing Escherichia coli and Klebsiella pneumoniae. Pharmacotherapy. 2001;21(5):583-92. [ Links ]
Manuscrito recibido el 08 de febrero de 2005 y aceptado para publicación el 15 marzo 2005.
Correspondencia:
Mg. Mario Monteghirfo Gomero
Centro de Investigación de Bioquímica y Nutrición, UNMSM
Av. Grau 755. Lima 1, Perú
Correo-e: mariomg@hotmail.com