Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Anales de la Facultad de Medicina
Print version ISSN 1025-5583
An. Fac. med. vol.69 no.4 Lima Dec. 2008
Relación del polimorfismo C-514T del gen de la lipasa hepática con indicadores nutricionales y lipoproteínas en una muestra poblacional peruana: una perspectiva nutrigenética
Relation of the hepatic lipase gene C-514T polymorphism with nutritional indicators and lipoproteins in a Peruvian population sample: a nutrigenetic perspective
Doris Huerta1, Oscar Acosta1, Cecilia Miranda2, Susan Polo1, Raquel Oré1, Amelia Bardales2, Jaime Pajuelo2
1 Centro de Investigación de Bioquímica y Nutrición. Facultad de Medicina, Universidad Nacional Mayor de San Marcos. Lima, Perú.
2 Facultad de Medicina, Universidad Nacional Mayor de San Marcos. Lima, Perú.
Resumen
Introducción: Las enfermedades cardiovasculares son una de las principales causas de muerte en todo el mundo. El promotor del gen de la lipasa hepática presenta un polimorfismo funcional C-514T que se relaciona con la actividad de la enzima, la variación de los niveles de lipoproteínas y un posible riesgo para desarrollar enfermedades cardiovasculares. Objetivos: Establecer la relación del polimorfismo C-514T del promotor del gen de la lipasa hepática con indicadores nutricionales y los niveles de lipoproteínas plasmáticas en una muestra de peruanos saludables. Diseño: Estudio descriptivo, transversal, asociativo. Lugar: Centro de Investigación de Bioquímica y Nutrición Alberto Guzmán Barrón, Facultad de Medicina, Universidad Nacional Mayor de San Marcos. Participantes: Noventiuna personas sanas de ambos sexos, cuyas edades fluctuaban entre 18 y 58 años, voluntarios con consentimiento informado. Intervenciones: Extracción del ADN genómico a partir de muestras sanguíneas según metodología estándar. Toma de medidas antropométricas, estableciéndose los indicadores nutricionales, determinación del perfil lipídico por el método enzimático. Análisis del polimorfismo C-514T mediante la técnica de PCR/RFLP, con primers específicos y digestión con la enzima de restricción NlaIII, detectándose los fragmentos de RFLP por electroforesis en gel de poliacrilamida (PAGE) y tinción con nitrato de plata. Principales medidas de resultados: Frecuencias genotípicas y alélicas del gen de la lipasa hepática y relación con parámetros lipídicos y nutricionales. Resultados: Se encontró las frecuencias genotípicas CC=0,143; CT=0,593 y TT = 0,264, siendo la distribución consistente con el equilibrio de Hardy-Weinberg (X2 =3,8024, g.l.=1, p = 0,086). Las frecuencias alélicas fueron alelo C = 0,4395 y el alelo T = 0,5605. Los niveles de colesterol, HDLc, LDLc, TG y los promedios de pliegue subcutáneo, el IMC y el porcentaje de grasa en los genotipos CC, CT y TT, no mostraron diferencias significativas (p > 0,05). Sin embargo, cuando se analizó solo al alelo T (genotipos CT y TT), según rangos de edad y sexo, se verificó diferencias significativas en algunos parámetros. Conclusiones: El genotipo heterocigoto CT y el alelo T, relacionado con una actividad baja de la lipasa hepática, fueron los más frecuentes en esta muestra peruana; además, la presencia del alelo T se asoció a variaciones en los niveles de lipoproteínas e indicadores nutricionales, lo que pudiera tener influencia sobre las enfermedades cardiovasculares.
Palabras clave: Genética; lipasa hepática; polimorfismo genético; lipoproteínas; enfermedades cardiovasculares.
Abstract
Introduction: Cardiovascular diseases are major causes de death in the world. The hepatic lipase (HL) gene promoter region presents a C-514T functional polymorphism related to enzyme activity, variation of lipoproteins levels and possible cardiovascular disease risk. Objectives: To determine the association of HL gene promoter region polymorphism with both nutritional indicators and lipoproteins levels in a healthy Peruvian sample. Design: Descriptive, transversal, associative study. Setting: Alberto Guzman Barron Biochemistry and Nutrition Research Center, Faculty of Medicine, Universidad Nacional Mayor de San Marcos. Participants: Ninety healthy male and female volunteers aged 18 to 58 years. Interventions: Genomic DNA was obtained from serum samples according to standard methodology. Anthropometric measurements and lipid profile by enzymatic methods were performed. Polymorphism C-514T in the HL gene was determined by polymerase chain reaction (PCR). PCR products were digested with NlaIII and fragments separated by polyacrilamyde gel electrophoresis (PAGE) and stained with silver nitrate. Main outcome measures: HL gene genotypes and alleles frequencies and relation with both lipid and nutritional parameters. Results: We found genotype frequencies CC=0,143; CT=0,593 and TT = 0,264, consistent with Hardy-Weinberg equilibrium (X2 =3,8024, g.l. = 1, p = 0,086). Alleles frequencies were C allele = 0,4395 and T allele = 0,5605. HDLc, LDLc, TAG cholesterol levels and subcutaneous fold, BMI and fat percentage averages in CC, CT and TT genotypes did not show significant differences (p>0,05). Nevertheless, when T allele was analyzed alone (genotypes CT and TT) according to age and sex there were significant differences (p<0,05) in some parameters. Conclusions: CT heterozygote and T allele, related to HL low activity were the most frequent genotypes in this Peruvian sample; in addition, presence of T allele was associated with variations in lipoprotein levels and nutritional factors which could have influence on cardiovascular diseases.
Key words: Genetic; hepatic lipase; polymorphism, genetic; lipoproteins; cardiovascular diseases.
INTRODUCCIÓN
Las enfermedades cardiovasculares son un conjunto de patologías del corazón y vasos sanguíneos, principales causas de muerte a nivel mundial. Se encuentran asociadas con ateroesclerosis e hipertensión y son el resultado de factores genéticos y ambientales. Se ha establecido una fuerte asociación con el tipo de dieta, considerando también que las interacciones genes-medio ambiente modulan las concentraciones de lipoproteínas plasmáticas y potencialmente el riesgo para estas enfermedades (1-3).
Diversos genes relacionados con la función y el metabolismo de las lipoproteínas, además de factores de transcripción que actúan como sensores de nutrientes en la célula, son investigados en el contexto de la nutrición, para la prevención de enfermedades cardiovasculares. Es muy conocido que el efecto provocado por los cambios en la dieta, principalmente de lipoproteínas, difiere significativamente entre individuos; algunos parecen ser insensibles a los cambios en la dieta y otros son muy sensibles, postulándose un componente genético. En esa perspectiva, surge una nueva disciplina denominada nutrigenética, que se centra en el estudio de genes asociados a la distinta respuesta fenotípica a la dieta (4).
El conocimiento actual, sin embargo, es muy limitado para su aplicación en la clínica, por lo que es necesario el estudio de polimorfismos genéticos relacionados con lipoproteínas. Una de las enzimas importantes es la lipasa hepática humana (EC 3.1.1.3), sintetizada por los hepatocitos y que tiene diversas funciones metabólicas, tales como la hidrólisis de los triglicéridos, la lipólisis de fosfolípidos, el modelado de las partículas de LDL y el transporte reverso del colesterol HDL. Además, la participación en la variación de los niveles plasmáticos de colesterol HDL tiene relación con el tipo de dieta y el origen étnico de las poblaciones (5). A nivel molecular, el promotor del gen de la lipasa hepática, localizado en el cromosoma 15, presenta un polimorfismo funcional que se relaciona con una menor o mayor actividad de la enzima y, por consiguiente, estaría relacionado con el tipo de dieta y con un posible riesgo para desarrollar enfermedades cardiovasculares (6).
El promotor del gen de la lipasa hepática presenta polimorfismo en la posición -514, en la cual una citosina puede ser reemplazada por una timina, cambio que afecta la transcripción; se generan los genotipos CC, CT y TT, relacionados con fenotipos de actividad enzimática alta y baja. Se ha comunicado que la presencia del alelo T (genotipos CT y TT) está asociado con actividad baja de la lipasa hepática -en personas saludables y pacientes con enfermedad cardiovascular-, niveles elevados de lipoproteínas -especialmente el HDLc (asociado con menor riesgo de enfermedad)- y con incremento de grasa abdominal, en mujeres (7-9).
Las investigaciones señalan que el polimorfismo en el promotor del gen de la lipasa hepática puede estar asociado, en interacción con otros genes y factores ambientales -como la dieta-, con el riesgo para desarrollar enfermedades cardiovasculares, además de estar involucrado con variaciones en los niveles de lipoproteínas e indicadores nutricionales, en personas aparentemente saludables (8-10). Desde que las primeras investigaciones establecieron relación entre el polimorfismo C-514T en el promotor del gen de la lipasa hepática y el HDLc (11), se hace necesario también estudiar su interacción con indicadores nutricionales, en muestras de personas aparentemente saludables, que es el objetivo del presente estudio. Y más aún, en nuestro país, caracterizado por su diversidad genética, y como una etapa previa para estudios de asociación con enfermedades cardiovasculares, ya que representan actualmente un gran problema de salud pública.
MÉTODOS
Se realizó un estudio observacional transversal. Los participantes fueron en total 91 personas, 70 mujeres y 21 varones, cuyas edades fluctuaban entre los 18 y 58 años, clínicamente saludables, sin antecedentes clínicos de dislipidemias ni enfermedades cardiovasculares, hepáticas, renales y diabetes. Todos los paticipantes firmaron voluntariamente un consentimiento informado, que incluía la finalidad de la investigación, procedimientos, riesgos y beneficios, condiciones y confidencialidad.
Se utilizó el suero de los participantes del estudio para la determinación del perfil lipídico: colesterol total, colesterol HDLc, colesterol LDLc, colesterol VLDL y triglicéridos (TAG), expresados en mg/dL, utilizando kits enzimáticos comerciales (Wiener). También, se efectuó medidas antropométricas; la composición corporal fue medida por impedancia bioeléctrica, como porcentaje de grasa corporal, y se calculó el índice de masa corporal (IMC = peso kg/talla m2). La circunferencia cintura cadera fue medida en posición supina; el índice cintura cadera se calculó como un índice de distribución de grasa corporal.
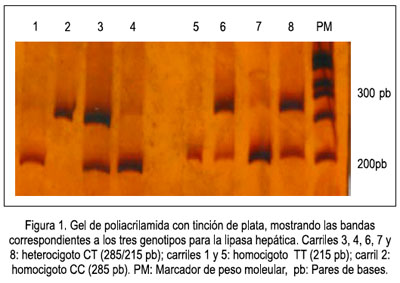
El ADN genómico fue aislado de sangre periférica, según el método estandar FTA Cards de Whatman. El genotipaje de lipasa hepática se realizó según metodología publicada por Fan y col (10,12). Una secuencia de 285 pares de bases del gen de la lipasa hepática se amplificó utilizando la técnica de reacción en cadena de la polimerasa (PCR), en un termociclador Perkin Elmer Gene Amp PCR System 2400, empleando primers específicos para lipasa hepática, forward AAG AAG TGT GTT TAC TCT AGG ATC A y reverse GGT GGC TTC CAC GTG GCT GCC TAA G, con las siguientes condiciones: denaturación a 95°C por 3 minutos y luego cada PCR fue de 35 ciclos, de 1 minuto de denaturación, a 95°C, 0,5 minutos de alineamiento a 63°C y 0,5 minutos de extensión a 72°C, extensión final de 7 minutos a 72°C. Los productos amplificados fueron digeridos con la enzima de restricción NlaIII. Los fragmentos fueron separados por electroforesis en geles de poliacrilamida al 8% y visualizados con tinción de plata, generándose bandas características para cada genotipo: CC (285 pb), CT (285/215 bp) y TT (215 pb) (figura 1).
Las frecuencias genotípicas y alélicas fueron obtenidas por conteo directo. Se aplicó la prueba de chi cuadrado (X2) para evaluar las frecuencias observadas, según lo esperado bajo la hipótesis del equilibrio de la genética de las poblaciones de Hardy-Weinberg. Se calculó la media y la desviación estandar de los datos antropométricos, nutricionales, bioquímicos y se les comparó según genotipo y/o alelo, considerando el sexo y la edad, aplicando las pruebas estadísticas t student y Anova. Para los cálculos respectivos, se utilizó el paquete estadístico SPSS 15.0 y programas de genética poblacional.
RESULTADOS
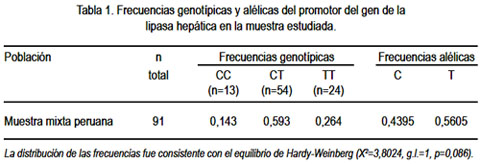
Se encontró las siguientes frecuencias genotípicas: CC=0,143 (14%); CT=0,593 (60%) y TT=0,264 (26%); la distribución fue consistente con el equilibrio de Hardy-Weinberg (X2 =3,8024, g.l.=1, p = 0,086). Las frecuencias alélicas encontradas fueron: alelo C = 0,4395 (43%) y para el alelo T = 0,5605 (56%) (tabla 1).
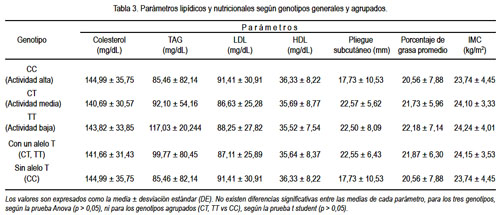
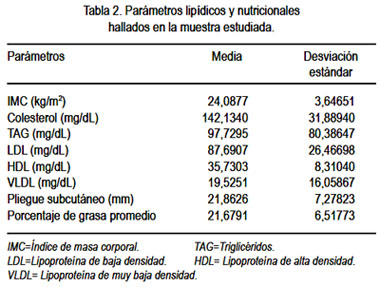
Los parámetros lipídicos y nutricionales investigados (IMC, colesterol total, TAG, LDL, HDL, VLDL, pliegue subcutáneo y posrcentaje de grasa) se describen en la tabla 2.
Los niveles de colesterol HDLc, LDLc, TAG y los promedios de pliegue subcutáneo, el IMC y el porcentaje de grasa comparados, según los genotipos CC, CT y TT o agrupando los genotipos según presencia o ausencia del alelo T, no mostraron diferencias significativas (p> 0,05), según prueba ANOVA y t student, respectivamente (tabla 3). Sin embargo, el análisis de los niveles de lipoproteínas y parámetros nutricionales considerando solo la presencia del alelo T (CT=54 y TT=24; n=78) revela diferencias significativas para los parámetros HDLc y pliegue subcutáneo según sexo (tabla 4), y para VLDL, colesterol total, TG, LDLc, pliegue subcutáneo, porcentaje de grasa corporal e IMC, según rango de edad menor o mayor de 40 años, destacándose que no existen diferencias para el HDLc, al aplicarse la prueba t student. Se usó el rango de edad (40 años) debido a que se presentan las diferencias más significativas en los parámetros estudiados (tabla 5).
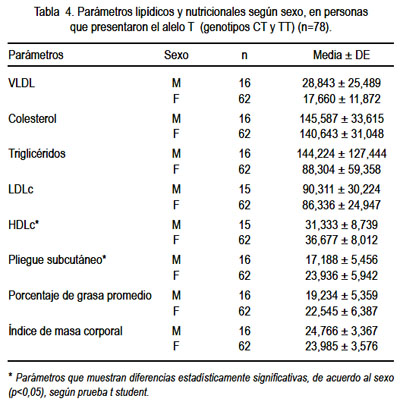
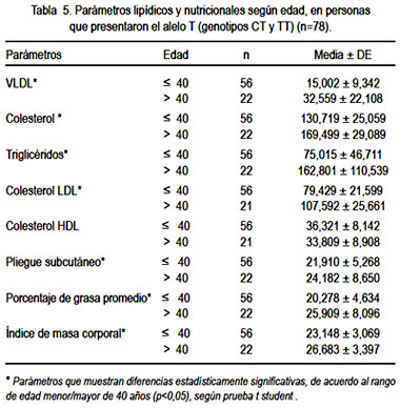
Estos resultados se mantienen, parcialmente, cuando se estudian otros rangos de edad, como 30 y 50 años, pero ya muestran diferencias significativas en los niveles de HDLc según rango de edad mayor y menor de 25 años (datos no mostrados).
DISCUSIÓN
Nuestro país tiene una serie de problemas de salud pública, siendo el manejo de las enfermedades prevalentes una tarea pendiente a la luz de los nuevos conocimientos en genética y medicina molecular. En el Perú, las principales enfermedades prevalentes son las cardiovasculares y por consiguiente es imperioso incluir el aspecto genético molecular para desarrollar las estrategias integrales de prevención, control y tratamiento.
El polimorfismo C-514T en el promotor del gen de la lipasa hepática puede estar asociado con niveles variables de lipoproteínas y con las enfermedades cardiovasculares.
Las frecuencias de los genotipos encontradas en esta población peruana (tabla 1) son las más bajas para el genotipo CC (14%) y las más altas para el genotipo CT (60%), si las comparamos con las frecuencias publicadas para muestras caucásicas, afroamericanas y asiáticas del mundo; mientras tanto, la frecuencia del genotipo TT (26%) es similar a muestras mixtas de los Estados Unidos y Japón, considerando que es variable en otras poblaciones y que este genotipo es muy raro en poblaciones caucásicas (8,9).
La frecuencia del alelo T (56%) en esta muestra peruana, es una de las más altas con respecto al promedio encontrado para algunas poblaciones del mundo (25,3%) y solo similar al 55% publicado para la población nativa Oji-Cree, de Canadá (9,13).
El 86% de los individuos de esta muestra tiene al menos un alelo T, lo cual podría ser relacionado con una actividad de LH baja, niveles altos de HDLc y probablemente un menor o mayor riesgo para enfermedades cardiovasculares.
Los parámetros lipídicos y nutricionales evaluados en esta muestra, según el genotipo, no muestran diferencias significativas entre las medias de cada parámetro para los tres genotipos (p> 0,05) ni para los genotipos agrupados, es decir CT, TT vs. CC (p> 0,05) (tabla 3). Estos resultados son similares a los encontrados para otras poblaciones (13-19), pero difieren de lo informado por otros investigadores, que encuentran asociación del polimorfismo, principalmente, con variaciones en los niveles de HDL según genotipo (5,6,11,20-23). Las discrepancias en los resultados pueden ser explicados por diversos factores. Pero, los estudios de metaanálisis demuestran la importancia de este polimorfismo en la actividad de la lipasa hepática y un efecto positivo sobre los niveles plasmáticos y el metabolismo de HDLc (8) y la dieta (9).
Cuando se agrupa solo los portadores del alelo T (genotipos CT y TT), predominantes en esta muestra, según sexo, se verifica diferencias significativas (p< 0,05) para los niveles de HDLc y valores de pliegue subcutáneo (indicador nutricional), ambos mayores en mujeres comparados con los hombres (tabla 4). Esto implicaría un efecto positivo sobre los niveles de HDLc en las mujeres de la muestra, aunque se encuentra en otras poblaciones efectos positivos tanto en hombres como en mujeres (6) y otros lo hallan solo en hombres (11). Con repecto al pliegue subcutáneo, podría tener relación con los niveles de HDLc, pero es importante considerar que el alelo T puede asociarse a un perfil lipídico aterogénico (efecto negativo), cuando el contenido en grasas excede el 30% del total de la dieta consumida (8).
Por otro lado, cuando se agrupa los portadores del alelo T según rango de edad mayor y menor de 40 años, se verifica diferencias significativas (p< 0,05) para todos los parámetros nutricionales y lipídicos, salvo para los niveles de HDLc (p> 0,05) (tabla 4). Estos parámetros, en general, son muy variables según grupo etáreo; sin embargo, puede verificarse un efecto del alelo T en la variación de los niveles de HDLc, aunque puede ser influenciado por el tipo de dieta y por la procedencia étnica (5,8). Esto sugiere un efecto del alelo T según la edad, inclusive asociado a riesgo para enfermedades cardiovasculares comunicado en otras poblaciones (24).
Se concluye que el genotipo heterocigoto CT y el alelo T, relacionado con una baja actividad de la lipasa hepática, son los más frecuentes en esta muestra peruana aparentemente saludable; además, la presencia del alelo T se asocia a variaciones en los niveles de lipoproteínas e indicadores nutricionales, considerando la edad y el sexo; todo ello podría tener influencia sobre las enfermedades cardiovasculares.
A pesar de la importancia del estudio de factores genéticos asociados a nutrición, niveles de lipoproteínas y enfermedades cardiovasculares, se debe tener en cuenta las limitaciones de la presente investigación, como por ejemplo el tamaño muestral, la predominancia de mujeres, la mixtura o mestizaje de los participantes; por lo que, los resultados e inferencias derivadas de las mismas deben ser consideradas preliminares y tomadas con cautela. Posteriores estudios de asociación de tipo cohortes o casos-controles para enfermedades cardiovasculares, una necesidad en nuestro país, deben tener en cuenta estos aspectos.
Debido a su relevancia, este polimorfismo C-154T en el promotor del gen de la lipasa hepática, en interacción con otros genes y con factores ambientales, en el contexto de la nutrigenética, debe ser considerado en los estudios de riesgo para enfermedades cardiovasculares, tanto en poblaciones mixtas como en nativas.
AGRADECIMIENTOS
Al Consejo Superior de Investigaciones de la Universidad Nacional Mayor de San Marcos, por la financiación del proyecto, FEDU 2007.
REFERENCIAS BIBLIOGRÁFICAS
1. Schaefer E. Lipoproteins, nutrition, and heart disease. Am J Clin Nutr. 2002; 75:191-212. [ Links ]
2. Hooper L, Summerbell CD, Higgins JPT, Thompson RL, Capps NE, Smith GD, et al. Dietary fat intake and prevention of cardiovascular disease: systemic review. Br Med J. 2001;322:757-63. [ Links ]
3. Loktionov A. Common gene polymorphisms and nutrition: emerging links with pathogenesis of multifactorial chronic diseases (Review). J Nutr Biochem. 2003;14:426-51. [ Links ]
4. Ordovas J. Nutrigenetics, plasma lipids, and cardiovascular risk. J Am Diet Assoc. 2006;106:1074-81. [ Links ]
5. Ordovas J, Corella D, Demissie S, Cupples A, Couture P, Coltell O, et al. Dietary fat intake determines the effect of a common polymorphism in the hepatic lipase gen promotor on high-density lipoprotein metabolism. Evidence of a strong dose effect in this gene-nutrient interaction in the Framingham study. Cicrculation. 2002;106:2315-21. [ Links ]
6. Couture P, Otvos J, Cupples A, Lahoz C, Wilson P, Schaefer E, et al. Association of the C-514T polymorphism in the hepatic lipase gene with variations in lipoprotein subclass profiles. Arterioscler Thromb Vasc Biol. 2000;20:815-22. [ Links ]
7. Perret B, Mabile L, Martinez L, Tercé F, Barbaras R, Collet X. Hepatic lipase: structure/function relationship, synthesis, and regulation. J Lipid Res. 2002;43:1163-9. [ Links ]
8. Tai E, Corella D, Deurenberg-Yap M, Cutter J. dietary fat interacts with the _514C>T polymorphism in the hepatic lipase gene promoter on plasma lipid profiles in a multiethnic asian population: The 1998 Singapore National Health Survey. J Nutr. 2003;133:3399-408. [ Links ]
9. Isaacs A, Sabed-Tabatabaei F, Njajou O, Witteman J, Van Duijn C. The -514C-T hepatic lipase promotor region polymorphism an plasma lipids: a meta-analyisis. J Clin Endocrinol Metab. 2004;89(8):3858-63. [ Links ]
10. Fan Y, Laaksonen R, Janatuinen T, Vesalainen R. Hepatic lipase gene variation is related coronary reactivity in healthy young men. Eu J Clin Invest. 2001;31:574-80. [ Links ]
11. Guerra R, Wang J, Grundy S, Cohen J. A hepatic lipase (LIPC) allele associated with high plasma concentrations of high density lipoprotein cholesterol. PNAS. 1997;94:4532-7. [ Links ]
12. Fan YM, Dastidar P, Jokela H, Punnonen R, Lehtimäki T. Hepatic lipase C-480T genotype-dependent benefit from long-term hormone replacement therapy for atherosclerosis progression in postmenopausal women. J Clin Endocrinol Metab. 2005;90:3786-92. [ Links ]
13. Hegele R, Harris S, Brunt J, Young T, Hanley A, Zinman B, et al. Absence of association between genetic variation in the LIPC gene promoter and plasma lipoproteins in three Canadian populations. Atherosclerosis. 1999;146:153-60. [ Links ]
14. Vega GL, Gao J, Bersot TP, Mahley RW, Verstraete R, Grundy SM, et al. The _514 polymorphism in the hepatic lipase gene (LIPC) does not influence androgen-mediated stimulation of hepatic lipase activity. J Lipid Res. 1998;39:1520-4. [ Links ]
15. Grundy SM, Vega GL, Otvos JD, Rainwater DL, Cohen JC. Hepatic lipase activity influences high density lipoprotein subclass distribution in normotriglyceridemic men. Genetic and pharmacological evidence. J Lipid Res. 1999;40:229-34. [ Links ]
16. Carr MC, Ayyobi AF, Murdoch SJ, Deeb SS, Brunzell JD. Contribution of hepatic lipase, lipoprotein lipase, and cholesteryl ester transfer protein to LDL and HDL heterogeneity in healthy women. Arterioscler Thromb Vasc Biol. 2002;22:667-73. [ Links ]
17. Fang DZ, Liu BW. Polymorphism of HL _1075C, but not _480T, is associated with plasma high density lipoprotein cholesterol and apolipoprotein AI in men of a Chinese population. Atherosclerosis. 2002;161:417-24. [ Links ]
18. Juo SH, Han Z, Smith JD, Colangelo L, Liu K. Promoter polymorphisms of hepatic lipase gene influence HDL(2) but not HDL(3) in African American men: CARDIA study. J Lipid Res. 2001;42:258-64. [ Links ]
19. St-Pierre J, Miller-Felix I, Paradis ME, Bergeron J, Lamarche B, Despres JP, et al. Visceral obesity attenuates the effect of the hepatic lipase _514C3T polymorphism on plasma HDL-cholesterol levels in French-Canadian men. Mol Genet Metab. 2003;78:31-6. [ Links ]
20. Hubacek JA, Waterworth DM, Pitha J, Humphries SE, Talmud PJ, Poledne R. Polymorphisms in the lipoprotein lipase and hepatic lipase genes and plasma lipid values in the Czech population. Physiol Res. 2001;50:345-51. [ Links ]
21. Murtomaki S, Tahvanainen E, Antikainen M, Tiret L, Nicaud V, Jansen H, et al. Hepatic lipase gene polymorphisms influence plasma HDL levels. Results from Finnish EARS participants. European Atherosclerosis Research Study. Arterioscler Thromb Vasc Biol. 1997;17:1879–84. [ Links ]
22. Chen W, Srinivasan SR, Boerwinkle E, Berenson GS. Hepatic lipase promoter C-514T polymorphism influences serial changes in HDL cholesterol levels since childhood: the Bogalusa Heart Study. Atherosclerosis. 2003;169:175-82. [ Links ]
23. Ko YL, Hsu LA, Hsu KH, Ko YH, Lee YS. The interactive effects of hepatic lipase gene promoter polymorphisms with sex and obesity on high-densitylipoprotein cholesterol levels in Taiwanese-Chinese. Atherosclerosis. 2004;172:135-42. [ Links ]
24. Fan Y, Lehtimaki T, Rontu R, Ilveskoski E, Goebeler S, Kajander O, et al. Age-dependent association between hepatic lipase gene C-480T polymorphism and the risk of pre-hospital suden cardiac death: The Helsinki Suden Death Study. Atherosclerosis. 2007;192(2):421-7. [ Links ]
Manuscrito recibido el 12 de octubre de 2008 y aceptado para publicación el 25 de noviembre de 2008.
Correspondencia:
Mg. Doris Huerta Canales
Centro de Investigación de Bioquímica y Nutrición
Facultad de Medicina - UNMSM
Av. Grau 755. Lima 1, Perú
Correo-e: dorishuerta@yahoo.com