Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Anales de la Facultad de Medicina
versión impresa ISSN 1025-5583
An. Fac. med. v.72 n.1 Lima ene./mar. 2011
ARTÍCULOS ESPECIALES SOBRE EL HOMBRE DE LA ALTURA
Divergencia genética en poblaciones peruanas detectada a partir de las frecuencias haplotípicas del mtDNA y del gen nuclear MBL
Peruvian populations genetic differences detected by both mtDNA and MBL nuclear gene haplotypical frequencies
Jesús H. Córdova1 ; Ricardo Fujita2 ; José Sandoval2 ; Jaime Descailleaux1 ; Margarita Velásquez1 ; Caleen Távara1 ; Claudia Barletta1
1 Laboratorio de Genética Humana, Facultad de Ciencias Biológicas, UNMSM.
2 Instituto de Genética y Biología Molecular, Facultad de Medicina, Universidad San Martín de Porras.
RESUMEN
Objetivos: Avanzar en el conocimiento del origen de las poblaciones peruanas estudiadas en un contexto filogeográfico. Diseño: Estudio genético poblacional. Instituciones: Laboratorio de Genética Humana, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos, e Instituto de Genética y Biología Molecular, Facultad de Medicina, Universidad San Martín de Porras, Lima, Perú. Participantes: Siete poblaciones peruanas. Metodología: Análisis comparativo de los resultados a partir del estudio del mtDNA y el gen nuclear MBL de siete poblaciones peruanas, procesados de manera separada y luego combinados, utilizando el programa PHYLYP 3.65, para obtener valores FST de diferenciación genética y la construcción de árboles de distancias por aplicación del algorritmo UPGMA y el análisis subsecuente de los agrupamientos (clusters) generados. Principales medidas de resultados: Árboles genéticos generados. Resultados: De manera separada, los árboles generados para cada marcador genético tuvieron topologías propias y diferentes entre sí. Procesados de manera combinada, el árbol resultante demostró que los mayores valores de diferenciación genética se hallaron en las Islas del Lago Titicaca (Puno, Perú) conocidas -Taquile, Amantani y Anapia-, que fue calificada como muy alta, porque mostró valores de FST de 0.3113, 0.2949 y 0.3348 respecto de las poblaciones estudiadas, tanto fuera del Departamento de Puno -como Chachapoyas, Pucallpa y Chiclayo, respectivamente-, así como a la de los Uro del mismo Puno y del mismo Lago Titicaca (0.2837). Fuera de Puno, el par de poblaciones Chachapoyas-Pucallpa fue el menos divergente, al alcanzar entre ellas un valor de FST de 0.0108, calificándosele de pequeña. Conclusiones: El árbol obtenido del procesamiento de los marcadores vía una matriz combinada demostró que las poblaciones que habitan las islas de Taquile, Amantani y Anapia, divergen notablemente de las restantes cuatro procesadas del Perú, incluyendo la más próxima a ellas dentro del mismo Lago Titicaca, como es la de los Uro. Explicar estos hallazgos será el siguiente objetivo de nuestras investigaciones, en principio, mediante la ampliación de los marcadores genéticos empleados y del número de poblaciones analizadas a nivel del Perú.
Palabras clave: mtDNA, MBL, filogeografía Perú, poblaciones peruanas.
ABSTRACT
Objectives: To advance in the knowledge of Peruvian populations origin in a phylogeographical context. Design: Population genetics study. Setting: Human Genetics Laboratory, Biological Sciences Faculty, Universidad Nacional Mayor de San Marcos, and Genetics and Molecular Biology Institute, Faculty of Medicine, Universidad San Martin de Porras, Lima, Peru. Participants: Seven Peruvian populations. Methods: Comparative analysis of mtDNA and MBL nuclear gene study results in seven Peruvian populations processed separately and then combined using PHYLYP 3.65 Program in order to obtain FST values of genetic differentiation; construction of distance trees by applying UPGMA algorithm and subsequent generated clusters analysis. Main outcome measures: Genetic trees. Results: Trees generated for each genetic marker had proper and distinct topologies among them. Combined processing resulted in a tree with higher values of genetic differentiation in Lago Titicaca Islands (Puno, Peru) Taquile, Amantani y Anapia, graded as very high because they showed 0.3113, 0.2949 y 0.3348 FST values with respect to the populations studied outside of Puno Department -like Chachapoyas, Pucallpa and Chiclayo-, as well as those of both Uros in same Puno and Lago Titicacas populations (0.2837). Out of Puno, the pair Chachapoyas-Pucallpa population was the least divergent with 0.0108 FST value between them, classifying as small. Conclusions: The tree obtained from markers by a combined matrix process determined that populations inhabiting in Taquile, Amantani y Anapia islands possess notable genetic divergence respect to the four remainders studied in Peru, including the Uros population geographically very close to them and within the same Lago Titicaca. Our next objective will be to explain these findings initially by increasing genetic markers and number of populations analyzed in Peru.
Key words: mtDNA, MBL, Peruvian phylogeography, Peruvian populations.
INTRODUCCIÓN
Uno de los estudios que vienen realizando los genetistas peruanos desde hace buen tiempo consiste en tratar de conocer la estructura genética, las relaciones de parentesco y la dinámica demográfica de las poblaciones peruanas, en especial las nativas o primigenias del Perú, principalmente porque se tiene la certeza a nivel global, de que sus resultados serán los insumos fundamentales para una planificación adecuada de las políticas de salud del estado peruano(1,2). Con este objetivo, se utiliza los avances que de manera creciente se vienen produciendo tanto en el conocimiento de nuevos y mejores marcadores genéticos como en los métodos existentes para su evaluación, como son aquellos que permiten hallar las relaciones genealógicas entre tales poblaciones (2-8).
A partir de la década del 90, uno de los marcadores más utilizados ha sido el de los haplotipos del ADN mitocondrial (mtDNA). Sus propiedades positivas y singulares para los estudios histórico-poblacionales o filogeográficos de la especie humana han aumentado exponencialmente desde el trabajo seminal de 1987 (9), y han sido tratados y proyectados a poblaciones peruanas por diferentes autores peruanos (2,7-8).
No obstante, es claro que a la fecha no existe evidencia alguna de que la evolución de una sola molécula pueda explicar la evolución de una especie (2). Así, en el presente estudio se trabaja el mtDNA, pero a un nivel de resolución mayor (de haplotipos a subhaplotipos) y se incluye los resultados de un nuevo marcador, el MBL (mannose binding lectin) o MBL2, un gen nuclear humano reconocido como polimórfico (10,11).
El gen MBL2 codifica una proteína involucrada en la inmunidad innata por su acción de reconocimiento de patógenos, interviniendo en su opsonización y activando el sistema del complemento. Las mutaciones de este gen son polimorfismos de un único nucleótido (single nucleotide polymophisms, o SNP) y producen haplotipos denominados HYPA, LYPA, LYQA y LXPA, que poseen diferentes niveles de producción de lectinas normales, mientras que LYPB, LYQC, LYPD e HYPD son defectuosos en su producción (10,12). Estos últimos, a su vez, pueden conferir resistencia para otros patógenos (10). La explicación de las frecuencias altas de ciertos haplotipos ha sido atribuida a un proceso de selección natural positiva o purificadora (10,13) o a eventos atribuibles a una neutralidad selectiva o codominancia (11,14). Lo cierto es que en las poblaciones humanas analizadas se ha detectado solo ocho de los 64 haplotipos teóricamente posibles, lo que permite hacer inferencias histórico-poblacionales (10,11).
Esperamos que nuestro trabajo, al analizar inicialmente por separado y luego simultáneamente un marcador del genoma mitocondrial (mtDNA) y otro del genoma nuclear (MBL), permita alcanzar niveles de inferencia evolutivos de mayor consistencia.
MÉTODOS
Con relación a los supuestos básicos sobre las muestras para el tipo de estudio y los tratamientos estadísticos para las inferencias a alcanzar, estos fueron:
-
Las muestras fueron obtenidas de poblaciones y/o localidades que se supone estarían lo más próximo al concepto de nativos de las mismas (por atributos geográficos e histórico-culturales, vg. etno-lingüísticos).
-
Las muestras fueron tomadas al azar dentro de cada población y, adicionalmente, todos los individuos de la muestra fueron adultos en edad reproductiva.
El tamaño de cada muestra fue representativo de su respectiva población (N).
Los tamaños efectivos (Ne) entre poblaciones muestreadas pueden no ser idénticos.
Los marcadores genéticos utilizados en el estudio fueron considerados selectivamente neutros o con valores muy próximos a tal calificación.
La ocurrencia de mutaciones fue ignorada a partir de la toma de las muestras.
Tampoco se aceptó la ocurrencia de flujogénico entre las poblaciones, durante el presente estudio.
No ha sido necesario admitir que la población ancestral esté en equilibrio de Hardy-Weinberg (H-W) o bajo efecto de la deriva genética.
Las poblaciones durante el estudio estuvieron en equilibrio H-W.
Las poblaciones nuevas se originaron de preexistentes por un proceso aleatorio, sin ocurrencia de selección.
Respecto a los procedimientos ético-legales para la obtención de las muestras, todos los individuos estudiados fueron informados sobre los alcances del proyecto y firmaron el consentimiento informado para su participación, en concordancia con la Declaración de Helsinki.
Las técnicas bioquímicas utilizadas para el reconocimiento de los subhaplotipos en el mtDNA, correspondieron a las comunicadas en 1995 (15) y modificadas en 2004 (8), mientras que las determinaciones de los haplotipos del MBL fueron realizadas según el protocolo publicado en 1995 (10).
La muestra biológica obtenida y procesada en el laboratorio nos permitió obtener datos cuantitativos, para lo cual aplicamos un protocolo estadístico que constó de dos etapas y cada una de tres pasos, los mismos que estuvieron orientados a obtener información suficiente para hacer inferencias evolutivas primarias sobre las poblaciones portadoras de los marcadores genéticos estudiados.
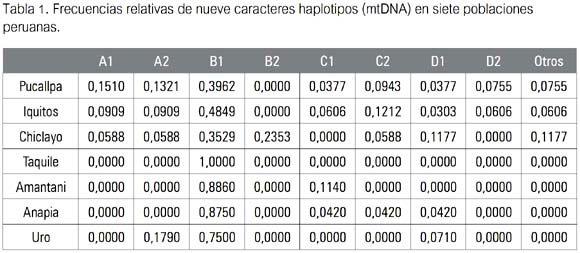
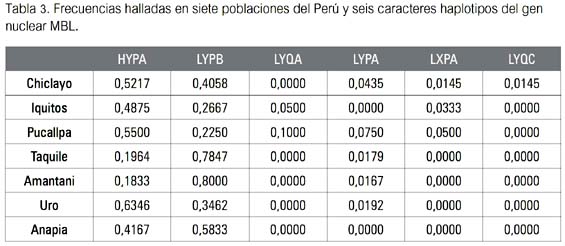
El primer paso de la primera etapa consistió en obtener las frecuencias de los haplotipos de los marcadores genéticos elegidos (mtDNA y MBL) para el presente estudio (ver las tablas 1 y 3, respectivamente). Cada una de estas tablas fue convertida en dos matrices separadas (una por cada marcador) del tipo localidades por haplotipos.
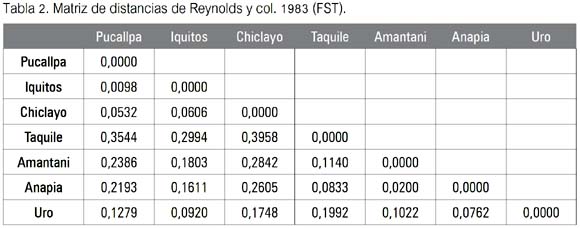
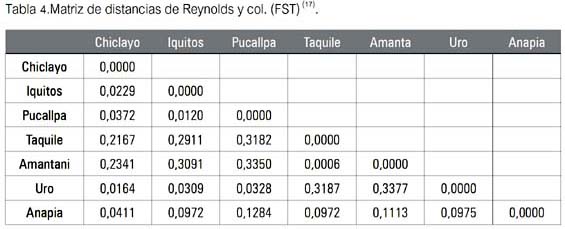
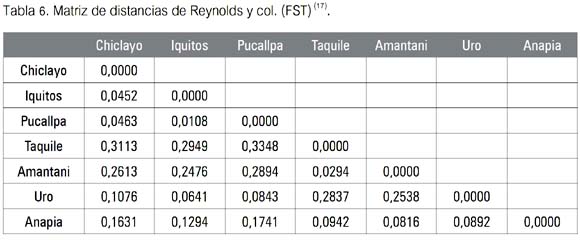
El segundo paso consistió en el procesamiento de la información por el paquete de programas PHYLIP 3.65 (16), específicamente utilizando el denominado GENDIST en la opción distancia, de Reynolds y col. (17), a fin de obtener valores de FST, conocidos también como de divergencia evolutiva, diferenciación genética, coeficiente de coancestría o coeficiente de de coalescencia (18) de cada marcador genético, a fin de apreciar la distribución de cada uno de ellos entre las poblaciones-localidades muestreadas (se observa en las tablas 2 y 4).
Los valores hallados fueron comparados con la tabla de calificación de valores FST, la misma que incluimos a continuación:
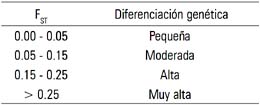
El rango de los valores FST está entre 0 y 1; el valor 0 indica que no existe divergencia genética y el valor 1, que existe fijación para alelos alternos en diferentes subpoblaciones.
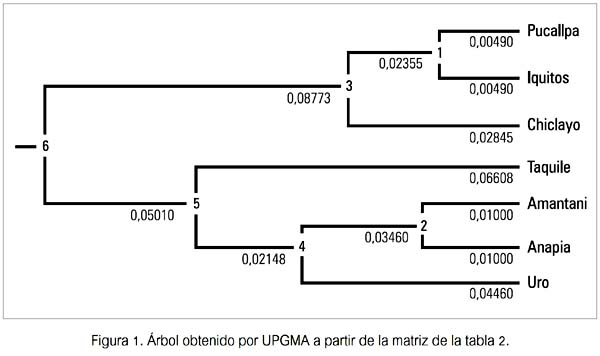
El tercer paso consistió en la aplicación del algorritmo UPGMA (unweighted pair group method with arithmetic mean) (20), del mismo paquete PHYLIP, para expresar gráficamente los valores FST entre pares de poblacioneslocalidades para cada marcador genético, mediante la construcción de un árbol de divergencia o diferenciación genética y hacer el análisis de los agrupamientos o clusters formados (figuras 1 y 2). Este procedimiento permite conocer la magnitud y dirección de la divergencia evolutiva de cada marcador entre las poblaciones estudiadas.
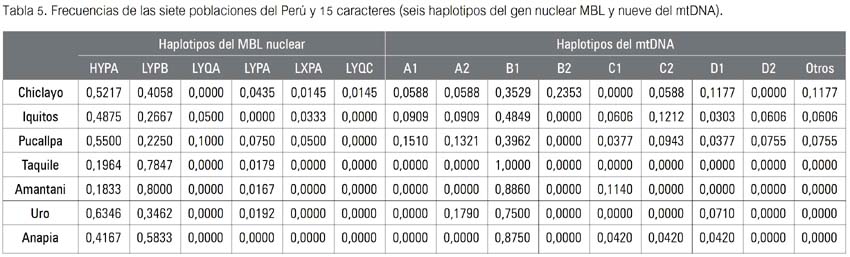
Sabiendo que es altamente improbable que desde matrices distintas se obtenga árboles idénticos y que se requiere información que permita, cuando menos, inferencias evolutivas primarias, se precisó de una segunda etapa, que consistió en realizar los tres pasos antes descritos, pero esta vez trabajando las matrices de frecuencia de manera integrada, que anteriormente habían sido procesadas por separado (tabla 5). Aquí, el producto final fue también un árbol de divergencia genética, pero con otro atributo: el análisis de los agrupamientos formados nos permitirá ahora conocer la contribución de ambos marcadores en el establecimiento de las relaciones de parentesco (distancias evolutivas) entre las poblaciones estudiadas y en las inferencias evolutivas a efectuar respecto a ellas.
RESULTADOS
Los resultados los presentamos en tablas y gráficas, ordenados por marcador genético. Primero los obtenidos del mtDNA y, en segundo lugar, los del gen nuclear MBL (primera etapa); y, finalmente, los de ambos marcadores integrados (segunda etapa), paso por paso.
Sobre el ADn mitocondrial
En la tabla 1 se observa las frecuencias relativas de los haplotipos mitocondriales procedentes de siete localidades-poblaciones ubicadas en la costa, sierra y selva del Perú. Por cada haplotipo del mtDNA, se formó un grupo, siendo detectados ocho. Estos fueron considerados polimorfismos y, por tanto, mutuamente excluyentes. Se les conoció como los haplotipos A1, A2, B1, B2, C1, C2, D1 y D2 que, por otra parte, se sabe son los que ocurren con notable mayor frecuencia en las poblaciones humanas sudamericanas (15). A fin de contabilizar aquellos individuos cuyos haplotipos no fue posible asignar a ninguno de los ocho hasta aquí mencionados, se formó un noveno grupo, que denominamos como Otros.
La tabla 1 también indica que la localidad más diversa para el mtDNA fue Pucallpa, pues su muestra comprendió todos los haplotipos, en tanto que la menos diversa fue Taquile, donde según la muestra el haplotipo B1 parece haberse fijado, pues estuvo presente en todos los individuos (1.00 o 100%).
En la tabla 2 puede observarse que los valores de diferenciación genética más altos fueron hallados en los pobladores de la localidad de Taquile (Puno), los cuales solo exhibieron el haplotipo B1, indicando una diferenciación genética muy alta, por ejemplo, respecto de las poblaciones muestreadas fuera del Departamento de Puno (valores de 0.3544, 0.2994 y 0.3958). Iquitos y Pucallpa fueron el par de poblaciones menos divergentes, al alcanzar entre ellas un valor de FST de 0.0098.
El tercer resultado obtenido se hizo evidente gráficamente y puede ser apreciado en la figura 1. Se observa los valores de divergencia genética hallados para el mtDNA, un árbol de distancias evolutivas donde se aprecia la formación de dos clusters, uno conformado por las poblaciones de Pucallpa, Iquitos y Chiclayo, y el otro por las de Taquile, Amantani, Anapia y Uro (todas del departamento de Puno). Cada cluster diverge del ancestro de ambos con un valor que le es propio. Así, el primero de los nombrados indica un valor de 0.08773 (entre ancestros 6 y 3), en tanto que el segundo lo hace con un valor de 0.05010 (entre ancestros 6 y 5). A ambos valores se les puede calificar según la tabla de calificación de Hart & Clark (19) como de divergencia moderada. El ancestro 6 se halló genética y evolutivamente más próximo al 5 que al 3.
Sobre el gen nuclear mannose
(mannan) binding-lectin (MBL)Los haplotipos hallados para el gen MBL fueron HYPA, LYPB, LYQA, LYPA, LXPA y LYQC y sus frecuencias pueden ser apreciadas en la tabla 3. Estos también son considerados polimorfismos y existen estudios que señalan a los dos primeros como los que ocurren con más frecuencia en las poblaciones humanas sudamericanas (10,21). Las muestras más diversas fueron las poblaciones de Chiclayo y Pucallpa, conteniendo cinco de los seis haplotipos en combinaciones diferentes, en tanto que la menos diversa fue la de Anapia, con solo dos de los seis haplotipos.
La tabla 4 muestra los valores de distancia hallados para los haplotipos del gen MBL. Se les comparó con la tabla de calificación de Hart & Clark (19) expuesta en II.I. Puede observarse entonces que los valores mayores de diferenciación genética fueron observados en los pobladores de la localidad de Amantani (Puno), indicando una alta y muy alta diferenciación genética; por ejemplo, alta respecto de las poblaciones muestreadas fuera del Departamento de Puno (valores de 0.2341, 0.3091 y 0.3350, respectivamente) y muy alta en relación a la de Uro, Puno (0.3377), mientras que Taquile y Amantani resultaron ser el par de poblaciones menos divergentes, al alcanzar entre ellas un valor de FST de 0.0006.
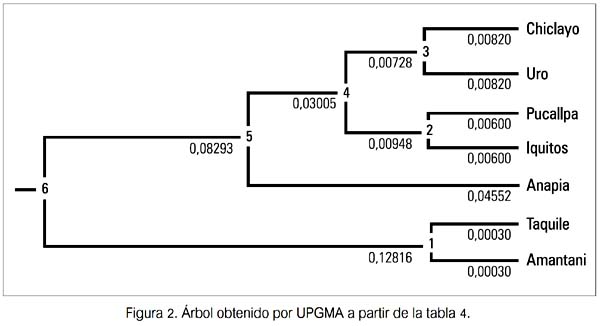
El tercer resultado sobre el gen MBL se hizo evidente cuando se expresó gráficamente (figura 2) los valores de divergencia genética mediante el análisis de agrupamiento o de cluster. Se obtuvo un árbol de distancias evolutivas, donde se aprecia también la formación de dos clusters, uno conformado por las poblaciones de Chiclayo, Uro, Iquitos, Pucallpa y Anapia y el otro por Taquile y Amantani (ambos del Departamento de Puno). Cada cluster diverge del ancestro de ambos con valores que les son propios. Así, para el primer cluster se indica un valor de divergencia de 0.08293 (entre ancestros 6 y 5), en tanto que para el segundo lo hace con el mayor valor (0.12816, entre ancestros 6 y 1). Individualmente, estos valores indican a su vez que se les puede calificar, según la tabla de calificación de Hart & Clark (19), como de presentar una divergencia moderada. En este caso, el ancestro 6 se halló genética y evolutivamente más próximo al 1 que al 5. La distancia entre los ancestros 1 y 5 -que dan origen a los dos principales clusters- fue de 0.21109, valor que es considerado alto.
Hasta este punto, es evidente que se tiene dos árboles o hipótesis diferentes sobre los patrones de diferenciación o distribución de sendos marcadores (los haplotipos del mtDNA y el gen nuclear MBL); o, de otro modo, tenemos la evolución de cada marcador entre las poblaciones estudiadas que han sido procesados por separado. Debido a que tales árboles no son idénticos, se les considera en conflicto para propósitos de las inferencias evolutivas, que es justamente lo que se espera hacer con las poblaciones humanas estudiadas.
Como un siguiente e inmediato paso para superar tal conflicto, se siguió la estrategia para casos donde existen árboles con topologías rivales (antagónicas) a partir de matrices distintas, y consiste en integrar o fusionar las matrices previas, para construir una matriz combinada y alcanzar a construir un árbol que tenga carácter consensual, puesto que no es el consensus de los cladistas (22), pero que fundamentalmente informa sobre la historia evolutiva de las poblaciones humanas peruanas.
Sobre la matriz combinada: gen nuclear MBL + mtDNA
El primer resultado obtenido fue la tabla de frecuencias (tabla 5) de los haplotipos del gen nuclear humano mannose (mannan) binding-lectin (MBL) y los del mtDNA. Proceden de las mismas siete localidades-poblaciones vistas líneas arriba. Las muestras más diversas y las que lo fueron menos obviamente fueron las mismas que las vistas en los puntos II y III.
La tabla 5 -ahora una matriz combinada - produjo los nuevos valores de divergencia hallados. Ellos fueron comparados con la tabla de calificación de Hart & Clark (19), expuesta en II.1. Puede observarse que los mayores valores de diferenciación genética se hallaron en la muestra de Taquile (Puno), indicando que tal diferenciación genética es muy alta respecto de las poblaciones estudiadas tanto fuera del Departamento de Puno (valores de 0.3113, 0.2949 y 0.3348), como a la de los Uro del mismo Puno (0.2837). En tanto que, esta vez Iquitos-Pucallpa fue el par de poblaciones menos divergente, al alcanzar entre ellas un valor de FST de 0.0108, calificándosele tal divergencia como pequeña.
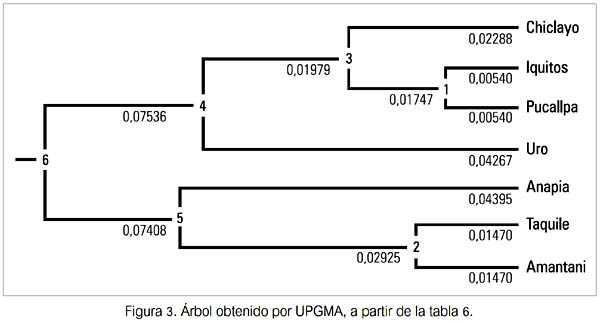
El tercer resultado de esta última etapa, esta vez sobre los marcadores mtDNA (ADNmt) y MBL trabajados de manera integral, se produjo cuando se expresó gráficamente (figura 3) los valores de divergencia genética seguidas de un análisis de agrupamiento o de cluster. En el árbol obtenido, se sigue apreciando la existencia de dos clusters, pero esta vez se tiene uno formado por las poblaciones de Chiclayo, Iquitos, Pucallpa y Uro, y el otro formado por Anapia, Taquile y Amantani (estas tres últimas del Departamento de Puno). Cada cluster diverge del ancestro de ambos con un valor que les es propio. Así, para el primer cluster nombrado, indica un valor de divergencia de 0.07536 (entre ancestros 6 y 4), en tanto que para el segundo lo hace con uno ligeramente menor, de 0.07408 (entre ancestros 6 y 5). Cada uno de tales valores indican a su vez que califican, según la tabla de calificación de Hart & Clark (19), como poseedores de una divergencia genético-evolutiva moderada, que implicaría también un tiempo de coalescencia estimado en moderado. En este caso, el ancestro 6 se halló genética y evolutivamente ligeramente más próximo al 5 que al 4, debiendo añadirse que el valor de la divergencia entre los ancestros 4 y 5 llegó a 0.14944, calificándola prácticamente como alta (límite inferior del rango para este es de 0.15). Es evidente, además, que el árbol aquí obtenido tiene una topología nueva, pues el primer cluster incluye a la población Uro de Puno, agrupándose con las que fueron estudiadas fuera del Departamento de Puno (Chiclayo, Iquitos y Pucallpa).
DISCUSIÓN
Primero nos referiremos a los marcadores genéticos mtDNA y MBL estudiados separadamente.
El patrón de herencia materna y los polimorfismos encontrados lo han convertido en alternativas genéticas selectivamente neutras (19). Su aporte para el establecimiento de relaciones de parentesco y la mayor tasa de mutación lo sustentan como un buen indicador evolutivo de reconocimiento universal. Los resultados encontrados en este trabajo invitan a realizar más estudios para avanzar hacia una hipótesis que permita entender cómo ocurrió la evidente fijación del haplotipo B1 en Taquile y su muy alta frecuencia relativa en Amantani y Anapia. Atribuir al azar tal fijación fue la primera hipótesis que se formuló para su explicación, pero ahora ella no parece suficiente (8). Además del azar, es posible que la fijación de tal haplotipo se deba también a una muy alta fecuencia del mismo en la población fundadora, la que bien pudo tener también un tamaño efectivo reducido (2).
Respecto al gen nuclear MBL, nuestra primera impresión fue que no calificaría para propósitos de suministro de información en estudios evolutivos, pues existen trabajos que indican que este gen estaría sometido a importantes presiones selectivas (10,13,21) y no cumpliría con el requisito de neutralidad razonable indicado en nuestro punto I. Pero, en esas mismas publicaciones dan a conocer que al menos dos de sus haplotipos mutantes defectivos del silvestre o normal HYPA -como son LYPB para Sudamérica y LYQC para África- podrían ser informativos desde el punto de vista demográfico-evolutivo, pues serían codominantes con ventajas y desventajas selectivas opuestas desde el punto de vista inmunológico respecto a la forma silvestre HYPA, que a su vez no sería ventajoso en todos los casos. Se debe reconocer que su estudio, tanto de manera separada como formando parte de la matriz combinada, fue retributivo, pues ayudó a reflexionar acerca de la simple aleatoriedad como explicación a la virtual fijación del haplotipo B1 mitocondrial en Taquile y la de su alta frecuencia en Amantani y Anapia.
Luego, los marcadores mtDNA y MBL fueron estudiados conjuntamente. Después de haber efectuado los tratamientos estadísticos por separado y de manera integral, nos encontramos con tres árboles con distinta topología (figuras 1, 2 y 3). Los dos primeros nos dieron información respecto a cómo se comportaron o evolucionaron los marcadores elegidos en cada población muestreada. Evidentemente, para las inferencias evolutivas entre poblaciones, los resultados obtenidos producto de la matriz combinada (tabla 6) son los más adecuados. Tomados en conjunto, los marcadores pueden darnos ahora alguna información sobre la evolución de las poblaciones estudiadas.
Así, el análisis de agrupamiento de la figura 3 nos remite necesariamente a la tabla 5, a fin de apreciar las frecuencias de cada marcador en cada población y hallar alguna explicación de porqué la población Uro (figura 4), que geográficamente está mucho más próxima a las tres isleñas de Puno -como son Anapia, Amantani y Taquile (ver tabla 7 y figura 5)-, sin embargo, forma un grupo con Chiclayo, Iquitos y Pucallpa. Su observación detenida nos da indicios sobre las probables causas: la población Uro tiene las frecuencias de los marcadores HYPA y LYPB invertidas respecto a las de sus vecinas isleñas de Puno y acusa en consecuencia un grado importante de miscigenación. En los Uro, HYPA casi dobla la frecuencia de LYPB, siendo muy similar a lo que ocurre en las poblaciones estudiadas fuera de Puno (Chiclayo, Iquitos y Pucallpa). En Amantani y Taquile ocurre a la inversa, el haplotipo LYPB es más de cuatro veces más frecuente que el HYPA, en tanto que en Anapia la frecuencia de LYPB es casi 50% mayor que la de HYPA. Por otra parte, el haplotipo mitocondrial B1 se encuentra virtualmente fijado en Taquile (constituye el 100% de la muestra) y alcanzando 88,6% y 87,5% en Amantani y Anapia, respectivamente. Algo distante se encuentra la de los Uro, con 75%, en tanto que las frecuencias para el citado haplotipo mitoncondrial para Chiclayo, Iquitos y Pucallpa se encuentran por debajo del 50% (rango 35,3% a 48,5%).

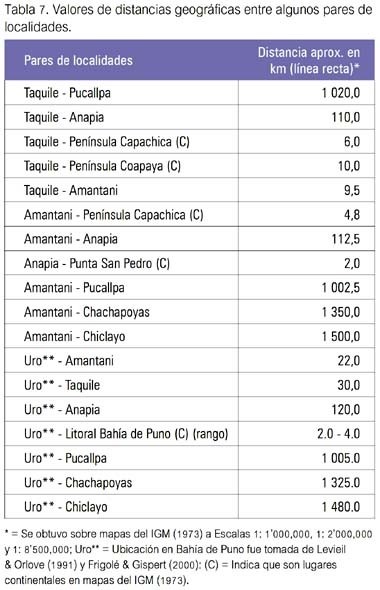
Los valores de divergencia genética calificada como alta, según la tabla de calificación de Hart & Clark (19), entre el primero con el segundo cluster formado por Anapia, Amantani y Taquile, parecería tener una explicación razonable cuando se observa en el mapa del Perú (23) (figura 6). Estas tres poblaciones habitan sendas islas geográficas dentro del Lago Titicaca y además su divergencia genética parece estar en relación directa a las distancias físicas que existen entre ellas. Taquile y Amantani están mucho más próximas entre sí (ambas casi en el extremo norte del Lago), en tanto que la de Anapia se encuentra virtualmente al extremo sur del Lago. También, parecen estar en relación directa a las distancias físicas que existen ente ellas y las orillas continentales del Lago. Taquile es la más distante de estas y Anapia la más próxima. Estos resultados sugieren que, desde hace un tiempo no precisado -que puede ser no muy lejano-, sus poblaciones estarían construyendo una historia natural incompatible con el modelo aislamiento por distancia (26-28) y donde se insinúa un proceso de coevolución en los marcadores estudiados, que permite formularse preguntas tales como ¿Amantani y Taquile parecen estar comportándose como aislados genéticos recientes, o bien son remanentes de poblaciones relícticas? ¿son parte terminal de un fenómeno clinal? ¿es un producto de tradiciones y/o costumbres respecto a la elección de la pareja? Se debe tener presente que las dos poblaciones siempre formaron el segundo cluster en los tres árboles obtenidos (figuras 1, 2 y 3).
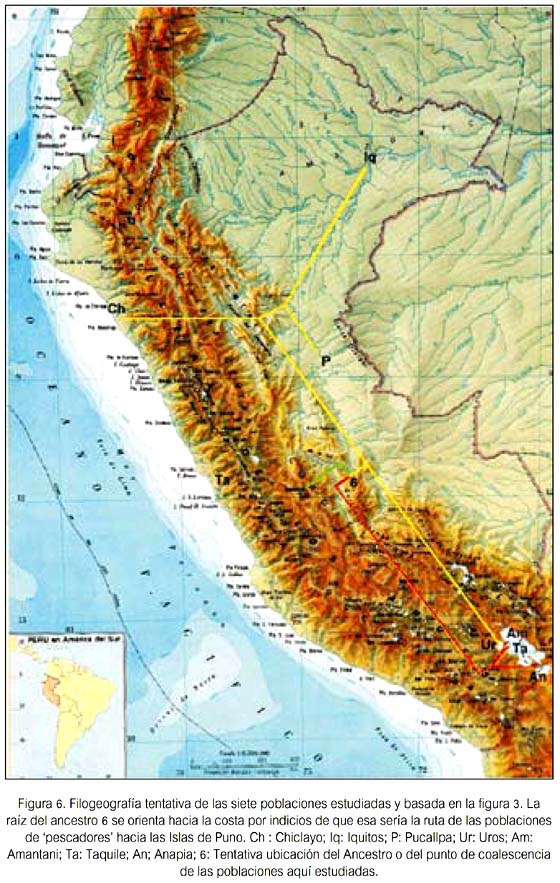
Finalmente, nuestros resultados señalan una virtual inexistencia de los Uro como entidad poblacional con características genéticas propias. El tipo de variantes genéticas que aportaron y la frecuencia en la que se hallaron fueron muy similares a las de Chiclayo, Iquitos y Pucallpa. La muestra de ellos aquí estudiada presenta signos evidentes de miscigenación, considerándose necesario efectuar más evaluaciones en las mismas poblaciones y en el mayor número posible de poblaciones del Perú, con estos y otros marcadores genéticos nucleares, para tener una visión más clara sobre cómo habría sido la secuencia histórica de la ocupación, asentamiento, migración y -porqué no- posibles extinciones locales de poblaciones humanas en el territorio nacional. Es pertinente señalar que la existencia de los Uro actuales, como descendientes de los Uru originales y sin mestizaje, ha sido negada por algunos autores del área etno-lingüista (24,28).
El tema de la divergencia genética significativa apreciada en Taquile y Amantani y el de la miscigenación de los Uro ameritan profundizarse, acompañados quizá de otras disciplinas, tales como la antropología física y la cultural, como la etnología. La de la lingüística parece desfasada de la genética desde su enfoque histórico, por ahora. El lugar físico de residencia de los Uro, al menos geográficamente, no es ubicable en los mapas (¿situación comparable a la de los gitanos?). Quizá sea el carácter artificial y sobre todo relativamente móvil de sus residencias lo que hasta cierto punto explique que no se reconozcan formalmente a las islas flotantes del Titicaca donde se encuentran actualmente los que las construyeron, y ahora son comunmente considerados los últimos descendientes de un linaje que aún no muestra indicios genéticos de su carácter ancestral.
REFERENCIAS BIBLIOGRÁFICAS
1. Goldstein DB, Chikhi L. Human migrations and population structure: what we kown and why it matters. Annu Rev Genomics Hum Genet. 2002;(3):129-52.
2. Córdova JH, Fujita R, Sandoval J, Velásquez M, Távara C, Cotos D, Vásquez J, Barletta C, Descailleaux J. Poblamiento del continente americano y del Perú sugerido de un análisis filogeográfico de haplogrupos del mtDNA en etnias nativas. I: inferencias primarias. Arch Biol Andina. 2008;14(1):23-39.
3. Descailleaux J, Orrillo M. Frecuencias de cromatina sexual en mujeres de diferentes edades.
Rev Ciencias. (UNMSM Perú) 1974; 70 (1): 52-8.4. Orrillo M, Descailleaux J, Pereda J, Hermoza P, Alvarado E. Numerical X-chromosome anomalies at the Maternity Hospital of Lima. J Genet Hum (Suiza). 1976;24(3):221-6.
5. Velásquez M, Descailleaux J. Cromatina sexual en escolares de Lima Metropolitana. Rev Ciencias (UNMSM , Perú). 1986;74(1):59-65.
6. Rodríguez-Delfín LA, Descailleaux J. Polimorfismo cromosómico en algunas poblaciones peruanas: Comparación cualitativa.
Magistri et Doctore. (UNMSM , Perú) 1994;2(9);9-11.7. Rodríguez-Delfin, LA, Rubin de Celis V, Zago MA. Genetic diversity of the Andean population from Peru and the regional migration patterns of Amerindians in South America: data from Y chromosome and mitochondria DNA. Hum Hered, 2001;51:97-106.
8. Sandoval J, Delgado B, Rivas L, Bonilla B, Nugent D, Fujita R. Variantes del DNA mt en isleños del lago Titicaca: máxima frecuencia del haplotipo B1 y evidencia de efecto fundador. Rev per biol. 2004;11(2):161-8.
9. Cann RL, Stoneking M, Wilson AC. Mitochondrial DNA and human evolution. Nature. 1987;325(6099):31-6.
10. Madsen HO, Satz ML, Hogh B, Svejgaard A, Garred P. Different molecular events results in low protein levels of mannan-binding lectin in populations from Southeast Africa and South America. J Inmunol. 1998;161(6):3169-75.
11. Boldt AB, Messias-Reason IJ, Meyer D, Schrago CG, Bertrand FL, Dietz LK, Kremsner PG, Petzl-Erler ML, Kun JFJ. Phylogenetic nomenclature and evolution of mannose-binding lectin (MBL2) haplotypes. BMC Genet. 2010;11:38.
12. Garred P. Mannose-binding lectin genetics: from A to Z. Biochem Soc Trans. 2008;36:1461-6.
13. Mukherjee S, Sarkar-Roya N, Wagener DK, Majumdera PP. Signatures of natural selection are not uniform across genes of innate immune system, but purifying selection is the dominant signature. PNAS. 2009;106(17):7073-8.
14. Bernig T, Taylor JG, Foster CB, Staats B, Yeager M, SJ Chanock SJ. Sequence analysis of the mannosebinding lectin (MBL2) gene reveals a high degree of heterozygosity with evidence of selection. Genes Immun. 2004;5(6):461-76.
15. Merriwheter DA, Rothhammer F, Ferrell RE. Distribution of the four founding lineages haplotypes in Native Americans suggest a single wave of migration for the New World. Am J Phys Anthropol. 2004;98:411-30.
16. Felsenstein J. PHYLIP (Phylogeny Inference Package) version 3.6. Distributed by the author. Department of Genome Sciences, University of Washington, Seattle; 2004. Disponible en: http://evolution.genetics.washington.edu/phylip.html
17. Reynolds J, Weir BS, Cockerham CC. Estimation of the coancestry coefficient basis for a short term genetic distance. Genetics. 1983;105(3):767-79.
18. Meyer D. Árbores Evolutivas Humanas: Uma discussão sobre inferência filogenética. Instituto Biociências. Universidade de São Paulo: 1996.
19. Hart DL, Clark AG. Principles of Population Genetics. 3th ed. Massachusetts: Sinauer Associates; 1997.
20. Michener CD, Sokal RR.
A quantitative approach to a problem in classification. Evolution. 1957;11(2):130-62.21. Turner MW, Heatley DL, Jack S, Boettcher DL, Lester B. McKluskey SJ. Roberton D. Restricted polimorphism of the Mannose-Binding Lectin gene of indigenous Australians. Hum Mol Genet. 2000;9(10):1481-6.
22. Simmons NB. 1990. The importance of Methods. 1. Pp. 3-61. En: MacPhee RDE. Primates in their Relation in Phylogenetic perspectives. New York: Plenum Press. 1990: 343 pp.
23. IGM Mapa Físico Político de la República del Perú. Instituto Geográfico Militar del Perú 1973: 284 pp.
24. Levieil D, Orlove BS. Importancia socio-económica de las macrofitas. VII 1d, En: Dejoux C, Iltis A. editores El Lago Titicaca: síntesis del conocimiento limnológico actual. Institut Français de Recherche Scientifique pour le Développement en Coopération-ORS TOM-HIS BOL (La Paz); 1991:509-16
25. Frigolé J, Gispert C. Razas y pueblos del mundo. Barcelona: Oceano Grupo E ditorial SA . 2000: 456 pp.
26. Wrigh S. Isolation by Distance. Genetics. 1943;38:139-56.
27. Kimura
M, Weiss GH. The stepping stone model of population structure and the decrease of genetic correlation with distance. Genetics. 1964;49:561-76.28. Slatkin M. Isolation by distance in equilibrium and non-equilibrium populations. Evolution. 1993;47(1):264-79.
29. Bouysse-Castagne T. Poblaciones humanas antiguas y actuales. VII 1a. En: Dejoux C, Iltis A. El Lago Titicaca: síntesis del conocimiento limnológico actual. Institut Français de Recherche Scientifique pour le Développement en Coopération. La Paz: ORS TOM – HIS BOL. 1991:481-98.
Trabajo recibido el 19 de noviembre de 2010 y aceptado para publicación el 10 de febrero de 2011.
Agradecimientos
Los autores desean expresar su profundo agradecimiento a todos los participantes en el presente estudio, que con su aporte voluntario hicieron posible la realización del trabajo.
Financiamiento
El estudio ha ido financiado parcialmente con los recursos otorgados por el Vicerrectorado de Investigación de la Universidad Nacional Mayor de San Marcos a los proyectos 101001021, 091001101 y 081001021 y el Instituto de Genética y Biología Molecular de la Facultad de Medicina de la Universidad San Martín de Porras.














