Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Anales de la Facultad de Medicina
versión impresa ISSN 1025-5583
An. Fac. med. v.73 n.3 Lima jul./set. 2012
SECCIÓN BIOQUÍMICA Y NUTRICIÓN
Variabilidad genética de la respuesta inflamatoria. I. Polimorfismo -511 C/T en el gen IL1β en diferentes subpoblaciones peruanas
Genetic variability of the inflammatory response. I. The -511 C/T IL1β polymorphism in different Peruvian subpopulations
Óscar Acosta1,2,3, Luis Solano2, Doris Huerta1, Daniel Oré3, José Sandoval3,4, José Figueroa3, Ricardo Fujita3
1 Centro de Investigación de Bioquímica y Nutrición, Facultad de Medicina, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Instituto de Medicina Tropical Daniel A. Carrión, Facultad de Medicina, Facultad de Medicina, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 Centro de Genética y Biología Molecular, Facultad de Medicina Humana, Universidad San Martín de Porres, Lima, Perú
4 Universidade Federal de Minas Gerais, Belo Horizonte, Brasil
Resumen
El polimorfismo -511 citosina/timina (-511 C/T) en la región promotora del gen interleuquina 1 beta (IL1β) está implicado en la producción diferencial de la citoquina y por tanto puede estar asociado a la respuesta inmuno-inflamatoria en obesidad, dislipidemias, cardiopatías, cáncer, infecciones, y el tratamiento con nutrientes y fármacos. Objetivos: Establecer la distribución de frecuencias de los genotipos y alelos del polimorfismo -511 C/T del gen IL1β en diferentes subpoblaciones peruanas. Diseño: Estudio descriptivo, observacional, transversal. Instituciones: Centro de Investigación de Bioquímica y Nutrición e Instituto de Medicina Tropical D.A. Carrión, Facultad de Medicina, UNMSM y Centro de Genética y Biología Molecular, Facultad de Medicina, USMP, Lima, Perú. Participantes: Pobladores peruanos. Intervenciones: Extracción de ADN genómico a partir de muestras sanguíneas o epitelio bucal según metodología estándar, de 168 individuos de 9 grupos subpoblacionales: 23 mestizos de Lima, 33 amazónicos (20 de Pucallpa y 13 de Amazonas) y 112 andinos (12 de Ancash, 10 de Cajamarca, 18 de Huarochirí-Lima, 25 de Puno-Taquile, 25 de Puno-Uros y 22 de Puno-Anapia). Análisis del polimorfismo -511 C/T mediante la técnica de PCR/RFLP, con primers específicos y digestión con la enzima de restricción AvaI, detectándose los fragmentos por electroforesis en geles de agarosa al 2% y tinción con bromuro de etidio. Principales medidas de resultados: Frecuencias genotípicas y alélicas del gen IL1β. Resultados: Se encontró las siguientes frecuencias genotípicas CC=0,024; CT=0,369 y TT=0,607, consistentes con el equilibrio de Hardy-Weinberg; y las frecuencias alélicas fueron alelo C=0,208 y aleloT= 0,792. La frecuencia del alelo T, considerado el mutante, fue muy alta en los Uros de Puno (0.940) y más baja en los mestizos de Lima (0.609). La comparación de las frecuencias genotípicas (TT versus CT+CC) y alélicas (T versus C) mostraron diferencias significativas (p<0,01) entre los pares Lima mestizos y las de Puno-Taquile y Puno-Uros. Existieron diferencias significativas (p<0,001) cuando se las comparó con otras poblaciones del mundo. Conclusiones: El alelo T mutante del polimorfismo -511 C/T en el gen IL1β, asociado a mayor producción de la citoquina, fue frecuente en las subpoblaciones estudiadas. Debido a las diferencias encontradas, es importante considerar la etnicidad en los estudios de asociación y de riesgo de este gen, como factor de respuesta inflamatoria, en obesidad, dislipidemias, cardiopatías, cáncer, infecciones, y el tratamiento con nutrientes y fármacos.
Palabras clave: Interleuquina 1 beta, gen, alelo, polimorfismo.
Abstract
The -511 citosyne/thymine (-511 C/T) polymorphism in the promoter region of human interleukin 1B (IL1β) gene is associated to differential cytokine synthesis and immuno-inflammatory response in obesity, dyslipidemias, cardiopathies, cancer, infections, other diseases, nutritional intervention and drug treatment. Objectives: To determine allelic and genotypic frequencies of -511 C/T polymorphism in the IL1β gene polymorphism in different Peruvian subpopulations. Design: Descriptive, cross-sectional study. Settings: Faculties of Human Medicine, Universidad Nacional Mayor de San Marcos and Universidad San Martin de Porres, Lima, Peru. Participants: Peruvian subjects. Interventions: Genomic DNA was extracted from 168 individuals from Lima (23 mestizos), 33 Amazonians (20 Pucallpa and 13 Amazonas) and 112 Andeans (12 Ancash, 10 Cajamarca, 18 Huarochiri-Lima, 25 Puno-Taquile, 25 Puno-Uros and 22 Puno-Anapia) according to standard methodology and amplification using PCR-RFLP technique. PCR products were digested with AvaI restriction enzyme and fragments were separated by 2% agarose gel electrophoresis and ethidium bromide stain. Main outcomes measures: Allelic and genotypic frequencies of the -511 C/T polymorphism in the IL1β gene. Results: CC=0,024, CT=0,369, and TT=0,607 genotypes frequencies were found, consistent with Hardy-Weinberg equilibrium, as well as alleles frequencies C = 0,208, and T= 0,792. The frequency of (mutant) T allele was very high in Puno-Uros (0,940) and low in Lima-mestizos (0,609). Comparison of genotypes (TT versus CT+CC) and alleles (T versus C) showed significant differences (p <0.01) between pairs Lima-mestizos and Puno-Uros and Puno-Taquile. Differences were significant (p <0.001) when compared to other world populations. Conclusions: T allele of IL1β gene -511 polymorphism related to increased production of the cytokine was frequent in these Peruvian subpopulations. Because of these differences, it is important to consider ethnicity in association studies and risk of this gene, factor of inflammatory response in obesity, dyslipidemias, cardiopathies, cancer, infections, other diseases, nutritional intervention and drug treatment.
Key words: Interleukin 1 beta, gene, allele, polymorphism.
Introducción
Las citoquinas son moléculas de señalización que contribuyen a la respuesta inflamatoria y componentes claves en la patogénesis de muchas enfermedades, como el cáncer, desórdenes metabólicos y condiciones inmuno-inflamatorias. El grupo de citoquinas de la interleuquina incluye tres citoquinas diferentes: interleuquina 1 beta, interleuquina 1 alfa y el antagonista del receptor de IL1 (IL-1Ra) (1).
La interleuquina 1 beta (IL1β) es una citoquina proinflamatoria potente cuya participación es relevante en inmunoregulación, inflamación y génesis de diferentes patologías y en la respuesta a los nutrientes y el tratamiento con fármacos. En ese panorama, se puede considerar que las variantes de IL1β pueden tener efectos directos en la respuesta inflamatoria y procesos metabólicos, afectando los niveles orgánico y sistémico (2).
El gen que codifica a IL1β está localizado en el brazo largo del cromosoma 2 (2q12), donde se ha encontrado sitios polimórficos tipo SNPs (polimorfismos de una sola base nucleotídica), en las regiones codificantes (exones) y no codificantes (intrones), así como en la región promotora del gen (3).
Una de las mutaciones más estudiadas en la región promotora es la correspondiente a un SNP del tipo citosina/timina en la posición -511 (-C511T) Eso significa que la citosina (C) es reemplazada por una timina (T), dando lugar a dos alelos denominados alelo C (o alelo complementario G, normal) y alelo T (o alelo complementario A, mutante) (4,5).
Estudios in vitro indican que células con al menos un alelo T presentan sobreexpresión (mayor actividad transcripcional), o sea mayores niveles de esta citoquina proinflamatoria, con respecto a las que tienen el alelo C (6). Además, diversos estudios sugieren que el alelo T está relacionado a la producción de altos niveles séricos de IL1β y con ello una respuesta inflamatoria diferencial en diferentes enfermedades y en el tratamiento con nutrientes y fármacos (1).
Numerosas investigaciones han intentado establecer asociaciones entre la respuesta inflamatoria en enfermedades y la presencia del polimorfismo -511 C/T de la IL1β, principalmente estudios casos y controles, asumiendo el alelo T como factor de riesgo, aunque los resultados han sido variables en diferentes grupos poblacionales del mundo, lo que implica que la etnicidad en un factor por considerar para establecer factores de riesgo genético (4). En el Perú, se ha publicado estudios sobre el polimorfismo -511 en el promotor de la IL1β y la susceptibilidad al cáncer gástrico en infectados con Helicobacter pylori, encontrándose asociación con el alelo C; sin embargo, no se menciona el origen étnico (7).
La distribución de la frecuencia de los polimorfismos de ADN, entre ellos los alelos ligados a inmuno-inflamación, pueden variar según el componente étnico de cada subpoblación humana, lo cual explica en parte la predisposición a enfermedades y/o respuesta a los nutrientes o al tratamiento con fármacos de algunas poblaciones (8).
En el contexto planteado, es de importancia conocer cuál es la distribución de este polimorfismo -511 C/T en la región promotora de la IL1β en diferentes subpoblaciones peruanas, caracterizada por su diversidad, por lo que se ha considerado determinar las frecuencias genotípicas y alélicas en diferentes grupos poblacionales de nuestro país. Este estudio representa una etapa inicial para caracterizar los perfiles y variabilidad de la respuesta inflamatoria e inmunológica a nivel genético molecular en la población peruana, es decir, establecer las bases poblacionales y, posteriormente, evaluar las asociaciones de este polimorfismo con diferentes patologías y respuesta a nutrientes y fármacos.
Métodos
Los participantes fueron 168 personas saludables, sin enfermedades crónicas, voluntarios, sin relación de parentesco, hombres y mujeres cuyas edades fluctuaban entre los 18 y 80 años. Estas personas representaron a 9 grupos subpoblacionales: 23 mestizos de Lima, 33 amazónicos (20 de Pucallpa y 13 de Amazonas) y 112 andinos (12 de Ancash, 10 de Cajamarca, 18 de Huarochirí-Lima, 25 de Puno-Taquile, 25 de Puno-Uros y 22 de Puno-Anapia).
Se consideraba la etnicidad de los participantes por autodeclaración y teniendo como mínimo tres generaciones habitando los respectivos lugares. Todos los participantes firmaron el consentimiento informado aprobado por el Comité de Ética de la Facultad de Medicina de la USMP.
El ADN genómico fue extraído de muestras sanguíneas o epitelio bucal, y procesado según técnicas estándar (9), con algunas modificaciones: las células fueron lisadas y tratadas con proteinasa K a 37 ºC, por 24 horas. Se determinó los diferentes genotipos del polimorfismo -511 C/T del gen IL1β mediante la técnica PCR-RFLP. Se amplificó una secuencia de 304 pb, empleando cebadores (primers) específicos y condiciones de amplificación de reportes previos (3,10). Los productos amplificados fueron digeridos con la enzima de restricción AvaI por 12 horas, sujetos a electroforesis en geles de agarosa al 2% y luego teñidos con bromuro de etidio, generándose bandas características para cada genotipo: CC (190/114 pb), TT (304 pb) y CT (304/190/114 pb) (figura 1).
Las frecuencias genotípicas y alélicas fueron obtenidas por conteo directo. Se evaluó las frecuencias genotípicas observadas según lo esperado bajo la hipótesis del equilibrio de Hardy-Weinberg. Para comparar y establecer diferencias o similitudes de las frecuencias genotípicas y alélicas de la IL1β -511 C/T entre los grupos subpoblacionales y entre la población peruana tomada como conjunto (n=168), con las encontradas para otras poblaciones del mundo, se utilizó la prueba X2 o la prueba exacta de Fisher, según el caso, con un α = 0,05. Para los cálculos respectivos, se utilizó el paquete estadístico SPSS 14.0 y el programa de genética poblacional Arlequín 3.11.
Resultados
La distribución de las frecuencias genotípicas y alélicas de la IL1β -511 en las muestras de los 9 grupos subpoblacionales peruanos, se muestra en la tabla 1. Las frecuencias genotípicas observadas en los tres grupos siguen una distribución consistente con el equilibrio de Hardy-Weinberg (p > 0,05, no significativa). Esto indica que las frecuencias observadas no mostraron diferencias significativas con las frecuencias esperadas, lo que significa que no estaban bajo la influencia de factor(es) que las modificaran.
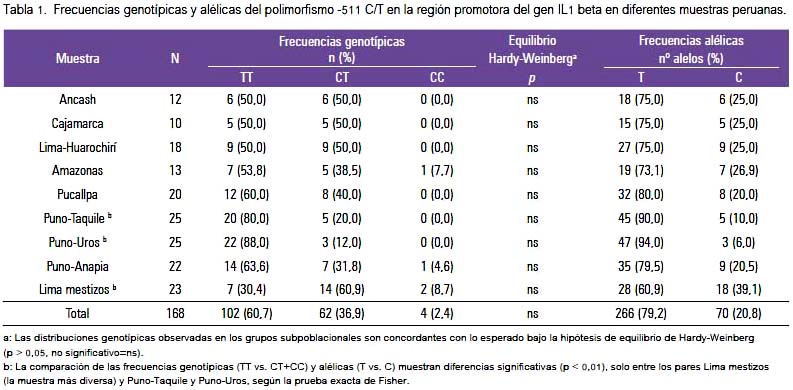
El alelo T (o el complementario A) fue el más común en el conjunto de los grupos poblacionales (n=168) estudiados (79,2%), siendo muy frecuente en Puno-Uros (94%) y Puno-Taquile (90%), mientras que el alelo C (o el complementario G), en general, tuvo una frecuencia menor (20,8%). El grupo de Lima mestizos es el que tuvo menor frecuencia del alelo T (60,9%). Los genotipos TT y CT fueron los más frecuentes en los 9 grupos (97,6% en conjunto), y el genotipo CC solo fue encontrado, en frecuencia baja, en las muestras de Amazonas, Puno-Anapia y Lima mestizos.
La comparación de las frecuencias genotípicas y alélicas, utilizando las pruebas X2 o la prueba exacta de Fisher, mostró diferencias significativas (p < 0,05) solo cuando se comparó la muestra de Lima mestizos (la más diversa) con las de Puno-Taquile y Puno-Uros, y no fue significativo (p > 0,05) cuando se comparó con los demás grupos.
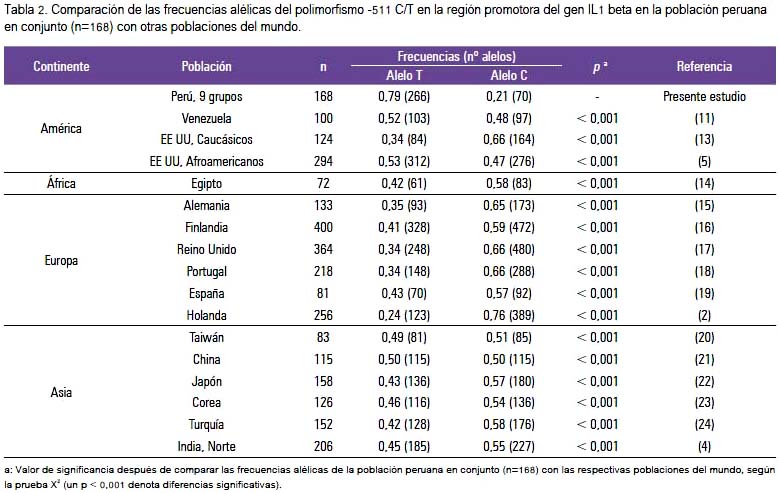
En la tabla 2 se compara las frecuencias alélicas de la IL1β -511 C/T en la población peruana (9 grupos, n=168) con las encontradas para otras poblaciones del mundo. El análisis de X2 o la prueba exacta de Fisher mostró diferencias significativas (p < 0,001) con todas las poblaciones de América, África, Europa y Asia.
Discusión
Esta investigación comunica las frecuencias genotípicas y alélicas de la IL1β -511 C/T en diferentes grupos subpoblacionales del Perú, considerando el origen étnico. Aceptando que hay una correlación entre genotipo, alelos y respuesta inflamatoria en diversas enfermedades y respuesta a nutrientes y fármacos (1), ello será importante si se establece una línea de base de estudios acerca del impacto de los diferentes genotipos y alelos en las subpoblaciones peruanas.
El genotipo TT, asociado con un mayor riesgo para distintas enfermedades según lo publicado para otras poblaciones del mundo (1,4,11), estuvo presente en frecuencia mayor de 60% en las muestras evaluadas, y puede ser considerada como una de las más altas con respecto a otras poblaciones del mundo (4,5,11). El genotipo heterocigoto CT fue detectado en todas las muestras, globalmente con un 39,6%, lo cual es ligeramente menor con lo observado en otras poblaciones de Europa, Asia y América, y por consiguiente el genotipo TT en esta muestra peruana es uno de los más bajos con respecto a estas poblaciones (4,5,11).
La frecuencia del alelo T, considerado el mutante, fue muy alta en los aymaras Uros (94%) y quechuas Taquile (90%) de Puno, y más baja en los mestizos de Lima (61%). La comparación de frecuencias genotípicas (TT versus CT+CC) y alélicas (T versus C) mostraron diferencias significativas (p<0,01) entre Lima mestizos y las de Puno-Taquile y Puno-Uros.
Las frecuencias del alelo T (79%) en la muestra de peruanos, fueron las más altas comparadas con poblaciones asiáticas (42-50%) y caucásicas (24-42%), donde el alelo T es considerado de riesgo para diversas enfermedades (1,12). Un estudio en nuestro país comunica que el C, y no el T, es un alelo de alto riesgo para cáncer gástrico en infectados con Helicobacter pylori (7).
Es importante considerar las diferencias encontradas entre las frecuencias genotípicas y alélicas de la muestra de Lima mestizos (con mayor componente caucásico) con las de otras subpoblaciones de nuestro país, principalmente con las de Puno-Taquile y Puno-Uros, con las frecuencias más altas para el alelo T para la IL1β -511. Ello podría tener implicancias en los estudios de asociación genética con enfermedades, específicamente los de tipo casos-controles y de cohortes, por ejemplo, al comparar subpoblaciones de distinta procedencia étnica (subestructura poblacional) y/o mezcladas (mestizas), y llegar a inferencias erróneas.
Específicamente, las frecuencias del alelo T de la población peruana comparadas con las de América, Asia, África y Europa, fueron las más altas y mostraron diferencias altamente significativas (p<0,00; ver tabla 2). Es posible que la alta frecuencia del alelo T en las poblaciones peruanas sea solo el reflejo de su distribución en toda la población mundial; sin embargo, es posible que se encuentren implicados otros mecanismos.
La frecuencia del alelo C, 21% en la población peruana, es una de las más bajas respecto a las encontradas en otras regiones del mundo (ver tabla 2). Así, el alelo C, que no es considerado como un factor de riesgo para diversas enfermedades en el mundo (1,8,11), presentó una frecuencia relativamente baja en población peruana.
A pesar que este estudio genético poblacional nos da un indicador del polimorfismo de la IL1β -511 C/T en poblaciones peruanas, se debe considerar como preliminar, pues es necesario incluir más poblaciones mestizas y nativas. Sin embargo, eso no lo excluye para futuros estudios tipo casos controles y de cohortes que consideran el origen étnico. Por otro lado, en las poblaciones peruanas podrían haber otras variantes desconocidas en el gen IL1β asociadas la respuesta inflamatoria en obesidad, dislipidemias, cardiopatías, cáncer, infecciones, y el tratamiento con nutrientes y fármacos, por lo que es necesario investigar mutaciones nuevas o propias en nuestra población.
Los genotipos TT y CT, y el alelo T, para el polimorfismo IL1β -511 C/T fueron los más frecuentes en todos los grupos poblacionales evaluados, con muy baja frecuencia del genotipo CC y el alelo C. Las frecuencias genotípicas y alélicas tomadas en conjunto como población peruana (n=168), muestran diferencias significativas con otras poblaciones del mundo, destacándose que el alelo T tiene una de las frecuencias más altas y el alelo C una de las más bajas respecto a otras regiones.
Este estudio sienta las bases poblacionales de la distribución del polimorfismo -511 C/T de la región promotora del gen de la IL1β en población peruana. Una implicancia práctica de la frecuencia baja del alelo C en nuestra población sería su utilidad para evaluar si una mayor frecuencia en pacientes podría estar asociada con una respuesta inflamatoria diferencial en diversas enfermedades y el tratamiento con nutrientes y fármacos. También, se sugiere identificar nuevas variantes en el gen de la IL1β, y otros genes, que podrían estar relacionadas a estas patologías o condiciones, y generar más información que permitirá aportar a la comprensión de los mecanismos de respuesta inflamatoria en nuestra población.
Agradecimientos
Al Consejo Superior de Investigaciones, Centro de Investigación de Bioquímica y Nutrición, y al Instituto de Medicina Tropical de la UNMSM, y al Centro de Genética y Biología Molecular de la facultad de Medicina Humana de la USMP por la financiación y apoyo para la realización del proyecto.
Referencias bibliográficas
1. Kornman KS. Interleukin 1 genetics, inflammatory mechanisms, and nutrigenetic opportunities to modulate diseases of aging. Am J Clin Nutr. 2006;83(suppl):475S-83S.
2. Stokkers PC, Van Aken Be, Basoski N, Reitsma PH, Tytgat GN, Van Deventer SJ. Five genetic markers in the interleukin 1 family in relation to inflammatory bowel disease. Gut. 1998;43:33-9.
3. Katila H, Hänninen K, Hurme M. Polymorphisms of the interleukin-1 gene in schizophrenia. Mol Psychiatry. 1999;4:179–81.
4. Manchanda PK, Bid HK, Mittal RD. Ethnicity greatly influences the interleukin-1 gene cluster (IL-1b promoter, exon-5 and IL-1Ra) polymorphisms: a pilot study of a north Indian population. Asian Pac J Cancer Prev. 2005;6(4):541-6.
5. Zabaleta J, Schneider BG, Ryckman K, Hooper PF, Camargo MC, Piazuelo MB. Ethnic differences in cytokine gene polymorphisms: potential implications for cancer development. Cancer Immunol Immunother. 2008;57:107-14.
6. Chen H, Wilkins L, Aziz N, Cannings C, Wyllie D, Bingle C, et al. Single nucleotide polymorphisms in the human interleukin-1B gene affect transcription according to haplotype context. Hum Mol Genet. 2006;15(4):519-29.
7. Gehmert S, Velapatiño B, Herrera P, Balqui J, Santivañez L, Cok J, Vargas G, Combe, Sijin J, Meyer F, Berg D, Gilman R. Interleukin-1 beta single-nucleotide polymorphisms C allele is associated with elevated risk of gastric cancer in Helicobacter pylori -infected Peruvians. Am J Trop Med Hyg. 2009;81(5):804-11.
8. González F, Christmas S, Middleton D, Jones A. Allele frequency net: a database and online repository for immune gene frequencies in worldwide populations. Nuc Acid Res. 2011;39:D913–D919.
9. Miller S, Dykes D, Polesky F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucl Acid Res. 1988;16(3):1915.
10. Papiol S, Rosa A, Gutierrez E, Martin B, Salgado P, Catalan R, Arias B, Fañanas L. Interleukin-1 cluster is associated with genetic risk for schizophrenia and bipolar disorder. J Med Genet. 2004;41:219-23.
11. Morán Y, Cañas M, Grimán P, Camargo M, Belén M, Chiurillo MA. Distribución de polimorfismos genéticos de Interleuquina-1 en individuos de la región centroccidental de Venezuela. Acta Biol Colomb. 2009;14(1):185-94.
12. Liou J, Lin J, Wang H, Huang S, Lee Y, Chiu H, Shun C, Wu M. IL1B -511 C/T polymorphism is associated with increased host susceptibility to Helicobacter pylori Infection in Chinese. Helicobacter. 2007;12:142–9.
13. Cantagrel A, Navaux F, Loubet-Lescoulie P, Nourhashemi F, Enault G, Abbal M. Interleukin-1 beta, interleukin-1 receptor antagonist, interleukin-4 and interleukin-10 gene polymorphisms: relationship to occurrence and severity of rheumatoid arthritis. Arthritis Rheum. 1999;42:1093-100.
14. Hegab AE, Sakamoto T, Saitoh W, Nomura A, Ishii Y, Morishima Y. Polymorphisms of TNF alpha, IL1beta, and IL1RN genes in chronic obstructive pulmonary disease. Biochem Biophys Res Commun. 2005;329:1246-52.
15. Heils A, Haug K, Kunz WS, Fernandez G, Horvath S, Rebstock J, Propping P, Elger CE. Interleukin-1beta gene polymorphism and susceptibility to temporal lobe epilepsy with hippocampal sclerosis. Ann Neurol. 2000;48:948–50.
16. Virta M, Hurme M, Helminen M. Increased frequency of interleukin-1beta (–511) allele 2 in febrile seizures. Pediatr Neurol. 2002;26:192–5.
17. Cavalleri GL, Lynch JM, Depondt C, Burley MW, et al. Failure to replicate previously reported genetic associations with sporadic temporal lobe epilepsy: where to from here?. Brain. 2005;128:1832–40.
18. Machado JC, Pharoah P, Sousa S, Carvalho R, Oliveira C, Figueiredo C. Interleukin 1B and interleukin 1RN polymorphisms are associated with increased risk of gastric carcinoma. Gastroenterol. 2001;121:823-9.
19. Pastor IJ, Laso FJ, Romero A, González-Sarmiento R. Interleukin-1 gene cluster polymorphisms and alcoholism in Spanish men. Alcohol. 2005;40:181-6.
20. Chou H, Shi Y, Hsu Y, Tsai F. Lack of Association of genetic polymorphisms in the interleukin-1β, interleukin-1 receptor antagonist, interleukin-4 and interleukin-10 genes with mitral valve prolapse in Taiwan Chinese. J Heart Valve Dis. 2003;12:38-44.
21. Jin L, Jia Y, Zhang B, Xu Q, Fan Y, Wu L, Shen Y. Association analysis of a polymorphism of interleukin 1β (IL-1β) gene with temporal lobe epilepsy in a Chinese population. Epilepsia. 2003;44(10):1306–9.
22. Kira R, Torisu H, Takemoto M, Nomura A, Sakai Y, Sanefuji M, Sakamoto K, Matsumoto S, Gondo K, Hara T. Genetic susceptibility to simple febrile seizures: interleukin-1beta promoter polymorphisms are associated with sporadic cases. Neurosci Lett. 2005;384(3):239-44.
23. Lee SH, Ihm CG, Sohn SD, Lee TW, Kim MJ, Koh G. Polymorphisms in interleukin-1 beta and interleukin-1 receptor antagonist genes are associated with kidney failure in Korean patients with type 2 diabetes mellitus. Am J Nephrol. 2004;24:410-4.
24. Haspolat S, Baysal Y, Duman O, Coskun M, Tosun O, Yegin O. Interleukin-1alpha, interleukin-1beta, and interleukin-1Ra polymorphisms in febrile seizures. J Child Neurol. 2005;20(7):565-8.
Correspondencia:
Biólogo Oscar Acosta Conchucos
Centro de Investigación de Bioquímica y Nutrición
Facultad de Medicina - UNMSM
Av. Grau 755. Lima 1, Perú
Correo-e: oacostac@yahoo.com













