Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Anales de la Facultad de Medicina
versión impresa ISSN 1025-5583
An. Fac. med. v.73 n.4 Lima oct./dic. 2012
ARTÍCULOS ORIGINALES
Variabilidad genética y recurrencia de Plasmodium vivax durante la malaria asintomática en Mazán, Iquitos, Perú
Genetic variability of Plasmodium vivax and patterns of recurrence in asymptomatic malaria at Mazan, Iquitos, Peru
Edward Valencia Ayala1,a, Maritza Calderón Sánchez1,b, Manuel Martín Fasabi Espinar1,c
1 Laboratorio de Investigación en enfermedades infecciosas LID, Universidad Peruana Cayetano Heredia.
a Biólogo, Maestro en Biología Molecular.
b Biólogo, Doctor en Ciencias Biológicas.
c Biólogo, Maestro en Bioquímica y Biología Molecular.
Resumen
El Plasmodium vivax muestra una alta variabilidad genética durante episodios recurrentes de la enfermedad. Objetivos: Determinar la variabilidad genética de P. vivax y los patrones de recurrencia durante la malaria asintomática. Diseño: Estudio descriptivo analítico. Institución: Laboratorio de Investigación en Enfermedades Infecciosas-Universidad Peruana Cayetano Heredia. Población: Individuos provenientes de Mazán-Iquitos, región endémica en malaria. Intervención: Se analizó 222 individuos con dos muestras de sangre secuenciales, entre junio de 2006 y noviembre de 2008. Principales medidas de resultados: Identificación de P. vivax, genotipificación en base al gen pvmsp3-α, variabilidad genética de P. vivax y patrones de recurrencia. Resultados: Primera evaluación: Positivos a P. vivax 191/222 (86%), a P. falciparum 2/222 (0,9%), con infección mixta 21/222 (9,5%) y negativos 8/222 (3,6%). Segunda evaluación: Permanecieron positivos a P. vivax 180/191 (94,2%). La genotipificación por nested PCR y digestión enzimática mostró haplotipos policlonales en 17/180 (9,4%) y monoclonales en 163/180 (90,6%). Se observó haplotipos diferentes (reinfección) en 88/180 (48,9%) y haplotipos idénticos (relapso) en 75/180 (41,7%). Conclusiones: Existió una alta variabilidad genética de P. vivax, y los patrones de recurrencia, basados en la genotipificación, indicaron diferencias entre reinfecciones y relapsos en individuos asintomáticos para malaria en Mazán, Iquitos.
Palabras clave: Malaria/vivax, plasmodium vivax, genotipo.
Abstract
Plasmodium vivax displays a high genetic variability for recurrent episodes of illness. Objectives: To determine the genetic variability of P. vivax and patterns of recurrence in asymptomatic malaria. Design: Descriptive analytical. Setting: Laboratory of Infectious Disease Research, Universidad Peruana Cayetano Heredia. Population: Individuals from Mazan, Iquitos, Peru, a malaria endemic region. Intervention: Between June 2006 and November 2008, 222 individuals were analyzed with two sequential blood samples. Main outcome measures: Identification of P. vivax, genotyping based on gene pvmsp3-α, genetic variability of P. vivax and patterns of recurrence. Results: First evaluation: Positive for P. vivax 191/222 (86%), P. falciparum 2/222 (0.9%), mixed infection 21/222 (9.5%) and negative 8/222 (3.6%). Second evaluation: 180/191 (94.2%) remained positive for P. vivax. Genotyping by nested PCR and enzymatic digestion showed polyclonal haplotypes in 17/180 (9.4%) and monoclonal in 163/180 (90.6%). Different haplotypes (reinfection) were observed in 88/180 (48.9%) and identical haplotypes (relapse) in 75/180 (41.7%). Conclusions: There was P. vivax high genetic variability and patterns of malaria recurrence based on genotyping showed differences between reinfection and relapse in asymptomatic individuals in Mazan, Iquitos.
Key words: Malaria/vivax, plasmodium vivax, genotype.
INTRODUCCIÓN
La malaria es un principal problema en salud pública y se ha convertido en una enfermedad reemergente en los lugares donde antes había sido controlada. La malaria ocasiona, en el mundo, alrededor de un millón de muertes cada año y más de tres billones de personas están en riesgo de infección, siendo considerada la enfermedad tropical más importante, según la Organización Mundial de la Salud (1). Entre las cuatro especies causantes de malaria, Plasmodium falciparum (P. falciparum) y Plasmodium vivax (P. vivax) tienen la mayor incidencia, considerando a P. falciparum como la más peligrosa, por su letalidad, por la dispersión mundial de sus estirpes resistentes a las drogas antimaláricas y por su predominio en África, el continente con mayor incidencia de malaria (2). En Sudamérica, P. vivax es el parásito más importante causante de malaria en términos de su morbilidad; hoy en día, Perú ocupa el tercer lugar después de Brasil y Colombia, siendo la región amazónica la más afectada (1). Los casos de malaria por P. falciparum ocurren mayormente en áreas de la jungla peruana, mientras que la malaria ocasionada por P. vivax es además endémica en la costa y valles interandinos, por lo que se presume que es la especie predominante (3,4).
En estas regiones endémicas, donde la transmisión de P. vivax es continua, la detección mediante vigilancia activa de casos asintomáticos podría no ser relevante, pero los datos sugieren la aparición de síntomas tempranos con escasa detección que llega al 0,1% (5). Esto puede atribuirse al hecho de que las personas experimentan síntomas rápidamente, lo cual conduce al incremento de pacientes con manifestaciones subclínicas no diagnosticadas, de los cuales solo algunos pasan a la detección por vigilancia pasiva en centros de salud y son sometidos a tratamiento, generalmente con fármacos como la cloroquina (6). La frecuencia de malaria asintomática no es conocida en regiones de transmisión baja. En Perú, los estudios se han limitado a diseños transversales, como los efectuados en las comunidades al sur de Iquitos, donde en un estudio de 998 individuos se encontró 13 con P. falciparum y 30 con P. vivax, detectados por microscopia; de estos, solo 8 y 19, respectivamente, comunicaron solamente fiebre. En zonas endémicas, el incremento de la inmunidad frente a las constantes infecciones, aunque no es protectora, evita las complicaciones de la enfermedad, incrementando el número de portadores asintomáticos (5, 6).
Claros ejemplos son los estudios realizados en Brasil, específicamente en las localidades ribereñas de Rondonia, Portuchuelo y Ji-Paraná, donde se diagnosticó infecciones por P. vivax y P. falciparum con formas asintomáticas y sintomáticas de la enfermedad, siendo estrechamente similares para ambas especies. En estas regiones geográficas, la prevalencia de infecciones asintomáticas fue 4 a 5 veces mayor que las sintomáticas, 20% y 4,6% para Portuchuelo, 49,5% y 10% para Ji-Paraná, respectivamente. La alta prevalencia de la malaria asintomática puede representar nuevos problemas para las estrategias de control adoptadas para la malaria en la región amazónica, la cual está basada esencialmente en el tratamiento de pacientes sintomáticos (7). Por otra parte, el incremento de la malaria asintomática se encuentra vinculado con las constantes infecciones o recurrencias de Plasmodium; así, en el estado de Acre (Granada), en la base de la Amazonía occidental de Brasil que limita con Perú y Bolivia, comunican índices de P. vivax = 0,39 y de P. falciparum = 0,13 infecciones/persona/temporada, con rangos de recurrencia postratamiento de 26% a 40% en 180 días de seguimiento (8).
La genotipificación de muestras secuenciales en individuos provenientes de regiones endémicas permite determinar la asociación genotípica entre distintos episodios de la infección, lo cual conlleva a determinar la recurrencia de la infección (9,10). Las reinfecciones y los relapsos son patrones de recurrencia poco conocidos; sin embargo, se sabe que los relapsos son una característica importante de P. vivax originados por estadios latentes en el hígado, conocidos como hipnozoitos. Los relapsos pueden ocurrir semanas o años después de registrado el primer episodio de parasitemia (11).
Las recurrencias de la infección por P. vivax generalmente son clasificadas por métodos de genotipificación, para lo cual se ha utilizado diversos marcadores genéticos, como los genes pvsc, pvmsp1 (12,13) y el pvmsp3-α, que codifica para la proteína de superficie 3α del merozoito del parásito, de mayor utilidad debido a su manejo, costo bajo y reproducibilidad alta; este marcador permite realizar una diferenciación precisa entre distintos genotipos del parásito (14). Es importante señalar que las recurrencias muestran diferencias notables entre diferentes cepas; así, las que provienen de zonas tropicales presentan infecciones primarias tempranas, con un corto periodo latente de 4 a 10 semanas antes de la aparición de frecuentes relapsos durante el siguiente año; en cambio, las cepas de zonas templadas presentan infecciones primarias de periodo variable, entre 5 a 10 meses antes del comienzo de los relapsos (13,15). En el 2009, en Guyana Francesa se encontró picos de relapsos de P. vivax similares a los de zonas tropicales, es decir, los relapsos se presentaron dentro de los tres meses después del primer episodio; después de este tiempo, los episodios secundarios fueron principalmente debidos a reinfecciones (16).
Estudios similares se han realizado en la Amazonía peruana desde el 2006, exactamente en el departamento de Loreto, donde se destaca la necesidad de efectuar análisis de genotipificación en zonas endémicas a malaria, por prevalencia de infecciones mixtas e incremento de recurrencias de la parasitemia principalmente por P. vivax (17). Por tales motivos, el presente estudio tuvo como principal objetivo determinar la variabilidad genética de P. vivax y los patrones de recurrencia en individuos con malaria asintomática, provenientes de la comunidad de Mazán, la cual actualmente está catalogada como una zona de riesgo alto en la transmisión de la malaria, principalmente por P. vivax.
MÉTODOS
El presente estudio se realizó con muestras colectadas durante los años 2006 a 2008 en la comunidad de Mazán, ubicada en la parte nororiental del Perú, a 50 km de la ciudad de Iquitos. Durante este periodo, mediante el sistema de vigilancia activa, se logró reclutar un total de 496 individuos asintomáticos. Para el estudio se consideró un tamaño muestral de 222 individuos, calculado con un nivel de confianza al 95% (Zα2 = 1.962), proporción esperada del 50% (p=0,5; q=1-p) y una precisión estadística en 5% (d=0,05) (18). Todos los individuos fueron previamente informados sobre el presente estudio. Se incluyó a todos los individuos comprendidos entre 0 a 75 años de edad, con firma de consentimiento informado, residentes en la zona endémica, que tuvieron dos muestras secuenciales, evaluados durante un año de seguimiento activo. Se excluyó a los individuos con tratamiento antimalárico previo a la evaluación.
A todos los individuos que formaron parte de la población muestral se les tomó 5 mL de sangre total (punción venosa). Todas las muestras fueron mantenidas a -20°C y posteriormente enviadas al área de Biología Molecular del Laboratorio de Investigación en Enfermedades Infecciosas de la Universidad Peruana Cayetano Heredia en Lima-Perú. Este estudio forma parte del proyecto Human reservoirs of Plasmodium vivax transmission in the Peruvian Amazon, el cual fue aprobado por el comité de ética de la universidad en mención (código SIDISI: 50457) y también aprobado por los comités de ética de la Universidad Johns Hopkins (USA), la Asociación Benéfica PRISMA y el Ministerio de Salud, a través de la Dirección Regional de Salud de Loreto.
Se realizó la extracción de ADN a partir de muestras de sangre con EDTA, usando el protocolo del kit comercial QIAamp DNA Blood (Qiagen Sciences, Maryland-USA). El volumen utilizado fue de 200 μL, del cual se obtuvo entre 20 a 50 ng de ADN, posteriormente empleado para las pruebas de diagnóstico y genotipificación molecular.
La identificación de las especies de Plasmodium se realizó mediante el nested-PCR (reacción en cadena de la polimerasa anidada), que consta de dos PCR consecutivos, realizados con la finalidad de aumentar tanto la especificidad como la sensibilidad del análisis. Las secuencias, los oligonucleótidos y las condiciones de reacción y amplificación fueron adaptadas a partir de un estudio epidemiológico efectuado previamente en la ciudad de Iquitos (6).
La primera PCR amplificó un segmento del gen que codifica el ARN ribosomal 18S (SSUrRNA), específico del género Plasmodiun, con los oligonucleótidos: Pgen1= 5´-CTTGTTGTTGCCTTAAACTTC-3´ y Pgen2= 5´-TTAAAATTGTTGCAGTTAAAACG-3´. El volumen final de reacción fue de 15 μL, conteniendo buffer 1X (Tris HCl 10 mM y KCl 50 mM), 2,0 mM de MgCl2, 0,2 mM deoxinucleótidos (dNTPs), 0,25 μM de cada oligonucleótido, 0,032 U/μL de Taq ADN polimerasa y 50 ng de ADN molde. Condiciones: 95ºC por 3 min., 19 ciclos cada uno de 94°C por 30 seg., 58°C por 30 seg., 72°C por 1 min. 30 seg. y 1 ciclo de extensión final de 72°C por 7 min.
La segunda PCR amplificó una región interna específica para P. vivax y para P. falciparum, con los oligonucleótidos para P. vivax Viv1= 5´-CGCTTCTAGCTTAATCCACATAACTGATAC-3´ y Viv2= 5´ -ACTTCCAAGCCGAAGCAAAGAAA-GTCCTTA-3´, los cuales amplificaron 120 pares de bases (pb), para P. falciparum: Fal1= 5´TTAAACTGGTTTGGGAAAACCAAATATATT-3´ y Fal2= 5´-ACACAATGAACTCAATCATGACTACCCGTC-3´, los cuales amplificaron 205 pb. El volumen final de reacción fue de 15 μL, conteniendo buffer 1X, 2,0 mM de MgCl2, 0,2 mM de dNTPs, 0,5 μM de cada oligonucleótido, 0,032 U/μL de Taq ADN polimerasa y 1 μL de ADN obtenido de la primera amplificación. Condiciones: 95ºC por 2 min, 34 ciclos cada uno de 94ºC por 30 seg., 60ºC por 1 min. 30 seg., 72ºC por 1 min. 30 seg., y finalmente 1 ciclo de extensión de 72ºC por 7 min.
La genotipificación de P. vivax se realizó mediante la técnica de PCR-RFLP, para la cual se utilizó el gen pvmsp3-α que codifica la proteína de superficie 3α del merozoito del parásito (MSP3-α); este marcador ha sido usado en otras investigaciones en zonas endémicas de Sudamérica como en nuestro país (19), Colombia (10) y en Brasil (20), también en lugares como Tailandia (14), Papúa Nueva Guinea (21) y la India (22). Las secuencias, los oligonucleótidos y las condiciones de reacción y amplificación fueron adaptadas a partir de estos estudios epidemiológicos.
Nested-PCR: Para la amplificación del gen pvmsp3-α, la primera PCR se realizó con los oligonucleótidos: P1V1= 5´CAGCAGACACCATTTAAGG-3´ y P2V1= 5´-CCGTTTGTTGATTAGTTGC-3´. El volumen final de reacción fue de 15 μL, conteniendo buffer 1X, 2,0 mM de MgCl2, 0,15 mM de dNTPs, 0,15 μM de cada oligonucleótido, 0,013 U/μL de Taq ADN polimerasa y 50 ng de ADN molde. Condiciones: 94ºC por 3 min., 34 ciclos cada uno de 94ºC por 30 seg., 56ºC por 30 seg., 68ºC por 2 min. 30 seg. y 1 ciclo de extensión final de 72ºC por 4 min.
Para la segunda PCR se utilizó los oligonucleótidos N1V1= 5´ -GACCAGTGTGATACCATTAACC-3´ y N2V1= 5´-ATACTGGTTCTTCGTCTTCAGG-3´, con los cuales se amplificó 1900 pb y 1500 pb, correspondientes a las secuencias alélicas A y B, respectivamente. Las reacciones de amplificación fueron similares a las usadas para la primera PCR con 1 μL de ADN agregado a cada reacción, obtenido de la primera amplificación. Condiciones: 94ºC por 3 min., 29 ciclos cada uno de 94ºC por 30 seg., 57ºC por 30 seg., 68ºC por 2 min. 30 seg. y 1 ciclo de extensión final de 68ºC por 4 min.
La digestión enzimática se realizó a partir de los productos de amplificación provenientes de la segunda PCR del nested PCR. Se utilizó dos enzimas de restricción denominadas Alu I y Hha I (New England BIOLabs), las cuales permitieron establecer el polimorfismo de la longitud de los fragmentos de restricción o RFLP, ampliamente utilizadas para estudios de genotipificación como los efectuados en Iquitos (Perú) (19), Colombia (10) y Brasil (20). Para la reacción de digestión se usó 8 μL del producto amplificado (ADN) conteniendo 0,5 unidades/enzima y se incubó a 37ºC por 3 horas. Los productos obtenidos (fragmentos de digestión) fueron visualizados en gel de agarosa al 2%, conteniendo 0,05 μg de bromuro de etidio (1 μg/mL). Se determinó los tamaños de los fragmentos usando un marcador de peso molecular de 100 pb. Los patrones enzimáticos fueron designados en base al tamaño de los fragmentos como haplotipos PA para Alu I y PH para Hha I. Se identificó las infecciones policlonales por la presencia de dos o más secuencias alélicas del producto de PCR (alelos A o B) o por la suma de los fragmentos, cuando excedían el tamaño del producto antes de la digestión (10).
La variabilidad genética de P. vivax fue evaluada mediante la determinación del nivel de la diversidad poblacional (PLD), típicamente definido como la variación de un gen en un loci antigénico, variable o neutral. Una medida de PLD es la heterozigocidad esperada He = 1/(1 − N) (1 – Σp2i), con p igual a la frecuencia de alelos y N igual al número de alelos detectados), demostrando una alta variabilidad genética con valores cercanos a la unidad. La complejidad de la infección (COI) es usada para evaluar el número mínimo de tipos de alelos presentes en un loci polimórfico de copia única; un COI>1 puede ser determinado por la observación de múltiples alelos dentro de una única muestra. Estos criterios fueron tomados en cuenta por estudios previos realizados en la Amazonía peruana (23).
Los patrones de recurrencia fueron establecidos a través de la genotipificación de P. vivax, durante dos periodos de evaluación. Se determinó los relapsos cuando se encontró haplotipos idénticos en ambos periodos y las reinfecciones cuando se halló haplotipos diferentes. En estudios de la Amazonía rural de Brasil (8), la genotipificación molecular permitió establecer los relapsos como la reactivación del mismo clon del parásito, es decir, se identificó genotipos idénticos tanto en la infección primaria como en la recurrencia de la enfermedad y en las reinfecciones se identificó genotipos diferentes. Asímismo, en Guyana Francesa (Camopi) (16) estiman las recurrencias como relapsos y reinfecciones tomando en cuenta rangos de tiempo antes y después de 90 días del primer episodio, respectivamente.
Para el análisis de datos, la identificación de los haplotipos se realizó independientemente con cada enzima y con la combinación de ambas. Para estimar la diferencia entre las medias de las frecuencias de haplotipos monoclonales, identificados en la primera y segunda evaluación, se realizó un análisis de varianza, con tablas de contingencia, el cual emplea la razón de las estimaciones o razón de Fisher (F). El valor estadístico de F se obtuvo dividiendo la estimación intermediante entre la estimación interna; luego, se comparó con el valor tabular de F, en un intervalo de significancia al 95% (α= 0,05). Para determinar la asociación entre los patrones de recurrencia y el rango de tiempo luego de la primera evaluación, se aplicó la prueba chi-cuadrado (X2), considerando una relación altamente significativa con una probabilidad del 99% cuando p < 0,01 (tabla X2) y con grados de libertad mayores o iguales a 1(GL ≥ 1).
RESULTADOS
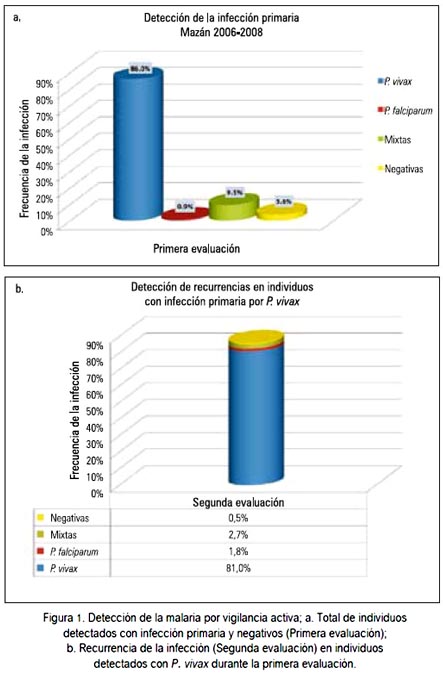
Durante la primera evaluación se detectó 191/222 (86%) casos con infección por P. vivax, 2/222 (0,9%) con infección por P. falciparum, 21/222 (9,5%) con infección mixta y 8/222 (3,6%) fueron negativos (figura 1a). Durante la segunda evaluación, considerando solo los casos con infección primaria por P. vivax, se detectó recurrencias de la infección por P. vivax en 180/191 (94,2%), los cuales fueron utilizados en los análisis posteriores de genotipificación (figura 1b).
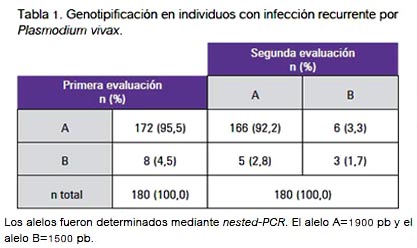
En la genotipificación de P. vivax en individuos con infección recurrente, mediante nested PCR se detectó 2 secuencias alélicas diferentes, el alelo A (1900 bp) y alelo B (1500 bp). En la primera evaluación, 172/180 (95,5%) presentaron el alelo A, siendo el de mayor frecuencia, mientras que 8/180 (4,5%) presentaron el alelo B. En la segunda evaluación 166/180 (92,2%) presentaron nuevamente el alelo A y 3/180 (1,7%) nuevamente el alelo B. Además, 5/180 (2,8%) con el alelo A y 6/180 (3,3%) con el alelo B fueron identificados en la primera evaluación como alelos B y A, respectivamente (tabla 1).
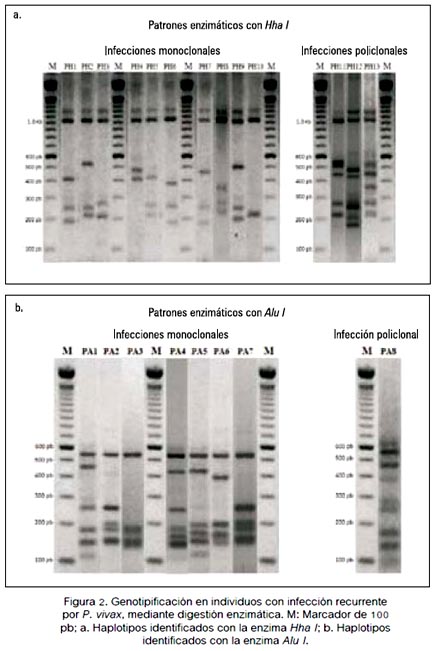
Mediante la digestión enzimática con Hha I, en el alelo A, se identificó nueve haplotipos monoclonales (PH1 a PH9) y tres haplotipos policlonales (PH11 a PH13). Con esta misma enzima, en el alelo B se identificó un haplotipo monoclonal (PH10) (figura 2a). Con Alu I, en el alelo A, se identificó seis haplotipos monoclonales (PA1PA6) y un haplotipo policlonal (PA8); con esta misma enzima, en el alelo B se identificó un haplotipo monoclonal (PA7) (figura 2b).
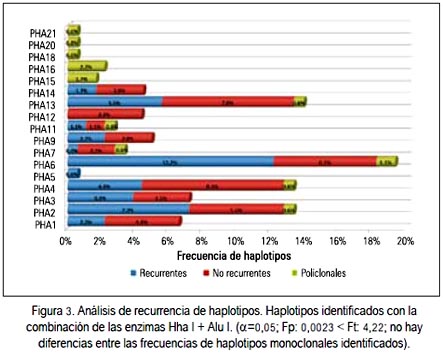
Con la combinación de ambas enzimas se logró una mayor variabilidad genética, identificando 14 haplotipos monoclonales (PAH1-PAH14) y siete haplotipos policlonales (PHA15PHA21). El análisis de recurrencia permitió identificar a los haplotipos PHA13, PHA2 y PHA6 como los más recurrentes, con 5,5 %, 7,2% y 12,2%, respectivamente. Los haplotipos PHA8, PHA10, PHA17 Y PHA19 no fueron identificados durante la primera evaluación; por lo tanto, no presentaron recurrencia. Se identificó haplotipos no recurrentes y policlonales, presentes como haplotipos diferentes en la segunda evaluación. La prueba F (α=0,05; Fp<Ft), indicó que no existe diferencias reales entre las frecuencias de haplotipos monoclonales identificados, mediante métodos de genotipificación, en los dos periodos de evaluación (figura 3). En el análisis estadístico, el 5,6% de los individuos presentó un COI>1 en la primera evaluación y 3,8% en la segunda evaluación. Los valores PLD en ambos periodos de evaluación fueron He=0,88 y 0,87, respectivamente.
En la determinación de los patrones de recurrencia de P. vivax, la combinación enzimática permitió identificar infecciones por haplotipos policlonales en 17/180 (9,4%) casos, en alguno de los dos periodos de evaluación. Se identificó infecciones por haplotipos monoclonales idénticos (relapsos) en 75/180 (41,7%) y diferentes (reinfecciones) en 88/180 (48,9%) casos, en ambos periodos de evaluación. En los relapsos, el haplotipo PHA6 fue el más frecuente con 12,2% y los haplotipos PHA5 y PHA7 fueron los menos frecuentes, ambos con 0,6%. En las reinfecciones, los haplotipos PHA4 y PHA13 tuvieron mayores porcentajes de haplotipos diferentes durante la segunda evaluación, con 8,4% y 7,8%, respectivamente. El haplotipo PHA12 solo presentó 4,4% de haplotipos diferentes, ninguno identificado como causante de relapso (figura 4a).
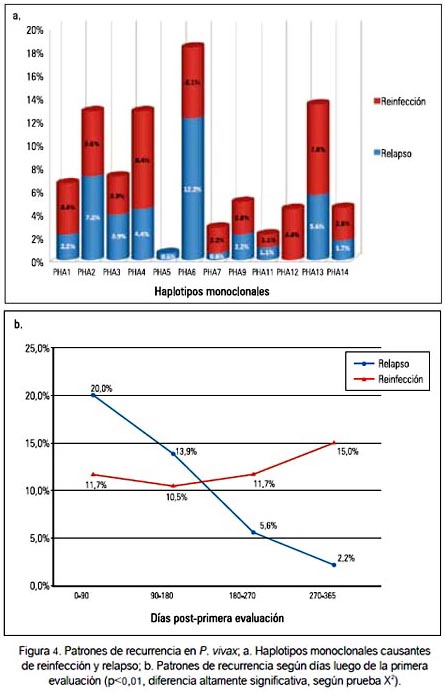
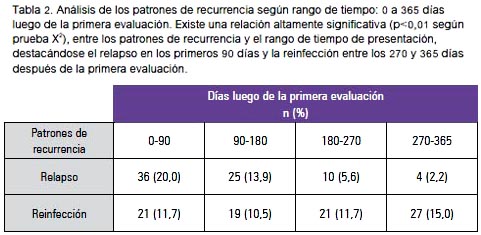
En la distribución de los patrones de recurrencias según el rango de tiempo se observó 36/180 (20%) relapsos y 21/180 (11,7%) reinfecciones entre 0 a 90 días. Los relapsos mostraron una disminución de 25/180 (13,9%), 10/180 (5,6%) y 4/180 (2,2%) casos entre 180, 270 y 365 días post-primera evaluación, respectivamente. En cambio las reinfecciones incrementaron mientras mayor era el rango de tiempo, de 19/180 (10,5%), 21/180 (11,7%) y 27/180 (15%) casos en los mismos periodos de tiempo (tabla 2 y figura 4b). Así mismo, la prueba X2 mostró una relación altamente significativa (p<0,01), entre los patrones de recurrencia y el rango de tiempo en que se presentaron estos patrones.
DISCUSIÓN
Las infecciones asintomáticas por Plasmodium son el principal problema en muchas regiones del mundo; la principal razón es que los individuos después de repetidas infecciones desarrollan inmunidad contra estos parásitos, por lo que se minimiza la intensidad de los síntomas, manteniendo además una parasitemia muy baja por largos períodos de tiempo (24). La inmunidad adquirida en forma natural a través de anticuerpos contra los estadios asexuales sanguíneos de Plasmodium, ha sido demostrada desde los años 1960, mediante técnicas serológicas como Elisa (25). Se ha comprobado que los anticuerpos persisten entre tres y seis meses después de la infección inicial (26). De esta manera, individuos de zonas endémicas podrían ser vinculados a un diagnóstico recurrente de la enfermedad, por la presencia continua de estos anticuerpos.
Técnicas novedosas como la PCR ofrecen alta sensibilidad y especificidad para la detección del ADN genómico de Plasmodium; además, es efectiva para la determinación de infecciones mixtas (27). Diferentes estudios usan variantes, como el nested PCR para el diagnóstico de Plasmodium (28); por lo general, este método es utilizado como prueba estándar. En Mazán-Iquitos, el diagnóstico por nested PCR ha demostrado la presencia de individuos asintomáticos con frecuencias altas de infecciones por P. vivax y bajas por P. falciparum. Condiciones similares han sido encontradas en Venezuela (29), indicando infecciones asintomáticas de 34,8% para P. vivax y 15,2% para P. falciparum. Otros estudios realizados en la Amazonía brasileña (8), donde existen bajos índices de transmisión tanto en P. vivax como en P. falciparum, indican un número alto de individuos asintomáticos no detectados.
El nested PCR también puede ser utilizado para realizar pruebas de genotipificación. En el presente estudio se detectó dos secuencias alélicas diferentes, una de 1900 pb (alelo A) y de 1500 pb (alelo B). Adicionalmente, investigaciones realizadas en Papúa Nueva Guinea (21, 26) informan sobre un tercer alelo de 1100 pb (alelo C); incluso estudios en la India (Chennai) (22), mencionan un cuarto alelo de 500 pb (alelo D). En todas estas investigaciones, la frecuencia del alelo A es mayor que la del alelo B, resultados similares a los encontrados en este estudio. Contrariamente, en regiones como Venezuela (30), se indica un 9,3% para el alelo A y 21,9% para el alelo B. Incluso contrastando con la mayoría de investigaciones de esta parte del mundo, un estudio realizado en el Sur de Irán (31) encuentra al alelo C como el más frecuente. Estos resultados nos muestran que existe una gran variabilidad genética de las poblaciones de P. vivax circulando en diferentes regiones geográficas; además, se observa un predominio de determinado tipo alélico, por determinada región.
Los métodos de nested PCR y digestión enzimática efectuados en Mazán permitieron identificar hasta 10 haplotipos monoclonales con la enzima Hha I y 7 con Alu I; algunos de estos haplotipos, seis con Hha I y tres con Alu I, ya han sido publicados en Tailandia (14), Papúa Nueva Guinea (21), India (22) e Irán (31), igual que en Perú (19), donde identificaron ocho y nueve patrones, respectivamente. Lo que sugiere que algunas de estas variantes alélicas tienen una distribución geográfica global. También, en Mazán se identificó tres haplotipos policlonales o mixtos con la enzima Hha I y 1 con la enzima Alu I, representando el 9,4% de todas las infecciones. Estos valores fueron menores a los encontrados en países como Tailandia(14) con 19,3%, Papúa Nueva Guinea (21, 26) con 23% y en Brasil (20) donde encontraron 14% de infecciones mixtas analizadas solo con la enzima Hha I. Incluso en otras localidades de la Amazonía peruana se registró 26,3% de infecciones mixtas por P. vivax, contrastando con este estudio, lo cual demuestra que en zonas como Mazán no hay una coinfección claramente establecida; sin embargo, la alta variabilidad genética determinada por los valores del PLD (cercano a la unidad) sí son bastante similares en estas regiones geográficas de la Amazonía peruana (23).
En las regiones endémicas, las recurrencias de malaria son bastante frecuentes, principalmente por las reinfecciones y por los relapsos propios de P. vivax. En el presente estudio, los métodos moleculares utilizados permitieron encontrar 41,7% de relapsos y 48,9% de reinfecciones. Frecuencias similares fueron halladas en estudios de genotipificación de la Amazonía brasileña (8), donde los rangos de recurrencias con haplotipos similares se encuentran entre 26% y 40%, con un intervalo de tiempo de 0 a 180 días postratamiento. Frecuencias más altas son informadas en soldados australianos (32), donde 99% de las recurrencias lo asociaron a un solo tipo alélico, con un intervalo de tiempo entre 29 y 343 días, el mismo periodo de tiempo utilizado en el presente estudio.
La importancia de realizar un diagnóstico preciso es determinar la especie de Plasmodium causante de la enfermedad. Del mismo modo, es necesario establecer la recurrencia de la infección, a fin de que el paciente reciba el tratamiento adecuado. Además, la variabilidad genética de P. vivax influye significativamente en el flujo de genes que pueden conferir resistencia a drogas, como la cloroquina. Esta resistencia ocasionada por la diversidad genética de P. vivax tiende a variar según el área geográfica y el vector. Estos conocimientos dirigidos a comprender la biología y diversidad genética son de utilidad para el tratamiento y diseño de vacunas efectivas, las cuales pueden ser dirigidas a una o varias fases específicas del parásito.
En conclusión, se estima una alta variabilidad genética de P. vivax circulante en regiones endémicas como Mazán-Iquitos, y los patrones de recu-rrencia, basados en la genotipificación, indican diferencias entre reinfecciones y relapsos, con una alta proporción de individuos infectados pero asintomáticos. Como recomendación se debe considerar necesario realizar estudios con un seguimiento más exhaustivo y detallado de los pacientes, así como con un mayor número de muestras junto con la utilización de otros marcadores y técnicas moleculares especializadas. El secuenciamiento y PCR en tiempo real podrían dar más luces sobre los resultados encontrados y ayudar a comprender mejor la dinámica de transmisión y la diversidad genética. Este estudio permitió conocer las frecuencias reales de la infección asintomática y los patrones de recurrencia. Esta información puede ser utilizada para establecer medidas que ayuden a las poblaciones asintomáticas a mejorar las condiciones de salud con un tratamiento eficaz, así como adecuados controles vectoriales que eviten las constantes recurrencias de la enfermedad.
AGRADECIMIENTOS
Agradecemos al Dr. R. Gilman, J. Vinetz y M. Kosek (Investigadores principales del proyecto Malaria en la UPCH); también, al Dr. C. Náquira, por su aporte en la revisión de la redacción del presente artículo.
REFERENCIAS BIBLIOGRÁFICAS
1. World Health organization. World malaria report. Switzerland: WHo Library Cataloguing in Publication Data. 2011.
2. Mendis K, Sina B, Marchesini P, Carter R. The neglected burden of Plasmodium vivax malaria. am J Trop Med Hyg. 2001;64:97-106.
3. Aramburu GJ, Ramal aC, Witzig R. Malaria reemergence in the Peruvian amazon region. Emerg InfectDis. 1999;5:209-15.
4. Roberts DR, Laughlin LL, Hsheih P, Legters LJ. Global strategies and malaria control crisis in Southamerica. Emerg Infect Dis. 1997;3:295-302.
5. Branch o, Casapia WM, Gamboa DV, Hernandez JN, alava FF, Roncal N, alvarez E, Perez EJ, Gotuzzo E. Clustered local transmission and asymptomatic Plasmodium falciparum and Plasmodium vivax malaria infections in a recently emerged,hypoendemic Peruvian amazon community. Malaria J. 2005;4:27.
6. Roshanravan B, Kari E, Gilman RH, Cabrera L, Lee E, Metcalfe J, Calderon M, Lescano aG, Montenegro SH, Calampa C, Vinetz JM. Endemic malaria in the Peruvian amazon region of Iquitos. am J Trop Med Hyg. 2003;69:45-52.
7. Alves FP, Durlacher RR, Menezes MJ, Krieger H, Silva LH, Camargo EP. High prevalence of asymptomatic Plasmodium vivax and Plasmodium falciparum infections in native amazonian populations. am J Trop Med Hyg. 2002;66:641-8.
8. Orjuela PS, da Silva N, da Silva M, Urbano FM. Recurrent parasitemias and population dynamics of Plasmodium vivax polymorphisms in rural amazonia. Department of Health Sciences, Federal University of acre, Rio Branco, Brazil. am J Trop Med Hyg. 2009;81:961-8.
9. Mallika I, Georges S, Sasithon P, Naowarat T, Jung RK, amitab N, Jean-Paul G, Francois N, Jane C, Sornchai L, Shalini N, Daniel S, Nicho-las PJ, Timothy JC, anderson JW. Relapses ofPlasmodium vivax infection usually result from activation of heterologous hypnozoites. J Infect Dis. 2007;195:27-33.
10. Cristiano Fa., Pérez Ma., Nicholls RS, Guerra aP. Polymorphism in the Plasmodium vivax msp 3α gene in field samples from Tierralta,Instituto Nacional de Salud, Bogotá, D.C., Colombia. Mem Inst oswaldo Cruz, Rio de Janeiro. 2008;103(5):493-6.
11. Vargas J, Elgegren J, San Miguel a, Cardoso R. Malaria en una población urbano marginal deIquitos. Rev Peru Epidemiol. 2003;11(1):1-6.
12. Imwong M, Pukrittayakamee S, Gruner aC, Renia L, Letourneur F, Looareesuwan S, White NJ, Snounou G. Practical PCR genotyping protocols for Plasmodium vivax using Pvcs and Pvmsp1. Malaria J. 2005;4:20.
13. Craig aa, Kain KC. Molecular analysis of strains of Plasmodium vivax from paired primary and relapse infections. J Infect Dis. 1996;174:373-9.
14. Cui L, Mascorro CN, Fan Q, Rzomp Ka, Khuntirat B, Zhou G, Chen H, yan G, Sattabongkot J. Geneticdiversity and multiple infections of Plasmodium vivax malaria in Western Thailand. am J Trop Med Hyg. 2003;68:613-9.
15. Kain KC, Craig aa, ohrt C. Single-strand conformational polymorphism analysis differentiates Plasmodium falciparum treatment failures from reinfections. Mol Biochem Parasitol. 1996;79(2):167-75.
16. Hanf M, Stéphani a, Basurko C, Nacher M, Carme B. Determination of the Plasmodium vivax relapse pattern in Camopi, French Guiana. Malaria J. 2009;8:278.
17. Ayala E, Lescano aG, Gilman RH, Calderón M,Pinedo V, Hilja T, Cabrera L, Vinetz JM. Polymerase chain reaction and molecular genotyping to monitor Parasitological response to anti-malarial chemotherapy in the Peruvian amazon. am J Trop Med Hyg. 2006;74(4):546-53.
18. Fernández PS. Metodología de la investigación. Unidad de Epidemiología Clínica y Bioestadística.Complejo Hospitalario Universitario de La Coruña. Cad aten Primaria. 1996;3:138-40.
19. Kosek M, Yori PP, Gilman RH, Calderon M, Zimic M, Chuquiyauri R, Jeri C, Pinedo-Cancino V, Matthias Ma, Llanos-Cuentas a, Vinetz JM. High Degree of Plasmodium vivax Diversity in the Peruvian amazon Demonstrated by Tandem Repeat Polymorphism analysis. am J Trop Med Hyg. 2012;86(4):580-6.
20. Ribeiro RS, Ladeira L, Rezende aM, Fernandes CJ,Carvalho LH, Ferreira C. analysis of the genetic variability of PvMSP-3α among Plasmodium vivax in Brazilian field isolates. Mem Inst oswaldo Cruz, Rio de Janeiro. 2011;6:27-33.
21. Bruce MC, Galinski MR, Barnwell JW, Snounou G, Day KP. Polymorphism at the merozoite surface protein-3alpha locus of Plasmodium vivax: global and local diversity. Am J Trop Med Hyg. 1999;61:518-25.
22. Prajapati SK, Joshi H, Valecha N. Plasmodium vivax merozoite surface protein-3α: a high-resolution marker for genetic diversity studies. J Vector BorneDis. 2010;47:85-90.
23. Sutton PL, Neyra V, Hernandez JN, Branch oH. Plasmodium falciparum and Plasmodium vivax infections in the Peruvian amazon: propagation of complex, multiple allele-type infections without super-infection. Am J Trop Med Hyg.2009;81(6):950-60.
24. Coura RJ, Suárez MM, andrade LS. a new challenge for malaria control in Brazil: asymptomatic Plasmodium infection - a review. Mem Inst oswaldo Cruz, Rio de Janeiro. 2006;101(3):229-37.
25. Ferreira MU. Distribución de la subclase IgG en anticuerpos adquiridos en forma natural contraPlasmodium falciparum, en relación a la exposición y a la severidad de malaria. TDR/oPS/oMS. Buenos Aires, Argentina. 2006;1:117-28.
26. Tangpukdee N, Duangdeev C, Wilaratana P, Krudsood S. Malaria diagnosis: a brief review. KoreanJ Parasitol. 2009;47:93-102.
27. Singh B, Bobogare a, Cox-Singh J, Snounou G, abdullah MS, Rahman Ha. a genus- and speciesspecific nested polymerase chain reaction malaria detection assay for epidemiologic studies. Am J Trop Med Hyg. 1999;60:687-92.
28. Andrade BB, Reis-Filho a, Souza-Neto SM, Clarêncio J, Camargo LM, Barral a, Barral-Netto M. Severe Plasmodium vivax malaria exhibits marked inflammatory imbalance. Fundação oswaldo Cruz Brazil. Malaria J. 2010;9:13.
29. Rodulfo H, De Donato M, Quijada I, Peña a. High prevalence of malaria infection in amazonas state, Venezuela. Rev Inst Med Trop. 2007;49:79-85.
30. ord R, Polley S, Tami a, Sutherland CJ. High sequence diversity and evidence of balancing selection in the Pvmsp3alpha gene of Plasmodium vivax in the Venezuelan amazon. Mol Biochem Parasitol. 1995;144:86-93.
31. Zakeri S, Barjesteh H, Djadid ND. Merozoite surfaceprotein-3alpha is a reliable marker for population genetic analysis of Plasmodium vivax. Malaria J. 2006;5:53.
32. Chen N, auliff a, Rieckmann K, Gatton M, Cheng Q. Relapses of Plasmodium vivax infection result from clonal hypnozoites activated at predeterminedintervals. J Infect Dis. 2007;195:934-41.
Artículo recibido el 30 de mayo de 2012 y aceptado para publicación el 26 de setiembre de 2012.
Financiado por: asociación Benéfica Prisma y Universidad Peruana Cayetano Heredia.
Correspondencia:
Edward Valencia Ayala
Laboratorio de investigación en enfermedades infecciosas lid Universidad Peruana Cayetano Heredia
Av. Honorio Delgado N° 430 Lima, Perú
Móvil: 954 668 623
Correo electrónico: Edu_5_7@hotmail.com













