Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Anales de la Facultad de Medicina
versión impresa ISSN 1025-5583
An. Fac. med. v.74 n.1 Lima ene. 2013
ARTÍCULOS ORIGINALES
Diagnóstico genético preimplantacional: análisis de aneuploidías únicas
Preimplantational genetic diagnosis: single aneuploidies analysis
Paul W. López1, Rosmary López1, Luis G. Noriega2, Soledad Sepúlveda2
1 Laboratorio Reprogenetics Latinoamérica, Lima, Perú.
2 Laboratorio de Reproducción asistida, Grupo PRANOR, Lima, Perú.
Resumen
Introducción: De las causas más conocidas en cuanto a la falta del éxito en el embarazo con tratamientos de reproducción asistida son aquellas relacionadas a las aneuploidías cromosómicas presentes en los embriones. El diagnóstico genético preimplantacional (PGD) es una técnica empleada en reproducción asistida para detectar estas anomalías, seleccionando aquellos que sean cromosómicamente normales, para luego transferirlos al útero de la paciente. Los embriones con aneuploidías únicas podrían tener la capacidad de sobrevivir y lograr la implantación, y por lo tanto, sin diagnóstico previo, estas podrían pasar desapercibidas. Objetivos: Determinar la incidencia de aneuploidías únicas en embriones de buena calidad embrionaria en el día 3 de desarrollo hasta blastocisto. Diseño: Estadístico y experimental. Instituciones: Reprogenetics Latinoamérica y Centro de Reproducción asistida, de la Clínica Concebir. Material Biológico: Muestras de biopsia embrionaria. Metodología: Análisis comparativo de resultados a partir de la evaluación de cada muestra obtenida por biopsia en el día tercero y día quinto de desarrollo embrionario, realizando el PGD por hibridación in situ (FISH) y genómica comparada (aCGH), respectivamente. Resultados: El 62,9% de embriones que presentaron monosomías únicas al tercer día de desarrollo embrionario resultaron ser de 8 células. Pero cuando se evaluó por aCGH en día cinco, 42,3% resultó anormal, y de estos 37,5% perteneció al estadio de 8 células. El índice de monosomías únicas en blastocisto resultó ser 57,9% de un total de 84,2% de aneuploidías únicas. Conclusiones: Los embriones de 8 células en el tercer día de desarrollo embrionario son los más probables de llegar al estadio de blastocisto, así como presentar aneuploidías únicas.
Palabras clave: Diagnóstico genético preimplantacional-PGD, genómica comparada - aCGH, hibridización in situ - FISH, aneuploidía, monosomía, trisomía, blastocisto.
Abstract
Background: Known causes of unsuccessful pregnancy in couples undergoing assisted reproduction treatment include embryo aneuploidies. Preimplantation genetic diagnosis (PGD) is a technique used in assisted reproduction in order to detect these abnormalities, select embryos chromosomally normal and subsequently transfer to the patients uterus. Embryos with single aneuploidies may have the ability to survive and achieve unnoticed implantation. Objectives: To determine incidence of single aneuploidies in good quality embryos in third day of development to blastocyst. Design: Statistical and experimental study. Setting: Reprogenetics Latin-America and Assisted Reproduction Center - Concebir. Biologic material: Samples of embryo biopsies. Methods: Comparative analysis of results from evaluation of each sample obtained by embryo biopsy on the third and fifth days of embryonic development, performing PGD by respectively in situ hybridization (FISH) and comparative genomics (aCGH). Results: On third day of embryonic development 62.9% of embryos with single monosomy had 8-cell morphology. Though when evaluated by aCGH in the blastocyst stage 42.3% were abnormal and 37.5% of these belonged to the 8-cell stage. Single monosomies index in the blastocyst stage was 57.9% in 84.2% of single aneuploidies. Conclusions: Eight-cell embryos on the third day of embryonic development are most likely to reach blastocyst stage and have single aneuploidies.
Key words: Preimplantation genetic diagnosis-PGD, comparative genomics -aCGH, in situ hybridization -FISH, aneuploidy, monosomy, trisomy, blastocyst.
INTRODUCCIÓN
Se conoce que más de la mitad de embriones obtenidos en los ciclos de fecundación in vitro (FIV) presenta células con aneuploidías (1-3), lo cual se ve reflejado en fallas de implantación, abortos espontáneos y el nacimiento de niños con trisomías (4-8).
Tratando de seleccionar los mejores embriones para transferirlos al útero de la paciente, se ha desarrollado técnicas de tamizaje genético preimplantacional (PGS, por sus siglas en inglés), que permiten analizar el contenido cromosómico de cada embrión.
Existen diferentes metodologías para el PGS. La primera técnica desarrollada fue la hibridización fluorescente in situ (FISH, por sus siglas en inglés), con la cual se puede analizar 5, 9 o 12 cromosomas (9-12). Luego, aparecieron la hibridación genómica comparada por microarreglos (array) (aCGH) (13-17) y la realizada en base a polimorfismos de nucleótido único (SNP, por sus siglas en inglés) (18-23), técnicas que permiten evaluar el total de cromosomas.
Se ha visto que todas estas metodologías tienen un efecto positivo en las tasas de implantación y en el progreso del embarazo, en pacientes con problemas de fertilidad (24-26).
La biopsia para el PGS puede ser hecha en el día 3 o 5 del desarrollo embrionario, analizándose una blástomera o un trozo de trofoectodermo (4 a 8 células), respectivamente (2,14,27,28).
La ventaja de realizar la biopsia en el día 3, es que la transferencia embrionaria puede realizarse en el mismo ciclo de estimulación ovárica. Pero, ello no asegura por completo un diagnóstico correcto, ya que al analizar solo una célula y de tenerse mosaisismo en el embrión, este no podría ser detectable. Por el contrario, cuando se biopsia en día 5, donde se analiza un grupo de células procedentes del trofoblasto, este riesgo disminuye (21). En el año 2011 (14), se encontró que con FISH, 75% de los embriones era informado como anormal, siendo 44% de estos mosaico y 56% presentaba uniformemente anomalías en todas sus células; por aCGH se detectó también que 60% de los eventos de aneuploidías tiende a la pérdida de cromosomas y el 40% restante a la ganancia de algún cromosoma. De este grupo, la FISH pudo detectar 41,2% de los eventos con un análisis de 9 cromosomas y 54,1% con el examen de 12 cromosomas. En general, la FISH apenas detecta 81% y 87% de los embriones anormales, con las pruebas de 9 y 12 cromosomas. Se ha demostrado también que la aCGH es robusta en cuanto a detección de aneuploidías, llegando a 2,9% de análisis sin resultado y 1,9% de tasa de error.
Así mismo, se sabe que una buena morfología embrionaria no garantiza una constitución cromosómica normal (30-31). Al llevar el cultivo de embriones al día 5 de desarrollo, la activación del genoma embrionaria ya ha ocurrido (32-33), pero ello no asegura que todos los embriones que llegan al estadio de blastocisto sean cromosómica-mente normales (34-36). Por lo tanto, se recomienda realizar análisis de aneuploidías.
El objetivo de este estudio ha sido correlacionar los datos de embriones obtenidos mediante fecundación in vitro (FIV) según su estado celular en el día 3 de cultivo, respecto a su normalidad cromosómica analizada en día 3 por FISH o en día 5 por aCGH.
MÉTODOS
Los procedimientos de fecundación in vitro fueron desarrollados en el laboratorio de Reproducción Asistida de la Clínica Concebir en Lima, de acuerdo a protocolos ya publicados (37). Se registró en una base de datos la información de clivaje de los embriones tempranos en día de 3 de cultivo, de parejas cuyos médicos indicaron que se realizara PGS, en donde los pacientes firmaron un consentimiento informado de manera voluntaria para la participación en el estudio. Al azar se seleccionó muestras de parejas para ser analizados por FISH de una blastómera en día 3 de desarrollo, así como muestras para biopsia de trofoectodermo en día 5 por aCGH. Todos los procedimientos de PGS fueron realizados por Reprogenetics Latinoamérica, Lima - Perú.
Para los casos de evaluación por FISH, luego de la biopsia embrionaria en día 3, la blastómera fue expuesta a una solución hipotónica [0,075 mol/L KCl suplementada con 0,6% BSA (p/v)] y luego fijada con solución Carnoy (38,39) sobre una lámina portaobjeto, para luego ser sometida al análisis por FISH para nueve (13, 15, 16, 17, 18, 21, 22, X e Y) o doce cromosomas (8,13, 14, 15, 16, 17, 18, 20, 21, 22, X e Y) (9).
Para el análisis de aCGH, la biopsia fue realizada con láser en día 5, en estadio de blastocisto. Se tomó una muestra de células del trofoectodermo mural y se la procesó de acuerdo a lo descrito por Gutiérrez y col., 2011. En breve, se la colectó en tubos de PCR estériles, libre de ADNsas y ARNsas. Para la amplificación genómica de cada muestra se empleó el Kit SurePlex, de acuerdo a las indicaciones de fabricante (BlueGnome). En cada amplificación se empleó como control positivo 1 ng de ADN genómico de una mujer y como control negativo se usó 2 μL del buffer NWB, en el que se realiza la biopsia (PBS 1X con PVA 0,1%). Las muestras fueron procesadas de acuerdo al protocolo Cytochip 2.0 de BlueGenome (disponible en www.cytochip.com). Las imágenes fueron analizadas con el software BlueFuse de acuerdo al algoritmo Cytochip 2.0.
Entraron al análisis de datos para el PGS en día 3 los resultados de 355 embriones diagnosticados como anormales, tomándose de este grupo aquellos resultados en donde como máximo se tuvo dos monosomías únicas, bajo el supuesto de que al menos una de estas monosomías fuera producto de una hibridación poco clara, como es la sobre-posición de señales por la cercanía de los cromosomas evaluados. Para el caso de las muestras para el análisis de PGS en día 5, se evaluó 52 muestras de trofoectodermo que resultaron tener un perfil genético analizable, lográndose obtener información de su estado embrionario en día 3 solo en 49 de estas muestras.
RESULTADOS
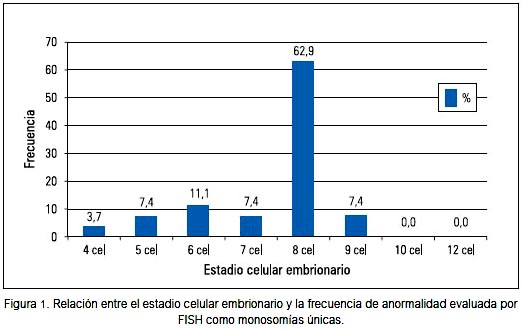
Se encontró, por el método de FISH que, de 355 embriones diagnosticados como anormales, 7,6% presentó hasta dos monosomías y 62,9% de estos pertenecía al estadio de 8 células (figura 1).
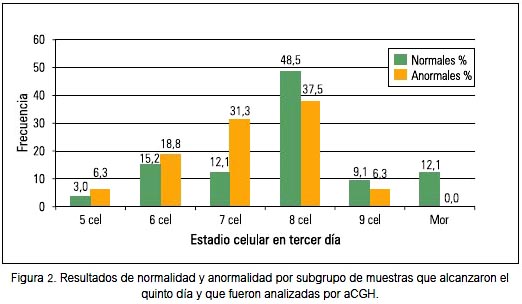
Para el caso en donde se realizó el PGS por aCGH, se encontró que 44,9% de los embriones que fueron biopsiados en el quinto día de desarrollo fue clasificado en día 3 como perteneciente al grupo de 8 células; de este grupo, 37,5% resultó anormal luego del aCGH (figura 2).
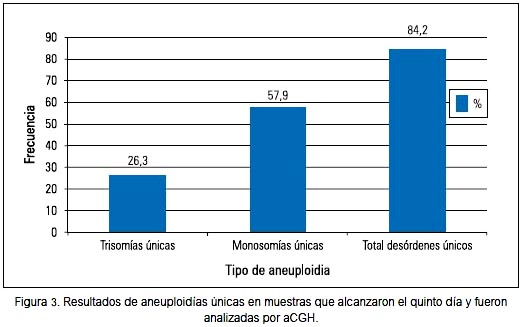
Al evaluar el total de embriones que resultaron anormales (42,3%) luego del aCGH, encontramos que 84,2% resultó tener aneuploidías únicas, distribuyéndose en 26,3% de trisomías únicas y 57,9% de monosomías únicas (figura 3).
DISCUSIÓN
Entre los embriones anormales cromosómicamente, aquellos que presentaron monosomías únicas con la prueba de FISH en día 3, 62,9% estuvo en grado de 8 células, coincidiendo con lo publicado anteriormente (14), donde se estima un 60%. De la misma manera, cuando evaluamos nuestros resultados por aCGH encontramos que cerca de la mitad (44,9%) de los embriones que llegaron a blastocisto correspondía a aquellos que fueron clasificados al tercer día de desarrollo como de buena calidad (8 células), lo que incluía a 57,9% de los embriones que presentó pérdidas cromosómicas.
Por lo tanto, aunque el embrión ya cuente con una propia activación genómica y una buena calidad morfológica, esto no asegura normalidad cromosómica, pudiendo pasar desapercibidas estas anomalías que conllevarían a fallos en el embarazo (32,33). Esto confirma la necesidad del PGS para evitar la transferencia de embriones aparentemente buenos, pero que son cromosómicamente anormales.
Los embriones en su tercer día de desarrollo embrionario en reproducción asistida, presentan un alto número de aneuploidías y mosaicismo (1-3,10,11) y quizás la morfología (en día 3) mejora la predicción para los embriones que tienen mayor posibilidad de seguir avanzando hasta llegar a blastocisto (día 5); pero, esto no ha de suponer normalidad cromosómica. Por otro lado, es también de esperar que se encuentre una mayor diversidad de anormalidades cromosómicas antes de que se llegue al estadio de blastocisto, tal como muestran nuestros primeros resultados, en donde solo un tipo anomalía única representa cerca del 8%.
REFERENCIAS BIBLIOGRÁFICAS
1. Vanneste E, Voet T, Le Caignec C, Ampe M, KoningsP, Melotte C, et al. Chromosome instability is common in human cleavage-stage embryos. Nat Med. 2009;15:577–83.
2. Fragouli E, Lenzi M, Ross R, Katz-Jaffe M, Schoolcraft WB, Wells D. Comprehensive molecular cytogenetic analysis of the human blastocyst stage. Hum reprod. 2008;23:2596–608.
3. Wells D, Delhanty JD. Comprehensive chromosomal analysis of human preimplantation embryos using whole genome amplification and single cell comparative genomic hybridization. Mol Hum reprod. 2000;6:1055–62.
4. Munne S, Magli C, Bahce M, Fung J, Legator M, Morrison L, et al. Preimplantation diagnosis of the aneuploidies most commonly found in spontaneous abortions and live births: XY, 13, 14, 15, 16, 18, 21, 22. Prenat Diagn. 1998;18:1459–66.
5. Warburton D, Kline J, Stein Z. Cytogenetic abnormalities in spontaneous abortions of recognized conceptions. In: Porter IH, Willey a, editors. Perinatalgenetics: diagnosis and treatment. new York: academic Press; 1986:133.
6. Bianco K, Caughey AB, Shaffer BL, Davis R, Nortonm E. History of miscarriage and increased incidence of fetal aneuploidy in subsequent pregnancy. Obstet Gynecol. 2006;107:1098–102.
7. Rubio C, Pehlivan T, Rodrigo L, Simon C, Remohi J, Pellicer A. Embryo aneuploidy screening forunexplained recurrent miscarriage: a minireview. Am J reprod Immunol. 2005;53:159–65.
8. Sullivan AE, Silver RM, La Coursiere DY, Porter TF, Branch DW. Recurrent fetal aneuploidy and recurrent miscarriage. Obstet Gynecol. 2004;104:784–8.
9. Colls P, Goodall N, Zheng X, Munneincreased S. Increased efficiency of preimplantation genetic diagnosis for aneuploidy by testing 12 chromosomes. Reprod Biomed Online. 2009;19(4):532–8.
10. Munné S, Magli C, Cohen J, Morton P, Sadowy S, Gianaroli L, Tucker M, Marquez C, Sable D, Ferraretti AP, Massey JB, Scott R. Positive outcome after preimplantation diagnosis of aneuploidy in human embryos. Hum reprod. 2009;14:2191–9.
11. Munné S, Sandalinas M, Escudero T, Velilla E, Walmsley R, Sadowy S, Cohen J, Sable D. Improved implantation after preimplantation geneticdiagnosis of aneuploidy. Reprod Biomed Online.2003;7:91–7.
12. Munné S, Fragouli E, Colls P, Katz-Jaffe m, Schoolcraft W, Wells D. Improved detection of aneuploid blastocysts using a new 12-chromosome FISH test. Reprod Biomed Online. 2010;20(1):92-7.
13. Bisignano a, Wells D, Harton G, munne S. PGD and aneuploidy screening for 24 chromosomes: advantages and disadvantages of competing platforms. reprod Biomed Online. 2011;23:677–85.
14. Gutiérrez-mateo C, Colls P, Sánchez-García J, Escudero T, Prates R, Ketterson K, Wells D, Munné S. Validation of microarray comparative genomic hybridization for comprehensive chromosome analysisof embryos. Fertil Steril. 2011;95(3):953-8.
15. Hellani A, Abu-amero K, Azouri J, El-akoum S. Successful pregnancies after application of arraycomparative genomic hybridization in PGS aneuploidy screening. Reprod Biomed Online. 2008;17:841–7.
16. Hu DG, Webb G, Hussey N. Aneuploidy detection in single cells using DNA array-based comparative genomic hybridization. mol Hum reprod.2004;10:283–9.
17. Le Caignec C, Spits C, Sermon K, De Rycke M, Thienpont B, Debrock S, et al. Single-cell chromosomal imbalances detection by array CGH. nucleic acids res. 2006;34:e68.
18. Brezina PR, Benner A, rechitsky S, Kuliev A, Pomerantseva E, Pauling D, Kearns WG. Single-gene testing combined with single nucleotide polymorphism microarray preimplantation genetic diagnosis for aneuploidy: a novel approach in optimizing pregnancy outcome. Fertil Steril. 2011;95:1786 (e5–8).
19. Handyside AH. PGD and aneuploidy screening for 24 chromosomes by genome-wide SnP analysis: seeing the wood and the trees. reprod Biomed Online. 2011;23:686–91.
20. Johnson DS, Gemelos G, Baner J, Ryan A, Cinnioglu C, Banjevic M, et al. Preclinical validation of a microarray method for full molecular karyotyping of blastomeres in a 24-h protocol. Hum reprod. 2010;25:1066–75.
21. Treff nr, Su J, Tao X, Levy B, Scott Jr rT. accuratesingle cell 24 chromosome aneuploidy screening using whole genome amplification and single nucleotide polymorphism microarrays. Fertil Steril. 2010;94(6):2017–21.
22. Treff N, Su J, Tao X, Miller K, Levy B, Scott R. A novelsingle-cell Dna fingerprinting method successfullydistinguishes sibling human embryos. Fertil Steril. 2010;94:477–84.
23. Treff NR, Northrop LE, Kasabwala K, Su J, LevyB, Scott Jr RT. Single nucleotide polymorphism microarray–based concurrent screening of 24chromosome aneuploidy and unbalanced translocations in preimplantation human embryos Fertil Steril. 2011;95():1606-12.e2
24. Donoso P, Platteau P, Papanikolaou EG, Staessen C, Van Steirteghem a, Devroey P. Does PGD for aneuploidy screening change the selectionof embryos derived from testicular sperm extraction in obstructive and non-obstructive azoospermic men? Hum reprod. 2006;21:2390–5.
25. Munné S, Sandalinas M, Magli C, Gianaroli L, Cohen J, Warburton D. Increased rate of aneuploid embryos in young women with previous aneuploid conceptions. Prenat Diagn. 2004;24:638–43.
26. Platteau P, Staessen C, Michiels A, Van Steirteghem A, Liebaers I, Devroey P. Preimplantation geneticdiagnosis for aneuploidy screening in patients with unexplained recurrent miscarriages. Fertil Steril. 2005;83:393–7.
27. Baart EB, Van Opstal D, Los FJ, Fauser BC, martini E. Fluorescence in situ hybridization analysis of twoblastomeres from day 3 frozen-thawed embryosfollowed by analysis of the remaining embryo onday 5. Hum reprod. 2004;19:685–93.
28. Grifo Ja. Preconception and preimplantacion gene-tic diagnosis: polar body, blastmere, and trophectoderm biopsy. En: Cohen J, malter HE, Talansky BE, Grifo Ja (eds). micromanipulation of Gametes and Embryos. new York: raven Press, 1992:223–49.
29. Sandalinas m, Sadowy S, alikani m, Calderon G, Cohen J, munné S. Developmental ability of chromo somally abnormal human embryos to develop to theblastocyst stage. Hum reprod. 2001;16:1954–8.
30. magli mC, Jones Gm, Gras L, Gianaroli L, KormanI, Trounson aO. Chromosome mosaicism in day3 aneuploid embryos that develop to morphologically normal blastocysts in vitro. Hum reprod. 2000;15:1781-6.
31. Voullaire L, Slater H, Williamson r, Wilton L. Chromosome analysis of blastomeres from human embryosby using comparative genomic hybridization. Hum Genet. 2000;106:210-7.
32. Gardner DK, Schoolcraft WB, Wagley L, Schlenker T,Stevens J, Hesla J. a prospective randomized trial ofblastocyst culture and transfer in in-vitro fertilization.Hum reprod. 1998;13:3434–40.
33. Papanikolaou EG, Dhaeseleer E, Verheyen G, Van de Velde H, Camus m, Van Steirteghem a, Devroey P, Tournaye H. Live birth rate is significantly higher after blastocyst transfer than after cleavage-stageembryo transfer when at least four embryos areavailable on day 3 of embryo culture. a randomizedprospective study. Hum reprod. 2005;20:3198–203.
34. Staessen S, Platteau P, Van Assche E, Michiels A, Tournaye H, Camus M, Devroey P, Liebaers I, Van Steirteghem A. Comparison of blastocyst transfer with or without preimplantation genetic diagnosis for aneuploidy screening in couples with advanced maternal age: a prospective randomized controlledtrial. Hum reprod. 2004;19:2849–58.
35. Fiegler H, Geigl JB, Langer S, Rigler D, Porter K, Unger K, et al. High resolution array-CGH analysis of single cells. nucleic acids res. 2007;35:e15.
36. Fragouli E, Katz-Jaffe M, Alfarawati S, Stevens J, Colls P, Goodall N, et al. Comprehensive chromosome screening of polar bodies and blastocystsfrom couples experiencing repeated implantation failure. Fertil Steril. 2010;94:875–87.
37. Sepúlveda S. Fecundación y desarrollo en humanos En: Lerner J, Urbina MT (eds). reproducción Asistida. Caracas: Editorial Médica Panamericana. 2008:119-23.
38. Munné S, Cohen J. Chromosome abnormalities in human embryos. Human reprod update.1998;4:842–55.
39. Velilla E, Escudero T, munné S. Blastomere fixation techniques and risk of misdiagnosis for preimplantation genetic diagnosis of aneuploidy Reproduction Biomed Online. 2002;4(3):210-7.
Artículo recibido el 17 de julio de 2012 y aceptado para publicación el 24 de octubre de 2012.
Correspondencia: Paul W. López
Correo electrónico: plopez@reprogenetics.com













