Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Anales de la Facultad de Medicina
Print version ISSN 1025-5583
An. Fac. med. vol.74 no.1 Lima Jan. 2013
ARTÍCULOS DE REVISIÓN
Staphylococcus aureus resistente a meticilina asociado a la comunidad: aspectos epidemiológicos y moleculares
Community-associated methicillin-resistant Staphylococcus aureus: molecular and epidemiological aspects
Daniel Angel Luján Roca1
1 Programa de Post-Grado en Infectología y Medicina Tropical. Facultad de Medicina, Universidade Federal de Minas Gerais, Minas Gerais, Brasil.
Resumen
El Staphylococcus aureus resistente a meticilina asociado a la comunidad (SARM-AC) está ampliamente diseminado en el mundo. Es epidémico en algunos países y posee dos marcadores moleculares característicos: el SCCmec IV y la leucocidina de Panton-Valentine, ambos escasamente encontrados en el SARM asociado a la asistencia médica.
Palabras clave: Staphylococcus aureus, SARM, SCCmec.
Abstract
Community-associated methicillin-resistant Staphylococcus aureus (CA-MRSA) is broadly disseminated in the world, is epidemic in some countries and possesses two characteristic molecular markers: SCCmec IV and Panton-Valentine leukocidin, both scarcely found in healthcare-associated MRSA.
Key words: Staphylococcus aureus, MRSA, SCCmec.
INTRODUCCIÓN
Entre 1880 y 1882, sir Alexander Ogston describió la enfermedad por estafilococos y su rol en la sepsis y formación de abscesos (1). Más de 130 años han pasado y Staphylococcus aureus continúa siendo uno de los patógenos más peligrosos que afecta a los humanos (2). El S. aureus es la principal causa de infecciones bacterianas que involucra el torrente circulatorio, el tracto respiratorio, la piel y los tejidos blandos (3). Es una de las causas más comunes de infecciones nosocomiales y comunitarias, manifestando una carga alta de morbilidad y mortalidad (4).
La penicilina demostró inicialmente una alta efectividad contra las infecciones estafilocócicas; pero, cepas de S. aureus productoras de penicilinasa emergieron a mediados de la década de 1940 y su prevalencia se incrementó dramáticamente en unos pocos años (5). Para afrontar este problema, en 1959, fue liberado el β-lactámico semisintético meticilina. Sin embargo, en 1961, se detectó la emergencia de S. aureus resistente a meticilina (SARM) (6). El mecanismo de resistencia a meticilina desarrollado por S. aureus se basa en la producción de una proteína de unión a la penicilina (PUP) adicional, la PUP2a, la cual es completamente funcional y no tiene afinidad por los antibióticos β-lactámicos (7). Esta proteína es codificada por el gen meca (8), que está localizado en un elemento genético móvil denominado casete cromosómico estafilocócico mec (SCCmec) (9).
Desde entonces, la prevalencia de SARM se ha incrementado (10-14). En los Estados Unidos se indica que en las unidades de cuidados intensivos el 59,5% de los S. aureus aislados son SARM (15) y que la tasa de mortalidad asociada con las infecciones por SARM invasivas es de aproximadamente 20% (5). Así, claramente se confirma la importancia de este patógeno, que se ha convertido en un problema mayor para los centros de asistencia médica.
En los últimos años, un cambio sorprendente en la epidemiología de SARM ha ocurrido con la emergencia en diversas partes del mundo de SARM asociado a la comunidad (SARMAC) (16-20), con el agravante de que estos SARM-AC pueden ocasionar infecciones severas o fatales (21).
En la presente revisión, brevemente, se aborda aspectos epidemiológicos y moleculares de SARM-AC, con el fin de informar al lector sobre este patógeno emergente.
EPIDEMIOLOGÍA
En el año 2000, los Centros de Control y Prevención de las Enfermedades (CDC) definieron una infección por SARM-AC como el aislamiento de una cepa SARM en un paciente externo o dentro de las 48 horas de hospitalización sin que existieran los factores de riesgo de los SARM nosocomiales: hemodiálisis, cirugía, residencia en centros de cuidados de largo plazo, hospitalización durante el año previo, presencia de un catéter intravenoso permanente, dispositivos transcutáneos o un aislamiento previo de SARM en el paciente (22). Todas las otras infecciones por SARM deben ser consideradas como ocasionadas por el SARM asociado a la asistencia médica (SARM-AH) (23).
El primer caso auténtico de SARMAC fue relatado en poblaciones indígenas, sin hospitalización previa, que habitaban en comunidades alejadas en Australia, a inicios de la década de 1990 (24). Posteriormente, en este mismo país, otros estudios han comunicado la presencia de SARM-AC (25,26), indicándose, además, que la frecuencia de estos aislados se ha incrementado de 4,7% en el 2000 a 7,3% en el 2004 (27).
En los Estados Unidos, el año de 1999 se notificó 4 casos de SARMAC en niños residentes en Minnesota y North Dakota, los cuales fallecieron, acontecimiento que reveló la potencial severidad de las infecciones por SARMAC (8). Seguidamente, ha sido referida la amplia presencia de SARM-AC en el territorio de la unión americana. En Nueva York, SARM-AC representó el 21,3% de todos los aislados SARM recuperados durante 3 meses en 15 hospitales participantes, resaltándose que en la región de más alta prevalencia hubo 4,6 casos por 100 000 habitantes, afectando principalmente a la población de las etnias negra e hispana, al sexo femenino y a los menores de 18 años (29). En Carolina del Norte, un estudio retrospectivo de 7 meses y medio en un centro de cuidados terciarios en pacientes menores de 18 años identificó como SARM-AC al 76% de los SARM aislados y resaltó que la frecuencia de SARM-AC entre la etnia negra fue mayor que la etnia blanca (OR=1,09) (30). En Hawái, un estudio retrospectivo entre 2001 y 2003 en 4 instituciones de asistencia médica concluyó que SARMAC representó el 28% de todos los aislados SARM, indicándose, además, que los residentes nativos hawaianos y con ancestros polinesios, micronesios y melanesios fueron los más afectados (31). En Rhode Island, un análisis retrospectivo entre 1997 y 2001 en un hospital de tercer nivel verificó que 40% de los aislados SARM en pacientes pediátricos fueron SARM-AC y que su frecuencia gradualmente se incrementó durante el período de investigación (32). Asimismo, en estados como Kansas (33), Alaska (34), Alabama (35), Ohio (36) y California (37) también ha sido registrada la presencia de SARM-AC.
En el continente europeo se ha constatado la diseminación de SARMAC. En España, un estudio prospectivo de un año en niños menores de 15 años atendidos en el departamento de emergencia de un hospital universitario determinó que el 13% de los participantes en la investigación presentaron SARM-AC (38). En Portugal, un amplio seguimiento de niños menores de 6 años en los que se aisló SARM durante los tres primeros meses de 2006, 2007 y 2009 encontró que <1% eran SARMAC, sugiriéndose que este grupo etario puede ser excluido como reservorio de tales aislados en la región estudiada (39). En Alemania se indicó la presencia de 5 casos de SARM-AC en un centro de solicitantes de asilo, recomendándose el uso de protocolos de descolonización para limitar su diseminación (40). En Suecia, una amplia vigilancia prospectiva en 2 hospitales, 35 clínicas de atención primaria y 2 locales para pacientes externos señaló que, entre 2004 y 2006, se aisló 58 casos por SARM-AC y que de estos 22 fueron infectados domiciliariamente y 36 fueron adquiridos en el exterior (41). En Finlandia, un estudio retrospectivo informó que entre 1997 y 1999 el 21% de los aislados SARM fueron SARM-AC y que la cuarta parte de estos provenían de niños menores de 15 años (42). En Francia, entre 1999 y 2003, un estudio prospectivo manifestó el incremento de SARM-AC de 0% a 6,8% en pacientes con edades de 3 meses a 100 años (43).
Entre los países asiáticos también existen datos de la presencia de SARMAC. En la China, en un hospital universitario se encontró que, en el período de un año, 1,5% de todos los aislados de S. aureus correspondió a SARM-AC (44). En Filipinas, en un centro médico renal terciario SARM-AC representó el 43% de los SARM aislados durante el año de estudio (45). En la India, un estudio prospectivo de 7 meses determinó que 28% de los SARM aislados fueron SARM-AC (46). En Camboya, en un hospital pediátrico se aisló 17 casos por SARM-AC en pacientes con edades de 8 meses a 14 años, entre 2006 y 2007 (47).
Algunos países africanos también han indicado la presencia de SARMAC. En Egipto, un estudio en adictos a drogas aisló SARM-AC en 51% de los participantes, informándose además, que a mayor tiempo de adicción mayor colonización por SARM-AC (48). En Costa de Marfil, un estudio prospectivo de corte transversal en escolares de 5 a 15 años en un período de 9 meses mostró que 14,8% de los aislados de S. aureus correspondía a SARM-AC (49); en Tunisia, un estudio retrospectivo en una unidad de cuidados intensivos pediátrica que abarcó de 2000 a 2009 encontró que SARM-AC representó el 14% de todos los S. aureus aislados (50).
Otros países de América han encontrado la emergencia de SARM-AC. En Canadá, un estudio prospectivo de 1999 al 2001 concluyó que SARM-AC representó el 60% de los SARM aislados (51). En Brasil, un análisis en individuos no hospitalizados encontró la presencia de SARM-AC en 7,5% de todos los aislados de S. aureus (52). En Argentina se informó que 42% de las infecciones por SARM en pacientes pediátricos entre 2004 y 2005 fueron por SARM-AC y que los infectados presentaban un alto índice de aglomeración en sus domicilios (53). En Chile se diagnosticó cinco casos que tuvieron como causante a SARM-AC, cuatro de los cuales probablemente fueron adquiridos en el extranjero (54). En Colombia se ha informado 12 casos de SARMAC en pacientes adultos y pediátricos, con una edad promedio de 32 años (55). En Uruguay, una investigación entre 2002 y 2004 en una institución de asistencia médica informó que los aislados SARM-AC se incrementaron de 4 a 23% durante el período de análisis (56). En el Perú, un estudio descriptivo multicéntrico entre 2005 y 2006 halló que 5,6% del total de aislados SARM fueron SARM-AC (57) y, en 2011, se ha publicado 3 casos más de pacientes infectados con SARM-AC (58).
Con respecto a los factores de riesgo para la adquisición de SARM-AC, estos han sido poco explorados. Algunos de los referidos en la literatura son: edad joven, ciertos grupos étnicos, aglomeración domiciliar, internos de centros penitenciarios, usuarios de drogas intravenosas, hombres que tienen sexo con hombres, atletas que practican deportes de contacto, personal militar, personas expuestas a uso frecuente de antibióticos y personas expuestas a un contacto directo con SARM (22,59,60,61).
CARACTERÍSTICAS MOLECULARES
Los SARM contienen el elemento SC-Cmec (10). SCCmec es un elemento genético móvil (21-67 kb) que se integra en el cromosoma de SARM en un sitio único (attBscc) localizado cerca al origen de replicación de S. aureus (62). El attBscc se encuentra ubicado al final del extremo 3´ de un marco de lectura abierta (ORF) de función desconocida, designado orfX (63). SCCmec transporta a los genes mecA, mecI (represor) y mecR1 (transductor de señal); estos dos últimos regulan la expresión de meca (64,65). Además, SCCmec transporta al complejo del gen ccr (ccrAB y ccrC), responsable por la movilidad de SCCmec en el cromosoma de S. aureus mediante eventos de integración y escisión (8,66).
La organización genética de los componentes adyacentes al mecA define al complejo del gen mec (10). Se ha definido tres principales clases de complejos mec: la clase A contiene al complejo mec completo (mecI-mecR1-mecA), las clases B y C tienen a los genes reguladores de mecA interrumpidos debido a la presencia de secuencias de inserción, ΨIS1272-ΔmecR1-mecA e IS431ΔmecR1-mecA, respectivamente (67).
Los tipos de SCCmec son definidos por la combinación del complejo del gen mec con el complejo del gen ccr (66). A la fecha, ocho tipos de SCCmec, designados del I al VIII, han sido descritos (63,68-75). Los genes ccrAB están presentes en SCCmec I-IV, VI y VIII, y el gen ccrC en SCCmec V y VII (76). Los SCCmec I, IV, V, VI y VII codifican exclusivamente resistencia a los β-lactámicos, en tanto que los SCCmec II, III y VIII contienen genes adicionales que les confieren resistencia a múltiples clases de antibióticos diferentes de los β-lactámicos, así como a metales pesados; esta resistencia es mediada por plásmidos (pUB110, pT181, pI258) y transposones (Tn554, ΨTn554) que se encuentran en las regiones J (joining), las cuales no son parte de los complejos mec y ccr (68,73,75).
Los SCCmec I, II y III son predominantemente encontrados en SARMAH (68,77,78), mientras que los SCCmec IV y V son principalmente observados en los SARM-AC (79-81) circulantes en el mundo entero. Los SCCmec IV y V son elementos más pequeños y mucho más móviles que los SCCmec I-III, lo que parece ser esencial para la emergencia y diseminación del SARM-AC (figura 1) (60,82).
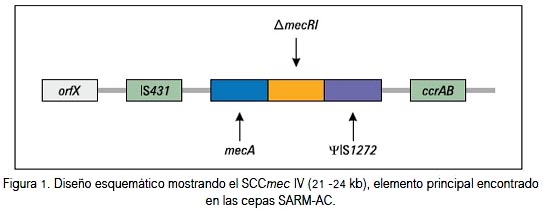
Frecuentemente, los SARM-AC presentan genes que codifican para la producción de la leucocidina de Panton-Valentine (LPV) (83-86), la cual puede ser en parte responsable de la incrementada virulencia de SARM AC (87-89). La LPV es codificada por lukF-PV y lukS-PV, dos genes contiguos y cotranscritos, los cuales producen proteínas de 32 y 38 kDa, respectivamente (90). Estos genes están localizados en cuatro fagos diferentes: φ108PVL, φPVL, φSLT y φSa2mw (90).
Actualmente, cinco clonas de SARM-AC se encuentran circulando mundialmente: ST1-IV (USA400), ST8-IV (USA300), ST30-IV (Pacífico/ Oceanía), ST59-IV y V (USA1000, Taiwán) y ST80 (Europea) (17,91). La clona ST8-IV es encontrada ampliamente en Estados Unidos (92-96) y una variante de esta se ha establecido en la región septentrional de Sudamérica (97). La clona ST80 es la más común en Europa (98-102) y esta puede haber surgido en el mediterráneo, oriente medio o el norte de África, ya que, muchos de los primeros infectados tuvieron historias de viajes a dichas regiones (103).
En cuanto al origen de las cepas SARM-AC, se debate dos posibilidades. La primera es que estas sean descendientes silvestres de cepas SARM AH(104); los estudios comparativos entre estos dos tipos de cepas son divergentes, ya que, han encontrado tanto similitud (105,106) como diferencias (107,108) entre las mismas. La segunda, es que, las cepas SARM-AC surgieron como una consecuencia de la transferencia horizontal del determinante de la resistencia a meticilina a un antecesor inicialmente susceptible (104); un estudio registró una alta prevalencia de SCCmec IV en cepas SARM-AC con diversos antecedentes genéticos, lo que sugeriría que este elemento se puede diseminar de una manera promiscua entre el S. aureus (109). Otro halló similitud entre cepas SARM-AC y cepas S. aureus sensible a meticilina asociado a la comunidad (SASM-AC), con la única diferencia de que en los SARM-AC se encontraba el SCCmec IV, infiriéndose que el SARM-AC surgió probablemente de la inserción de este elemento en un SASM-AC (110). Por otra parte, recientemente se confirmó que la clona de S. aureus resistente a la penicilina PT80/81, la cual estuvo ampliamente diseminada durante los años de 1950, es positiva para la LPV y que, entre las clonas actuales de SARM-AC, la conocida como ST30-IV pertenece al linaje de esta PT80/81. Así, los descendientes de la PT80/81 habrían adquirido resistencia a meticilina (111) y estarían re-emergiendo en el mundo entero como sus ancestros lo hicieron en la década de 1950 (112).
En conclusión, indudablemente que S. aureus demuestra una versatilidad genómica excepcional. A través de varias décadas se ha adaptado a la diferente terapéutica enviada por la industria farmacéutica. Una prueba más de la plasticidad de su genoma es su actual diseminación al ambiente comunitario. La presencia de SARM-AC se encuentra ampliamente diseminada en el orbe, llegando a constituirse en un preocupante problema de salud pública. Se hace necesario entender la epidemiología de SARM-AC para poder implementar medidas de control adecuadas. Aumentar tecnologías para el estudio molecular del SARM-AC nos permitirá conocer su evolución. Según Chambers y de Leo (75), esta sería la cuarta ola de resistencia a los antibióticos de S. aureus, tal vez, la de mayor desafío para médicos y microbiólogos involucrados en una labor clínica diaria.
REFERENCIAS BIBLIOGRÁFICAS
1. Lowy FD. Staphylococcus aureus infections. n Eng J Med. 1998;339:520-32.
2. Ardura MI. Staphylococcus aureus: old bugwith new tricks [editorial]. Rev Chil Infect. 2009;26:401-5.
3. Echevarría J, Iglesias D. Estafilococo meticilino resistente, un problema actual en la emergencia de resistencia entre los Gram positivos. Rev Med Hered. 2003;14:195-203.
4. Gelatti LC, Bonamigo RR, Becker AP, d´Azevedo PA. Staphylococcus aureus resistentes à meticilina: disseminação emergente na comunidade. An Bras Dermatol. 2009;84:501-6.
5. De Leo FR, Chambers HF. Reemergence of antibiotic-resistant Staphylococcus aureus in the genomics era. J Clin Invest. 2009;119:2464-74.
6. Jevons MP. Celbenin"-resistant staphylococci. Br Med J. 1961;1:124-5.
7. Hartman BJ, Tomasz A. Low-affinity penicillin binding protein associated with beta-lactam resistance in Staphylococcus aureus. J Bacteriol. 1984;158:513-6.
8. Song MD, Wachi M, Doi M, Ishino F, Matsuhashi M. Evolution of an inducible penicillin-target protein in methicillin-resistant Staphylococcus aureus by gene fusion. FEBS Lett. 1987;221:167-71.
9. Katayama y, Ito T, Hiramatsu K. A new class of genetic element, staphylococcus cassettechromosome mec, encodes methicillin resistance in Staphylococcus aureus. Antimicrob Agents Chemother. 2000;44:1549-55.
10. Simões RR, Aires de Sousa M, Conceição T, Antunes F, da Costa PM, de Lencastre H. High prevalence of EMRSA-15 in portuguese public buses: a worrisome finding. PLoS One. 2001;6(3):e17630.
11. Velázquez-Meza ME, Aires de Sousa M, Echaniz AG, Solórzano SF, Miranda nG, Silva SJ, de Lencastre H. Surveillance of methicillin-resistant Staphylococcus aureus in a pediatric hospital in Mexico city a 7 year period (1997 to 2003): clonal evolution and impact of infection control. J Clin Microbiol. 2004;42:3877-80.
12. Luján DA. Evaluación de Staphylococcus aureus multirresistente en pacientes hospitalizados en el Instituto de Enfermedades neoplásicas. Rev Per Enferm Infecc Trop. 2003;2:10-3.
13. Mamani E, Luján DA, Pajuelo GR. Perfil de sensibilidad y resistencia de Staphylococcus aureus. Experiencia en el Hospital nacional Hipólito Unanue. An Fac med. 2006;67:120-4.
14. Seas C, Hernandez K, Ramos R, Bazan E, Rodriguez I, Torres A, Zamudio C, Gotuzzo E. oxacillinresistant and multidrug-resistant Staphylococcus aureus in Lima, Peru. Infect Control Hosp Epidemiol. 2006;27:198-200.
15. Shinefield HR, Ruff nL. Staphylococcal infections: a historical perspective. Infect Dis Clin n Am.2009;23:1-15.
16. Groom AV, Wolsey DH, naimi TS, Smith K, JohnsonS, Boxrud D, Moore KA, Cheek JE. Communityacquired methicillin-resistant Staphylococcus aureus in a rural American Indian community. J Am Med Assoc. 2001;286:1201-5.
17. Tattevin P. Les infections à Staphylococcus aureus résistant à la méticilline (SARM) d´ acquisition communautaire. Med Mal Infect. 2011;41:167-75.
18. Broseta A, Chaves F, Rojo P, otero JR. Emergencia de un clon de Staphylococcus aureus resistente a meticilina de origen comunitário en la población pediátrica del sur de Madrid. Enferm Infecc Microbiol Clin. 2006;24:31–5.
19. Kulkarni GB, Pal PK, Veena-Kumari HB, Goyal M,Kovoor JME, nadig S, Arakere G. Communityacquired methicillin-resistant Staphylococcus aureus pyomyositis with myelitis: A rare ocurrence with diverse presentation. neurol India. 2009;57:653-6.
20. nazareth R, Gonçalves-Pereira J, Tavares A,Miragaia M, de Lencastre H, Silvestre J, Freitas P, Gonçalves E, Martins F, Mendes V, Tapadinhas C, Povoa P. Infeção por Staphylococcus aureus meticilina-resistente da comunidade em Portugal. Rev Port Penumol. 2012;18:34-8.
21. García C. Staphylococcus aureus meticilino resistente adquirido en la comunidad. Acta Med Per. 2011;28:159-62.
22. File TM. Impact of community-acquired methicillinresistant Staphylococcus aureus in the hospital setting. Cleveland Clin J Med. 2007;74(Suppl. IV):S6-S11.
23. David MZ, Daum RS. Community-Associated Me-thicillin-Resistant Staphylococcus aureus: Epidemiology and clinical consequences of an emerging epidemic. Clin Microbiol Rev. 2010;23:616-87.
24. Udo EE, Pearman JW, Grubb WB. Genetic analysis of community isolates of methicillin-resistant Staphylococcus aureus in Western Australia. J Hosp Infect. 1993;25:97-108.
25. Munckhof WJ, Krishnan A, Kruger P, Looke D.Cavernous sinus thrombosis and meningitis from community-acquired methicillin-resistant Staphylococcus aureus infection. Intern Med J. 2008;38:283–7.
26. Nimmo GR, Playford EG. Community-acquired MRSA bacteraemia: four aditional cases including one associated with severe pneumonia. Med J Aus. 2003;178:245.
27. nimmo GR, Coombs GW. Community-associated methicillin-resistant Staphylococcus aureus (MRSA) in Australia. Int J Antimicrob Agents.2008;31:401-10.
28. CDC. Four pediatric deaths from communityacquired methicillin-resistant Staphylococcus aureus - Minnesota and North Dakota, 1997-1999. MMWR. 1999;48:707-10.
29. Bratu S, Landman D, Gupta J, Trehan M, Panwar M,Quale J. A population-based study examining theemergence of community-associated methicillinresistant Staphylococcus aureus USA300 in new york City. Ann Clin Microbiol Antimicrob. 2006;5:29.
30. Shapiro A, Raman S, Johnson M, Piehl M. Community-acquired MRSA infections in North Carolinachildren: prevalence, antibiotic sensitivities, and risk factors. NC Med J. 2009;70:102-7.
31. CDC. Community-associated methicillin-resistant Staphylococcus aureus infections in Pacific Islanders - Hawaii, 2001-2003. MMWR. 2004;53:767-70.
32. Dietrich DW, Auld DB, Mermel LA. Communityacquired methicillin-resistant Staphylococcus aureus in southern new England children. Pediatrics. 2004;113:347-52.
33. El Fakih Ro, Moore TA, Assi MA. Sinusitis and orbital cellulitis due to community-associatedmethicillin-resistant Staphylococcus aureus. KS J Med. 2008;1:85-8.
34. Baggett HC, Hennessy TW, Rudolph K, Bruden D, Reasonover A, Parkinson A, Sparks R, Donlan RM, Martínez P, Mongkolrattanothai K, Butler JC. Community-onset methicillin-resistant Staphylococcus aureus associated with antibiotic use and the cytotoxin Panton-Valentine leukocidin during a furunculosis outbreak in rural Alaska. J Infect Dis. 2004;189:1565–73.
35. Hasty M, Klasner A, Kness S, Denmark TK, Ellis D, Herman MI, Brown L. Cutaneous communityassociated methicillin-resistant Staphylococcus aureus among all skin and soft-tissue infections in two geographically distant pediatric emergencydepartments. Acad Emerg Med. 2007;14:35–40.
36. Woods SE, Beiter E, Drake B, Engel A. Theprevalence of asymptomatic methicillin-resistant Staphylococcus aureus in school-age children. East J Med. 2011;16:18-21.
37. Outbreaks of community- associated methicillin-resistant Staphylococcus aureus skin infections-Los Angeles County, California, 2002-2003. MMWR. 2003;52:88.
38. Daskalaki M, Rojo P, Marin-Ferrer M, Barrios M, Otero JR, Chaves F. Panton-Valentine leukocidinpositive Staphylococcus aureus skin and soft tissue infections among children in an emergency department in Madrid, Spain. Clin Microbiol Infect. 2010;16:74–7.
39. Tavares DA, Sá-Leão R, Miragaia M, de Lencastre H. Large screening of CA-MRSA among Staphylococcus aureus colonizing healthy young children living in two areas (urban and rural) of Portugal. BMC Infect Dis. 2010;10:110.
40. Dudareva S, Barth A, Paeth K, Krenz-Weinreich A, Layer F, Deleré Y, Eckmanns T. Cases of community-acquired methicillin-resistant Staphylococcus aureus in an asylum seekers center in Germany, November 2010. Euro Surveill. 2011;16(4):pii=19777.
41. Christenson B, Ardung B, Sylvan S. Methicillinresistant Staphylococcus aureus infections in Uppsala county, Sweden. Open Infect Dis J. 2011;5:107-14.
42. Salmenlinna S, Lyytikäinen O, Vuopio-Varkila J. Community-acquired methicillin-resistant Staphylococcus aureus, Finland. Emerg Infect Dis. 2002;8:602-7.
43. Del Giudice, Blanc V, Durupt F, Bes M, Martinez JP, Counillon E, Lina G, Vandenesch F, Etienne J. Emergence of two population of methicillin-resistant Staphylococcus aureus with distinct epidemiological clinical and biological features, isolated from patients with community-acquired skin infections. Br J Dermatol. 2006;154:118–24.
44. Ma XL, Chen FU, Zhou X, Chang WJ, Daí YY. Molecular characteristics of Staphylococcus aureus isolates in a Chinese teaching hospital. Afr J Microbiol Res. 2011;5:2969-74.
45. Arakama MH, Mendoza M, Fernandez P, Belmonte L, Galapia Y, Pile M. Emergence of methicillinresistant Staphylococcus aureus among patients in a tertiary renal medical center. Phil J Microbiol Infect Dis. 2010;39:28-33.
46. Krishna BVS, Patil AB, Chandrasekhar MR. Community-acquired methicillin-resistant Staphylococcus aureus infections in a South Indian city. Southeast Asian J Trop Med Public Health. 2004;35:371-4.
47. Chheng K, Tarquinio S, Wuthiekanun V, Sin L, Thaipadungpanit J, Amornchai P, Chanpheaktra N, Tumapa S, Putchhat H, Day NPJ, Peacock SJ. Emergence of community-associated methicillinresistant Staphylococcus aureus associated with pediatric infection in Cambodia. PLoS One. 2009;4(8):e6630.
48. El Sharif A, Ashour HM. Community-acquired methicillin-resistant Staphylococcus aureus (CAMRSA) colonization and infection in intravenous and inhalational opiate drug abusers. Exp Biol Med. 2008;233:874-80.
49. Kacou-Ndouba A, Kazali A, Koffi KS, Ekaza E, Kouablan A, Kangah T, Okpo S, Elogne-Kouamé C, Dosso M. Community-acquired skin infections in children in Abidjan: methicillin-resistant Staphylococcus aureus and exfoliative toxin production. J Microbiol Antimicrob. 2011;3:201-5.
50. Menif K, Bouziri A, Khaldi A, Hamdi A, Belhadj S, Borgi A, Fitouri Z, Ben Jaballah N. Communityassociated methicillin-resistant Staphylococcus aureus infections in a pediatric intensive care unit. J Infect Dev Ctries. 2011;5:587-91.
51. Campbell AL, Bryant KA, Stover B, Marshall GS. Epidemiology of methicillin-resistant Staphylococcus aureus at a childrens hospital. Infect Control Hosp Epidemiol. 2003;24:427–30.
52. Menegotto FR, Picoli SU. Staphylococcus aureus oxacilina resistente (MRSA): incidência de cepas adquiridas na comunidade (CA-MRSA) importância da pesquisa e descolonização em hospital. Rev Bras Anal Clin. 2007;39:147-50.
53. Paganini H, Verdaguer V, Rodríguez AC, Latta PD, Hernández C, Berberian G, Pinheiro JL, Rosanova MT. Infecciones causadas por Staphylococcus aureus resistentes a la meticilina en niños provenientes de la comunidad en niños de la Argentina. Arch Argent Pediatr. 2006;104:293-8.
54. Noriega LM, González P, Hormazábal JC, Pinto C, Canals M, Munita JM, Thompson L, Marcotti A, Pérez J, Ibáñez D, Araya C, Canals C, Vial P. Staphylococcus aureus communitario resistente a cloxacilina: comunicación de los primeros cinco casos descritos en Chile. Rev Med Chile. 2008;136:885–91.
55. Pérez N, Baquero HG, Rojas SP, Torres HM, Forero LC, Gutiérrez FM, García MR, Bustos JA, Abello RF Gómez, Castell SF, Moreno AM, Arango GK, Sánchez L, Pavas NC, Briceño JM, Rodríguez EI. Staphylococcus aureus resistente a la meticilina asociado a la comunidad en la Orinoquía colombiana; reporte de casos. Acta Col Cuidado Intensivo. 2010;10:181-91.
56. Benoit SR, Estivariz C, Mogdasy C, Pedreira W, Galiana A, Bagnulo H, Gorwitz R, Fosheim GE, McDougal LK, Jernigan D. Community strains of methicillin-resistant Staphylococcus aureus as potential cause of healthcare-associated infections, Uruguay 2002–2004. Emerg Infect Dis. 2008;14:1216–23.
57. Tamariz J, Agapito J, Horna G, Tapia E, Vicente W, Silva M, Zerpa R, Guerra H. Staphylococcus aureus resistente a meticilina adquirido en la comunidad aislado en tres hospitales de Lima-Perú. Rev Med Hered. 2010;21:4-10.
58. García C, Deplano A, Denis O, León M, Siu H, Chincha O, Samalvides F, Jacobs J. Spread of community- associated methicillin-resistant Staphylococcus aureus to Peru. J Infect. 2011;63:482-3.
59. Drews TD, Temte JL, Fox BC. Community-associated methicillin-resistant Staphylococcus aureus: review of an emerging public health concern. Wis Med J. 2006;105:52-7.
60. Echevarría J. Estado actual de la resistencia bacteriana. Diagnóstico. 2008;47:164-74.
61. Scheurich D, Woeltje K. Skin and soft tissue infections due to CA-MRSA. Mo Med. 2009;106:274-6.
62. Kuroda M, Ohta T, Uchiyama I, Baba T, Yuzawa H, Kobayashi I, Cui L, Oguchi A, Aoki K, Nagai Y. Whole genome sequencing of methicillin-resistant Staphylococcus aureus. Lancet. 2001;357:1225- 40.
63. Ito T, Katayama Y, Hiramatsu K. Cloning and nucleotide sequence determination of the entire mec DNA of pre-methicillin resistant Staphylococcus aureus N315. Antimicrob Agents Chemother. 1999;43:1449-58.
64. Hiramatsu K, Asada K, Suzuki E, Okonogi K, Yokota T. Molecular cloning and nucleotide sequence determination of the regulator region of mecA gene in methicillin-resistant Staphylococcus aureus (MRSA). FEBS Lett. 1992;298:133-6.
65. Matsuhashi M, Song MD, Ishimoto F, Wachi M, Doi M, Inoue M, Ubukata K, Yamashita N, Konno M. Molecular cloning of the gene of a penicillinbinding protein supposed to cause high resistance to β-lactam antibiotics in Staphylococcus aureus. J Bacteriol. 1986;167:975-80.
66. Castellano MJ, Perozo-Mena A. Mecanismos de resistencia a antibióticos β-lactámicos en Staphylococcus aureus. Kasmera. 2010;38:18-35.
67. De Lencastre, Oliveira D, Tomasz A. Antibiotic resistant Staphylococcus aureus: a paradigm of adaptive power. Curr Op Microbiol. 2007;10:428- 35.
68. Ito T, Katayama Y, Asada K, Mori N, Tsutsumimoto K, Tiensasitorn C, Hiramatsu K. Structural comparison of three types of staphylococcal cassette chromosome mec integrated in the chromosome in methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2001;45:1323-36.
69. Ma XX, Ito T, Tiensasitorn T, Jamklang M, Chongtrakool P, Boyle-Vavra S, Daum RS, Hiramatsu K. Novel type of staphylococcal cassette chromosome mec identified in community-acquired methicillin-resistant Staphylococcus aureus strains. Antimicrob Agents Chemother. 2002;46:1147-52.
70. Ito T, Ma XX, Takeuchi F, Okuma K, Yuzawa H, Hiramatsu K. Novel type V staphylococcal cassette chromosome mec driven by a novel cassette chromosome recombinase ccrC. Antimicrob Agents Chemother. 2004;48:2637-51.
71. Oliveira DC, Milheirico C, de Lencastre H. Redefining a structural variant of staphylococcal cassette chromosome mec, SCCmec type VI. Antimicrob Agents Chemother. 2006;50:3457-9.
72. Berglund C, Ito T, Ikeda M, Ma XX, Söderquist B, Hiramatsu K. Novel type of staphylococcal cassette chromosome mec in a methicillin-resistant Staphylococcus aureus strain isolated in Sweden. Antimicrob Agents Chemother. 2008;52:3512–6.
73. Zhang K, McClure JA, Elsayed S, Conly JM. Novel staphylococcal cassette chromosome mec type, tentatively designated type VIII, harboring class A mec and type 4 ccr gene complexes in a Canadian epidemic strain of methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2009;53:531–40.
74. Chongtrakool P, Ito T, Ma XX, Kondo Y, Trakulsomboon S, Tiensasitorn C, Jamklang M, Chavalit T, Song JH, Hiramatsu K. Staphylococcal cassette chromosome mec (SCCmec) typing of methicillinresistant Staphylococcus aureus strains isolated in 11 Asian countries: a proposal for a new nomenclature for SCCmec elements. Antimicrob Agent Chemother. 2006;50:1001–12.
75. International Working Group on the Classification of Staphylococcal Cassette Chromosome Elements (IWG-SCC). Classification of Staphylococcal Cassette Chromosome mec (SCCmec): Guidelines for Reporting Novel SCCmec Elements. Antimicrob Agents Chemother. 2009;53:4961-7.
76. Chambers HF, de Leo FR. Waves of resistance: Staphylococcus aureus in the antibiotic era. Nat Rev Microbiol. 2009;7:629-41.
77. Holden MTG, Feil EJ, Lindsay JA, Peacock SJ, Day NPJ, Enright MC, Foster TJ, Moore CE, Hurst L, Atkin R, Barron A, Bason N, Bentley SD, Chillingworth C, Chillingworth T, Churcher C, Clark L, Corton C, Cronin A, Doggett J, Dowd L, Feltwell T, Hance Z, Harris B,Hauser H, Holroyd S, Jagels K, James KD, Lennard N, Line A, Mayes R, Moule S, Mungall K, O rmond D, Quail MA, Rabbinowitsch E,Rutherford K, Sanders M, Sharp S, Simmonds M, Stevens K, Whitehead S, Barrell BG, Spratt BG, Parkhill J. Complete genomes of two clinical Staphylococcus aureus strains: evidence for the rapid evolution of virulence and drug resistance. PNAS. 2004;101:9786-91.
78. Reiter KC, Machado ABMP, de Freitas ALP, Barth AF. High prevalence of methicillin-resistant Staphylococcus aureus with SCCmec type III in cystic fibrosis patients in southern, Brazil. Rev Soc Bras Med Trop. 2010;43:377-81.
79. Palombarani S, Gardella n, Tuduri A, Figueroa S, Sly G, Corazza R, Gutkind G, Almuzara M, Mollerach M. Infecciones adquiridas en la comunidad por Staphylococcus aureus resistente a meticilina en un hospital de agudos. Rev Arg Microbiol.2007;39:151-5.
80. O´Brien FG, Coombs GW, Pearson JC, Christiansen KJ, Grubb WB. Type V staphylococcal cassettechromosome mec in community staphylococcifrom Australia. Antimicrob Agents Chemother.2005;49:5129-32.
81. Wong VWy, Cheung yS, Wong J, Lee KF, LaiPBS. A community-acquired methicillin-resistant Staphylococcus aureus liver abscess. Hong Kong Med J. 2010;16:227-9.
82. Amatsu K, Katayama Y, Yuzawa H, Ito T. Molecular genetics of methicillin-resistant Staphylococcus aureus. Int J Med Microbiol. 2002;292:67-74.
83. Ton PN, Valentine FCo. Staphylococcal toxin. Lancet. 1932;219:506–8.
84. Vandenesch F, Naimi T, Enright MC, Lina G, Nimmo GR, Heffernan H, Liassine N, Bes M, Greenland T, Reverdy ME, Etienne J. Community-acquiredmethicillin-resistant Staphylococcus aureus carrying Panton-Valentin leukocidin genes: worldwideemergence. Emerg Infect Dis. 2003;9:978-84.
85. Main CL, Jayaratne P, Haley A, Rutherford C, Smaill F, Fisman DN. Outbreaks of infection caused by community-acquired methicillin-resistant Staphylococcus aureus in a Canadian correctional facility. Can J Infect Dis Med Microbiol. 2005;16:343-8.
86. Harbarth S, François P, Schrenzel J, Frankhouser-Rodriguez C, Hugonnet S, Koessler T, Huyghe A, Pittet D. Community-associated methicillin-resistant Staphylococcus aureus, Switzerland. Emerg Infect Dis. 2005;11:962-5.
87. Gillet y, Issartel B, Vanhems P, Fournet JC, Lina G, Bes M, Vandenesch F, Piémont y, Brousse N, Floret D, Etienne J. Association between Staphylococcus aureus strains carrying gene for Panton-Valentine leukocidin and highly lethal necrotising pneumonia in young immunocompetent patients. Lancet. 2002;359:753–9.
88. Labandeira-Rey M, Couzon F, Boisset S, Brown El, Bes L, Benito Y, Barbu EM, Vazquez V, Höök M, Etienne J, Vandenesch F, Bowden MG.Staphylococcus aureus Panton-Valentine leukocidin causes necrotizing pneumonia. Science. 2007;315:1130–3.
89. Genestier AL, Michallet MC, Prévost G, Bellot G, Chalabreysse L, Peyrol S, Thivolet F, Etienne J, Lina G, Vallette FM, Vandenesch F, Genestier L. Staphylococcus aureus Panton-Valentin leucocidin directly targets mitochondria and induces Baxindependent apoptosis of human neutrophils. J Clin Invest. 2005;115:3117-27.
90. Deurenberg RH, Stobberingh EE. The evolution of Staphylococcus aureus. Infect Genet Evol. 2008;8:747-63.
91. Skov RL, Jensen KS. Community-associatedmethicillin-resistant Staphylococcus aureus as a cause of hospital-acquired infections. J Hosp Infect. 2009;73:364-70.
92. Shurland SM, Stine OC, Venezia RA, Johnson JK, Zhan M, Furuno JP, Miller RR, Johnson T, Roghmann MC. Colonization sites of USA300 methicillin-resistant Staphylococcus aureus in residents of extended care facilities. Infect Control Hosp Epidemiol. 2009;30:313-8.
93. Kourbatova EV, Halvosa JS, King MD, Ray SM, White N, Blumberg HM. Emergence of community associated methicillin-resistant Staphylococcus aureus USA300 clone as a cause of health care associated infections among patients with prosthetic joint infections. Am J Infect Control. 2005;33:385–91
94. King MD, Humphrey BJ, Wang YF, Kourbatova EV, Ray SM, Blumberg HM. Emergence of community acquired methicillin-resistant Staphylococcus aureus USA 300 clone as the predominant cause of skin and soft-tissue infections. Ann Intern Med. 2006;144:309-17
95. Diep BA, Chambers HF, Graber CJ, Szumowski JD, Miller LG, Han LL, Chen JH, Lin F, Lin J, Phan TH, Carleton HA, McDougal LK, Tenover FC, Cohen DE, Mayer KH, Sensabaugh GF, Perdreau-Remington F. Emergence of multidrug-resistant, community associated, methicillin-resistant Staphylococcus aureus clone USA300 in men who have sex with men. Ann Intern Med. 2008;148:249-57.
96. Roberts JC, Krueger RL, Peak KK, Veguilla W, Cannons AC, Amuso PT, Cattani J. Community associated methicillin-resistant Staphylococcus aureus epidemic clone USA300 in isolates from Florida and Washington. J Clin Microbiol. 2006;44:225-6.
97. Reyes J, Rincón S, Díaz L, Panesso D, Contreras GA, Zurita J, Carrillo C, Rizzi A, Guzmán M, AdachiJ, Chowdhury S, Murray BE, Arias CA. Dissemination of methicillin-resistant Staphylococcus aureus USA300 sequence type 8 lineage in Latin America. Clin Infect Dis. 2009;49:1861-7.
98. Robert J, Etienne J, Bertrand X. Methicillin-resistant Staphylococcus aureus producing Panton-Valentine leukocidin in a retrospective case series from 12 French hospital laboratories, 2000–2003. Clin Microbiol Infect. 2005;11:585–7.
99. Köck R, Mellmann A, Schaumburg F, Friedrich AW, Kipp F, Becker K. The epidemiology of methicillinresistant Staphylococcus aureus (MRSA) in Germany. Dtsch Arztebl Int. 2011;108:761–7.
100. Vourli S, Vagiakou H, Ganteris G, Orfanidou M, Polemis M, Vatopoulos A, Malamou-Ladas H. High rates of community-acquired, Panton-Valentine leukocidin (PVL)-positive methicillin-resistant S aureus (MRSA) infections in adult outpatients in Greece. Euro Surveill. 2009;14(2):pii=19089.
101. Stam-Bolink E, Mithoe D, Baas W, Arends J, Moller A. Spread of a methicillin-resistant Staphylococcus aureus ST80 strain in the community of the northern netherlands. Eur J Clin Microbiol Infect Dis. 2007;26:723–7.
102. Fang H, Hedin G, Li G, nord CE. Genetic diversity of community-associated methicillin-resistant Staphylococcus aureus in southern Stockholm, 2000–2005. Clin Microbiol Infect. 2008;14:370–6.
103. Otter JA, French GL. Molecular epidemiology of community-associated methicillin-resistant Staphylococcus aureus in Europe. Lancet Infect Dis. 2010;10:227-39.
104. Chambers HF. The changing epidemiology of Staphylococcus aureus. Emerg Infect Dis. 2001;7:178-82.
105. Charlebois ED, Perdreau-Remington F, Kreiswirth B, Bangsberg DR, Ciccarone D, Diep BA, ng VL, Chansky K, Edlin B, Chambers HF. origins of community strains of methicillin-resistant Staphylococcus aureus. Clin Infect Dis. 2004;39:47-54.
106. Jung SI, Shin DH, Park KH, Shin JH. Antimicrobial susceptibility and clonal relatedness between community- and hospital-acquired methicillin-resistant Staphylococcus aureus from blood cultures. J Microbiol. 2006;44:336-43.
107. Fey PD, Saïd-Salim B, Rupp ME, Hinrichs SH, Boxrud DJ, Davis CC, Kreiswirth Bn, Schlievert PM.Comparative molecular analysis of community- or hospital-acquired methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2003;47:196-203.
108. Okuma K, Iwakawa K, Turnidge JD, Grubb WB, Bell JM, o´Brien FG, Coombs GW, Pearman JW,Tenover FC, Kapi M, Tiensasitorn C, Ito T, Hiramatsu K. Dissemination of new methicillin-resistant Staphylococcus aureus clones in the community.J Clin Microbiol. 2002;40:4289-94.
109. Daum RS, Ito T, Hiramatsu K, Hussain F, Mongkolrattanothai K, Jamklang M, Boyle-Vavra S. A novel methicillin-resistance cassette in communityacquired methicillin-resistant Staphylococcus aureus isolates of diverse genetic backgrounds. J Infect Dis. 2002;186:1344-7.
110. Mongkolrattanothai K, Boyle S, Kahana MD, Daum RS. Severe Staphylococcus aureus infections caused by clonally related community-acquiredmethicillin-susceptible and methicillin-resistant isolates. Clin Infect Dis. 2003;37:1050-8.
111. Robinson DA, Holmes A, Morrison D, GrundmannH, Edwards G, o'Brien FG, Tenover FC, McDougal LK, Monk AB, Enright MC. Re-emergenceof early pandemic Staphylococcus aureus as a community-acquired meticillin-resistant clone. Lancet. 2005;365:1256-8.
112. Ho PL. Community-associated methicillin-resistantstaphylococcus aureus (CA-MRSA) is emerging or just re-emerging? [editorial]. Hong Kong Med Diary. 2007;12:2-3.
Artículo recibido el 14 de mayo de 2012 y aceptado para publicación el 16 de octubre de 2012.
Conflictos de interés: ninguno.
Fuentes de financiamiento: autofinanciado.
Correspondencia:
Daniel Angel Luján Roca
Rua Pedra Bonita, 1.131/ 33 -Alto Barroca, CEP30.431-065, Belo Horizonte, Minas Gerais, Brasil
Teléfono: (55) (31) 97943495
Correo electrónico: d_lujan@starmedia.com













