Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Anales de la Facultad de Medicina
Print version ISSN 1025-5583
An. Fac. med. vol.79 no.1 Lima Jan./Mar. 2018
http://dx.doi.org/10.15381/anales.v79i1.14599
CARTAS AL EDITOR
Genotipificación molecular de las cepas del virus sarampión en el Perú, 2018
Molecular genotyping of measles virus strains in Perú, 2018
Fredy J. Condori-Yujra 1,a,b, Víctor Jimenez-Vasquez 2,4,b, Giancarlo W. Enríquez-Alva 3, Sara M. Gordillo-Vilchez 2,a, Néstor E. Cabezudo-Pillpe 1,a,c
1 Laboratorio Nacional de Sarampión y Rubeola, Centro de Nacional de Salud Pública, Instituto Nacional de Salud. Lima, Perú.
2 Facultad de Ciencias Biológicas, Escuela de Posgrado, Facultad de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos. Lima, Perú.
3 Facultad de Ciencias Naturales y Matemáticas, Escuela Profesional de Biología, Universidad Nacional Federico Villareal. Lima, Perú.
a Biólogo Microbiólogo b Especialista en Genética y Biología Molecular c Epidemiólogo.
Sr. Editor, en el año 2017, cuatro países de la región de las Américas notificaron casos confirmados de sarampión, a saber: Argentina (3 casos), Canadá (45 casos), los Estados Unidos de América (120 casos) y la República Bolivariana de Venezuela (727 casos). En los primeros meses de 2018, los siguientes 9 países han notificado casos confirmados: Antigua y Barbuda (1 caso), Brasil (14 casos), Canadá (4 casos), Colombia (1), Estados Unidos de América (13 casos), Guatemala (1 caso), México (4 casos), Perú (2 casos) y Venezuela (159 casos)(1).
La epidemiología molecular de los virus del sarampión es de vital importancia en la vigilancia de cepas de sarampión en la circulación mundial, especialmente durante los brotes. La genotipificación del virus del sarampión puede jugar un papel importante en el seguimiento de las vías de transmisión durante la investigación del brote, por lo que, los resultados de genotipificación ayudan a confirmar, refutar o detectar las conexiones entre casos. Si dos casos tienen genotipos coincidentes, estos pueden estar conectados incluso si dicho nexo no es evidente(2).
De acuerdo con el análisis de laboratorio realizado por la Fundación Oswaldo Cruz (Fiocruz/RJ), el genotipo identificado en todos los casos confirmados es el D8. Este genotipo es idéntico al identificado en Venezuela en 2017(3). En nuestro país fueron confirmados dos casos en 2018 (Juliaca y Callao), de estos se obtuvieron aislamientos virales con el objetivo de caracterizarlos molecularmente.
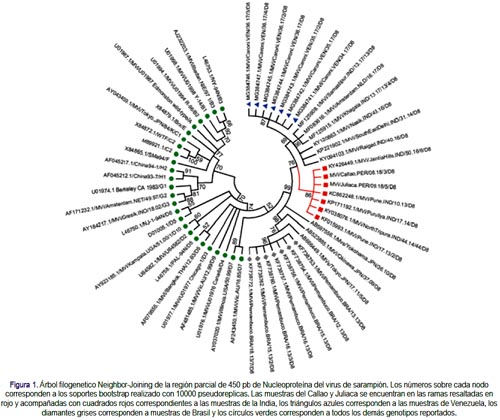
Las muestras de hisopado nasal y faríngeo, procedentes de Callao (paciente de 46 años) y Juliaca (paciente de 16 años), ambos con ELISA IgM positivo y PCR en tiempo real positivo, fueron inoculadas en líneas celulares Vero, tras lo cual se realizaron pasajes cada 7 días. Las muestras fueron de pacientes con síntomas clínicos de sarampión recepcionadas en el Instituto Nacional de Salud (INS) para diagnóstico durante los meses de febrero y marzo del presente año. Se extrajo el ARN viral de las muestras seleccionadas utilizando un Kit de extracción QIAamp Viral RNA Mini, el cual fue almacenado a -40°C hasta su uso. Se realizó una PCR por transcripción reversa (RT-PCR) del gen de Nucleoproteína viral de sarampión (NP), los productos fueron revelados en un gel de agarosa al 2%, con ello se obtuvo un fragmento de 450 pb. Los amplicones fueron posteriormente purificados con el método Exosap y secuenciados con el método de Sanger, en un analizador genético Applied Biosystems 3500. Las secuencias forward y reverse de cada amplicón fueron editadas y ensambladas con el software SeqTrace versión 0.9.0 y luego contrastadas con secuencias de nucleoproteína de sarampión depositadas en la base de datos GeneBank(4). Se contó con secuencias de todos los genotipos conocidos, pero con mayor énfasis en secuencias del genotipo D8. Las secuencias fueron alineadas con el programa MAFFT versión 7. Se realizó una reconstrucción filogenética con la metodología de máxima verosimiltud (MV) con el programa RaXML version 8.0.0 con una búsqueda inicial de 50 árboles. Obtenido el árbol MV, se identificaron en él las secuencias más próximas a las muestras peruanas y se procedió con una última reconstrucción con la metodología Neighbor-Joining (NJ), con un número menor se secuencias, opción de pairwise deletion y 10000 repeticiones bootstrap, esto último para estimar los soportes estadísticos de los nodos del árbol. (Figura 1).
El árbol inicial obtenido con la metodología de máxima verosimilitud logró agrupar a las muestras peruanas en un clado conformado por muestras provenientes de la India, con aquella información se obtuvo un árbol más pequeño que permitió una mejor visualización de los vínculos entre las muestras de Perú, Venezuela, Brasil e India. Este último árbol estimado con la metodología NJ permitió corroborar el vínculo entre las muestras de Perú y la India, pertenecientes al genotipo D8, con un soporte estadístico bootstrap de 86%." Los análisis realizados, por tanto, permitieron caracterizar genotípicamente a las muestras de Perú como perteneciente al genotipo D8 y vinculada con brotes de la India.
REFERENCIAS BIBLIOGRÁFICAS
1. Boletín Semanal de Sarampión/Rubéola: Organización Panamericana de la Salud. OPS/OMS.Vol.24, n.9 marzo de 2018. Disponible en: http://bit.ly/2HeaPZt. [ Links ]
2. World Health Organization: Standardization of the nomenclature for describing the genetic characteristics of wild-type measles viruses WHO/Wkly. Epi Rec. 1998; 73, 265-269. [ Links ]
3. World Health Organization, Regional Office forEurope. Press Release: Europe observes a 4-fold increase in measles cases in 2017 compared to previous year. Copenhagen. Acceso 19 febrero2018. Disponible en: http://bit.ly/2ETCTnd. [ Links ]
4. GeneBank del Centro Nacional de Información Biotecnológica de los Estados Unidos. Disponible en: https://www.ncbi.nlm.nih.gov/genbank/. [ Links ]
Conflictos de interés: Ninguno.
Fuentes de financiamiento: Estudio financiado por la Vigilancia de Sarampión y Rubéola CNSP/INS/MINSA.
Correspondencia:
Fredy Condori Yujra
fcondori@ins.gob.pe
Recibido: 6 abril 2018
Aceptado: 7 abril 2018