Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Anales de la Facultad de Medicina
Print version ISSN 1025-5583
An. Fac. med. vol.79 no.3 Lima July/Set. 2018
http://dx.doi.org/10.15381/anales.v79i3.15313
ARTÍCULO ORIGINAL
Resistencia a colistín mediado por el gen mcr-1 identificado en cepas de Escherichia coli y Klebsiella pneumoniae. Primeros reportes en el Perú
Resistance to colistin mediated by the mcr-1 gene identified in strains of Escherichia coli and Klebsiella pneumoniae. First reports in Peru
Ruth Giovanna Ugarte Silva1,a, José Maria Olivo López1,b, Alejandra Corso2, Fernando Pasteran2,Ezequiel Albornoz2, Zaida Patricia Sahuanay Blácidoc
1 Instituto Nacional de Salud del Niño. Lima, Perú.
2 Laboratorio Regional de Resistencia a los Antimicrobianos, Instituto Nacional de Enfermedades Infecciosas "Dr. Carlos G. Malbran". Buenos Aires, Argentina.
a Licenciado tecnólogo médico en laboratorio clínico y anatomía patológica, especialista en microbiología clínica. ORCID: orcid.org/0000-0003-2465-1732
b Licenciado tecnólogo médico en laboratorio clínico y anatomía patológica, especialista en microbiología clínica. ORCID: orcid.org/0000-0002-0688-16071
c Licenciado tecnólogo médico en laboratorio clínico y anatomía patológica. ORCID: orcid.org/0000-0002-4748-6648
RESUMEN
Introducción. Ante la aparición de reportes de la presencia del gen mcr-1 y su posible diseminación por plásmidos en los países de la región y dado que este gen confiere resistencia a colistín, fármaco que es la última línea de tratamiento contra bacterias multirresistentes, es importante conocer su presencia en nuestro país en microorganismos que lo expresen. Métodos. Se realizó un estudio descriptivo y transversal. Se incluyeron microorganismos aislados de urocultivos de pacientes ambulatorios de un centro de salud privado en Lima, Perú, en agosto del año 2017. De 326 urocultivos positivos se seleccionaron 10 aislamientos entre cepas de Escherichia coli y Klebsiella pneumoniae que presentaron concentración mínima inhibitoria ≥ 4µg/ mL (interpretado como resistente para colistín) por el sistema automatizado Microscan Walkaway 96 plus. Se utilizaron los siguientes métodos: colistín agar spot, predifusión con tabletas de colistín, microdilución en caldo colistin y PCR para el gen mcr-1. Resultados. Se determinó que 7 aislamientos, todas Escherichia coli, expresaron la presencia del gen mcr-1 por PCR, el cual confiere resistencia plasmídica a polipéptidos. De las cepas restantes, dos Escherichia coli y una Klebsiella pneumoniae, resultaron positivos para resistencia a colistín en las pruebas fenotípicas pero no en la PCR para gen mcr-1 lo cual sugiere un mecanismo de resistencia a colistín no asociado a gen mcr-1. Conclusiones. Se obtuvieron 7 aislamientos de Escherichia coli resistentes a colistín y con expresión del gen mcr-1.
Palabras clave: Colistín; Farmacorresistencia microbiana; Escherichia coli; Klebsiella pneumoniae.
ABSTRACT
Introduction. Given the appearance of reports of the presence of the mcr-1 gene and its possible dissemination by plasmids in the countries of the region and given that this gene confers resistance to colistin, the drug that is the last line of treatment against multiresistant bacteria, it is important to know its presence in our country in microorganisms that express it. Methods. Descriptive and cross-sectional study was carried out. Microorganisms isolated from urine culture of outpatients from a private health center in Lima, Peru, were included in august 2017. Out of 326 positive urine cultures, 10 isolates were selected between strains of Escherichia coli and Klebsiella pneumoniae that had a minimum inhibitory concentration ≥4µg/mL (interpreted as resistant for colistín) by the automated system Microscan Walkaway 96 plus. The following methods were used: colistin agar spot, prediffusion with colistin tablets, microdilution in colistin broth and PCR for the mcr-1 gene. Results. It was determined that 7 isolates, all Escherichia coli, expressed the presence of the mcr-1 gene by PCR, which confers plasmid resistance to polypeptides. Of the remaining strains, two Escherichia coli and one Klebsiella pneumoniae, were positive for resistance to colistin in the phenotypic tests but not in the PCR for mcr-1 gene, which suggests a mechanism of colistin resistance not associated with the mcr-1 gene. Conclusions. Seven isolates of Escherichia coli resistant to colistin and with expression of the mcr-1 gene were obtained.
Keywords: Colistin; Drug resistance, microbial; Escherichia coli; Klebsiella pneumonia.
INTRODUCCIÓN
La resistencia a los antibióticos constituye un problema global, creciente y preocupante en la actualidad. Nuestro país no es ajeno a ello. Uno de los grupos de antimicrobianos que en su mayoría se ven afectados por este fenómeno han sido el grupo de betalactámicos1; no obstante, ningún grupo está exento a este fenómeno. Quedan pocas opciones de antimicrobianos para tratamiento sin que se desarrolle pronta resistencia a ellos. Uno de los compuestos usados como única y última alternativa contra microorganismos multirresistentes es colistín2.
Recientemente se ha reportado la presencia de microorganismos aislados de muestras biológicas que expresan resistencia plasmídica a colistín mediada por el gen mcr-1 o sus variantes. Este suceso cobra relevancia, puesto que colistín es la última opción de tratamiento ante microorganismos multirresistentes; así, el gen transferible mcr-1 representa un alto riesgo para la diseminación de la resistencia a colistín2,3,4.
Colistín es una polimixina del grupo de los polipéptidos, descubierto en la década de los cuarenta y usada entonces para el tratamiento de infecciones por bacilos Gram negativos, que cayó en desuso por su alta toxicidad3. Recientemente, este compuesto ha resurgido como medicamento de última línea para el tratamiento de infecciones por gérmenes multirresistentes. De los diferentes tipos de polimixinas, solo la B y E (colistín) son de uso clínico4,5,6. Su estructura química está conformada por un anillo peptídico policatiónico que contiene entre 8 y 10 aminoácidos, este decapéptido contiene un lazo cíclico de siete aminoácidos entre el grupo amino de la cadena lateral del ácido di-amino-butírico (Dab) en posición 4 y el grupo carboxilo del carbono terminal del residuo 10 de treonina 5,6, este lazo se une al ácido graso amino terminal mediante una cadena de tres aminoácidos. Tiene como característica ser anfipático debido a la carga positiva sobre los residuos Dab y la cola del ácido graso, una mezcla de grupos hidro y lipofílico. Esta característica le proporciona la misma facilidad para disolverse en agua (sangre) que en la bicapa lipídica de membranas procariotas y eucariotas. El sitio blanco de acción de colistín es el lípido A del lipopolisacárido de las membranas bacterianas y su efecto es bactericida5.
En el año 2015 en China, Liu y col.7 reportaron el primer mecanismo plasmídico de resistencia a colistín en Enterobactriaceae denominado mcr-1 (mobile colistín resistance) que corresponde a una fosfoetanolamina transferasa que es capaz de modificar el sitio blanco disminuyendo la afinidad de colistín por el lípido A. En este caso, se hallaron 260 muestras de E. coli portadoras de mcr-1 recuperadas de animales para consumo, alimentos y muestras clínicas de pacientes hospitalizados.
A la fecha se ha confirmado la presencia del gen mcr-1 en países de 4 continentes incluido América8. Entre noviembre de 2012 y noviembre del 2013 se hizo un estudio en muestras de turistas holandeses tras retornar a su país provenientes de América Latina, aislándose en ellos 6 cepas de E coli mcr -1 positivo9 .
En el año 2015, el Laboratorio Regional de Resistencia a los Antimicrobianos del Servicio Antimicrobianos del Instituto Nacional de Enfermedades Infecciosas "Dr. Carlos G. Malbrán" de Argentina, realizó un estudio retrospectivo de cepas que mostraron resistencia a colistín durante los años 2012 a 2016, encontrándose 9
E. coli positivas a mcr-110. En el año 2016 se reportó en Colombia 3 aislamientos de Salmonella entérica positivas al gen mcr1 aislados de alimentos procedentes de Bogotá.19,29. También el año 2016, la OPS y OMS alertaron que: "Aunque es posible la portación asintomática de bacterias con este gen, existe riesgo de que se disemine a través de plásmidos a otras cepas virulentas o clones hiperepidémicos".9
Descrito brevemente el panorama, y dado que nuestro país no cuenta con datos al respecto, el presente estudio tiene por objetivo describir los primeros hallazgos de microorganismos que expresan resistencia a colistín mediada por el gen mcr-1.
MÉTODOS
Se realizó un estudio de tipo descriptivo y transversal. Los aislamientos se obtuvieron de un centro de salud privado de Lima, Perú, centro de mediana afluencia que atiende pacientes de manera ambulatoria, los cuales pertenecen a todos los estratos socioeconómicos provenientes de variados puntos del país y cuya población consta de adultos jóvenes y en su mayoría adultos mayores.
Durante el mes de agosto del año 2017, se obtuvieron un total 1027 uro-cultivos. Todas las muestras fueron sembradas en placas de agar sangre y agar McConkey con asas calibradas de 1µL, incubadas a 35 ° C por 24 horas y posteriormente evaluadas bajo los criterios del manual de la Sociedad Americana de Microbiología para clasificarlos como positivos o negativos, según se aprecia en el flujograma de la figura 1. Se obtuvieron 326 urocultivos positivos, todos ellos fueron procesados por el sistema automatizado Microscan Walkaway Plus 96 (Beckman y Coulter) para identificación del microorganismo y la respectiva prueba de sensibilidad, utilizando los paneles NUC 66. Figura 1.
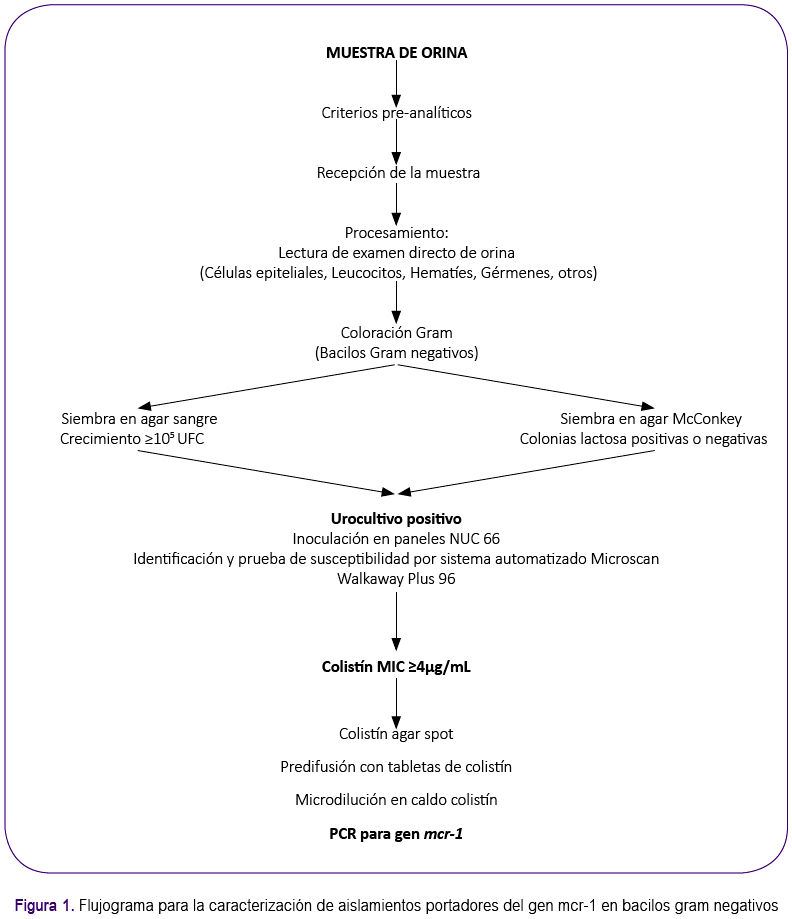
Se consideraron importantes todos los bacilos Gram negativos que no presentaron resistencia natural a colistín y con una concentración mínima inhibitoria (MIC) ≥4µg/mL para este mismo antimicrobiano (interpretado como resistente). Sólo se consideró el primer aislamiento de cada uno de los pacientes en el periodo de tiempo señalado.
Se obtuvieron así 10 cepas, 9 Escherichia coli y 1 Klebsiella pneumoniae, las cuales fueron sometidas a las siguientes pruebas para la confirmación de la resistencia a colistín10: colistín agar spot 11 , predifusión con tabletas de colistín 12, microdilución en caldo colistín 13 y PCR para gen mcr-1 14,15 (figura 1). Los aislamientos fueron conservados en TSA y luego derivados al Laboratorio Regional de Resistencia a los Antimicrobianos del Servicio Antimicrobianos del Instituto Nacional de Enfermedades Infecciosas de Argentina "Dr. Carlos G. Malbrán", manteniendo las medidas de bioseguridad requeridas por la International Air Transport Association, donde se realizaron las pruebas mencionadas. La identificación de todos los aislamientos se realizó mediante espectometría de masas: matrix assisted laser desorption/ionization time of flight mass spectrometry (MALDI-TOF MS).
RESULTADOS
Se obtuvieron 1027 urocultivos, 326 de ellos positivos. Se determinó que 7 aislamientos, todas Escherichia coli, expresaron la presencia del gen mcr-1 por PCR, el cual confiere resistencia plasmídica a polipéptidos (Tabla 1). De las cepas restantes, dos Escherichia coli y una Klebsiella pneumoniae, resultaron positivos para resistencia a colistín en las pruebas fenotípicas pero no en la PCR para gen mcr-1 lo cual sugiere un mecanismo de resistencia a colistín no asociado a gen mcr-1.

DISCUSIÓN
Se ha descrito la presencia del gen mcr-1 en países de 4 continentes incluido América15,16,17,19. Nuestros resultados confirman la presencia del gen mcr-1 en nuestro país; sin embargo, esto se considera un hallazgo dado que según manuales de procedimientos y normas internacionales no se encuentra contemplado el uso de colistín en pruebas de sensibilidad de rutina en aislamientos urinarios, a excepción de aislamientos de Pseudomonas aeruginosa en pacientes hospitalizados o en microorganismos con multidrogorresistencia. En esos casos, los puntos de corte y el método idóneo a usar son controversiales por las limitantes del método y las características de la molécula previamente señaladas18,20. El hecho de no tener como protocolo de rutina la búsqueda de este tipo de resistencia en enterobacterias se consideraría un factor limitante para conocer la prevalencia real en nuestro medio; así, a la fecha Perú no cuenta con indicadores o datos estadísticos comparados con otros países europeos que ya señalan incluso un 2,9% de aislamientos resistentes a colistín21.
Sin embargo, consideramos que nuestros hallazgos aportan a la investigación de la resistencia bacteriana en nuestro país.
La prueba de colistín agar spot es considerada una prueba de tamizaje que comparada con el método microdilución presenta una concordancia de 99,5%11 dado que microdilución en caldo para colistín (BMD) es considerado el método de referencia. En la tabla de resultados se puede observar que todos los aislamientos dieron resultados positivos para colistín agar spot.
El método de predifusión con tabletas de colistín está fundamentado en mejorar la difusión de colistín en el medio, ya que éste es un antimicrobiano de alto peso molecular22. En los resultados obtenidos todos los aislamientos presentan halos ≤ 11mm lo que los categoriza como resistentes11, a excepción de la cepa 22964 que presenta como resultado 16mm. En cuanto a los resultados de microdilución en caldo, todos los aislamientos presentaron MIC ≥ 4µg/mL que los posiciona en la categoría resistente23,24.
La PCR para el gen mcr-1 es la prueba molecular incluida en el esquema trabajado y por lo obtenido podemos señalar que los 3 aislamientos mcr-1 negativos resultaron resistentes a colistín pero su resistencia no se encontró asociada a la presencia de este gen en particular. Es posible que en estos aislamientos la resistencia se encuentre asociada a otros genes que confieren resistencia cromosómica a polipéptidos o a otras variantes de mcr-1 ya descritas (mcr-2 a mcr-8)25,26,31.
Las limitaciones del presente estudio fueron que durante el desarrollo del mismo, no se estudiaron los mecanismos cromosómicos y/o plasmídicos de las cepas mcr-1 negativas27,28,29.
Concluimos que de 326 urocultivos positivo, se obtuvieron 7 aislamientos de Escherichia coli resistentes a colistín y con expresión del gen mcr-1. Si bien este tipo de resistencia ya había sido identificado en países lejanos24,27,28 e incluso vecinos al nuestro19, estos representan los primeros aislamientos confirmados en enterobacterias en el Perú asociados a la presencia del gen mcr-1. Recomendamos continuar el estudio para la búsqueda de aislamientos con esta resistencia e implementar pruebas en los laboratorios para su detección temprana30, con la finalidad de generar fuentes de información para detectar la presencia de este gen y sus variantes23,24.
AGRADECIMIENTOS
Al médico patólogo clínico Elena Tapia Egoavil. A los licenciados tecnólogos médicos Blanca Cutis Onsihuay, Lacey Prado Duran, Heydi García Leyva, Manuel Melo Espinoza por su colaboración en el presente estudio.
REFERENCIAS BIBLIOGRÁFICAS
1. Instituto Nacional de Salud. Informe de la Resistencia Antimicrobiana en Hospitales en Perú. Lima: Instituto Nacional de Salud. 2007:20-28. [ Links ]
2. Jian Li, Nation R, Turnidge J, Milne R.,Kingsley C, Rayner C, Paterson D. colistín: The re-emerging antibiotic for multidrug-resistant Gram-negative bacterial infections. Lancet Inf. 2006; 6:589-60. DOI: 10.1016/S1473-3099(06)70580-1. [ Links ]
3. Yahav D, Farbman L, Leibovici L. Colistín: New lessons on an old antibiotic. Clinic Microbi Infec. 2012; 18(1):18-29. DOI: 10.1111/j.1469-0691.2011.03734.x. [ Links ]
4. Velkov T, Roberts KD, Nation RL, Thompson PE, Li J. Pharmacology of polymixins: new insights into an old class of antibiotics. NIH Public Access. 2013; 8(6):711-24. DOI: 10.2217/fmb.13.39. [ Links ]
5. Aguayo A, Mella S, Riedel G, Nello H, Domínguez M, Gonzales-Rocha G. Colistín en la era post-antibiótica. Rev Chil Infectol. 2016;33(2):166-176. DOI: http://dx.doi.org/10.4067/S0716-10182016000200006. [ Links ]
6. Tran TB, Velkov T, Nation RL, Forrest A, Tsuji BT, Bergen PJ, et al. Pharmacokinetics/pharmacodynamics of colistín and polymixin B: are we yet?. Int J Antimicrob Agents. 2016; 48(6): 592-597. DOI: 10.1016/j.ijantimicag.2016.09.010. [ Links ]
7. Liu YY, Wang Y, Walsh TR, Yi LX, Zhang R, Spencer J, et al. Emergence of plasmid-mediated colistín resistance mechanism mcr-1 in animal and human beings in China: a microbiological and molecular biological study. Lancet Infect. Dic. 2016;16(2):161-168. DOI: 10.1016/S14733099(15)00424-7. [ Links ]
8. Wang R, Van Dorp L, Shaw L, Bradley P, Wang Q, Wang X, et al. The global Distribution and spread of the mobilized colistín resistance gene mcr-1. Nature communication.2018;9(1):1179. DOI: 10.1038/s41467-018-03205-z. [ Links ]
9. Organización Panamericana de la Salud/Organización Mundial de La Salud [internet]. Alerta Epidemiológica: Enterobacterias con resistencia transferible a colistín, implicaciones para la salud pública en las Américas. Washington D.C.: OPS/OMS. 2016. [Fecha de acceso 20 de julio de 2018]. Disponible en: https://www.paho.org/hq/dmdocuments/2016/2016-jun-10-alerta-epienterob-resist.pdf. [ Links ]
10. Programa Nacional de Control de Calidad en Bacteriología del Insituto Nacional de Enfermedades Infecciosas "Dr. Carlos Malbrán". Emergencia de Resistencia Plasmídica (Transferible) a colistín/Polimixina B MCR-1 en Argentina. Boletín informativo Nº3. Buenos Aires: Insituto Nacional de Enfermedades Infecciosas "Dr. Carlos Malbrán". 2016. [ Links ]
11. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS" Dr Carlos G. Malbrán" [Internet]. Métodos de Screening "colistín Agar-Spot". [Fecha de acceso 24 de julio 2018]. Disponible en: http://antimicrobianos.com.ar/ATB/wp-content/uploads/2017/09/Protocolo-Agar-spot-COL-2017-version2-Agosto2017.pdf. [ Links ]
12. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS "Dr Carlos G. Malbrán" [Internet]. Métodos depredifusión con Tabletas Rosco-Neosensitabs. [Fecha de acceso 24 de julio 2018]. Disponible en: http://antimicrobianos.com.ar/ATB/wpcontent/uploads/2017/09/Protocolo-Predifusion-TabletasCOL-Rosco-version2-Agosto2017.pdf. [ Links ]
13. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS "Dr Carlos G. Malbrán" [Internet]. Protocolo para determinación de Concentración Inhibitoria Mínima por el Método de Microdilución. Aplicación para determinar la sensibilidad a colistín. [Fecha de acceso 24 de julio 2018]. Disponible en: http://antimicrobianos.com.ar/ATB/wp-content/uploads/2017/09/Protocolo-CIM-microdiluci%C3%B3n-COLversion2-Agosto-2017.pdf. [ Links ]
14. Servicio Antimicrobianos, Laboratorio Nacional de Referencia en Antimicrobianos, INEI-ANLIS "Dr Carlos G. Malbrán" [Internet]. Protocolo de PCR para la detección del gen mcr-1 en aislamientos Gram-negativos. [Fecha de acceso 24 de julio 2018]. Disponible en: http://antimicrobianos.com.ar/2016/01/deteccion-de-resistencia-transferiblea-colistín-gen-mcr-1/. [ Links ]
15. Wong S, Tse H, Chen J, Cheng V, Ho Pak-Leung, Yuen Kwok-Yung. Colistin-Resistant Enterobacteriaceae Carrying the mcr-1 Gene among Patients in Hong Kong. Emerg Infect Dis. 2016; 22(9):16671669. DOI: 10.3201/eid2209.160091. [ Links ]
16. Chan WS, Au CH, Ho DN, Chan TL, Ma ES, Tang BS. Prospective study on human fecal carriage of Enterobacteriaceae possessing mcr-1 and mcr-2 genes in a regional hospital in Hong Kong. BMC Infectious Diseases. 2018;18(1):81. DOI: 10.1186/s12879-018-2987-y. [ Links ]
17. Zhang J, Chen L, Wang J, Butaye P, Huang K, Qiu H et al. Molecular detection of colistín resistance genes (mcr-1 to mcr-5) in human vaginal swabs. BMC Res Notes. 2018;11:143. DOI: 10.1186/s13104-018-3255-3. [ Links ]
18. García L. Clinical Microbiology Procedures Handbook. 3rd Edition. Washington D.C.: American Society for Microbiology. 2010.
19. Instituto Nacional de Vigilancia de Medicamentos y Alimentos – INVIMA. Alerta por la primera detección del gen mcr-1 de resistencia al antibiótico colistín en aislamientos de Salmonella typhimurium y Salmonella give en alimentos en Colombia. Bogotá. Agosto 2017. [ Links ]
20. American Society for Microbiology. Clinical Microbiology Pocedures Handbook, 3rd edition, vol 1, 2010. [ Links ]
21. European Society of Clinical Microbiology and Infectious Diseases [Internet]. EUCAST guidelines for detection of resistance mechanisms and specific resistances of clinical and/or epidemiological importance. Version 2.0. Jul 2017;pp:25-26. [Fecha de acceso 24 de julio 2018]. Disponible en: http://www.eucast.org/fileadmin/src/media/PDFs/EU-CAST_files/Resistance_mechanisms/EUCAST_detection_of_resistance_mechanisms_170711.pdf. [ Links ]
22. Gao R, Hu Y, Li Z, Sun J, Wang Q, Lin J, et al. Dissemination and Mechanism of the MCR-1 Colistín Resistance. Plos Pathog. 2016;12(11):e1005957. DOI: https://doi.org/10.1371/journal.ppat.1005957. [ Links ]
23. Belaynehea K, Won S, Yoon K, Young J, Geun H, Joong I, Sang H. Emergence of mcr-1 and mcr-3 variants coding for plasmid-mediated colistín resistance in Escherichia coli isolates from food- producing animals in South Korea. InterJour Infec Dis. 2018; 72: 22–24. DOI: https://doi.org/10.1016/j.ijid.2018.05.011. [ Links ]
24. Srijan A, Margulieux KR, Ruekit S, Snesrud E, Maybank R, Serichantalergs O, et. al. Genomic Characterization of Nonclonal mcr-1-Positive Multidrug-Resistant Klebsiella pneumoniae from Clinical Samples in Thailand. Microbial DrugResistance. 2018;24(4):403-410. DOI: 10.1089/mdr.2017.0400. [ Links ]
25. Xu Y, Wei W, Lei S, Lin J, Srinivas S, Fenj Y. An Evolutionarily Conserved Mechanism for Intrinsic and Transferable Polymyxin Resistance. Mbio. 2018;9(2):1 – 16. DOI: 10.1128/mBio.02317-17. [ Links ]
26. Ye H, Li Y, Li Z, Gao R, Zhang H, Wen R, et. al. Diversified mcr-1-Harbouring Plasmid Reservoirs Confer Resistance to colistin in Human Gut Microbiota. Mbio. 2016; 7(2): 1-16. DOI: 10.1128/mBio.00177-16. [ Links ]
27. Principe L, Piazza A, Mauri C, Anesi A, Bracco S, Brigante G, et. al. Multicenter prospective study on the prevalence of colistín resistance in Escherichia coli: relevance of mcr-1-positive clinical isolates in Lombardy, Northern Italy. Infect Drug Resist.2018;11: 377–385. DOI: 10.2147/IDR.S160489. [ Links ]
28. Grondahl-Yli-Hannuksela K, Lonnqvist E, Kallonen T, Lindholm L, Jalava J, Rantakokko-Jalava K, et. al. The first human report of mobile colistín resistance gene, mcr-1 in Finland. APMIS. 2018;126(5): 413–417. DOI: 10.1111/apm.12834. [ Links ]
29. Venter H, Henningsen M, Begg S. Antimicrobial resistance in healthcare, agriculture and the environment: the biochemistry behind the headlines. Essaysin Biochemistry. 2017;61(1):1-10. DOI: 10.1042/EBC20160053. [ Links ]
30. Hernán C, Maza J, Vay C, Detección Rápida de Resistencia adquirida a colistín en Enterobacteriacea. Facultad de Farmacia y Bioquímica. Cátedra de Microbiología Clínica. Clínica Hospital de Clínicas-UBA. Laboratorio de Bacteriología .Universidad de Buenos Aires. [ Links ]
31. Wang X, Wang Y, Zhou Y, Li J, Yin W, Wang S, et. al. Emergence of a novel mobile colistín resistance gene, mcr-8, in NDM-producing Klebsiella pneumonia. Wang et al. Emerging Microbes & Infections.2018; 7:122. DOI: DOI 10.1038/s41426-018-0124-z. [ Links ]
Conflictos de interés: Los autores declaran no tener conflictos de interés
Fuentes de financiamiento: Autofinanciado
Correspondencia:
Ruth Giovanna Ugarte Silva
rugarte@insn.gob.pe
Recibido: 24 de julio 2018
Aprobado: 20 de setiembre 2018














