LOS CORONAVIRUS
Los coronavirus (reino: Riboviria; Filo: Incertae sedis; y orden: Nidovirales; família Coronaviridae; subfamília Orthocoronavirae; géneros: Alphacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus) 1 son virus envueltos (120 nm de diámetro) con simetría helicoidal que consisten en un genoma de ARN monocatenario de polaridad positiva con un tamaño que oscila entre 26 a 36 kb. La envoltura presenta una morfología variable de esférica a pleomórfica y es rica en glicoproteínas proyectadas en forma de espigas de 20 nm lo que le confiere las propiedades características del nombre - corona 2.
PRINCIPALES MECANISMOS ASOCIADOS CON LA EVOLUCIÓN DE LOS CORONAVIRUS EN LA NATURALEZA
Los animales como reservorio o huéspedes evolutivos
Los animales salvajes, especialmente los mamíferos y las aves, han sido identificados como reservorios de una gran diversidad de virus, de la mayoría de los cuales no tenemos ningún conocimiento, a pesar de saber que estos virus circulan con facilidad en sus nichos específicos. Generalmente, estos virus son silenciosos o asintomáticos en sus huéspedes naturales y experimentan frecuentes eventos de cambio de huésped entre especies de animales o entre especies de animales y humanos, un proceso que puede implicar cambios en las células o tejidos que infectan los virus (tropismo viral) 3 (tabla 1). Dichos cambios han resultado en nuevas enfermedades animales (como la enfermedad por coronavirus bovino 6 y la enfermedad por coronavirus canino 7) y enfermedades humanas (como OC438 y 229E9). Se propone que el agente etiológico del COVID-19, el SARS-CoV-2, se originó en los murciélagos y pasó a los humanos a través de un huésped reservorio intermedio 4.
Se sabe que siete coronavirus infectan a los humanos, SARS-CoV, MERS-CoV y SARS-CoV-2 que causan enfermedades graves, mientras que HKU1, NL63, OC43 y 229E causan síntomas más leves en la mayoría de los pacientes 5. Un nuevo estudio, realizado por la Escuela de Salud Pública de la Universidad de Michigan, en Estados Unidos, señala que cuatro tipos de coronavirus que infectan humanos son responsables por causar problemas respiratorios y que suelen ser estacionales, lo que significa que tienen un pico de casos en temporadas específicas como en invierno. Los siete tipos de coronavirus, que han sido reconocidos como patógenos respiratorios humanos, históricamente se registran en enfermedades respiratorias más leves.
El SARS-CoV-2, junto con el SARS-CoV y el MERS-CoV, forman parte del género Betacoronavirus. Un estudio mostró que el SARS-CoV-2 posee una mayor similitud (88% de identidad) con dos coronavirus similares al SARS derivados de murciélagos (bat-SL-CoVZC45 y bat-SL-CoVZXC21), recolectados en 2018 en Zhoushan, este de China, que con SARS-CoV (alrededor del 79 %) y MERS-CoV (alrededor del 50 %) 6. Otro estudio ha demostrado que el genoma del SARS-CoV-2 es un 91,02 % similar al de Pangolin-CoV, a pesar de tener una mayor identidad del 96,2 % entre el SARS-CoV-2 y otro coronavirus de murciélago (RaTG13) 7. Esto sugiere que el SARS-CoV-2 se originó en los murciélagos y podría tener pangolines como especie huésped intermediaria, ya que cinco residuos de aminoácidos clave (LFQNY) en el dominio de unión al receptor (RBD) de la proteína S, involucrados en la interacción con humanos El receptor ACE2, son similares entre Pangolin-CoV y SARS-CoV-2, pero no con RaTG13 7.
Existe un riesgo anticipado de que el SARS-CoV-2 se propague a otros mamíferos silvestres, incluyendo los grandes simios 8, los murciélagos en diferentes ubicaciones geográficas 9, y los felinos 10, amenazando la industria del turismo de vida silvestre en muchos países. Aunque los huéspedes intermediarios, como las civetas, se han asociado con los brotes de SARS-CoV, estos animales exhibieron edema pulmonar e inflamación en respuesta a la infección por coronavirus relacionados con el SARS-CoV-2 4,11, lo que sugiere que no son verdaderos reservorios de estos coronavirus. Por otro lado, los murciélagos no muestran síntomas clínicos de la enfermedad cuando se infectan con la mayoría de los virus, aunque existen algunas raras excepciones.
Varios hallazgos confirman características biológicas particulares de los murciélagos que permiten que estos mamíferos actúen como reservatorios excepcionales albergando más patógenos zoonóticos que cualquier otra especie de mamífero conocida 9,12. Los murciélagos poseen varias adaptaciones metabólicas y patrones de flujo de aire optimizados para eludir los gastos de alta energía que de otro modo podría conducir al hambre y la muerte. Una adaptación clave es la marcada alteración de la frecuencia cardíaca, que aumenta de 4 a 5 veces durante vuelo a un máximo de 1.066 pulsaciones por minuto. Para compensar altos niveles de estrés cardíaco, se induce bradicardia cíclica durante 5 a 7 min varias veces por hora durante el descanso, que puede conservar hasta un 10% de energía disponible 13,14. Por otro lado, estudios recientes sobre la inmunidad innata y adaptativa de los murciélagos proporcionan información de como la tolerancia inmune de estos animales puede ser responsable sobre la relación entre los murciélagos y los virus y como los humanos huéspedes se tornan susceptibles a las infecciones 15,16,17.
Una hipótesis propuesta sugiere que los murciélagos pueden controlar replicación viral al principio de la respuesta inmune, a través de mecanismos antivirales que involucran genes estimulados por interferón, conocidamente por los iniciales en inglés ISGs. En comparación con los humanos, muchas especies de murciélagos expresan constitutivamente IFNα basal y varios ISGs por IFN antes de la estimulación. Esto puede estar regulado por factores reguladores de IFN (IRF) que lleva a respuestas antivirales en murciélagos. Además, varios genes antivirales, como RNASEL, son inducida por IFN en murciélagos, pero no en otros mamíferos o han sufrido presión de selección para alterar potencialmente la función 18,19,20.
No obstante, durante el brote actual de COVID-19 se observó que existe el riesgo de repercusiones o transmisión zoonótica (antropozoonótica) 'inversa' de humano a animal, como lo demostraron los brotes de COVID-19 en visones de granjas en los Países Bajos, seguidos por la transmisión de animal a humano del virus SARS-CoV-2 21. También se han observado infecciones antropozoonóticas de SARS-CoV-2 de dueños de mascotas a perros y gatos domésticos 22,23, y a tigres y leones alojados en zoológicos 24.
El coronavirus SARS-CoV-2 se detectó en más del 35% de los 360 venados de cola blanca (Odocoileus virginianus) testados en el noreste de Ohio (EE.UU.). Se encontraron al menos tres cepas virales humanas en los animales junto con evidencia de transmisión de venado a venado. Estos hallazgos sugieren que estos animales pueden ser un reservorio evolutivo de SARS-CoV-2 y podrían facilitar la aparición de nuevas variantes 25.
Mecanismos genéticos virales arrollados en la generación de variantes virales
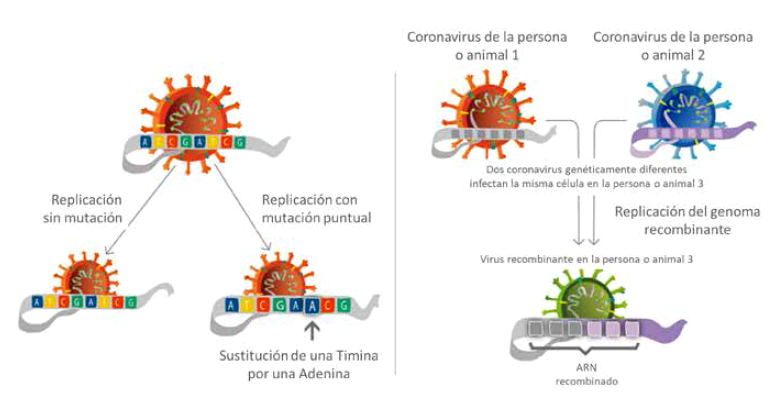
Muchos son los mecanismos genéticos asociados a la evolución de patógenos en la naturaleza, dentro de ellos las mutaciones puntuales y las recombinaciones son las más comunes (figura 1). Las mutaciones corresponden a cambios puntuales en el genoma viral, y debido a la presión selectiva ejercida por el sistema inmunológico del huésped, podrán modificar las características del virus.
Otro mecanismo más común en la evolución de los coronavirus es la recombinación, que es un proceso natural que reúne nuevas combinaciones de material genético y, por lo tanto, nuevas cepas virales, a partir de dos cepas de virus parentales similares no idénticas. Esta recombinación ocurre cuando diferentes cepas coinfectan un animal individual, con secuencias de cada cepa parental en la composición genética del virus de la progenie. La recombinación homóloga se ha demostrado previamente en muchos virus importantes, como el virus de la inmunodeficiencia humana (VIH), el virus de la peste porcina clásica y en toda la familia Coronaviridae. La recombinación en la región génica que codifica la proteína espiga S se ha implicado en la generación de SARS-CoV-2, aunque las investigaciones aún están en curso 26.
Las mutaciones y las recombinaciones ocurren millones de millones de veces en cada huésped que fue infectado por dos linajes o variantes de coronavirus. Linaje es un grupo de virus estrechamente relacionados con un ancestro en común y variante es un genoma viral (código genético) que puede incluir una o más mutaciones. En algunos casos, un grupo de variantes con cambios genéticos similares, como una linaje o grupo de linajes, puede ser definido por las organizaciones de salud pública como variante bajo monitoreo (VBM, por sus siglas en inglés), variante de preocupación (VOC, por sus siglas en inglés) o variante de interés (VOI, por sus siglas en inglés) debido a atributos y características compartidas que pueden requerir medidas de salud pública 27.
El hombre como principal fuerza motriz de la generación de variantes de coronavirus en la naturaleza
Las zoonosis, enfermedades naturalmente transmisibles entre animales y humanos, han existido siempre y actualmente han sido alarmas por las pandemias recientes, como la del ébola con varios brotes desde 1976, con posible aparición en murciélagos; la "enfermedad de las vacas locas" en la década de 1990, en la que se cree que el ganado se infectó al alimentarse con harina de carne y huesos de otro ganado; gripe aviar (2005) y porcina (2009); y también los recientes brotes de Dengue, Fiebre Amarilla, Fiebre Mayaro, Zika y Chikungunya, todos vinculados al mosquito Aedes aegypti.
La pandemia del SARS-CoV-2, es la tercera de coronavirus en los últimos 20 años. El primero fue el SARS-CoV en 2003, originado en gatos civetas infectados por murciélagos y la segunda fue el MERS-CoV en 2012 originado en vendedores ambulantes. Así, se observa la importancia de la investigación en la seguridad y bioseguridad en el área de salud, en especial la de los animales, como una tecnología gubernamental con alcance global.
Se cree que ciertos factores estresantes en los murciélagos, como enfermedades o pérdida de hábitat para ejemplo, puedan causar la interrupción de la coexistencia virus-sistema inmunológico que ellos poseen. Por lo tanto, trastornar este equilibrio puede inducir la multiplicación del virus, aumentando su virulencia y permitiendo la transmisión a otros huéspedes 28. Entre las intervenciones humanas observadas está el manejo de animales, refiriéndose a zoológicos, grandes ferias y mercados populares, en los que la propagación de patógenos es alta, por ser espacios con animales silvestres de diferentes regiones confinados en pequeñas áreas. La bioseguridad en estos lugares es sumamente importante para que no se transmitan nuevas enfermedades a humanos y a otros animales. Además, la deforestación descontrolada deja a los humanos más vulnerables a las enfermedades, ya que los animales presentes en el ambiente destruido migran a las regiones urbanas.
Avanzando en este tema, llama la atención el hecho de que los animales salvajes pueden ser el punto de origen del 70% de los patógenos zoonóticos conocidos, y aún pueden ser asintomáticos. Por lo tanto, se destaca la importancia de discutir aspectos relacionados con la bioseguridad, ya que es un término que se refiere a minimizar los riesgos de transmisión de agentes biológicos o microbiológicos a animales o humanos. En el contexto actual, es necesario profundizar la discusión sobre hasta qué punto el ser humano es capaz de interferir en la naturaleza, ya que la destrucción de los recursos naturales para expandir los sectores agrícola, industrial y urbano, dio lugar al inicio de lo que los científicos llaman la sexta extinción masiva de la vida en la Tierra en cuatro mil millones de años en el planeta. Por lo tanto, se hacen necesarias acciones efectivas sobre las causas de la interferencia humana en la naturaleza, a través de propuestas y soluciones capaces de mitigar los impactos ambientales.
Figura adaptada de: Margarida Duarte, Teresa Nogueira, Ana Botelho, Sandra Cavaco, Ana Duarte, Patrícia T. Santos, Rita de Sousa. Instituto Nacional de Investigação Agrária e Veterinária. Coronavírus dos animais e do Homen. Evolução e patogenia do vírus e prevenção de doença. Disponible en: https://projects.iniav.pt/coronavirus/images/CORONAVI%CC%81RUS%20dos%20animais%20e%20do%20Homem_high3.pdf.
SI NO HAY CAMBIOS EN LAS ACCIONES HUMANAS, TENDREMOS MÁS CORONAVIRUS RECOMBINANTES CON POTENCIAL PANDÉMICO EN NUESTRA GENERACIÓN
Además de instigar el cambio de huésped, la recombinación en diferentes regiones del genoma del virus también podría introducir nuevos fenotipos en las cepas de coronavirus que ya son infecciosas para los humanos. Hay al menos siete regiones potenciales para recombinación en las regiones de la replicasa y de la espiga en el genoma del SARS-CoV, con posibles virus asociados de recombinación de una variedad de otros coronavirus de mamíferos y humanos. Los eventos de recombinación entre dos cepas asociadas compatibles en un huésped compartido podrían conducir a futuros coronavirus novedosos, ya sea al permitir que las cepas de mamíferos preexistentes infecten a los humanos o al agregar nuevos fenotipos que surgen de diferentes alelos a las cepas preexistentes que afectan a los humanos 26. Estudios están siendo realizados para predecir anfitriones mamíferos de múltiples coronavirus, con el objetivo de determinar qué especies pueden ser reservorios no identificados de coronavirus, cuáles son los huéspedes más probables, en que especies puede ocurrir la recombinación, qué coronavirus tienen más probabilidades de coinfectar anfitriones, y por lo tanto actúan como fuentes para futuros virus nuevos 26.
El hombre es el centro de la dinámica de circulación de los patógenos en la naturaleza (figura 2). De esta manera, sus acciones pueden cambiar las fuerzas evolutivas asociadas a la emergencia de nuevos patógenos con potencial pandémico. Por lo tanto, así como podría ocurrir para otros agentes patogénicos, la generación de nuevos coronavirus podrá ser evitada con la implementación de políticas públicas que propongan acciones de salud única. Solamente existirá salud humana si el hombre contribuye con la salud ambiental y la salud animal.
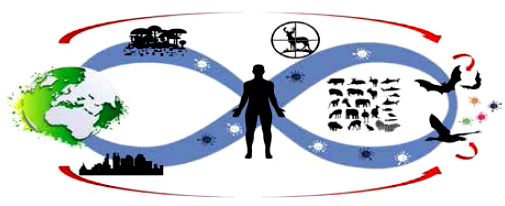
Figura 2 La dinámica de circulación de los patógenos en la naturaleza. El hombre es el centro de un ciclo, que podrá ser virtuoso o vicioso. La destrucción del medio ambiente, la urbanización demasiada y la interferencia en los nichos ecológicos de animales claves como aves y murciélagos son esenciales para la emergencia de patógenos con potencial pandémico.