Sr. Editor,
A dos años de la pandemia de la COVID-19, a finales de junio del 2022 los peruanos estamos enfrentando la cuarta ola causada por la variante Ómicron del virus SARS-CoV-2 (Severe acute respiratory syndrome coronavirus). A diferencia de la primera ola de la COVID-19 en marzo del 2020, causada por el linaje viral B.1.1, la tercera y cuarta ola han sido causadas por los linajes y sublinajes de la variante Ómicron 1,2, los cuales se caracterizan por ser más transmisibles, pero causan la enfermedad menos grave y menor mortalidad, comportamiento observado durante la evolución epidemiológica de la enfermedad. Con el objetivo de evaluar el comportamiento in vitro en la replicación viral del linaje B.1.1, linaje de mayor frecuencia y persistencia en el tiempo durante la primera ola del 2020 3, con el linaje BA.1 (variante Ómicron) de la tercera ola en el Perú, se comparó la replicación de 10 generaciones sucesivas de las 2 variantes (B.1.1 y BA.1), observándose que el linaje del 2020 se replica rápidamente in vitro, con alto título viral y causando daño celular, a diferencia de la variante Ómicron (linaje BA.1) que presenta replicación lenta causando leve daño celular. Este hallazgo podría correlacionar con la gravedad de la enfermedad observada en las olas ocasionadas por las mencionadas variantes, y contrasta con la capacidad de infección observada por las mismas, aunque otro factor que juega un rol importante es la protección inmune, sea por infecciones previas a lo que se suma la vacunación de la población.
Para el estudio de comparación de las variantes, se utilizaron muestras de hisopado nasofaríngeo correspondientes a los linajes B.1.1 y BA.1, identificado mediante secuenciamiento genómico con código de acceso: EPI_ISL_1111366 y EPI_ISL_7877459 en GI-SAID, respectivamente. Las muestras fueron inoculadas en células VERO-81 (ATCC(r) CCL 81TM), incubadas a 37 °C / 5 % CO24 por 5 días y el aislamiento del virus confirmado por RT-PCR. El aislamiento de cada variante fue denominado Pasaje 0 (P0) y utilizado para realizar los pasajes sucesivos del virus en células VERO-81, en las mismas condiciones de tiempo e incubación, realizado a cada 5 días para la obtención de 10 pasajes (P1,....P10) o generaciones continuas de los linajes B.1.1 y BA.1 5. Posteriormente, los pasajes de cada virus fueron cuantificados por el método de titulación en placa, observándose las unidades formadoras de placa (UFP) 6. Asimismo, a partir del pasaje con mejor definición de las UFP, se realizó una producción de antígeno para cada virus, determinándose la dosis infectiva y registrándose el efecto citopático después de 24, 48 y 72 horas, para cada virus.
El resultado de confirmación de aislamiento por RT-PCR muestra alta carga viral semejante para ambos linajes, con un umbral de ciclos (Ct) equivalente a 12,31 y 12,64 para el gen S, correspondiente al linaje B.1.1 y Ómicron (linaje BA.1), respectivamente. La cuantificación de los pasajes con igual tiempo de cultivo se muestra en la figura 1A, observándose alto título viral con destrucción de la monocapa celular a partir del pasaje 3 para el linaje B.1.1; a diferencia, la variante Ómicron (linaje BA.1) muestra menor carga viral en el mismo tiempo de replicación, observándose las UFP solo hasta la dilución 10-2. La figura 1B muestra la gráfica del título viral de los pasajes para cada virus, donde el linaje B.1.1 fue significativamente mayor en comparación a la variante Ómicron (linaje BA.1), siendo estas diferencias significativas en los pasajes: P2, P4, P6 a P9, según el análisis de ANOVA de 2 vías.
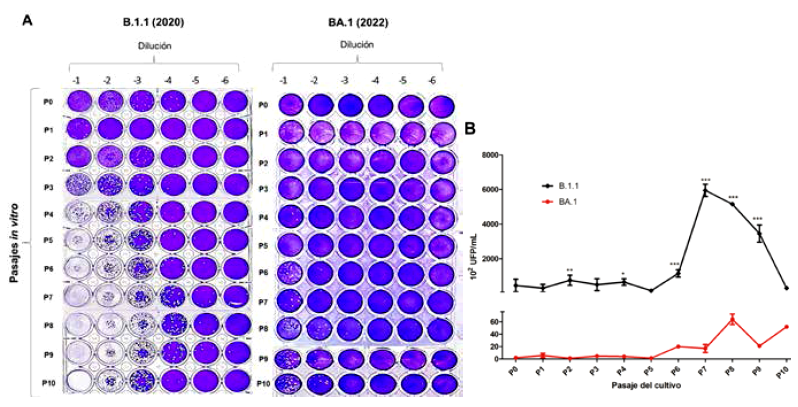
Figura 1 Comparación de 10 pasajes in vitro de los linajes B.1.1 y BA.1 (Ómicron). A. Titulación en placa de los pasajes 0 al 10 (P0 - P10) de B.1.1 y BA.1. La muestra de cada pasaje fue diluida y una alícuota utilizada para inocular en células Vero-81. Las células fueron coloreadas con cristal violeta y las UFP contabilizadas. B. Curva representativa de la cuantificación de las UFP/ mL de los pasajes de cada virus. El análisis estadístico se realizó utilizando ANOVA de dos variables, seguido de una posterior prueba de Bonferroni, *p<0,05, **p<0,01, ***p<0,001, ****p<0,0001.
A partir de un stock de virus con carga viral semejante, se realizaron diluciones seriadas y cultivadas para evaluación de la dosis infectiva y aparición del efecto citopático. Los cultivos en célula VERO-81 mostraron aparición del efecto citopático (recuadro en amarillo en la figura 2) a las 48 horas posterior a la infección, con progresivos aumentos a las 72 horas, dependiente de la carga viral. Ómicron (linaje BA.1) mostró efecto citopático leve a las 72 horas para la mayor concentración viral, según se muestra en la figura 2.

Figura 2 Aspecto morfológico comparativo del cultivo de las diluciones de los linajes B.1.1 y BA.1 (Ómicron).
Nuestros hallazgos muestran que el virus del linaje B.1.1, correspondiente a la primera ola de la COVID-19 en el Perú en el año 2020, presenta mayor virulencia y capacidad de replicación in vitro en comparación a la variante Ómicron (linaje BA.1) de la tercera ola a fines del año 2021. Las diferencias entre los linajes B.1.1 y BA.1 también pueden ser evidenciadas por su capacidad de infección y efecto citopático en cultivo in vitro en células VERO-81, siendo la variante Ómicron (linaje BA.1) menos infectivo en cultivo y mínimo efecto citopático en condiciones de misma carga viral que el linaje B.1.1. La atenuación de la virulencia en ómicron en comparación al virus de inicios de pandemia, observado in vitro, puede ser extrapolado al contexto actual de la enfermedad de la COVID-19, que después de 2 años de pandemia se ha visto la aparición de diversas variantes y nuevos linajes del virus SARS-CoV-2, siendo las ultimas variantes como la Ómicron de menor mortalidad en la población, a pesar de su alto índice de transmisibilidad; sin embargo, se debe considerar además que esta menor severidad y mortalidad también puede estar asociada a la protección inmune inducida por infecciones previas y la vacunación en el huésped humano 7. La replicación lenta de la variante Ómicron también es descrita por otro estudio in vitro en células Vero E6, explicando que el comportamiento de menor capacidad replicativa de Ómicron en comparación a la variante delta estaría asociada a diferente afinidad a receptores y vías de ingreso a la célula, extrapolándose en las manifestaciones clínicas de la enfermedad 8. Si bien la mutación en la proteína S del virus SARS-CoV-2 representa una ventaja por su mayor capacidad de transmisión entre las personas, al mismo tiempo pierde la virulencia celular, representando las dos caras de una moneda.















