INTRODUCCIÓN
A fines de diciembre del 2019, en la ciudad de Wuhan en la República Popular de China, apareció el nuevo coronavirus denominado SARS-CoV-2, el cual es la causa de la nueva enfermedad llamada COVID-19 1. Este nuevo virus respiratorio se extendió rápidamente a muchos países y causó casos de neumonía atípica, que en asociación con una serie de factores de riesgo, generaron casos de mayor severidad y mortalidad 2,3. El 11 de marzo del 2020 la infección fue declarada pandemia por la Organización Mundial de la Salud (OMS) 4.
En el Perú, el Instituto Nacional de Salud reportó el 6 de marzo del 2020 el primer caso confirmado de la COVID-19. A pesar de las estrictas medidas de inmovilización social adoptadas por el gobierno, los casos de infección por el SARS-CoV-2 fueron aumentando considerablemente, el mayor pico de infección de la primera ola fue en julio del 2020. El Perú fue el país con la mayor tasa de mortalidad per cápita en el mundo 5.
Las constantes mutaciones genéticas del virus SARS-CoV-2 ha originado la aparición de diversas variantes que se han extendido por todo el mundo 6, observándose una competencia evolutiva entre ellas, con el desplazamiento de una sobre otras. Dichas mutaciones son las responsables de los diversos picos de contagio observados en todos los países 7,8. Según su impacto en la salud pública, la OMS las ha clasificado en variantes de preocupación (VOC, por sus siglas en inglés) y variantes de interés (VOI, por sus siglas en inglés) 9. Actualmente las variantes con bajos niveles de circulación o no detectadas son clasificadas como variantes de bajo monitoreo, mientras que ómicron pertenece a las VOC y VOI.
El Perú ha registrado cuatro olas; la primera en abril del 2020 generada por la variante B.1.1 como linaje predominante 10,11; la segunda ola durante el primer semestre del 2021 fue causada por la variante lambda/gamma (C37/P1) 12; la tercera ola en enero del 2022 fue causada por la variante ómicron, principalmente (BA.1/BA.2) 12,13; y la cuarta ola en junio del 2022 fue causada por los sublinajes/subvariantes de ómicron (BA.4 y B.A.5) 13,14.
El aislamiento, la tipificación, la caracterización del comportamiento del virus y la determinación de la respuesta humoral a la infección natural y a la vacuna son logros científicos derivados del conocimiento de la biología de los virus. Entre las aplicaciones del aislamiento del virus SARS-CoV-2 tenemos la elaboración de paneles de evaluación de la calidad para garantizar el diagnóstico molecular del SARS-CoV-2; la evaluación de los principios activos con potencial terapéutico; y el desarrollo de diversos métodos de ensayo como las pruebas inmunoenzimáticas que detectan anticuerpos IgM e IgG, la prueba de neutralización por reducción de placas (PRNT) para la titulación de los anticuerpos neutralizantes, y la inmunofluorescencia indirecta (IFI) para la detección de virus y anticuerpos totales. Por lo descrito, este estudio tuvo como objetivo el aislamiento y caracterización el virus SARS-CoV-2 a partir de muestras de hisopado nasal y faríngeo obtenidas al inicio de la pandemia de la COVID-19 en el Perú.
MÉTODOS
Muestras
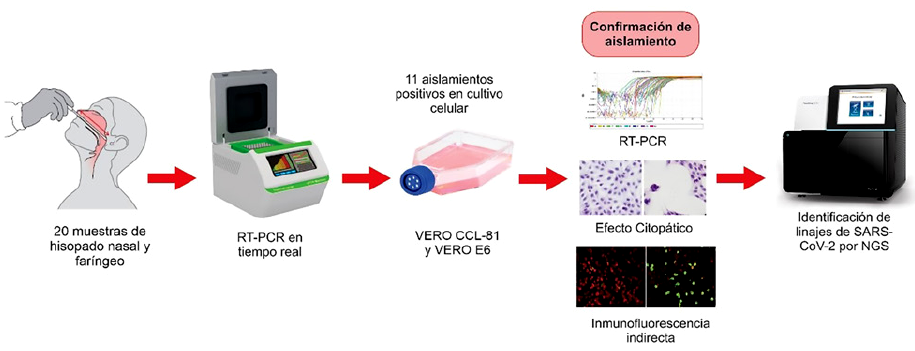
Para el aislamiento viral fueron seleccionadas 20 muestras de hisopado nasal y faríngeo con resultado positivo según el método de RT-PCR para SARS-CoV-2 15. Se consideró valores de umbral de ciclo (Ct) menores a 30 para una carga viral adecuada 16. Las muestras fueron recibidas por el Laboratorio de Referencia Nacional de Virus Respiratorios (LRNVR) del Instituto Nacional de Salud (INS) (Lima-Perú) en abril del 2020. El resumen del aislamiento e identificación de la cepa prototipo se muestra en la figura 1.
Cultivo celular
Las líneas celulares Vero ATCC CCL81 y E6 se utilizaron para el aislamiento del virus de SARS-CoV-2 17,18. Las células crecieron en medio esencial mínimo de Earle (EMEM) suplementado con suero bovino fetal (FBS, marca SIGMA) al 10% y penicilina-estreptomicina-anfotericina al 1%, se cultivó a 37 °C en una atmósfera humidificada que contenía 5% de CO2 18.
Aislamiento y pasajes del virusSARS-CoV-2
El aislamiento del virus SARS-CoV-2 se realizó en las instalaciones del laboratorio de nivel de bioseguridad 3 (NBS-3) del Laboratorio de Microbiología y Biomedicina del INS). Se siguieron los protocolos internacionales establecidos 19 y procedimientos institucionales para el aislamiento de virus respiratorios 20. Las muestras de hisopado positivas a SARSCoV-2 fueron filtradas con membrana de poro de 0,22 µm e inoculadas en la línea celular Vero ATCC CCL-81 y E6 con 90% de confluencia de células, luego de 1 hora de adsorción viral a 37°C, se adicionaron 3 mL de medio EMEM con 2% SBF e incubaron a 37 °C con 5% de CO2. Las células fueron observadas diariamente en microscopio invertido para detectar la aparición de efecto citopático, hasta siete días después de la infección 21,22. Los sobrenadantes se colectaron para la confirmación por RT-PCR, y las células adherentes se usaron para confirmación por IFI.
Titulación viral
La cuantificación del virus SARS-CoV-2 se realizó mediante el ensayo en placa. Se utilizaron placas de cultivo celular con fondo plano de 24 pocillos; las placas fueron sembradas con una suspensión celular de 2,5 x 105/mL de células (Vero ATCC CCL-81) incubadas a 37 °C con 5 % de CO2 durante una noche para lograr una confluencia del 100% 23.
Las células fueron infectadas con el virus realizándose diluciones seriadas en base diez (10-1, 10-2,10-3, 10-4 ,10-5 y 10-6) y se incubaron a 37 °C con 5 % de CO2 durante 60 min. Seguidamente se adicionó a cada pozo 1 mL de medio overlay (medio MEM 2X suplementado con L-glutamina a 2 nM), suero fetal bovino al 10%, 100 unidades/mL de antibiótico y carboximetilcelulosa (CMC de SIGMA al 3% estéril). Las células fueron incubadas a 37°C con 5% de CO2. Después de 5 días, el medio fue retirado, las células se fijaron y colorearon con cristal violeta/formaldehído. Los títulos de SARS-CoV-2 fueron expresados como unidades formadoras de placas por mililitro (UFP/mL) 24,25.
Pruebas de confirmación viral
Se colectó 200 μL del sobrenadante del cultivo, el cual fue inactivado a 56 °C y 450 rpm en termobloque durante 60 minutos 26. Luego fue transportado al laboratorio de nivel bioseguridad 2 (NBS-2). La extracción de RNA fue realizada utilizando el kit de extracción viral de ácido nucleico Zybio (China), a través del método de perlas magnéticas, siguiendo las instrucciones del fabricante 22,27. Las muestras de ARN purificadas se procesaron mediante RT-PCR. El proceso de amplificación se realizó utilizando el kit de un solo paso SuperScript(tm) IV (Invitrogen) siguiendo condiciones estandarizadas 15,28.
Prueba complementaria de inmunofluorescencia indirecta
Se desarrolló la técnica de inmunofluorescencia indirecta (IFI) para detectar el virus SARS-CoV-2 aislado en el cultivo celular. Se utilizó muestra de suero de un paciente en fase convaleciente (diagnóstico positivo para SARS-CoV-2 por RT-PCR), caracterizada por la prueba de neutralización por reducción de placas (PRNT), como fuente de anticuerpos policlonales al virus SARS-CoV-2.
A los 7 días posteriores a la infección de las células Vero ATCC CCL-81 y Vero E6, se colectó el sobrenadante y las células fueron lavadas y resuspendidas con PBS 1X. Se obtuvo una suspensión celular adecuada y se dispensaron en láminas para inmunofluorescencia, estas láminas fueron fijadas con acetona -20 °C/20 minutos; para la detección del virus se adicionó por duplicado 20 µL de una dilución de suero (suero control positivo, suero control negativo a dilución 1/20 en PBS), y control de diluyente PBS, se incubó en cámara húmeda a 37 °C/30 min, se lavó 2 veces con PBS y se dejaron secar a temperatura ambiente; seguidamente, se agregó 20 µL de conjugado anti-IgG humano marcado con FITC (isotiocianato de fluoresceína) + Azul de Evans (100X) en una dilución de (1/160) a todos los pocillos y se incubó en cámara húmeda a 37 °C/30 min protegidas de la luz, se lavaron 2 veces con PBS y se adicionó el líquido de montaje, pH 7,2 - 7,4 22,27. La lectura se realizó en el microscopio de fluorescencia, las láminas se pueden mantener hasta 24 h a 4 °C en la oscuridad. La interpretación se basa en la presencia de fluorescencia (positivo), la ausencia de fluorescencia se considera negativo.
Modelo de dinámica evolutiva in vitro y caracterización
Se utilizó el modelo de la dinámica de evolución viral propuesto por Wood 29, que sugiere 10 pasajes sucesivos en cultivo de la misma línea celular para conocer la adaptación del virus al nuevo entorno del huésped en relación a la virulencia. A partir del aislamiento de una de las muestras positivas, a la que denominamos cepa prototipo (PE/B.1.1/28549/2020), se le realizó 10 pasajes continuos en la línea celular Vero ATCC CCL-81. El cultivo del virus en cada pasaje fue observado hasta la aparición del efecto citopático (5 a 7 días). El sobrenadante fue colectado y centrifugado a 2000 rpm por 10 min a 4°C y se realizaron alícuotas para el análisis por titulación viral y secuenciación genómica.
Secuenciación del genoma completo de aislados de SARS-CoV-2
Las muestras de los aislamientos y de los pasajes de la cepa prototipo (PE/B.1.1/28549/2020) del virus SARSCoV-2, fueron extraídas con el kit de extracción de ADN/ARN viral por inmunomagnetismo MAGBEAD (ZymoBIO-MICS) en la plataforma automatizada Opentrons OT-2. La preparación de la biblioteca se realizó utilizando la prueba COVIDSeq de IIlumina y se secuenció en NextSeq 550 (Illumina(r)) siguiendo las instrucciones del fabricante 30
Análisis bioinformático
La calidad de las lecturas y la eliminación de la contaminación de las lecturas se realizó utilizando FastQC v0.11.9 (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/), y Kraken2 v2.0.8 respectivamente 29. Las lecturas del filtro se asignaron a la secuencia de referencia NCBI (NC_045512) aislada de Wuhan a través del programa BWA v.0.7.17 31. La secuencia de consenso se obtuvo utilizando Samtools v.1.9 e IVAR v.1 32,33. Finalmente, la anotación de secuencias consenso se realizó utilizando NextClade (https://clades.nextstrain.org/), y la designación de linaje se definió utilizando el programa Pangolin v.3.1.2 (https://pangolin.cog-uk.io/). El resumen de mutaciones se calculó con el programa Snipit (https://github.com/aineniamh/snipit) con edición manual, se utilizó como referencia el primer aislado. Los genomas fueron alineados con el programa MAFFT v7.310 (https://mafft.cbrc.jp/alignment/software/) contra la referencia del SARS-CoV-2 (NC_045512). El alineamiento múltiple fue usado para la inferencia filogenética de los aislados, esta fue calculada con el programa RaxML v8.2.12 34 con 1000 de boostrap.
RESULTADOS
Aislamiento viral y efecto citopático
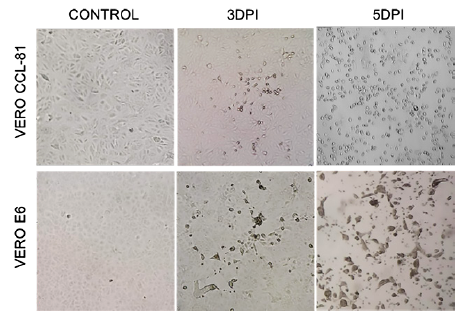
Se aisló el virus SARS-CoV-2 en las dos líneas celulares Vero ATCC CCL-81 y Vero E6. La valoración de la infección viral se realizó por la observación del efecto citopático (ECP). Se observó replicación viral más rápida en células Vero ATCC CCL-81 que en Vero E6, desde el tercer día posterior a la infección. El ECP en células Vero ATCC CCL-81 se caracterizó por focos de células redondas en la superficie de la monocapa celular, expandiéndose a un desprendimiento gradual y homogéneo. Las células Vero E6 mostraron formación de sincitios y desprendimiento solo en algunas muestras, con lento desarrollo del ECP a partir de los 3 días posteriores a la infección.
Se consideró a las células Vero ATCC CCL-81 como óptimas para el aislamiento del virus SARS-CoV-2, por presentar un fenotipo de replicación viral más homogéneo y característico. En la figura 2 se observa el ECP comparativo entre las células Vero ATCC CCL-81 y Vero E6 después de 3 y 5 días posteriores a la infección, con la misma muestra de hisopado nasal y faríngeo. Se observó las células redondas y los sincitios, respectivamente, comparado con la monocapa de las células control.
Confirmación del aislamiento por RT-PCR e IFI
Se confirmaron 11 (55,0%) aislamientos de 20 muestras de hisopado nasal y faríngeo por el método RT-PCR, a partir de los cultivos en la línea celular Vero ATCC CCL-81. Los valores de Ct de los aislamientos se encontraron en el rango de 11 a 14, indicando que hubo replicación viral en comparación al Ct inicial del RTPCR diagnóstico (Ct<30).
La IFI confirmó los mismos 11 (55,0%) aislamientos en las 20 muestras de hisopado nasal y faríngeo, sin diferencias entre las dos líneas celulares Vero ATCC CCL-81 y Vero E6. La figura 3 muestra la identificación del aislamiento del virus SARS-CoV-2 por IFI en células Vero ATCC CCL-81, observándose las células control sin infección (rojas) y las células infectadas con el virus SARS-CoV-2 con fluorescencia (verde).
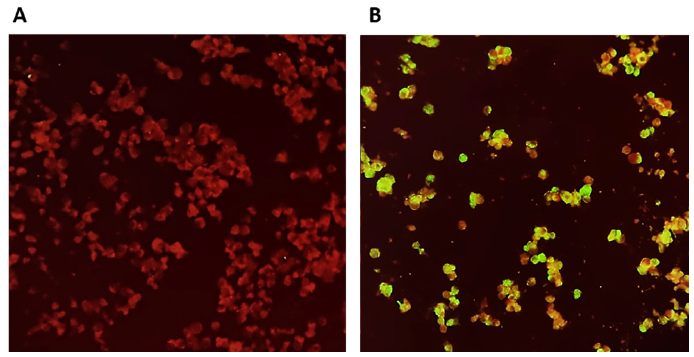
Figura 3 Identificación de aislamiento del virus SARS-CoV-2 por inmunofluorescencia indirecta (IFI). A. Reacción negativa para IFI. B. Reacción positiva para IFI.
Se resalta la muestra con código 28549 cuyo aislamiento fue uno de los que presentaron menores valores de Ct, indicando alta carga viral, además de mostrar el ECP característico y confirmación por IFI. Esta fue seleccionada como cepa prototipo y denominada PE/B.1.1/28549/2020 (Tabla 1).
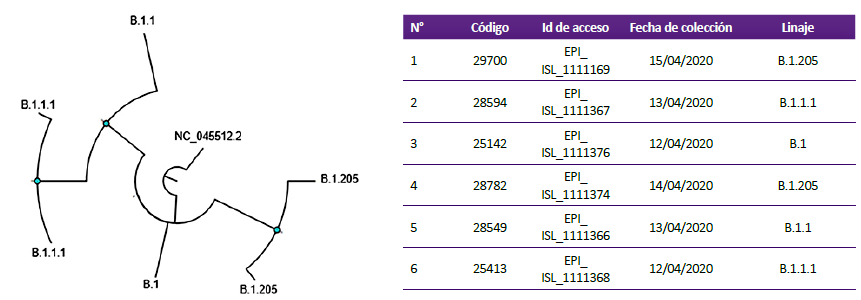
Tabla 1 Resultado del diagnóstico, aislamiento y secuenciación de las 20 muestras de SARS-CoV-2.
| Muestras | Hisopado (nasal y faríngeo) | Pruebas confirmatorias para aislamiento viral | Secuenciación | |||||
|---|---|---|---|---|---|---|---|---|
| N° | Código de muestra | RT-PCR | RT-PCR del aislamiento en Vero ATCC CCL-81 | Vero ATCC CCL-81 y Vero E-6 | NGS | |||
| Ct | Resultado | Ct | Resultado | Efecto citopático | IFI | Linaje (PANGO) | ||
| 1 | 28549 | 21.42 | Positivo | 9.56 | Positivo | Positivo | Positivo | B.1.1 |
| 2 | 28594 | 20.56 | Positivo | 10.45 | Positivo | Positivo | Positivo | B.1.1.1 |
| 3 | 8720 | 19.07 | Positivo | 13.63 | Positivo | Positivo | Positivo | * |
| 4 | 25413 | 16.04 | Positivo | 12.93 | Positivo | Positivo | Positivo | B.1.1.1 |
| 5 | 9116 | 22.3 | Positivo | 12.89 | Positivo | Positivo | Positivo | * |
| 6 | 27171 | 23.7 | Positivo | 33.61 | Positivo | Negativo | Negativo | - |
| 7 | 27148 | 28.52 | Positivo | 36.63 | Positivo | Negativo | Negativo | - |
| 8 | 25400 | 20.3 | Positivo | 12.51 | Positivo | Positivo | Positivo | * |
| 9 | 28755 | 20.79 | Positivo | 10.05 | Positivo | Positivo | Positivo | * |
| 10 | 27195 | 23.58 | Positivo | 35.43 | Positivo | Negativo | Negativo | - |
| 11 | 28782 | 16.83 | Positivo | 12.27 | Positivo | Positivo | Positivo | B.1.205 |
| 12 | 25618 | 29.97 | Positivo | 37.04 | Positivo | Negativo | Negativo | - |
| 13 | 25480 | 28.4 | Positivo | 38.43 | Positivo | Negativo | Negativo | - |
| 14 | 29700 | 21.54 | Positivo | 10.8 | Positivo | Positivo | Positivo | B.1.205 |
| 15 | 25303 | 21.81 | Positivo | 31.91 | Positivo | Negativo | Negativo | - |
| 16 | 25142 | 24.03 | Positivo | 10.59 | Positivo | Positivo | Positivo | B.1 |
| 17 | 284674 | 22.23 | Positivo | 10.71 | Positivo | Positivo | Positivo | * |
| 18 | 28520 | 19.38 | Positivo | 29.92 | Positivo | Negativo | Negativo | - |
| 19 | 23611 | 24.95 | Positivo | 35.98 | Positivo | Negativo | Negativo | - |
| 20 | 25262 | 22.19 | Positivo | 33.05 | Positivo | Negativo | Negativo | - |
*No se secuenció por muestra insuficiente.
NGS: Secuenciación de nueva generación, RT-PCR: Reverse transcription PCR, IFI: Inmunofluorescencia indirecta
Secuenciación del genoma completo de muestras de SARS-CoV-2 y análisis filogenético
De las 11 muestras con aislamiento confirmado del virus SARS-CoV-2 se logró obtener el genoma completo de 6 muestras que fueron analizadas y clasificadas en 4 diferentes linajes PANGO (un B.1, un B.1.1, dos B.1.1.1 y dos B.1.205). El linaje de la cepa prototipo PE/B.1.1/28549/2020 corresponde al B.1.1 (Tabla 1). El análisis filogenético muestra las distancias genéticas entre los diferentes linajes aislados al inicio de la pandemia en comparación al basal NC_045512.2 (Figura 4).
Dinámica evolutiva in vitro
Los pasajes sucesivos de la cepa prototipo PE/B.1.1/28549/2020 muestra la adaptación del virus SARS-CoV-2 en la línea celular Vero ATCC CCL-81, con aumento progresivo del título viral a mayor número de pasajes y una posterior caída en los últimos pasajes (Figura 5A). Esta adaptación muestra el aumento progresivo de la virulencia en la línea celular Vero ATCC CCL-81, observada desde la formación de unidades formadoras de placas (UFP) deficientes, con bordes no definidos en los primeros pasajes, que progresivamente se definen en los pasajes 3 y 4; mientras que en el pasaje 8 se muestran placas de lisis translúcidas, circulares y simétricas con bordes regulares (Figura 5B). La cuantificación de las UFP muestra el aumento progresivo del título viral proporcional al número de pasajes, teniendo como pico el pasaje 7 con un promedio de 59,5 x 105 UFP/mL y posterior declive en los pasajes siguientes (Figura 5C).
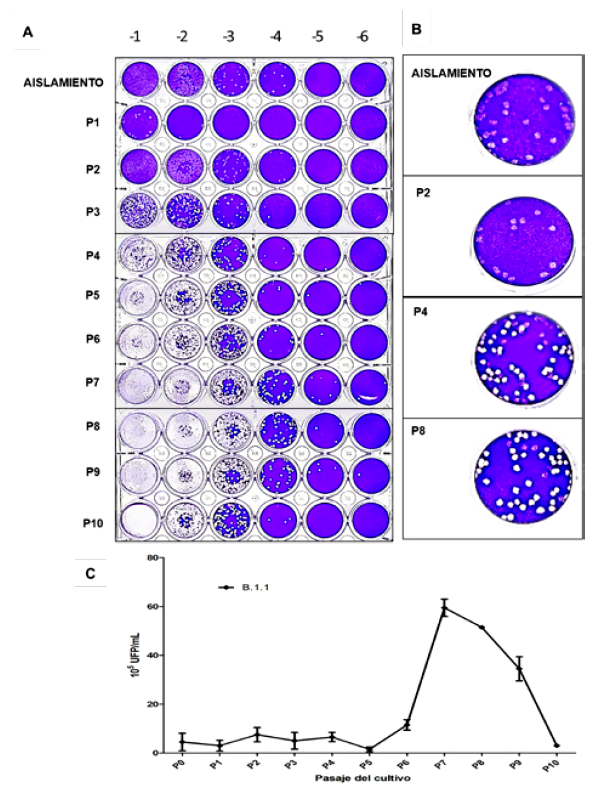
Figura 5 Dinámica mutacional del linaje B.1.1 del virus SARS-CoV-2 (PE/B.1.1/28549/2020) en 10 pasajes sucesivos en células Vero ATCC CCL-81. A. Titulación de los 10 pasajes del virus SARS-CoV-2. B. Aspecto morfológico de las placas virales de los pasajes del virus SARS-CoV-2. C. Cuantificación del título viral en los 10 pasajes. Los datos muestran la media y desviación estándar de 4 repeticiones.
En complemento a las características fenotípicas observadas en los pasajes de la cepa prototipo, la secuenciación en cada pasaje mostró alteraciones en el genoma del linaje asociadas con el fenotipo y carga viral (Figura 6). En el pasaje 2 (P2) de la cepa prototipo se encontró una deleción de 27 pares de base en la posición 21764 - 21790 y mutaciones a nivel de la proteína de la espiga en el pasaje 4 (P4) y pasaje 8 (P8). La mutación del pasaje 4 en la posición 23014 corresponde a un cambio aminoacídico de ácido glutámico por ácido aspártico (E484D) y la segunda mutación en la posición 24507 corresponde al cambio de una serina por leucina (S982L).
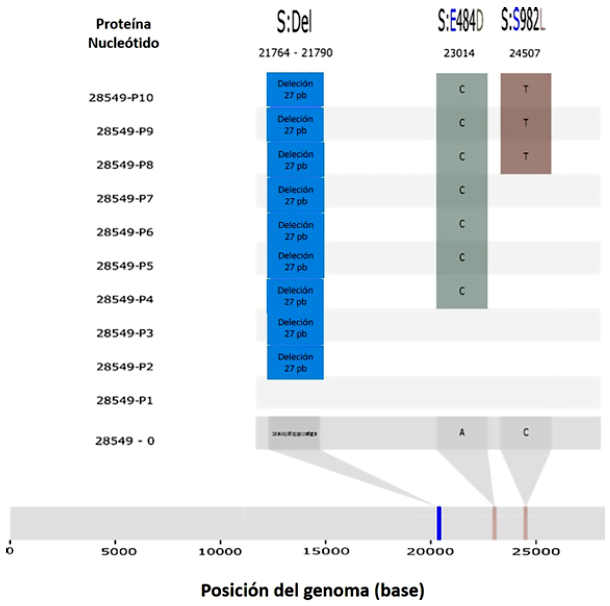
Figura 6 Secuenciación de los 10 pasajes del linaje B.1.1 del virus SARS-CoV-2. Se observa en el pasaje 2 (P2) una deleción de 27 pares de base en la posición 21764 - 21790 y mutaciones a nivel de la proteína de la espiga en el pasaje 4 (P4) en la posición 23014 (E484D) y el pasaje 8 (P8) corresponde a la posición 24507 (S982L).
DISCUSIÓN
Nuestros resultados muestran concordancia con otros reportes que mencionan la mayor probabilidad de aislamiento del virus SARS-CoV-2 asociado a la carga viral de las muestras, con Ct< 24 se logra 80% de recuperación del virus 35. También se muestra la utilidad de las líneas celulares Vero ATCC CCL-81 y Vero E6 en la replicación del virus SARS-CoV-2, con preferencia por la célula Vero ATCC CCL81 como lo reportan algunos estudios 36, a diferencia de otros que indican mayor replicación celular en Vero E6, siendo la más utilizada 17. Para este estudio se utilizó el aislamiento clásico que sigue siendo un estándar de comparación, aunque actualmente se han desarrollado métodos como el de aislamiento automatizado de alta velocidad a gran escala de SARS-CoV-2, a partir de muestras clínicas mediante cocultivo miniaturizado 37.
En esta investigación se confirmó el aislamiento del virus SARS-CoV-2 de 11 muestras de hisopado nasal y faríngeo por el método de RT-PCR e IFI, se observó que en el RT-PCR del aislamiento los valores de Ct fueron menores en comparación con el RT-PCR diagnóstico. Las 9 muestras no aisladas a pesar de tener un RT-PCR de aislamiento positivo los valores de Ct son mayores al PCR diagnóstico, lo cual indica que no hubo replicación viral, esto fue complementado con los resultados negativos del efecto citopático e IFI.
La selección de la cepa prototipo del virus SARS-CoV-2 denominada PE/B.1.1/28549/2020 y la generación de 10 pasajes sucesivos in vitro mostró mutaciones del virus para una mejor adaptación a la célula huésped Vero ATCC CCL-81, en este caso, específicamente a nivel de la proteína de la espiga, causando deleciones y mutaciones. La mutación nucleotídica del pasaje 4 (P4) en la posición 23014 (E484D) permitió a la cepa adaptarse a la línea celular e infectar satisfactoriamente, con aumento de su virulencia. Esta mutación fue reportada en la cepa original de Wuhan (WuhanHu-1, número de acceso de GenBank NC_045512) 38
La segunda mutación del pasaje 8 corresponde a la posición 24507 (S982L), esta mutación disminuye su virulencia, pero demuestra la adaptación genética a la línea celular. Investigaciones con resultados similares indican que el virus SARSCoV-2 cuando es cultivado está bajo una fuerte presión selectiva para adquirir estas mutaciones en diversos ORF del genoma, entre ellos el gen S 39,40. Dichos hallazgos confirman que estas mutaciones se fijan durante la adaptación del virus a las condiciones del ambiente, como lo muestran estudios de ARN in vitro 40,41.
En conclusión, se aisló el virus del SARSCoV-2 en células Vero ATCC CCL-81. El aislamiento se realizó de muestras obtenidas durante la primera ola de la pandemia de la COVID-19 en el Perú. En abril del 2020 fueron identificados 4 linajes PANGO (B.1, B.1.1, B.1.1.1 y B.1.205) circulantes. Los pasajes sucesivos de la cepa prototipo PE/B.1.1/28549/2020 presentó mutaciones en la proteína de la espiga indicando su adaptación a la célula hospedera, que le permitió tener más éxito de supervivencia. La obtención de dicho aislado nos permitió realizar la transferencia del material biológico a otras instituciones de investigación que desarrollaban tecnologías para enfrentar la pandemia de la COVID-19.