INTRODUCCIÓN
La unión consanguínea es una práctica muy controversial por los desenlaces éticos y médicos que implica. Este tipo de relación está presente desde los tiempos bíblicos y su incidencia se ha incrementado en algunas culturas. Actualmente, el 10,4% de la población mundial (500800 millones de personas) tiene uniones consanguíneas 1. A la fecha, en Medio Oriente y el norte de África se observa en el 20 a 50% de su población, entre el uno y el 5% en el sur de Europa, América del Sur y Japón, y en menos del 1% en Europa Occidental, Oceanía y América del Norte 2. Esta práctica es más frecuente en países como Qatar, Arabia Saudita, Pakistán y zonas del sur de la India (54,6%-80,6%) 3.
Estas uniones parentales se fundamentan en diversos factores como religiosos, sociales, conveniencias económicas, aislamiento geográfico, entre otros 4,5. Entre los distintos tipos de consanguinidad la más frecuente a nivel mundial es la relación establecida entre primos hermanos, que corresponde a un coeficiente de endogamia (F) de 1/8 o tercer grado de consanguinidad. Las consecuencias de esta práctica en la descendencia son perjudiciales, observándose un mayor riesgo de presentar trastornos multifactoriales (ej. diabetes mellitus, psicosis, obesidad), partos prematuros, bajo peso al nacer, malformaciones congénitas, errores innatos del metabolismo y trastornos del neurodesarrollo 4.
Los trastornos del neurodesarrollo son alteraciones en el proceso madurativo a nivel motor, habla-lenguaje, socioemocional y cognitivo. Incluyen al retraso del desarrollo psicomotor, discapacidad intelectual, parálisis cerebral, epilepsia, trastorno del espectro autista, discapacidades especificas en el aprendizaje, trastorno del lenguaje y trastorno de déficit de atención/ hiperactividad. Representa el 19% de las atenciones de un hospital pediátrico de referencia nacional peruano 5. Los trastornos del neurodesarrollo poseen un rasgo heredable importante, con herencias de tipo dominante o recesiva 6-9.
Las regiones de homocigosidad (ROH) son segmentos cromosómicos amplios que presentan haplotipos idénticos; y si sobrepasan el 0,5% de los cromosomas autosómicos, podría deberse a uniones consanguíneas 10. Del 2010 al 2019, el análisis cromosómico por micromatrices o CMA (del inglés chromosomal microarray analysis) fue la prueba de elección para la determinación de variantes en el número de copias en pacientes con anomalías congénitas y algunos trastornos del neurodesarrollo 11-13. Adicionalmente, algunas plataformas que realizan CMA también establecen las ROH basado en los polimorfismos de un solo nucleótido o SNP (del inglés single nucleotide polymorphism), el cual indica consanguinidad parental y disomías uniparentales 12 ) .
La consanguinidad no declarada se da durante la elaboración de la historia clínica, cuando no se menciona algún grado de parentesco entre los padres. Esta suele ser descubierta con pruebas genéticas como el CMA. Esta consanguinidad no declarada varía según las poblaciones, en Brasil y en el sur de Florida se encontró el 3,5% (n=21) de casos con un ROH que estimaba un coeficiente de endogamia de 1/4 (primer grado) 14. Chaves et al. al sur de Brasil mostró aproximadamente que el 8,5% eran consanguíneos, de los cuales el primer grado fue el 0,2% y el quinto grado el 4,2% de la población estudiada 15.
El objetivo del presente estudio fue determinar las frecuencias absolutas y relativas de la consanguinidad no declarada según distribución geográfica; así como establecer la diferencia de sus proporciones, en los pacientes con trastornos del neurodesarrollo que acudieron al Servicio de Genética y Errores Innatos del Metabolismo del Instituto Nacional de Salud del Niño-Breña, entre el 2016 y 2021.
MÉTODOS
Diseño de estudio
Se realizó un estudio observacional descriptivo, basado en la revisión de las historias clínicas de los pacientes menores de 18 años que acudieron al Servicio de Genética y Errores Innatos del Metabolismo del Instituto Nacional de Salud del Niño (INSN) entre los años 2016 y 2021. El INSN es un centro especializado de referencia nacional pediátrico y es la única institución que en la actualidad realiza el CMA en Perú.
Población y muestra
Los pacientes incluidos fueron aquellos que se realizaron el CMA por presentar algún trastorno del neurodesarrollo, fueron incluidos los diagnósticos de retraso en el desarrollo psicomotor, trastorno del espectro autista y discapacidad intelectual.
La inclusión de los participantes fue definida mediante el programa ChAS (Chromosome Analyze Suite). Fueron incluidos aquellos pacientes con ROH igual o mayor a 0,5% y con un límite umbral o superior a 5 Mb. Fueron excluidos los que presentaban al menos dos ROH con un tamaño superior de 10 Mb en cromosomas autosómicos, por sospecha de disomía uniparental.
Variables
El coeficiente de endogamia (F) fue medido según las siguientes categorías: primer (ROH = 19 - 38%, F = 1/4), segundo (ROH = 9,4 - 18,9%, F = 1/8), tercer (ROH = 4,7 - 9,3%, F = 1/16), cuarto (ROH = 2,4-4,6%, F = 1/32) o quinto grado (ROH = 0,5 - 2,39%, F = 1/64) 12,16.
Otras variables fueron los departamentos de procedencia de los padres y los abuelos. La procedencia de los abuelos se dividió en tres regiones: costa, sierra y selva. Se dividió el grado de parentesco en cercano (F = 1/32 y 1/64) y muy cercano (F = 1/4 a 1/16).
El cálculo de la consanguinidad poblacional media de cada departamento y de la población en general se realizó a través de esta fórmula 17: Α (α) = ∑ Fi f i , donde; f i = frecuencia de los sujetos con consanguinidad F i y Fi = coeficiente de consanguinidad.
Análisis de datos
Se utilizó datos del último censo de Perú, se extrajo la población total de cada departamento, para luego calcular una frecuencia relativa total (según α) y parcial (según la frecuencia de cada grado de consanguinidad) 18. Para uniformizar los datos y realizar las comparaciones, se hizo el cálculo por cada 10 000 habitantes. Posteriormente, se dividió en tres grupos (baja, moderada y alta) según los percentiles 25 y 75 de los valores obtenidos según grado de consanguinidad y región. Otras variables analizadas fueron sexo, edades de participantes y parentales.
El análisis fue realizado en el pro-grama STATA (Stata Corp. versión 15) y se consideró un valor significativo de p menor a 0,05. El análisis descriptivo fue con frecuencias absolutas y relativas. Se evaluó la distribución normal de las variables numéricas mediante la prueba de Shapiro-Wilk, calculando posteriormente las medidas de tendencia central (media o mediana) o de dispersión (desviación estándar o rangos intercuartílicos), percentiles, valores máximos y mínimos. Se usó la prueba de chi cuadrado con bon-dad de ajuste para comparar las proporciones del grado de consanguinidad del presente estudio con otros estudios.
RESULTADOS
Entre enero del 2016 y enero del 2021, 1143 pacientes se sometieron al CMA, de los cuales 176 fueron excluidos por tener el diagnóstico de anomalías congénitas, talla baja u otras condiciones (ej. sexo ambiguo). Fueron seleccionados 967 pacientes con el diagnóstico de trastorno del neurodesarrollo: retraso del desarrollo psicomotor sindrómico (41,3%), discapacidad intelectual sindrómica (26,6%), discapacidad intelectual aislada (14,8%), retraso del desarrollo psicomotor aislado (14,5%) y trastorno del espectro autista (2,9%). El 57,4% fueron varones, con una mediana de edad de 4 años y rango intercuartílico de 6.
De estos pacientes, 288 (29,8%) tenían un ROH mayor al 0,5%, con una media de 85 Mb. El 58,3% fueron varones (p<0,001), la media de la edad del padre y la madre fue de 37,6 y 34,2 años, respectivamente y la mediana de edad de los pacientes fue de 4 años (Tabla 1).
Tabla 1 Información sociodemográfica, grado de consanguinidad, características y regiones de homocigosidad en niños con trastornos del neurodesarrollo

ROH = regiones de homocigosidad, Kpb = kilos pares de bases; Mpb= mega pares de bases.
La procedencia de los abuelos separada en los grupos de consanguinidad cercano y muy cercano según regiones naturales se encontró que, en ambos conjuntos, el origen de la sierra era el más frecuente (52,8% y 52,2%) con relación a la costa y la selva (Tabla 2).
Tabla 2 Procedencia de los abuelos de 288 niños con trastorno del neurodesarrollo según grado de parentesco (N = 1152)

El grado de consanguinidad parental de mayor frecuencia fue el de quinto grado (25,9%), y en menor frecuencia la relación fue del segundo grado (0,1%). En base al censo de 2017 en Perú, se determinó la frecuencia relativa por población de cada departamento. El departamento con mayor densidad de abuelos de pacientes consanguíneos de primer grado fue Huancavelica (0,86 por 10 000 habitantes), mientras que el departamento con menor frecuencia relativa fue Lima (0,04 por 10 000 habitantes) (Tabla 3 y Figura 1).
Tabla 3 Frecuencias absolutas y relativas (por cada 10 000 habitantes) de consanguinidad no declarada según la procedencia de los abuelos de niños con trastorno del neurodesarrollo
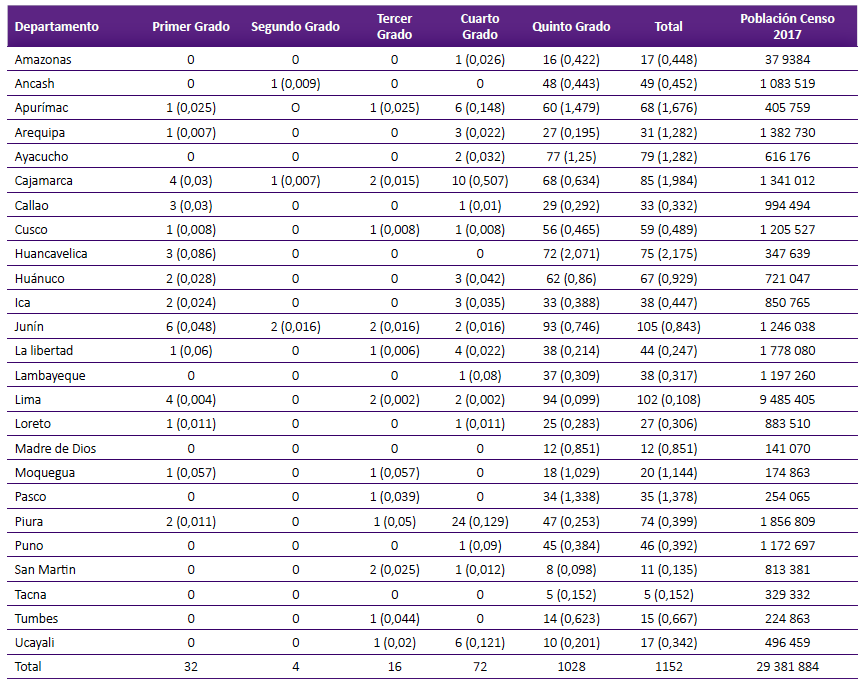
Figura 1 Distribución departamental de la consanguinidad no declarada de primer a tercer grado normalizado según el CENSO de Perú 2017.
El grado de consanguinidad parental declarado previo a la realización del CMA fue del 1,0% (10/967), los cuales representaron al 3,5% de los pacientes con ROH superior al 0,5%. Las uniones consanguíneas declaradas se encontraron en tres de cuarto grado y siete de quinto grado de parentesco (Tabla 4).
Tabla 4 Grado de consanguinidad parental y coeficiente de endogamia de los pacientes con trastornos del neurodesarrollo

a El porcentaje considera como denominador a los 967 pacientes con trastornos del neurodesarrollo incluidos en el presente estudio.
La procedencia departamental de los abuelos maternos, descartando Lima, fueron en mayor frecuencia de Cajamarca (25/288) y en menor cantidad procedieron de Lambayeque (8/288); además, la mayoría de las abuelas maternas de los pacientes provenían de Huánuco (22/288) y la minoría del Callao (10/288).
Respecto a la procedencia de los abuelos paternos, descartando Lima, Junín fue el más frecuente (31/288), mientras que Cusco presentó el menor número (9/288); al analizar la procedencia departamental de las abuelas paternas se observó que Junín presentó un mayor número de casos (30/288) y en menor frecuencia, el departamento de Arequipa (9/288).
DISCUSIÓN
La frecuencia de pacientes con trastornos del neurodesarrollo con consanguinidad no declarada es alta. Las uniones consanguíneas son un factor de riesgo para que la descendencia padezca una enfermedad recesiva autosómica, esto porque existe una alta probabilidad de que la pareja posea alelos idénticos, los cuales estarán ubicados en varios fragmentos a lo largo del genoma, denominadas regiones de homocigosidad.
Las regiones de homocigosidad (ROH) que involucran tramos largos significa que la endogamia es reciente, y esto es debido a una menor tasa de recombinación genética, lo que provoca que los segmentos en común desaparezcan. En cambio, ROH con tamaños menores significaría un evento muy antiguo, y por lo tanto, una mayor posibilidad de recombinación, en consecuencia, menores segmentos cromosómicos compartidos 19. La identificación de dichas regiones se realizan a través de diversas pruebas, las cuales tienen metodologías y puntos de corte diferentes 19. La tecnología de micromatrices detecta las regiones carentes de heterocigosidad. Luego de descartar aquellos casos con disomías uniparentales (DUP), el porcentaje de ROH de los cromosomas autosómicos puede indicar consanguinidad 20-22.
En los estudios en pacientes con trastornos del neurodesarrollo la frecuencia de consanguinidad estuvo entre 3,45% y 35,14% 14,23. En dichos estudios, los umbrales de los tamaños de los segmentos cromosómicos que indican las ROHs fueron distintos; por ejemplo, en Túnez, Brasil, Brasil - EE. UU. y sólo EE. UU, los valores usados fueron de 0,5 Mb, 3 Mb, 5 Mb y 10 Mb, respectivamente 14,24,23,15.
Esto significa que mientras más alto sea el umbral, el número de pacientes con ROH mayor a 0,5% del total de autosomas será menor. En nuestro estudio, con un tamaño de las ROH por encima de 5 Mb se encontró consanguinidad en el 28,7% de los pacientes, frecuencia superior a lo reportado en Brasil, EE. UU. y Brasil - EE. UU. (Tabla 5) 14,15,24. Estas frecuencias son superadas por el hallazgo en Túnez (35,2%), el cual a su vez ha tenido como margen superior para establecer una ROH en 10 Mb 23.
Tabla 5 Detalle del umbral de ROH, grados de consanguinidad y consanguinidad de la población de diversos estudios
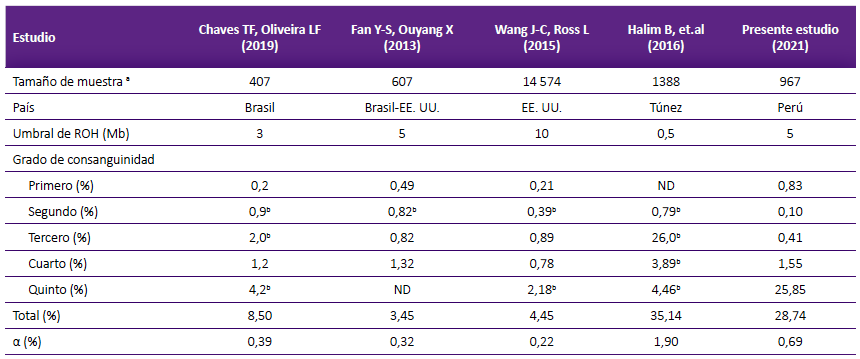
a Pacientes con trastorno del neurodesarrollo, b comparación de proporción con respecto al presente estudio (valor de p < 0,05 en prueba de chi cuadrado con bondad de ajuste). α= Consanguinidad poblacional media, ND= No determinado.
El presente estudio muestra que el primer grado de consanguinidad (padres-hijos, hermanos) tiene una mayor frecuencia en relación con lo observado en los estudios realizados en Brasil (0,2%) y EE.UU. (0,49%). Esto indicaría que las relaciones incestuosas en nuestro medio serían aparentemente más frecuentes 15,24. Al evaluar el segundo grado de consanguinidad (ej. primos hermanos), se encontró por debajo de lo reportado (0,10% vs 0,39-0,9%) 14,15,23,24.
La frecuencia de personas con trastornos del neurodesarrollo que tuvieron padres con un cuarto grado de consanguinidad fue similar a lo publicado anteriormente (1,55 vs 3,89%). Mientras que la consanguinidad parental no declarada de quinto grado en nuestro estudio (28,74%) sólo es superada por lo observado en Túnez (35,14%) y se encuentra muy por encima de lo descrito en Brasil y EE. UU. (3,45 - 8,5%) 14,15,23,24.
El promedio del tamaño de las ROH es muy variable según los diferentes continentes, en América (Central y Sur) se observó un tamaño de aproximadamente 70 Mb, Europa de 3 Mb y en África de 95 Mb 19,23. En nuestro estudio se evidenció que el promedio de ROH fue de 85 Mb, el cual es similar a lo observado previamente, lo cual podría ser explicado porque hay una mayor diversidad genética en la población europea en contraste a África o América donde este efecto es mucho menor, y, por ende, mayor el tamaño de los tramos largos de ROH.
El coeficiente de consanguinidad poblacional (α) en nuestro estudio es superado por reportes previos, es así que en Egipto el α fue 1,45%; en Libia, 2,0%; en el norte de Túnez, 2,1% 25; en Uruguay, 2,2%; en Brasil, 5,0%; en Irán, 37,4%; Emiratos Árabes Unidos, 50,5%; en Qatar, 54,0%; en Pakistán, de 55 a 59%, en Arabia Saudita, 56,0%; y en Kuwait, 68,0% 26,27. Se tiene que precisar que la metodología utilizada en estos estudios fue encuestas en pacientes con enfermedades particulares y en la población general.
La consanguinidad poblacional en Perú (0,69%) es superior a los estudios realizados en EE. UU. (n=14 574; α = 0,22%), Brasil (n=407; α = 0,39%) y en Brasil - EE. UU. (n=607; α = 0,32%). Aunque, se debe precisar que este último no presenta datos de quinto grado de consanguinidad. Podríamos concluir que un factor que modifica el α es la distribución de los casos según el grado de consanguinidad, es así mientras mayor porcentaje de uniones consanguíneas más cercanas (primer al tercer grado) la consanguinidad poblacional será mayor.
El departamento de Junín tenía la mayoría de los casos de primer grado (coeficiente de endogamia de 1/4); y, por otro lado, los departamentos de Arequipa, Apurímac, Cusco, La Libertad, Moquegua y Loreto tuvieron una menor proporción. Sin embargo, al aplicar los últimos datos censales departamentales (2017) y haciendo un cálculo de la frecuencia relativa, nos muestra que el departamento con mayor consanguinidad fue el de Huancavelica.
Dentro de las limitaciones del estudio se encuentra la pérdida de información parcial o total de las historias clínicas. Los SNPs que se utilizaron para el CMA estuvo basado en datos de población extranjera, por lo tanto, los datos respecto al grado de consanguinidad podrían ser eventualmente diferentes, aunque los estudios previos que utilizaron CMA se basaron en estos mismos SNPs.
En conclusión, en Perú los pacientes con trastornos del neurodesarrollo presentan una alta frecuencia de consanguinidad parental no declarada, la cual es más frecuente en departamentos como Huancavelica, Apurímac, Junín y Cajamarca y en la región natural de la sierra; por lo que es importante implementar tecnologías como la secuenciación de segunda o tercera generación, para poder identificar las variantes asociadas a enfermedades con herencia recesiva autosómica. Es necesario formar un sistema multidisciplinario destinados a la prevención y que brinde asesoramiento genético sobre el riesgo de las uniones consanguíneas y el impacto en la salud.















