Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Revista de Investigaciones Veterinarias del Perú
Print version ISSN 1609-9117
Rev. investig. vet. Perú vol.21 no.2 Lima July/dic. 2010
ARTÍCULOS PRIMARIOS
Fenotipo y genotipo del virus de la diarrea viral aislado de bovinos en el Perú
Phenotype and genotype of bovine viral diarrhoeavirus isolated in Peruvian cattle
Mariluz Araínga R.1,2, Hermelinda Rivera G.1.3, Juan Carlos Huamán G.1, Alberto Manchego S.1
1 Laboratorio de Microbiología y Parasitología Veterinaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima
2 E-mail: mariluz.arainga@gmail.com
3 E-mail: hriverag2005@yahoo.es
RESUMEN
El objetivo del presente estudio fue determinar el fenotipo y genotipo de cepas del Virus de la Diarrea Viral Bovina (VDVB) aisladas de bovinos de diversos lugares del Perú. Entre 1998 y 2007 se colectó muestras de tejidos de fetos abortados, sangre, suero o plasma de bovinos con infección aguda o portadores del virus procedente de las principales cuencas lecheras del país para la búsqueda del antígeno del VDVB mediante inmunofluorescencia y ELISA de captura. Las muestras positivas al antígeno viral fueron cultivadas en monocapas de células de cornete nasal de feto bovino (CNB), libre del VDVB hasta el tercer pasaje, para determinar el biotipo. Las muestras positivas, tanto de los tejidos como del tercer pasaje, fueron procesadas por la técnica RT-PCR en tiempo real, para determinar el genotipo viral. Se aislaron 41 cepas del VDVB en CNB. El 85.4% (35/41) correspondieron al fenotipo no citopático y 14.6% (6/41) al citopático. El 45.5% (25/55) y el 74.5% (41/55) de las muestras de tejidos y del 3er pasaje en CNB, respectivamente, resultaron positivas al VDVB mediante la técnica de RT-PCR en tiempo real. Todas las cepas pertenecieron al VDVB-1. Los resultados enriquecen la epidemiología de la DVB en el país y sugieren la ausencia o baja prevalencia de cepas del VDVB-2 en la población bovina de las principales cuencas lecheras.
Palabras clave: virus de la diarrea viral bovina, ARN, fenotipo, genotipo, PCR en tiempo real, cultivo celular
ABSTRACT
The aim of this study was to determine the phenotype and genotype of Bovine Viral Diarrhoea Virus (BVDV) in Peruvian cattle. During the period 1998-2007, tissues of aborted foetus, blood, serum or plasma samples of bovine with acute infection or persistently infected from dairy herds of various locations in the country were collected and the BVDV antigen was detected by immnunofluorescence or capture ELISA tests, and positive samples were inoculated en BVDV free BT cells and cultured until third passage to determine the biotype. Both animal tissues and third passage of positive samples were tested by a Real Time Reverse Transcription-Polymerase Chain Reaction (RT-PCR) for genotyping the strains. Forty one BVDV strains were isolated in BT cells. Eighty five percent (35/41) of the isolates were non-cytopathic and 14.6% (6/41) cytophatic. Forty five and a half percent (25/55) and 74.5% (41/55) of the animal tissues and from the 3rd passage samples respectively, were positive to BVDV by Real Time RT-PCR test. All isolates belonged to BVDV-1 genotype. These results contribute to a better understanding of the epidemiology of the bovine viral diarrhoea disease in Peru and suggest that BVDV-2 is absent or has low prevalence in dairy herds.
Key words: bovine viral diarrhea virus, RNA, phenotype, genotype, real time PCR, cell Cultura
INTRODUCCIÓN
La industria lechera en el Perú está concentrada en las cuencas de Arequipa, Lima y Cajamarca (MINAG, 1996), aunque también se encuentra en zonas como Trujillo (La Libertad), valle del Mantaro (Junín) y valles interandinos con disponibilidad de recursos forrajeros. La ganadería lechera afronta serias limitantes que afectan su desarrollo, entre ellas, enfermedades infecciosas como el virus de la diarrea viral bovina (VDVB).
La DVB es una enfermedad ampliamente distribuida en el país, con prevalencias mayores al 50%, siendo causa importante de fallas reproductivas y un componente del complejo respiratorio en terneros (Rivera et al., 1994). Las pérdidas estimadas a causa de esta enfermedad son muy variables, dependiendo de las condiciones de cada establo; pues ocasiona reducción en la producción de leche, menor tasa de concepción, desórdenes respiratorios, abortos, defectos congénitos y retardo en el crecimiento, entre otros (Houe, 2003). El virus puede cruzar la barrera placentaria, infectar al feto y ocasionar el nacimiento de un ternero con infección persistente durante la vida postnatal, convirtiéndose en el principal diseminador del virus (Becher et al., 1997). Los animales persistentemente infectados (PI) posibilitan la generación de los dos fenotipos virales, citopático y no citopático, pudiendo, además, desarrollar una enfermedad de carácter mortal, denominada Enfermedad de las Mucosas (Baker, 1987).
Los métodos de diagnóstico, como serología con anticuerpos policlonales o monoclonales, pruebas de neutralización viral, pruebas de Reacción en Cadena de la Polimerasa (PCR) y análisis filogenéticos sostenidos por estudios de secuenciamiento genético de pestivirus, han identificado varios genotipos pestivirales: virus de la diarrea viral bovina genotipo 1 (VDVB-1), virus de la diarrea viral bovina genotipo 2 (VDVB-2), virus de la enfermedad de la frontera (VEF), virus de la peste porcina clásica (VPPC) y una quinta especie tentativa, representada por pestivirus de animales silvestres y atípicos (Ridpath et al., 1994; Paton 1995; Becher et al., 1997, 2003).
EL VDVB ha sido aislado en varios países europeos (Wolfmeyer et al., 1997; Letellier et al., 1999; Vilcek et al., 1999, 2005; Falcone et al., 2001; Tajima et al., 2001), EEUU y Canadá (Ridpath et al., 1994; Chul et al., 2005; Ridpath, 2005), Japón (Sakoda et al., 1999), y en América Latina (Flores et al., 2000; Jones et al., 2001; Pizarro-Lucero et al., 2006). Asimismo, se han desarrollado estudios de genotipificación y cuantificación del VDVB, usando para ello técnicas más rápidas, con alta sensibilidad y especificidad, como es el PCR en Tiempo Real (Letellier y Kerkhofs, 2003).
En el país, la información sobre el VDVB se limita a la prevalencia de la enfermedad, pero no se conocen las características biomoleculares de las cepas que circulan en la población bovina. El objetivo del presente estudio fue conocer el fenotipo y genotipo del virus y su distribución en la población bovina de varios lugares del país como una vigilancia de la emergencia de cepas patógenas y para un mejor conocimiento de la epidemiología de la enfermedad.
MATERIALES Y MÉTODOS
Lugar de Estudio
El estudio se llevó a cabo en el Laboratorio de Virología y el Laboratorio de Diagnóstico Molecular de la Facultad de Medicina Veterinaria (FMV) de la Universidad Nacional Mayor de San Marcos (UNMSM), Lima.
Muestras
Se seleccionaron 55 muestras de bazo, timo, riñón, pulmón de fetos bovinos abortados y muestras de sangre, suero o plasma de bovinos con sospecha de infección aguda o PI, procedentes de establos lecheros de Arequipa, Cajamarca, Junín, La Libertad, Lima y Puno, que fueron remitidas para la detección de antígenos del VDVB entre 1998 y 2007 y que resultaron positivas o sospechosas a antígenos del VDVB mediante pruebas convencionales (Inmunofluorescencia y ELISA de captura).
Determinación del Antígeno Viral
Se determinó la presencia del antígeno viral en los tejidos mediante la técnica de inmunofluorescencia directa, usando un conjugado comercial (Ames, Iowa, USA), siguiendo el protocolo disponible en el Laboratorio de Virología de la FMV; mientras que las muestras de sangre y suero sanguíneo fueron analizadas mediante ELISA de captura (kit comercial Moredun-UK o IDEXX-USA). Todas las muestras positivas y sospechosas se conservaron en congelación a -35 ó -70 ºC hasta el momento del aislamiento viral. Las muestras de sangre que resultaron positivas a virus fueron centrifugadas a 3000 g x 10 minutos para la obtención de leucocitos y plasma, antes de ser congeladas.
Aislamiento Viral y Determinación del Fenotipo
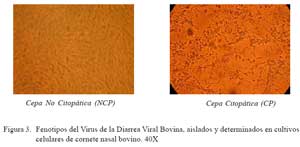
Las muestras de tejido que resultaron positivas a virus fueron homogenizadas al 10% Peso/Volumen en medio MEM, y se centrifugaron para obtener el sobrenadante. Este, así como leucocitos y suero sanguíneo se inocularon en monocapas de cornete nasal de feto bovino (CNB), cultivadas en placas de 24 hoyos en una incubadora a 37 ºC con una atmósfera de 5% de CO2, durante 5 días, y observadas diariamente en busca de lesión celular para determinar el fenotipo (biotipo) viral. Cumplidos los 5 días, se cosecharon las células infectadas, para luego hacer pasajes ciegos (hasta por tercera vez) en nuevas monocapas de células. En cada pasaje se realizaron pruebas de identificación de antígenos virales con la técnica de inmunoperoxidasa (kit comercial Trop-IIP Pestivirus, Trop-Bio, Australia). Las muestras positivas al virus y que no mostraron efecto citopático fueron consideradas cepas No Citopáticas (NCP) y las que presentaron lesiones celulares fueron consideradas cepas Citopáticas (CP).
Determinación del Genotipo Viral
Se realizó la extracción del ARN de las 55 muestras originales y de las muestras del tercer pasaje en cultivo celular, utilizando el kit comercial de extracción y purificación de ARN SV Total RNA Isolation System (Promega, EEUU). Además, se extrajo el ARN de las cepas virales controles NADL (VDVB genotipo 1) y cepa 125 (VDVB genotipo 2), y del cultivo celular normal (cornete nasal bovino), así como de las muestras de suero de bovinos de un hato negativo al VDVB. El ARN aislado fue cuantificado usando un espectrofotómetro de luz UV visible (Varian - Casy 50 Bio) y conservado en congelación a -70 °C.
Para la reacción de RT-PCR en Tiempo real se utilizó el kit DyNamo SYBR Green 2-Step qRT-PCR F-430L (Finnzymes, Finlandia), considerando, además, las especificaciones para cada set de primers específicos (Cuadro 1). Brevemente, la técnica consistió en:
Extracción del ARN viral y de los controles
Todos los ARN fueron extraídos de 100 μl de la muestra utilizando 175 μl del buffer de lisado del ARN siguiendo las instrucciones del fabricante del kit SV (Promega). La lisis se realizó a 70 °C por 3 minutos y los tubos enfriados en hielo. Los tubos conteniendo el lisado fueron centrifugados a 14 000 x g a 20-25 °C por 10 minutos continuándose con la purificación del ARN por centrifugación. Finalmente el ARN purificado fue eluido con 100 μl de agua libre de nucleasas y fue almacenado a -70 °C hasta su procesamiento.
PCR en Tiempo Real en 2 pasos
a) Transcripción Reversa (Síntesis de ADNc). Para la Transcripción Reversa se utilizó el kit DyNAmo SYBR Green 2-step qRT-PCR F430L (Finnzymes, Finlandia) que contiene los reactivos necesarios para la síntesis de ADNc y para la amplificación del ADN (PCR) por los primers específicos. Para la síntesis del ADNc se preparó la mezcla de reactivos que contiene el kit en un volumen de 20 μl al cual se le añadió el ARN extraído y purificado colocándose en el equipo termociclador PTC 200 (Peltier Therme Cycler) Chromo 4 (detector continuo de fluorescencia) (MJ Research, UK) por 10 minutos a 25 °C, 30 minutos a 37 °C, 5 minutos a 85 °C , y una vez finalizado se mantuvo a 4 °C.
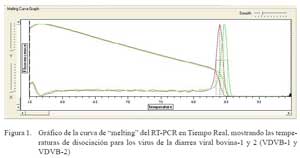
b) PCR en Tiempo Real. Se preparó una mezcla de reacción final de 20 μl/muestra/ genotipo por cada set de primers (VDVB-1 y VDVB-2) y se programó en el termociclador de acuerdo a la temperatura de hibridización (annealing) de los primers específicos. El programa constó de 15 minutos a 95 °C y 35 ciclos de: 10 segundos a 94 °C, 30 segundos a la temperatura promedio de los primers (52 °C), 30 segundos a 72 °C, luego 10 minutos a 72 °C. Se continuó con 55 °C con incremento de 0.2 °C cada 2 segundos hasta 95 °C y al finalizar se mantuvo a 4 °C. Al término de la prueba, se visualizó las curvas de amplificación (CT) en forma logarítmica y se analizó las temperaturas de disociación (TD) para cada genotipo viral de referencia VDVB-1 y VDVB-2 que estuvieron entre 84 y 85 °C, respectivamente (Fig. 1), comparando con la TD de los aislados de campo (Fig. 2).
Análisis de Datos
La positividad al VDVB-1 y VDVB-2 fue determinada por el análisis de la temperatura de disociación (Melting), del programa Opticon Monitor del equipo del PCR en Tiempo Real. El número de muestras positivas a antígenos virales, fenotipos y genotipos se representan mediante porcentajes.
RESULTADOS
Las 55 muestras que resultaron positivas o sospechosas al VDVB por inmunofluorescencia (IF) o ELISA de captura, así como al tercer pasaje en cultivo celular de CNB se muestran en el Cuadro 2, en tanto que los resultados de las muestras originales y del tercer pasaje en CNB mediante la técnica molecular de RT-PCR en tiempo real, usando primers para la detección de Pestivirus, se muestran en el Cuadro 3.
La genotipificación del VDVB usando el ARN extraído del tercer pasaje en cultivo celular de las muestras, determinada mediante RT-PCR en tiempo real, usando primers específicos para cada genotipo viral, indicó que 14 de las 55 muestras fueron positivas el genotipo VDVB-1 y ninguna al genotipo VDVB-2.
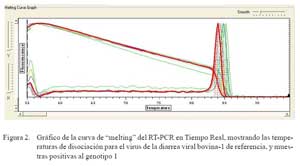
La Fig. 1 muestra las curvas de disociación para las cepas del VDVB de referencia, y genotipos 1 y 2 observándose temperaturas de disociación de 84 y 85 ºC, respectivamente. Asimismo, en la Fig. 2, se compara los productos amplificados del VDVB- 1 de referencia y de las cepas aisladas, usando primers específicos para el genotipo 1.
En relación al fenotipo viral de los aislados, el 85.4% (35/41) fue de cepas NCP y el 14.6% (6/41) fue CP (Fig. 3 y fig. 4).
DISCUSIÓN
Las diferencias a nivel molecular han permitido agrupar las cepas como genotipo 1 y 2 y en subgenotipos dentro de cada genotipo. Se sabe que las cepas del VDVB exhiben una amplia variación antigénica, que son citopáticas y no citopáticas. En el Perú, tal clasificación no se había realizado, aunque en un análisis preliminar se ha reportado la presencia del genotipo 1b (Ståhl et al., 2009).
En este estudio, el 65.5% (36/55) de muestras positivas y 34.5% sospechosas del VDVB, diagnosticadas mediante las pruebas de IF y ELISA de captura, representan cepas de campo aisladas de animales de diver sos lugares y situaciones climáticas del país, entre 1998-2007 (Cuadro 2). La utilización de las pruebas de IF o ELISA permite la identificación rápida y económica del VDVB. Sin embargo, la sensibilidad y especificidad de estas pruebas pueden ser influenciadas por factores como la gran diversidad antigénica viral, calidad de los reactivos utilizados, y tipo de muestras, entre otros, por lo que un resultado positivo confirma la presencia del antígeno viral, pero un resultado sospechoso o negativo no descarta definitivamente la presencia del mismo (Saliki y Dubovi, 2004; Mars y Van Maanen, 2005).
Una de las razones de inocular las 55 muestras en una línea celular secundaria de CNB libre de VDVB endógeno fue definir los resultados obtenidos directamente de los tejidos. Luego de tres pasajes sucesivos en las células de CNB, la identificación del antígeno pestiviral utilizando mAbs contra la proteína P125/NS23 (región muy conservada entre los pestivirus) evidenció un 78.2% (43/55) de muestras positivas a Pestivirus (Cuadro 2). La técnica de aislamiento del virus en cultivo celular, a pesar de los avances en técnicas de diagnóstico del VDVB, continúa siendo la técnica estándar de oro, sobre todo si se utilizan células de CNB y de testículo bovino, que son las más sensibles para aislar VDVB (Saliki y Dubovi, 2004).
El análisis molecular de un fragmento de la región 5´ UTR del ARN extraído de las muestras originales indicó que solo el 45.5% (25/55) fueron positivas al VDVB, pero el análisis de esta misma región del ARN extraído de las muestras del tercer pasaje en CNB, mostró una positividad al VDVB de 74.5% (41/55), indicando que en el trabajo no solo se aisló ARN viral, sino también virus viable (Cuadro 3). El mayor porcentaje de positividad detectado en las muestras del tercer pasaje evidencia recuperación viral producto de la replicación en los sucesivos pasajes en CNB. El menor número de muestras originales positivas obtenido al analizar este fragmento podría deberse a la degradación del ARN viral, debido a factores como tiempo de permanencia en el laboratorio (desde 1998) e inadecuada conservación de las muestras, sobre todo, durante los primeros años de colección (a -20 ó -30 ºC). Las moléculas del ARN viral son más fácilmente degradadas por las RNasas presente en los tejidos comparados con las moléculas de ADN.
Todos los aislados positivos al VDVB pertenecieron al genotipo 1 y no se detectó alguno del genotipo 2, según las Curvas de Melting. El genotipo 1 es el más difundido en poblaciones bovinas de muchos países como EEUU (Vilcek et al., 1999; Chul et al., 2005), de donde el Perú importó bovinos sin restricción respecto al VDVB, sobre todo a fines de la década del 80.
Las cepas del VDVB-1 son conocidas como las cepas clásicas utilizadas en la elaboración de vacunas, en pruebas diagnósticas, y en investigación. Ejemplo de este genotipo son los virus NADL, Singer, C24V, entre otros, aislados en EEUU, pero distribuidos globalmente (Fulton et al., 2005). En el Perú, el único tipo de vacuna utilizada para el control del VDVB es la vacuna a virus inactivado, por lo que sería poco probable que el virus de origen vacunal forme parte del ejambre viral en el país. En un estudio preliminar con pocas muestras se determinó la presencia del genotipo 1, subgenotipo 1b del VDVB en el Perú (Ståhl et al., 2009); además, se conoce que el subgenotipo 1b es el más predominante en la población bovina mundial (Ridpath et al., 1994, 2000; Fulton et al., 2003, 2005).
La no detección del VDVB-2 pude deberse a una baja prevalencia o al bajo número de muestras de campo analizadas, comparado con el número de cepas analizadas en otros estudios donde se ha detectado ambos genotipos (Ridpath et al., 1994; Sakoda et al., 1999; Vilcek et al., 1999; Flores et al., 2000; Falcone et al., 2001; Jones et al., 2001; Tajima et al., 2001; Chul et al., 2005; Pizarro- Lucero et al., 2006), aún cuando el VDVB-1 es más frecuentemente detectado en comparación al VDVB-2 (Chul et al., 2005). El VDVB-2 ha sido aislado de brotes agudos de DVB o síndrome hemorrágico en EEUU y Canadá a fines de la década del 80 (Corapi et al., 1990; Pellerin et al., 1994; Carman et al., 1998). Sin embargo, no todas las cepas del VDVB-2 aisladas han sido asociadas con el síndrome hemorrágico, ya que fueron aisladas de terneros PI nacidos de vacas vacunadas con VDVB-1 (Ridpath et al., 1994, 2000). Una de las razones de la ausencia o baja prevalencia del VDVB-2 en el país se debería a que la vacuna empleada es a virus inactivado y la vacunación no es una práctica común. No obstante, y si bien el VDVB-2 está presente en Norte América, también se ha detectado en países de América Latina como Brasil (Wageck et al., 1998; Flores et al., 2000), Argentina (Jones et al., 2001) y Chile (Pizarro-Lucero et al., 2006).
La diversidad genética del VDVB, también expresada como diversidad antigénica (Fulton et al., 2003), tiene impacto en el control de la enfermedad a través del uso de vacunas, pues la mayoría de ellas emplea vacunas a virus vivo modificado o muerto, y tiene como antígeno al VDVB-1a y VDVB 2a. Se ha demostrado que anticuerpos dirigidos contra el VDVB-1b no neutralizan eficientemente al VDVB-1a (Ridpath, 2005). Las potenciales cepas del VDVB y su diversidad genética deben ser consideradas cuando se diseña programas de control, tanto a nivel de hatos como a nivel nacional (Bolin y Grooms, 2004), por lo que las 41 cepas del VDVB-1 aisladas deben ser secuenciadas para conocer los subgenotipos presentes y establecer su distribución en las zonas de procedencia.
La técnica de RT-PCR en Tiempo Real cualitativo que es la característica del SYBR Green 2 step, ha sido establecido en el laboratorio para diagnóstico e investigación del VDVB en el país. Esta técnica amplifica el producto de PCR y lo detecta durante cada ciclo por la liberación de un colorante fluorescente, provee más información permitiendo una mejor estandarización de la técnica comparado con el PCR convencional, evita el consumo de reactivos para análisis posteriores al PCR, y disminuye los riesgos de contaminación post PCR (Wirz et al., 1993). Las secuencias de nucleótidos en la región 5' UTR y en la región que codifica la proteína NS3, son las más conservadas de los pestivirus; por lo que la RT-PCR usando cebadores específicos para estas regiones ofrece una alta sensibilidad (Sandvik, 1999). Diversos estudios sugieren que la detección y el análisis filogenético de la región 5' UTR, es útil para una rápida caracterización de aislados de campo (Letellier et al., 1999; Sakoda et al., 1999). El desarrollo de la prueba de PCR en tiempo real ha permitido la rápida detección de otros pestivirus, tal como el VPPC (Risatti et al., 2005); así como la genotipificación del VDVB en pools de muestras de sangre (Letellier y Kerkhofs, 2003).
El cultivo de las muestras en CNB permitió determinar el fenotipo viral, observándose que el 14.6 y 85.4% de las muestras positivas fueron citopáticas y no citopáticas, respectivamente (Fig. 3 y fig 4), similar a lo reportado en la literatura (Fulton et al., 2000; Chul et al., 2005). La presencia de animales PI y de cepas citopáticas en la población bovina del país sugiere la existencia de riesgos de la ocurrencia de la enfermedad de las mucosas (EM), que es una de las formas clínicas de la DVB de carácter fatal, no reportada aún en el país. Está bien documentado que el VDVB-NCP es la fuente para la aparición del VDVB-CP, como resultado de una mutación espontánea de virus NCP dentro de un animal PI (Brownlie, 1991; Meyers et al., 1992; Bolin, 1995; Tautz et al., 1998).
Los cultivos celulares utilizados durante el aislamiento viral fueron comprobados de ser libres del VDVB por diferentes pruebas convencionales y finalmente por pruebas moleculares como el RT-PCR en tiempo real, demostrando ser negativos al virus en el cornete nasal bovino y el suero fetal bovino, descartando la probabilidad de contaminación con las cepas analizadas durante el estudio por lo que todos los virus aislados en este estudio son cepas de campo genuinas al VDVB. Esta evaluación fue necesaria debido a que el VDVB es un contaminante frecuente de productos biológicos derivados de bovinos, en particular, el suero fetal bovino comercial, componente de los medios de cultivos celulares (Saliki y Dubovi, 2004).
Hay una tendencia mundial para erradicar al VDVB, debido a que se conoce bien la patogénesis y las características biomoleculares de este virus; por tanto, los estudios epidemiológicos y la disponibilidad de herramientas diagnósticas modernas podrían servir de base para la elaboración de un programa nacional de control-erradicación del VDVB en el Perú, que al inicio podría ser de tipo voluntario como está ocurriendo actualmente en dos hatos en Arequipa.
CONCLUSIONES
-
Se determinaron 41 cepas del VDVB de bovinos procedentes de diversos lugares del Perú.
-
Se encontró los fenotipos virales no citopático (85.4%) y citopático (14.6%) entre las cepas de campo del VDVB aisladas.
-
Todas las cepas de campo del VDVB fueron de genotipo 1.
Agradecimiento
El trabajo fue parcialmente financiado por el Consejo Superior de Investigaciones de la UNMSM. Los autores agradecen al Dr. Raúl Rosadio por sus valiosas sugerencias en la redacción del manuscrito.
LITERATURA CITADA
1. Baker J. 1987. Bovine viral diarrhoea virus: A review. JAVMA 190: 1449-1458.
2. Becher P, Orlich M, Shannon A, Horner G, Konig M, Thiel H-J. 1997. Phylogenetic analysis of pestivirus from domestic and wild animals. J Gen Virol 78: 1357-1366.
3. Becher P, Avalos Ramirez R, Orlich M, Cedillo Rosales S, König M, Schweizer M, Stalder H, Schirrmeier H, Thiel H-J. 2003. Genetic and antigenic characterization of novel pestivirus genotypes: implications for classification. Virology 311: 96-104.
4. Bolin S. 1995. The pathogenesis of mucosal disease. Vet Clin N Am-Food A 11(3): 489-500.
5. Bolin S, Grooms D. 2004. Origination and consequences of bovine viral diarrhea virus diversity. Vet Clin N Am- Food A 20: 51-68.
6. Brownlie J. 1991. The pathways for bovine diarrhoea virus biotypes in the pathogenesis of disease. Arch Virol 3: 79-96.
7. Carman S, Van Dreumel T, Ridpath J, Hazlett M, Alves D, Dubovi E, Tremblay R, Bolin S, Godkin A, Anderson N. 1998. Severe acute bovine viral diarrhea in Ontario. J Vet Diagn Invest 10: 27-35.
8. Chul B, Walz P, Kennedy G, Kapil S. 2005. Byotipe, genotipe, and clinical presentation associated with bovine viral diarrhea virus (BVDV) isolates from cattle. Inter J Appl Res Vet Med 3: 319- 326.
9. Corapi WV, Elliott RD, Frech TW, Arthur DG, Bezek DM, Dubovi EJ. 1990. Trombocitopenia and hemorrhages in veal calves infected with bovine viral diarrea virus. J Am Vet Med Assoc 196: 590- 596.
10. Falcone E, Cordioli P, Sala G, Tarantino M, Tollis M. 2001. Genotyping of bovine viral diarrhoea viruses isolated from cattle in Northern Italy. Vet Res Commun 25: 161-167.
11. Flores E, Gil L, Botton S, Weiblen R, Ridpath J, Kreutz L, Pilati C, Driemeyer D, et al. 2000. Clinical, pathological and antigenic aspects of bovine viral diarrhoea virus type 2 isolates identified in Brazil. Vet Microbiol 77: 175-183.
12. Fulton R, Purdy C, Confer A, Saliki J, Loan R, Briggs R, Burge L. 2000. Bovine viral diarrhoea viral infections in feeder calves with respiratory disease: Interactions with Pasteurella spp., parainfluenza-3 virus, and bovine respiratory syncytial virus. Can J Vet Res 64: 151-159.
13. Fulton R, Ridpath J, Confer A, Saliki J, Burge L, Payton M. 2003. Bovine viral diarrhoea virus antigenic diversity: impact on disease and vaccination programmes. Biologia 31: 89-95.
14. Fulton RW, Ridpath JF, Ore Sh, Confer AW, Salike JT, Burge LJ, Payton ME. 2005. Bovine viral diarrhea virus subgenotypes in diagnostic laboratory accessions: distribution of BVDV 1a, 1b, and 2a subgenotypes. Vet Microbiol 111: 35-40.
15. Houe H. 2003. Economic impact of BVDV infection in diaries. Biologia 31: 137-143.
16. Jones L, Zandomeni R, Weber L. 2001. Genetic typing of bovine viral diarrhea virus isolates from Argentina. Vet Microbiol 81: 367-375.
17. Letellier C, Kerkhofs P, Wellemans G, Vanopdenbosch E. 1999. Detection and genotyping of bovine diarrhea virus by reverse transcription-polymerase chain amplification of the 5 untranslated region. Vet Microbiol 64: 155-167.
18. Letellier C, Kerkhofs P. 2003. Realtime PCR for simultaneous detection and genotyping of bovine viral diarrhea virus. J Virol Meth 114: 21-27.
19. Mars M, Van Maanen C. 2005. Diagnostic assays applied in BVDV control in the Netherlands. Prev Vet Med 72: 43-48.
20. Meyers G, Tautz N, Stark R, Brownlie J, Dubovi E, Collet M, Thiel H-J. 1992. Rearrangement of viral sequences in cytopathogenic pestiviruses. Virology 191: 368-386.
21. [MINAG] Ministerio de Agricultura, Perú. 1996. Producción pecuaria e industria avícola. Presidencia de la República. Documento de consulta. Lima: MINAG.
22. Paton JD. 1995. Pestivirus diversity. J Comp Path 112: 215-236.
23. Pellerin C, van den Hurk J, Lecomte J, Tijssen P. 1994. Identification of a new group of bovine viral diarrhea virus strains associted with severe outbreak and high mortalities. Virology 203: 260- 268.
24. Pizarro-Lucero J, Celedon M, Aguilera M, de Calisto A. 2006. Molecular characterization of pestiviruses isolated from bovines in Chile. Vet Microbiol 115: 208-217.
25. Ridpath J, Bolin S, Dubovi E. 1994. Segregation of bovine viral diarrhea virus into genotypes. Virology 205: 66-74.
26. Ridpath J, Neill J, Frey M, Landgraf L. 2000. Phylogenetic, antigenic and clinical characterization of type 2 BVDV from North America. Vet Microbiol 77: 145-155.
27. Ridpath J. 2005. Practical significance of heterogeneity among BVDV strains: Impact of biotype and genotype on U.S. control programs. Prev Vet Med 72: 17-30.
28. Risatti G, Holinka L, Lu Z, Kitish G, Callahan J, Nelson W, Brea Tió E, Borca M. 2005. Diagnostic evaluation of a real time reverse transcriptase PCR Assay for detection of classical swine fever virus. J Clin Microbiol 43: 468-471.
29. Rivera H, Manchego A, Sandoval N, Morales C, Flores E. 1994. Complejo respiratorio bovino en terneros del Valle de Lima. Rev Inv Pec, IVITA 7(1): 35-38.
30. Saliki J, Dubovi E. 2004. Laboratory diagnosis of bovine viral diarrhea virus infections. Vet Clin N Am-Food A 20: 69-83.
31. Sakoda Y, Damrongmotonapakin S, Sato M, Ishikan E, Fukushu E. 1999. Genetic heterogeneity of porcine and rumiant pestiviruses mainly isolated in Japan. Vet Microbiol 65: 75-86.
32. Sandvik T. 1999. Laboratory diagnostic investigations for bovine viral diarrhoea virus infections in cattle. Vet Microbiol 64: 123-134.
33. Ståhl K, Benito A, Felmer R, Zuñiga J, Reinhardt G, Rivera H, Baule C, Moreno-López J. 2009. Genetic diversity of bovine viral diarrhea virus (BVDV) from Peru and Chile. Pesq Vet Bras 29(1): 41-44.
34. Tajima M, Frey H, Yamato O, Maede Y, Moenning V, Scholz H, Greiser- Wilke I. 2001. Prevalence of genotypes 1 and 2 of bovine viral diarrhea virus in Lower Saxony, Germany. Virus Res 76: 31-42.
35. Tautz N, Meyers G, Thiel H-J. 1998. Pathogenesis of mucosal disease, a deadly disease of cattle caused by a pestivirus. Clin Diag Virol 10: 121-127.
36. Vilcek S, Drew T, McGoldrick A, Paton D. 1999. Genetic typing of bovine pestiviruses from England and Wales. Vet Microbiol 69: 227-237.
37. Vilcek S, Durkovic B, Kolesarova M, Paton DJ. 2005. Genetic diversity of BVDV: Consequences for classification and molecular epidemiology. Prev Vet Med 72: 31-35.
38. Wageck C, Strasser M, Hertig Ch, Masuda A, Peterhans E. 1998. Detection of antibodies to bovine viral diarrhea virus and characterization of genomes of BVDV from Brasil. Vet Microbiol 63: 85-97.
39. Wirz B, Tratschin JD, Muller HK, Mitchell DB. 1993. Detection of hog cholera virus and differentiation from other pestivirus by Polymerase Chain Reaction. J Clin Microbiol 31: 1148-1154.
40. Wolfmeyer A, Wolf G, Beer M, Strube W, Hehnen HR, Schmeer N, Kaaden OR. 1997. Genomic (5´-URT) and serologic differences among German BVDV field isolates. Arch Virol 142: 2049-2057.