Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú v.23 n.2 Lima abril./jun 2012
CARACTERIZACIÓN Y DISTRIBUCIÓN DE CEPAS DE ESCHERICHIA COLI POTENCIALMENTE PATÓGENAS AISLADAS DE POLLOS BROILER DE EXPLOTACIONES AVÍCOLAS EN EL PERÚ
CHARACTERIZATION AND DISTRIBUTION OF POTENTIALLY AVIAN PATHOGENIC ESCHERICHIA COLI ISOLATES FROM BROILERS IN PERU
Claudia Carranza1,4, Ricardo León1, Néstor Falcón2, Anthony Neumann3, Christopher Kromm3
1 Departamento de Dirección Técnica, Innova Andina S.A., Lima, Perú
2 Facultad de Veterinaria y Zootecnia, Universidad Peruana Cayetano Heredia, Lima, Perú
3 Danisco, USA
4 E-mail: claudia.carranza76@gmail.com
RESUMEN
La colibacilosis aviar producida por infecciones de Escherichia coli es causa de elevados niveles de mortalidad y morbilidad en la industria avícola. El objetivo de este trabajo fue identificar las cepas patógenas de explotaciones avícolas en el Perú en base a la presencia de cinco genes codificadores de los factores de virulencia: iss, iucC, tsh, cvaC e irp2, mediante la técnica de Multiplex PCR. Se muestrearon 36 aves entre los 14 y 31 días de edad, provenientes de tres centros avícolas ubicados en el norte, centro y sur del país. Se hicieron aislados intestinales y se obtuvieron tres colonias sospechosas de E. coli por ave (n=108 colonias), que fueron evaluadas bioquímicamente con TSI, SIM, LIA y CITRATO, y adicionalmente con agar EMB para confirmación del diagnóstico. Las 41 colonias positivas a E. coli fueron analizadas mediante Multiplex PCR, resultando 65.9% de colonias potencialmente patógenas (portadoras de dos o más genes codificadores de virulencia); asimismo, se demostró que el 9.8% de los aislados contenían los cinco genes. Adicionalmente, los tres patotipos que contienen los genes irp2, iucC y tsh fueron encontrados en las zonas de La Libertad y Lima.
Palabras clave: Escherichia coli, patógena, genes, virulencia, broilers, Perú
ABSTRACT
Avian colibacillosis produced by Escherichia coli infections cause high mortality and morbidity in poultry farms. The objective of this study was to identify the pathogenic isolates of Peruvian broiler farms based in the presence of five genes encoding the virulence factors: iss, iucC, tsh, cvaC and irp2 by the Multiplex PCR. A total of 36 chicks between 14 and 31 days old from three poultry farms located in the north, centre, and south of Peru were sampled. Three colonies suspected to be E. coli were isolated from the intestine of each bird (n=108 colonies), and were biochemically tested with TSI, SIM, LIA, CITRATE and EMB mediums for the confirmation of the diagnostic. The 41 positive colonies to E. coli were analysed by Multiplex PCR, resulting 65.95% potentially pathogenic (carriers of two or more virulence genes); moreover, 9.8% of the isolates had all five genes. Three pathotypes containing irp2, iucC and tsh genes were found in La Libertad and Lima area.
Key words: Escherichia coli, pathogenic, genes, virulence, broilers, Peru
INTRODUCCIÓN
La avicultura peruana ha experimentado un crecimiento sostenido en la última década. La venta de pollos de carne en los centros de acopio de Lima Metropolitana y el Callao ha incrementado de 26 329 TM en enero de 2006 a 35 720 TM en enero del 2009, llegando a 40 440 TM en julio del mismo año (Actualidad Avipecuaria, 2009). Si bien se presenta un crecimiento sostenido de la producción, aún se tienen limitaciones de tipo sanitario. Dentro de los patógenos bacterianos de mayor importancia se encuentra Escherichia coli, bacilo Gram negativo (Barnes et al., 2008; Lee y Nolan, 2008), causante de la colibacilosis aviar y de cuantiosas pérdidas económicas a nivel mundial (Baltzley y Rehberg, 2006).
La colibacilosis es un síndrome complejo caracterizado por lesiones multiorgánicas, tales como aerosaculitis asociada a pericarditis, perihepatitis y peritonitis, que resulta en alta morbilidad y mortalidad (Jansen et al., 2003). Este síndrome, causado por cepas patogénicas de E. coli (APEC), puede presentarse tanto de forma local como sistémica (Barnes et al., 2008).
Se sabe que la colibacilosis aviar es una enfermedad secundaria y que el agente etiológico es un oportunista; sin embargo, nuevas evidencias sugieren que cepas APEC pueden actuar como agentes primarios y producir la enfermedad (Ewers et al., 2003; Kariyawasam et al., 2006; Barnes et al., 2008) e incluso, las cepas apatógenas, mediante el intercambio de material genético, podrían tornase patógenas si adquieren códigos genéticos para factores de virulencia específicos (Jansen et al., 2003). Los resultados de Skyberg et al. (2003) y Kariuki et al. (2002) sugieren que cepas intestinales aisladas de aves aparentemente sanas podrían causar enfermedad en huéspedes susceptibles, dependiendo de la combinación de los factores de virulencia que se presenten.
Las cepas patógenas o potencialmente patógenas, además de asociarse a causas de mortalidad embrionaria (Jeffrey et al., 2002; Skyberg et al., 2003; Gibbs et al., 2004), pueden ser identificadas por su resistencia al suero aviar debido a la lipoproteína externa de membrana ISS, codificada por el gen iss (Lynne et al., 2005, 2006; Johnson et al., 2002, 2008). El crecimiento bacteriano en suero aviar de cepas mutantes pAPEC-O2-colV iss- se vio notablemente reducido, pero se restituyó al insertar nuevamente el gen iss en el genoma de la cepa mutante, dando a entender la importancia de este factor para la supervivencia de la bacteria en el suero (Lynne et al., 2007).
Otro gen recientemente descubierto es el tsh (hemaglutinina sensible a la temperatura), que codifica una proteína similar a las proteasas de IgA de Haemophilus spp y Neisseria spp. Se postula como parte de la familia de las proteasas autotransportadoras y puede ser vital para la colonización del tracto respiratorio superior (Dozois et al., 2000; Ngeleka et al., 2002). Se caracteriza por su capacidad hemoaglutinante en eritrocitos de pollo, no provocar proteólisis de IgA humana o aviar (Stathopoulos et al., 1999) y producir lesiones fibrinosas en los sacos aéreos; sugiriendo que la proteína Tsh aumenta la tasa de colonización y el desarrollo de aerosaculitis (Dozois et al., 2000).
El gen irp2 se encuentra en cepas de enterobacterias altamente patógenas y codifica la síntesis del sideróforo yersiniabactina (Carniel et al., 1992; Bultreys et al., 2006; Barnes et al., 2008). El gen iuc C está vinculado al último paso de la síntesis del sideróforo conocido como aerobactina, el cual está relacionado a la captación y transporte de hierro (Lorenzo et al., 1986; Ngeleka et al., 2002; Barnes et al., 2008). Ambos genes forman parte vital de la supervivencia de la bacteria y se les considera importantes marcadores de virulencia debido a su capacidad de lograr la permanencia del patógeno en ambientes agrestes del huésped, como los carentes de hierro de libre disponibilidad.
El gen cvaC se encuentra dentro del operón ColV y sintetiza a la colicina V, proteína pequeña que interfiere con la formación del potencial de membrana requerido para la producción de energía (Johnson et al., 2006a,b). Se infiere que la colicina V como tal, no es totalmente relevante para la patogenicidad de las cepas APEC, pero el gen cvaC, al ser considerado un gen estructural del operón ColV y pertenecer al plásmido del mismo nombre (en donde se encuentran el operón aerobactina y los genes iss y tsh), es distinguido como un factor para la determinación de virulencia (Barnes et al., 2008; Mellata et al., 2009). Adicionalmente, se infiere que la mayor resistencia de las cepas virulentas en el intestino, en comparación con las avirulentas, se debe a la producción de colicina V, aunque se desconoce el mecanismo de acción (Gonçalvez et al., 2002).
Ngeleka et al. (2002) aisló cepas APEC a partir de brotes clínicos. Las muestras resultaron positivas a genes como tsh e iucC. Adicionalmente, los genes iss, iucC y tsh se encontraban en cepas de ponedoras sanas y enfermas de Francia, Bélgica y España (Gibert et al., 2006). Otros estudios, asimismo, han demostrado la presencia de irp2, iss, tsh y cvaC, tanto en aislados intestinales de aves sanas como de aves enfermas (Rodriguez-Siek et al., 2005; Trampel et al., 2007; Yaguchi et al., 2007). Por otro lado, se ha demostrado que los factores de virulencia Iss, aerobactina y producción de Colicina V se encuentran en más del 80% de los aislamientos de colibacilosis (Jeffrey et al., 2002), mientras que en Brasil se encontró el 36.5% de prevalencia del gen estructural de la Colicina V (Gonçalves et al., 2002).
Ante la evidencia que las cepas que causan colibacilosis en aves provienen del tracto gastrointestinal y se eliminan a través de las heces, para luego ser inhaladas por los animales, y la ausencia de información en el país, el objetivo del presente estudio fue caracterizar y evaluar la distribución geográfica de las cepas potencialmente patógenas de Escherichia coli presentes en el tracto gastrointestinal de pollos broiler aparentemente sanos, mediante el estudio de genes portadores de virulencia presentes.
MATERIALES Y MÉTODOS
Lugar de Estudio
Se realizó un estudio de tipo descriptivo en tres explotaciones de pollos de engorde ubicadas en el norte, centro y sur del país (La Libertad, Lima y Arequipa, respectivamente). El aislamiento e identificación de las colonias se realizó en el Laboratorio de Investigación y Desarrollo de la empresa Innova Andina S.A., Lima, y el análisis molecular de las colonias positivas a E. coli se efectuó en los laboratorios de investigación de Agtech Products Inc, Waukesha, EEUU.
Toma de Muestras
El tamaño de la muestra se determinó en base a los trabajos de Skyberg et al. (2003), Banach et al. (2004) y Ewers et al. (2005). Se tomaron muestras de 12 pollos de engorde entre 14 y 31 días de edad, aparentemente sanos, de cada centro avícola, para obtener 108 colonias (tres colonias por ave) sospechosas de E. coli. Se siguieron los criterios establecidos para el aislamiento de la bacteria en aves jóvenes (Banach et al., 2003, 2004; Lago et al., 2006). Las aves fueron sacrificadas mediante desarticulación cervical, minutos antes de la toma de muestras.
Para la colección de las porciones de intestino, se tomaron como referencia dos puntos anatómicos: 10 cm a distal desde el píloro y 10 cm a craneal desde la unión iliocecal. Las porciones fueron retiradas y lavadas con PBS estéril (0.15 M) hasta eliminar todo residuo intestinal. Ambas secciones fueron colocadas en un frasco de vidrio de 50 mL (Boeco) conteniendo 20 mL de PBS estéril y fueron homogenizadas por 30 s (Banach et al., 2003, 2004; Ewers et al., 2005; Barnes et al., 2008). Se inoculó 1 ml del sobrenadante en un tubo de ensayo conteniendo 10 mL de caldo lactosado (Merck 1.07661.0500).
Procesamiento de Muestras
El procesamiento se realizó en un ambiente libre de contaminación bacteriana, en una sala de ambiente controlado y dentro de una cabina de bioseguridad clase II (Labconco Corp.), logrando una pureza de aire del 98- 99%. Los tubos de ensayo con caldo lactosado fueron incubados por 24 h a 37 ºC y luego fueron inoculados en placas de Agar McConkey (BBL, 6163276) e incubados bajo las mismas condiciones. Seguidamente, de cada placa se escogieron tres colonias lactosa positivas sospechosas de E. coli para su análisis bioquímico y confirmación del aislamiento. Los medios utilizados fueron SIM o sulfato, indol y movilidad (Criterion, C6941), TSI o agar triple azúcar hierro (Merck KGaA, 1.01342.0500), agar lisina-hierro (Merck KGaA, 1.11640.0500), agar citrato Simmons (Merck KGaA, 1.02501.0500) (Barnes et al., 2008; Lee y Nolan, 2008) y Agar EMB Levine (Merck KGaA, 1.01342.0500).
Se escogieron las colonias que resultaron móviles, Indol positivas, negativas a la degradación del citrato, negativas a sulfuro de hidrógeno, fermentadoras de glucosa, lactosa y sacarosa, positivas a la descarboxilación de lisina y negativas a la desaminación de lisina, así como productoras de gas (Blanco et al., 2002; Jeffrey et al., 2004; Barnes et al., 2008; Lee y Nolan, 2008). Paralelamente, se inocularon las muestras en agar diferencial EMB (Eosina y Azul de metileno) y se contrastaron los resultados obtenidos de las pruebas bioquímicas con las colonias que dieran un tono verde tornasolado.
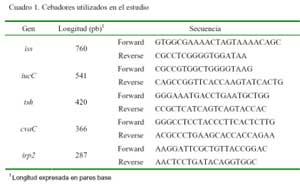
Las colonias que resultaron positivas a E. coli fueron colocadas en tarjetas FTA clásicas (Whatman Ltda.) y enviadas en condiciones de esterilidad al laboratorio para la determinación antigénica mediante MULTIPLEX PCR. El DNA fue aislado usando un High Pure PCR Template Preparation Kit (Roche Diagnostics GMBH, Mannheim, Alemania). El procedimiento MULTIPLEX PCR fue realizado según lo descrito por Banach et al. (2003). Este contenía 5 μl 10x buffer sin MgCl2, 2 μl 10nM dNTP mix, 0.3 5U/μl Taq Polimerasa Platinum (Invitrogen, Carlsbad, CA), 4 μl 50 mM MgCl2, 0.5 μl iss cebadores Forward y Reverse, 0.3 μl tsh, iucC, cvaC, irp2 cebadores Forward y Reverse, 31 μl de dH2O estéril y 5 μl de DNA genómico, para tener un volumen final de 50 μl. La secuencia de los cebadores se muestra en el Cuadro 1.
El programa de PCR comenzó con una pre-incubación de 95 ºC por 5 min, luego 9 ciclos de 95 ºC por 59 s, 55 ºC por 30 s, 72 ºC por 59 s; 28 ciclos de 94 ºC por 30 s, 55 ºC por 30 s, 72 ºC por 30 s; y se terminó con una incubación a 72 ºC por 7 min. Los productos del PCR fueron identificados con electroforesis en un gel de agarosa Un-Sieve al 3% (Bio-Rad).
Análisis de Datos
Se determinó la frecuencia de genes presentes por zona geográfica y se analizó la asociación entre el tipo de gen encontrado y la zona geográfica mediante la prueba de Chi cuadrado.
RESULTADOS
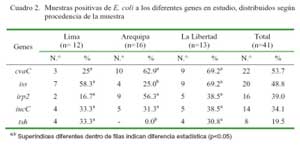
Se obtuvieron 41 colonias positivas (41/ 108) a Escherichia coli, correspondiendo el 39.0, 31.7 y 29.3% a Arequipa, La Libertad y Lima. El gen de mayor presentación fue el cvaC, encontrándose en el 53.7% de las cepas de E. coli aisladas. La distribución de este gen, al igual que los genes iucC e irp2 fue estadísticamente similar entre los tres departamentos. Contrariamente, los genes iss y tsh tuvieron una menor presencia en Arequipa (p<0.05), siendo el tsh el de menor frecuencia (19.5%) en el estudio (Cuadro 2).
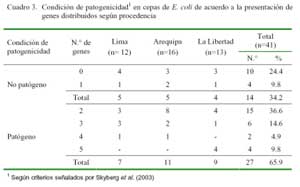
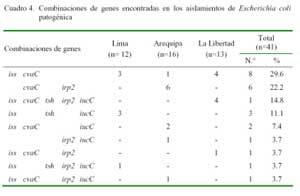
El 65.9% (27/41) de colonias positivas a E. coli podría ser considerado patógeno, ya que en su estructura genómica poseen dos o más genes codificadores de virulencia (Cuadro 3). El patotipo de mayor frecuencia fue la combinación de genes iss + cvaC (29.6%), la misma que estuvo presente en los tres departamentos; seguido por cvaC + irp2 (22.2%), presente únicamente en Arequipa, y el patotipo que combina los 5 genes (14.5%), hallado solamente en La Libertad (Cuadro 4).
Adicionalmente, el 41.6% de los aislados de aves de 14 días representan patotipos con marcadores de alta patogenicidad (tsh o irp2), en tanto que sólo ocurrió en el 9.1% de los aislados de aves de 25 días. Por otro lado, los cuatro aislados de aves de 31 días resultaron ser patógenos y presentaron los cinco genes estudiados.
La presencia del gen cvaC en el 53.7% de los aislados, así como su relación con 6 de los 9 patotipos encontrados, puede deberse a que este gen se encuentra ubicado dentro del plásmido ColV, como parte del operón del mismo nombre y codificando a la proteína estructural de la Colicina V (Johnson et al., 2006b). Este plásmido es característico y frecuente en las cepas patógenas, al igual que su producto final, la Colicina V. Otros estudios, asimismo, han demostrado una alta frecuencia de aislamiento de este gen y de otros del mismo operón (Baltzley y Rehberg, 2006; Gebert et al., 2006; Lago et al., 2006).
De igual manera, el gen iss está fuertemente relacionado al plásmido ColV ya que se ubica dentro de esta sección del genoma (Lynne et al., 2006). Dada la alta presentación de este plásmido en los aislamientos, se podría sospechar una incidencia similar del gen iss en conjunto con el gen estructural de la Colicina V, tal como se dio en este estudio. Johnson et al. (2006a) y Lynne et al. (2007) postulan que este gen puede también estar contenido en el plásmido ColBM, sugiriendo a este último como una evolución de su par ColV, lo que explicaría los presentes resultados donde la presencia del gen iss se da en patotipos donde el cvaC no está presente.
Los genes relacionados con la captación de hierro (iucC e irp2) tuvieron una similar frecuencia de presentación (4/9). Esta doble presencia puede ser un indicador de la importancia de los sideróforos para el desarrollo de patogenicidad de la bacteria en ambientes carentes de hierro de libre disponibilidad (Rodriguez-Siek et al., 2005).
Al igual que el iss, el gen iucC está también codificado dentro del operón ColV (Lorenzo et al., 1986; Johnson et al., 2006b); sin embargo, no hay estudios que detallen que este puede ser encontrado en otras partes del genoma bacteriano, lo que dificulta aún más la determinación de un patotipo común o frecuente para todos los APEC y, a la vez, queda sin poder relacionar la presentación de este gen en los patotipos donde el cvaC estaba ausente.
La presentación del gen tsh en los aislados se limitó al 19.5% y estuvo en sólo 3 de los 9 patotipos encontrados. Sin embargo, su relación con uno o dos de los genes captadores de hierro se dio en todos los casos. Esto podría explicarse debido a que tanto el gen tsh como cualquiera de los codificadores de sideróforos están presentes sólo en las cepas más patógenas (Carniel et al., 1992; Dozois et al., 2000; Ngeleka et al., 2002; Dozois et al., 2003), lo que implicaría que estas combinaciones serían las que representarían el mayor riesgo para el ave de todos los patotipos encontrados en el tracto gastrointestinal de las aves muestreadas; sobre todo, tomando en cuenta que el gen irp2 es también un marcador de alta patogenicidad para especies como Yersinia spp, Pseudomonas spp, e incluso las cepas de E. coli de procedencia humana (Xu et al., 2000; Bultreys et al., 2006).
De acuerdo a los patotipos encontrados, podría inferirse que 3 de los 9 patotipos representan el mayor riesgo para las aves portadoras, debido a que estos poseen las combinaciones que acarrean los genes catalogados como marcadores de alta patogenicidad (irp2, iucC y tsh). La combinación de genes es perfecta para la invasión de los tejidos, su supervivencia y la evasión a las defensas del huésped (Ngeleka et al., 2002; Mellata et al., 2003; Lynne et al., 2005).
Skyberg et al. (2003) demostraron que aislados gastrointestinales de aves sanas conteniendo los cinco genes lograron matar a la mayoría de embriones en las pruebas de ELA (Prueba de letalidad embrionaria). Esta prueba es considerada (en conjunto con los desafíos en aves jóvenes) como prueba tradicional para la determinación de patogenicidad de las cepas aisladas (Lee y Nolan, 2008). El departamento de La Libertad es el único cuya población de aves presentó colonias positivas a los cinco genes en estudio, de allí que las explotaciones avícolas de la zona tendrían un alto mayor riesgo para la enfermedad.
El conocimiento de la presentación de estos genes en el país es un buen inicio para el mejor entendimiento del mecanismo de la enfermedad y para desarrollar formas más eficaces de prevención y control. Una de las opciones que mejor se perfila es la producción de vacunas. Las vacunas experimentales están dirigidas contra serogrupos específicos, muchos de los cuales sólo presentan protección contra desafíos homólogos (Kariyawasan et al., 2003; Roland et al., 2004), lo cual es una seria limitación debido a la variedad tan extensa que poseen los APEC.
Lynne et al. (2006) sugieren la creación de vacunas contra las estructuras comunes a la mayoría de cepas APEC, como los genes codificadores de virulencia. Sus estudios revelan que la creación de una vacuna experimental a partir de la proteína Iss, codificada por el gen iss, fue capaz de conferir protección ante el desafío heterólogo. Esta protección conferida por un solo factor de virulencia, podría verse multiplicada en vacunas multivalentes dirigidas contra varios productos de genes específicos marcadores de virulencia, como los descritos en el presente estudio, sobre todo aquellos que sean de mayor presentación y alta patogenicidad. La edad más apropiada para la vacunación, según los resultados del presente estudio, estaría antes de los 14 días de edad. A esto se suman los resultados obtenidos por Gomis et al. (2007) que indican que broilers de 10 días vacunados con bacterina de E. coli mostraron anticuerpos específicos contra esta bacteria a los 30 días post vacunación.
CONCLUSIONES
-
Se encontraron nueve patotipos de cepas APEC potenciales, de los cuales tres representan combinaciones de genes ideales para la invasión y supervivencia en los tejidos, y evasión a las defensas del huésped.
-
Los tres patotipos que contienen los genes irp2, iucC y tsh fueron encontrados en las zonas de La Libertad y Lima.
-
Las aves de 14 y 31 días fueron las que presentaron mayor cantidad de patotipos con marcadores altamente patógenos.
Agradecimientos
Los autores agradecen a la empresa Innova Andina S.A. por las facilidades proporcionadas para el desarrollo del estudio, así como a las explotaciones avícolas que brindaron las aves de estudio.
LITERATURA CITADA
1. Actualidad Avipecuaria. 2009. Estadísticas. Actualidad Avipecuaria, Perú 3(14): 84-86.
2. Baltzley T, Rehberg T. 2006. Characterization of avian pathogenic E. coli in a layer production facility. Agtech Products Inc. Waukesha, Wisconsin. [Internet]. Disponible en: http://www.agtechproducts.com/resources/2.PDF
3. Banach S, Lago F, Wiard T, Rehberger T. 2004. Molecular typing of avian pathogenic Escherichia coli (APEC) from turkey poults. In: 2004 American Society for Microbiology General Meeting. Idaho, USA.
4. Banach C, Lago F, Rehberger T. 2003. Distribution and diversity of pathogenic E. coli in commercial turkey poult production. Agtech Products Inc., Waukesha, WI. [Internet]. Disponible en: http://www.agtechproducts.com/resources/research_poultry_18.pdf
5. Barnes H, Nolan L, Vaillancourt JP. 2008. Colibacillosis. In: Saif Y (ed). Diseases of poultry. 12th ed. Iowa: Blackwell Publishing. p 691-732.
6. Blanco J, Blanco M, Blanco J, Mora A, Alonso M, Gonzáles E, et al. 2002. Enterobacterias: características generales. Género Escherichia. En: Vadillo S, Durán S, Mateos E (eds). Manual de microbiología veterinaria. Madrid: McGraw Hill/Interamericana. p 301-325.
7. Bultreys A, Gheysen I, Hoffmann E. 2006. Yersiniabactinn production by Pseudomonas syringae and Escherichia coli, and description of a second yersiniabactin locus evolutionary group. Appl Environ Microb 72: 3814-3825.
8. Carniel E, Guivoule A, Guilvout I, Mercereau-Puijalon O. 1992. Molecular cloning, iron-regulation and mutagenesis of the irp2 gene encoding HMWP2, a protein specific for the highly pathogenic Yersinia. Mol Microbiol 6: 379-388.
9. Dozois C, Daigle F, Curtiss III R. 2003. Identification of pathogen-specific and conserved genes expressed in vivo by an Avian Pathogenic Escherichia coli strain. P Natl Acad Sci USA 100: 247-252.
10. Dozois C, Dho-Moulin M, Brée A, Fairbrother J, Desautels C, Curtiss R. 2000. Relationship between the Tsh autotransporter and pathogenicity of avian Escherichia coli and localization and analysis of the tsh genetic region. Infect Immun 68: 4144-4154.
11. Ewers C, JanBen T, Kiebling S, Philipp H, Wieler L. 2005. Rapid detection of virulence-associated genes in avian pathogenic Escherichia coli by Multiplex Polymerase Chain Reaction. Avian Dis 49: 269-273.
12. Ewers C, Janssen T, Wieler LH. 2003. Avian pathogenic Escherichia coli (APEC). Berl Munch Tierarztl Wochenschr 116: 381-395.
13. Gibbs P, Petermann S, Wooley P. 2004. Comparison of several challenge models for studies in avian colibacillosis. Avian Dis 48: 751-758.
14. Gebert S, Kromm C, Rehberger T. 2006. Development of a direct fed microbial to control pathogens associated with turkey poult production. In: Poultry Science Association Annual Meeting. Edmonton, Canada.
15. Gibert M, Téllez S, García N, Tejedor J, Comerón M, López G, et al. 2006. Caracterización molecular de factores de virulencia por PCR y comparación entre Escherichia coli septicémicos (APEC) y Escherichia coli fecales (AFEC) en gallinas ponedoras. En: XLIII Simposio Científico de Avicultura EXPOAVIGA. Barcelona, España.
16. Gomis S, Babiuk L, Allan B, Willson P, Waters E, Hecker R, et al. 2007. Protection of chickens against a lethal challenge of Escherichia coli by a vaccine containing CpG oligodeoxynucleotides as an adjuvant. Avian Dis 51: 78-83.
17. Gonçalvez A, Bernardes A, Guimaraes B, Luiz de Souza H, Pontes A, Cristine M. 2002. Virulence factors of avian pathogenic Escherichia coli isolated from broilers from the south of Brazil. Avian Dis 46: 749-753.
18. Jansen T, Philipp HC, Voss M, Preisinger R, Wieler L. 2003. Multiplex PCR is the first technique to allow the specific and sensitive detection of avian pathogenic Escherichia coli (APEC). Lohmann Information 28: 1-5.
19. Jeffrey J, Nolan L, Tonooka K, Wolfe S, Giddings C, Horne S, et al. 2002. Virulence factors of Escherichia coli from cellulitis or colisepticemia lesions in chickens. Avian Dis 46: 48-52.
20. Jeffrey J, Singer R, OConnor R, Atwill E. 2004. Prevalence of pathogenic Escherichia coli in the broiler house environment. Avian Dis 48: 189-195.
21. Johnson T, Giddings C, Horne S, Gibbs P, Wooley R, Skyberg J, et al. 2002. Location of increased Serum Survival gene and selected virulence traits on a conjugative R plasmid in an avian Escherichia coli isolate. Avian Dis 46: 342-352.
22. Johnson T, Johnson S, Nolan L. 2006. Complete DNA sequence of a ColBM plasmid from Avian Pathogenic Escherichia coli suggests that it evolved from closely related ColV virulence plasmids. J Bacteriol 188: 5975-5983.
23. Johnson T, Siek K, Johnson S, Nolan Lisa. 2006. DNA sequence of a ColV plasmid and prevalence of selected plasmid-encoded virulence genes among avian Escherichia coli strains. J Bacteriol 188: 745-758.
24. Johnson T, Wannemuehler Y, Nolan L. 2008. Evolution of the iss gene in Escherichia coli. Appl Environ Microbiol 74: 2360-2369.
25. Kariyawasam S, Wilkie B, Gyles C. 2003. Construction, characterization and evaluation of the vaccine potential of three genetically defined mutants of avian pathogenic Escherichia coli. Avian Dis 48: 287-299.
26. Kariyawasam S, Johnson T, DebRoy C, Nolan L. 2006. Ocurrence of Pathogenicity Island IAPEC-01 genes among Escherichia coli implicated in avian colibacillosis. Avian Dis 50: 405-410.
27. Lago F, Gebert S, Rehberger T. 2006. Diversity and relationship among APEC within the GI tract and APEC of diseased turkey poults. In: Poultry Science Association Annual Meeting. Edmonton, Canada.
28. Lee M, Nolan L. 2008. Colibacillosis. In: Dufour-Zavala L, et al. (eds). A laboratory manual for the isolation, identification and characterization of avian pathogens. 5th ed. Jacksonville (FL): American Association of Avian Pathologists. p 10-11.
29. Lynne A, Foley S, Nolan L. 2005. Immune response to recombinant Escherichia coli Iss protein in poultry. Avian Dis 50: 273-276.
30. Lynne A, Foley S, Nolan L. 2006. Characterization of monoclonal antibodies to Avian Escherichia coli Iss. Avian Dis 50: 445-449.
31. Lynne A, Skyberg J, Logue C, Doetkott C, Foley S, Nolan L. 2007. Characterization of a series of transconjugant mutants of an Avian Pathogenic Escherichia coli isolate for resistance to serum complement. Avian Dis 51: 771-776.
32. Lorenzo V, Bindereif A, Paw B, Neilands J. 1986. Aerobactin biosynthesis and transport genes of plasmid Col V-K30 in Escherichia coli K-12. J Bacteriol 165: 570-578.
33. Mellata M, Dho-Moulin M, Dozois C, Curtiss R, Lehoux B, Fairbrother J. 2003. Role of avian pathogenic Escherichia coli virulence factors in bacterial interaction with chicken heterophils and macrophages. Infect Immun 71: 494-503.
34. Mellata M, Touchman J, Curtiss R III. 2009. Full sequence and comparative analysis of the plasmid pAPEC-1 of avian pathogenic E. coli x7122 (O78:K80:H9). PLoS ONE 4: e4232. doi:10.1371/journal.pone.0004232
35. Ngeleka M, Brereton L, Brown G, Fairbrother J. 2002. Pathotypes of avian Escherichia coli as related to tsh-, pap-, pil-, and iuc- DNA sequences, and antibiotic sensitivity of isolates from internal tissues and the cloacae of broilers. Avian Dis 46: 143-152.
36. Roland K, Karaca K, Sizemore D. 2004. Expression of Escherichia coli antigens in Salmonella typhimurium as a vaccine to prevent airsacculitis in chickens. Avian Dis 48: 595-605.
37. Rodriguez-Siek K, Giddings W, Doetkott C, Johnson T, Nolan L. 2005. Characterizing the Apec pathotype. Vet Res 36: 241-256.
38. Skyberg J, Horne S, Giddings C, Wooley R, Gibbs P, Nolan L. 2003. Characterizing avian Escherichia coli isolates with Multiplex Polymerase Chain Reaction. Avian Dis 47: 1441-1447.
39. Stathopoulos C, Provence D, Curtiss III R. 1999. Characterization of the Avian Pathogenic Escherichia coli hemagglutinin Tsh, a member of the immunoglobulin A protease-type family of autotransporters. Infect Immun 67: 772-781.
40. Trampel D, Wannemuehler Y, Nolan L. 2007. Characterization of Escherichia coli isolates from peritonitis lesions in commercial laying hens. Avian Dis 51: 840-844.
41. Xu J, Cheng B, Wen X, Cui S, Ye C. 2000. High-pathogenicity island of Yersinia sp in Escherichia coli strains isolated from diarrhea patients in China. Clinic Microbiol 38: 4672-4675.
42. Yaguchi K, Ogitani T, Osawa R, Kawano M, Kokumai N, Kaneshige T, et al. 2007. Virulence factors of Avian Pathogenic Escherichia coli strains isolated from chickens with colisepticemia in Japan. Avian Dis 51: 656-622.