Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú v.23 n.3 Lima ago. 2012
Genotipificación, evaluación toxigénica in vitro y sensibilidad a antibióticos de cepas de escherichia coli aisladas de casos diarreicos y fatales en alpacas neonatas
Genotypification, in vitro toxigenic evaluation, and antibiotic sensitivity of escherichia coli strains isolated from diarrheic and fatal cases in neonatal alpacas
Luis Luna E.1, Lenin Maturrano H.1,2, Hermelinda Rivera G.2, Víctor Zanabria H.3, Raúl Rosadio A.1,2,4
1 CONOPA – Instituto de Investigación y Desarrollo de Camélidos Sudamericanos, Lima
2 Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima
3 Universidad Nacional del Altiplano, Puno
4 E-mail: rrosadio@gmail.com
RESUMEN
Se analizaron 27 hisopos diarreicos rectales y 24 contenidos patológicos del intestino de 51 alpacas de 1 a 7 semanas de edad para aislar y genotificar Escherichia coli, y caracterizar su sensibilidad antimicrobiana. Los aislamientos de E. coli fueron genotipificados por PCR múltiple para detectar genes de virulencia: stx1 y stx2 (shigatoxina 1 y 2), eae (intimina) y bfp (Bundle-Forming Pili), lt (toxina termolábil), sta y stb (toxina termoestable A y B), presentes en E. coli enterohemorrágica (EHEC), enteropatógena (EPEC) y enterotoxigénica (ETEC). Paralelamente, cepas positivas a stx1 o stx2 fueron analizadas en células Vero para detectar efectos verocitotóxicos. Posteriormente, todas las cepas aisladas fueron evaluadas para determinar su sensibilidad antimicrobiana a ocho antibióticos frecuentemente utilizados en ganaderías alpaqueras altoandinas. Los análisis microbiológicos y moleculares determinaron aislamientos de E. coli potencialmente patógenas en 19/27 (70.4%) de los hisopados clínicos y en 11/24 (45.8%) de los contenidos analizados. En crías padeciendo diarreas clínicas, se identificaron 18/19 (94.7%) cepas EPEC y la restante EHEC positiva al gen stx2 de baja actividad verocitotóxica (1:16). Once de las cepas EPEC contenían ambos genes eae y bfp y las siete restantes solamente el gen eae. De las crías muertas con antecedentes diarreicos, en 6/11 (54.5%) se aislaron cepas EPEC positivos al gen eae, mientras que las cinco restantes (45.5%) fueron EHEC. Tres de las 5 EHEC tenían el gen stx1 (dos con alta actividad verocitotóxica), la cuarta presentó el gen stx2 expresando alta actividad verocitotóxica, pero en el quinto aislado, positivo a ambos genes stx1 y stx2, no pudo evaluarse la actividad verocitotóxica. El 80% del total de E. coli (patogénicas y no patogénicas) aisladas en el estudio demostraron resistencia a la neomicina, y el 25% a la oxitetraciclina. Los resultados evidencian que las alpacas albergan cepas de E. coli potencialmente patógenas y probablemente causantes de patologías intestinales en crías de alpacas andinas.
Palabras clave: E. coli, genotipificación, diarreas, antibióticos, alpaca
ABSTRACT
Clinical rectal diarrheic swabs (n=27) and pathological intestinal contents (n=24) from 51 alpacas between 1 to 7 weeks of age were used to isolate and genotype Escherichia coli, and to test for antimicrobial sensibility. All the E. coli isolates, confirmed by the API system, were genotyped by Multiplex PCR to determine the presence of virulence genes: stx1 and stx2 (shigatoxin 1 and 2), eae (intimin), bfp (Bundle-Forming Pili), lt (heat-labile toxin), sta and stb (heat-stable toxin A and B), in enterohemorrhagic (EHEC), enteropathogenic (EPEC) or enterotoxigenic (ETEC) E. coli strains. Positive stx1 and stx2 strains were tested on Vero cells for verocytotoxicity, and all isolates were tested for antimicrobial sensibility to the eight most frequent used antibiotics in Andean livestock. Microbiological and molecular analysis revealed the presence of pathogenic E. coli strains in 19/27 (70.4%) of the clinical diarrhea cases and 11/24 (45.8%) of the pathological intestinal content samples. In crias suffering from diarrhea, 94.7% (18/19) of the pathogenic isolates were EPEC strains while the remaining was stx2 EHEC with a low expression of verocytoxic activity (1:16). Eleven of the 18 EPEC were positive for both eae and bfp, while the remaining seven were eae positive only. Six of the 11 (54.5%) isolates from intestinal contents were EPEC eae positive only, while the remaining five (45.5%) were EHEC isolates. Three of the 5 EHEC isolates tested positive to stx1 with two expressing high verocytotoxicity, while the fourth tested positive for stx2 with similar high expression of verocytotoxicity, and the fifth was positive for both stx1 and stx2 genes but could not be evaluated the verocytotoxicity. Eighty percent of the pathogenic and non-pathogenic E. coli strains showed resistance to neomycin, and 25% to oxytetracycline. These results indicate that alpacas harbor potentially pathogenic E. coli strains that might cause clinical and fatal intestinal disorders in young animals.
Key words: E. coli, genotyping, diarrhea, antibiotic sensitivity, alpaca
INTRODUCCIÓN
Las diarreas neonatales son causantes de elevadas morbilidades y mortalidades durante las primeras semanas de vida de las alpacas andinas (Ameghino, 1990). Se sospecha que uno de los principales agentes causales de estas patologías, sobretodo en animales muy jóvenes, sean cepas patógenas de Escherichia coli, que a diferencia de las cepas comensales no patógenas, contienen elementos génicos adicionales responsables de su patogenicidad. Estos genes codifican factores de virulencia, presentes en el genoma bacterial y adquiridos mediante trasferencia de elementos génicos móviles (islotes de patogenicidad, ADN plasmídico o introducidos por bacteriófagos o transposones) (Dobrint et al., 2003). En el pasado, la determinación de patogenicidad de estas cepas dependía de la serotipificación que inicialmente era solamente del antígeno O (Margall et al., 1997) y posteriormente, de una batería antigénica (O, K y H) o inoculando experimentalmente a animales de laboratorio. Actualmente, todo esto se obvia utilizando técnicas moleculares para identificar los genes responsables de factores de virulencias (Margall et al., 1997).
Estudios iniciales identificaron cepas patogénicas de E. coli productores de diarreas en alpacas peruanas. Las cepas aisladas eran productoras de toxinas termoestables y termolábiles, capaces de producir cambios patológicos en intestinos ligados de conejo, ratones y lechones lactantes (Ellis et al., 1983; Ramírez, 1990). Recientemente, y utilizando PCR múltiple se han aislado cepas de E. coli conteniendo genes codificantes de shigatoxinas y de intimina en muestras diarreicas de crías de alpacas y de vicuñas adultas (Bravo de Rueda, 2006; Arainga et al., 2008). Similarmente, de un brote de diarreas catarrales en crías de guanacos se logró aislar una cepa de E. coli productora de shigatoxinas con actividad citotóxica para células Vero (Mercado et al., 2004).
La presencia de genes productores de toxinas patológicas es utilizada para agrupar o diferenciar genotípicamente las cepas de E. coli productores de diarreas y de infecciones sistémicas. Las cepas que producen toxinas shigatoxinas de tipos 1 y 2 (Stx1 y 2) y, opcionalmente, intimina (eae) sirven para agrupar a las E. coli enterohemorrágica (EHEC), mientras que las productoras de toxinas termoestables a y b (Sta y Stb, respectivamente) y termolábil (lt) congregan a las E. coli enterotoxigénicas. La presencia de adhesinas, denominadas intimina (eae), y el Boundle-forming Pili (bpf) caracterizan a las cepas de E. coli enteropatógena (EPEC) con capacidad de adherirse a las vellosidades intestinales al inicio del proceso de infección (Nataro y Kaper, 1998).
El presente estudio utilizó la técnica de PCR múltiple para identificar genes codificantes de las diferentes toxinas patogénicas es decir: termolábil (lt), termoestable (sta/stb), shigatoxina 1 y 2 (stx 1 y 2), intimina (eae) y el Bundle-forming Pili (bfp) en cepas de E. coli aisladas de casos clínicos de diarrea y en animales muertos con antecedentes diarreicos en crías de alpacas. Paralelamente, estas mismas cepas fueron enfrentadas a diversos antibióticos buscando información de resistencia o susceptibilidad a antibióticos usados frecuentemente en explotaciones altoandinas. Finalmente, las cepas EHEC fueron analizadas para determinar su actividad citotóxica in vitro en células Vero.
MATERIALES Y MÉTODOS
Se muestrearon 51 crías de alpacas con procesos diarreicos (n=27) o muertas con antecedentes de diarreas (n=24). Los animales tenían entre 1 a 7 semanas de edad, nacieron durante la campaña de parición de enero a marzo de 2007, y pertenecieron a centros de crianza de los departamentos de Arequipa, Puno y Cuzco.
Las muestras patológicas consistentes en hisopados rectales (animales vivos) o de contenido intestinal (animales muertos) fueron conservadas en medio de transporte Cary Blair (Eurotubo, Deltalab, España) hasta su procesamiento en el laboratorio. Posteriormente, los hisopos fueron sembrados en caldo Luria Bertani (LB) (Lennox L Broth, Acumedia, Michigan, EEUU) e incubados a 37 ºC durante 18 h. Se tomó un inóculo (ansada) del caldo y se sembró por agotamiento en Agar MacConkey (Merck, Alemania), incubándose por 18 h a 37 ºC. Luego se seleccionaron 2-3 colonias para realizar pruebas bioquímicas confirmatorias de E. coli (Koneman et al., 2004).
Cepas individuales confirmadas de E. coli fueron cultivadas en caldo LB en similares condiciones de incubación para producir alícuotas de 1 ml. Estas se colocaron en tubos eppendorf de 2 ml y se centrifugaron a 13 000 rpm por 10 min. Se descartó el sobrenadante y se realizó la extracción de ADN a partir de la biomasa del pellet siguiendo el método de fenol/cloroformo (Sambrook et al., 1989) y precipitación de los ácidos nucleicos con isopropanol al 100% y etanol al 70%. El ADN extraído se diluyó en 100 μl de buffer TE (100 mM Tris, 1 mM EDTA).
Reacción de PCR
Se realizó un PCR múltiple para la detección de los genes stx-1, eae, lt, sta, stb y stx-2 (codificantes de la shigatoxina 1, intimina, toxina termolábil, toxina termoestable A, toxina termoestable B y shigatoxina 2, respectivamente) utilizando los cebadores diseñados por Pass et al. (2000). La mezcla de reacción consistió en 1.0 pmol de cada cebador forward y reverse para la detección del gen stx1, 1.0 pmol de stx2, 0.5 pmol de sta, 1.0 pmol de stb, 0.5 pmol de lt, 0.5 pmol de eae, 100 mM de KCl, 20 mM de Tris-HCl (pH: 8.3), 3 mM de MgCl2, 0.5 mM de cada dNTP, 2.5 U de Taq Polimerasa y 1 μl de ADN bacteriano (25-30 ng). Las condiciones de PCR usados fueron de 30 s a 95 ºC, 72 ºC por 1 min durante 5 ciclos; 95 ºC por 30 s, 63 ºC por 30 s y 72 ºC por 30 s durante 30 ciclos; y una extensión final de 72 ºC por 5 min.
En las cepas eae positivas se realizó, paralelamente, otra reacción de PCR para buscar la presencia simultánea del gen bfp, codificadores del Bundle-forming Pili, utilizando los cebadores diseñados por Gunzburg et al. (1995), en una mezcla consistente de 0.5 μM de cada cebador, 1.5 μl de buffer PCR 10X, 1.5 mM de MgCl2, 100 μM de cada dNTP, 1 U de Taq polimerasa y 1 μl de ADN bacteriano (25-30 ng) en temperaturas desnaturalizantes iniciales de 94 ºC por 5 min, 30 ciclos de 94 ºC por 1 min, 60 ºC por 1 min y 72 ºC por 1 min, y una extensión final de 72 ºC por 1 min. La amplificación de ambas reacciones de PCR se realizó en un termociclador modelo 2720 Thermal Cycler (Applied Biosystems, EEUU). Los productos de PCR fueron separados mediante electroforesis en gel de agarosa (Cambrex, EEUU) al 2.5% diluido en buffer TBE 0.5X (Tris Base 0.0445 M, ácido bórico 0.0445 M y EDTA 0.001 M, pH 8) a 100 V por 2 h. Para la observación de las bandas de ADN amplificadas, el gel fue teñido por inmersión en una solución de bromuro de etidio (0-5 μg/ ml) por 2 min y visualizado en un transluminador UV. Para la estimación del tamaño de los amplicones se utilizó un marcador de peso molecular (100 bp DNA ladder, Promega, EEUU).
Detección de Verotoxicidad
Las seis cepas EHEC fueron expuestas a cultivos de células Vero (células derivadas de riñón de mono verde africano) en busca de expresión de efecto citotóxico (Konowalchuk et al., 1977). Para este efecto, las cepas se procesaron de acuerdo al protocolo descrito por Karmali et al. (1985). Las cepas se cultivaron en caldo LB (Acumedia) por 18 h a 37 ºC, y los pellets se concentraron por centrifugación a 13 000 rpm por 10 min. El pellet celular fue resuspendido en una solución de polimixina B (0.5 mg/ml) e incubado por 30 min a 37 ºC y sometido a una nueva centrifugación a 13 000 rpm por 10 min. Se recuperó el sobrenadante y se pasó por filtros de 0.22 μm de porosidad. El filtrado se almacenó a -20 ºC hasta su análisis. Para la prueba, los filtrados fueron diluidos desde 1:2 hasta 1:1024, y expuestos al cultivo celular, incubados a 37 ºC en una incubadora húmeda con 5% de CO2, monitoreadas cada 24 h hasta por 120 h o hasta observar un efecto citotóxico (Karmali et al., 1985).
Resistencia de Escherichia coli a Antibióticos
El método de Disco difusión o de Kirby- Bauer, descritos en el manual de procedimientos para la prueba de sensibilidad antimicrobiana y el método de disco difusión del Instituto Nacional de Salud, Perú (Sacsaquispe y Velásquez, 2002) fueron utilizados para determinar la resistencia de antibióticos del total de cepas patogénicas y no patogénicas aisladas. Los discos de ocho antibióticos fueron seleccionados tomando en cuenta informaciones sobre las drogas antibacterianas frecuentemente usadas en las zonas donde se tomaron las muestras, así como los más utilizados en pruebas de sensibilidad antibiótica para cepas de E. coli (DeFrancesco et al., 2004).
RESULTADOS
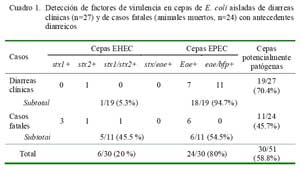
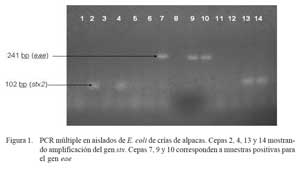
En el 58.8% (30/51) de las muestras se logró aislar cepas de E. coli enteropatógenas, siendo 24 cepas enteropatogénicas (EPEC) y 6 cepas enterohemorrágicas (EHEC), sin haberse detectado cepas con genes codificantes de las toxinas termodependientes (Sta, Stb y Lt) (Fig. 1). La frecuencia de muestras positivas a determinados genes se presenta en el Cuadro 1.
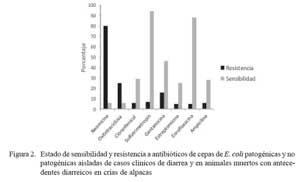
El 80% de cepas aisladas de E. coli demostró resistencia a la neomicina, mientras que el 25% a la oxitetraciclina (Fig. 2). Se observó también que el 60% de cepas demostró resistencia a un solo antibiótico. Asimismo, las cepas mostraron tener alta sensibilidad a la enrofloxacina (88%) y a sulfatrimetropin (94%) (Fig. 2).
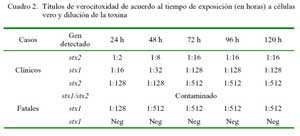
En tres de las 4/6 cepas EHEC (una de caso clínico y tres de casos fatales) se de tectó producción de toxinas con efectos citotóxicos para células Vero (verocitotoxinas), y dos de ellas expresaron citotoxicidad hasta en títulos de 1:512 (Cuadro 2).
DISCUSIÓN
El complejo diarreico neonatal es comúnmente asociado con alta morbilidad neonatal en la mayoría de las especies productivas. La infección del tracto digestivo es comúnmente observada en animales que nacen o se manejan en condiciones insalubres, pobremente protegidos por anticuerpos calostrales y expuestos a cepas de alta patogenicidad (Garmendia et al., 1987).
La disponibilidad de herramientas moleculares está permitiendo identificar factores de virulencia en cepas de E. coli en crías de alpacas padeciendo diarreas clínicas o en crías muertas con antecedentes de procesos similares (Arainga et al., 2008; Rosadio et al., 2008). En el 58.8% (30/51) de los casos analizados en este estudio se aisló cepas conteniendo genes codificantes de factores patogénicos (Cuadro 1), en concordancia con dichos estudios. La predominancia en la detección de cepas con genotipo EPEC de casos de diarreas y en menor grado a EHEC es similar a resultados encontrados en crías padeciendo cuadros similares en el sur del Perú (Bravo de Rueda, 2006) y en otras especies de animales (Blanco et al., 1997); sin embargo, la presencia de cepas EHEC y EPEC en casos fatales no ha sido reportada.
La presencia de 11/18 cepas EPEC en animales padeciendo diarreas, positivas para los genes de eae y bfp, evidencian la enorme capacidad patogénica de estas cepas para causar diarreas. La presencia del factor de virulencia codificada por el gen Bundleforming Pilus (bfp), provee capacidad a la bacteria para adherirse a los enterocitos e iniciar una infección activa (adherencia focalizada) y facilitar, posteriormente, una unión mucho más firme para desencadenar la lesión denominada adhesión y barrido (A/ E), características de las cepas EPEC (Donnenberg y Kaper, 1992).
La mayor proporción de aislados pertenecientes a cepas EHEC en animales muertos por diarreas sugiere a estas cepas una mayor patogenicidad, y que podría explicar las infecciones fatales en el estudio. La capacidad patogénica de estos aislados se expresa por una alta actividad verocitóxica observada en 2/3 cepas EHEC analizadas en el estudio (Cuadro 2). Sin embargo, llama la atención que estas cepas EHEC carezcan del gen de la intimina (eae), evidenciando incapacidad de producir la lesión de adhesión y barrido y tal vez una patogenicidad disminuida. La presencia de cepas EHEC positivas para stx1 y stx2 en elevados porcentajes (50- 60%) ha sido igualmente detectada en animales padeciendo de diarreas en hatos comunales (Arainga et al., 2008). Similarmente, una cepa EHEC distinta (stx1/eae positivas) ha sido aislada en un brote de diarreas catarrales en crías (chulengos) de guanacos patagónicos, donde esta cepa demostró expresión de citotoxicidad en células Vero (Mercado et al., 2004). La infección de animales jóvenes por cepas EHEC podría deberse a que los animales adultos serían reservorios naturales de estas cepas, de manera similar a la observada en otras especies de animales domésticos (Beutin et al., 1993; Blanco et al., 1997).
La no detección en el estudio de cepas codificantes de toxinas termoestables y termolábiles, comúnmente causantes de diarreas en el hombre, corderos y terneros (Mouricot, 1991), contrasta con lo previamente reportado por Ellis et al. (1983). Estos autores, utilizando la prueba biológica del intestino ligado, demostraron la presencia de cepas termosensibles en crías de alpacas padeciendo de diarreas. La no detección de cepas conteniendo genes termodependientes, tal vez sea consecuencia de un bajo número de plásmidos codificantes de estos genes, pero no es producto de una aparente inespecificidad de los cebadores utilizados, pues las cepas positivas patrones para estos genes siempre fueron detectadas en las pruebas moleculares.
La alta resistencia de las cepas hacia antibióticos como la neomicina y la oxitetraciclina, puede explicarse por el uso indiscriminado de estas drogas en el campo y por la aplicación de dosis inapropiadas. Esto es sugerente, pues similares comportamientos se observaron en cepas patógenas y apatógenas, indicando la posibilidad de que la resistencia a dichos antibióticos circulen por conjugación genética (DeFranceso et al., 2004).
CONCLUSIONES
-
Las alpacas hospedan cepas enteropatógenas (EPEC) y enterohemorrágicas (EHEC) potencialmente productoras de procesos diarreicos, y algunas de ellas son capaces de desencadenar infecciones fatales en crías a edades muy tempranas.
-
Las cepas EPEC serían potencialmente productoras de diarrea recurrentes a través del gen eae, bpf o ambos factores de virulencia identificados, mientras que las EHEC serían causantes de infecciones fatales.
Agradecimientos
Los autores agradecen a CONOPA – Instituto de Investigación y Desarrollo de Camélidos Sudamericanos, Consejo Superior de Investigaciones (CSI - UNMSM), INCAGRO y FINCyT por el financiamiento parcial del estudio; a la Dra. Dolores Cid Vásquez de la UCM por proporcionar las cepas controles; asimismo, al personal del IVITA-Maranganí, del CIP La Raya de la UNA-Puno, y a todas las personas que colaboraron con la realización del presente estudio.
LITERATURA CITADA
1. Ameghino E. 1990. Avances sobre investigación en salud animal: camélidos sudamericanos. Bol Div N.º 23 IVITAUNMSM: 1-66.
2. Arainga MA, Taguchi T, Morales SM, Portilla KV, Villacaqui ER, Valencia N, Rivera H, Yamasaky S. 2008. Detección de genes de E. coli enterohemorrágica productora de toxina Stx1 y Stx2 en alpacas (Lama pacos) con diarrea. En: XIX Congreso Nacional de Ciencias Veterinarias. Huancavelica, Perú.
3. Beutin, L, Geier D, Steinruck H, Zimmermann S, Scheutz F. 1993. Prevalence and some properties of verotoxin (Shiga-like toxin)-producing Escherichia coli in seven different species of healthy domestic animals. J Clin Microbiol 31: 2483-2488.
4. Blanco M, Blanco JE, Blanco J, Mora A, Prado C, Alonso MP, Mourino M, et al. 1997. Distribution and characterization of faecal verotoxin producing Escherichia coli (VTEC) isolated from healthy cattle. Vet Microbiol 54: 309-319.
5. Bravo de Rueda C. 2006. Caracterización molecular de los genes stx1, stx2 y eae en aislados de Escherichia coli de crías de alpaca con diarrea. Tesis de Médico Veterinario Zootecnista. Lima: Universidad Peruana Cayetano Heredia.73 p.
6. DeFrancesco K, Cobbold R, Rice D, Besser D. 2004. Hancock. Antimicrobial resistance of commensal Escherichia coli from dairy cattle associated with recent multi-resistant salmonellosis outbreaks. Vet Microbiol 98: 55-61.
7. Dobrint U, Agerer F, Michaelis K, Janka A, Buchrieser C, Samuelson M, Svanborg C, et al. 2003. Analysis of genome plasticity in pathogenic and commensal Escherichia coli isolates by use of DNA arrays. J Bacteriol 185: 1831-1840.
8. Donnenberg S, Kaper J. 1992. Enteropathogenic Escherichia coli. Infect Immun 60: 3953-3961.
9. Ellis R, Wilson R, Ramirez A. 1983. Characteristics of enteropathogenic Escherichia coli isolated from neonatal alpaca. In: Proc IV International Symposium on Neonatal Diarrhea. Saskatoon, Canada.
10. Garmendia A, Palmer G, DeMartini J, McGuire T. 1987. Failure of passive immunoglobulin transfer: a major determinant of mortality in newborn alpacas (Lama pacos). Am J Vet Res 48: 1472-1476.
11. Gunzburg S, Tornieporth N, Riley L. 1995. Identification of enteropathogenic Escherichia coli by PCR-based detection of the bundle-forming pilus gene. J Clin Microbiol 33: 1375-1377.
12. Karmali M, Petric M, Lim C, Cheung R, Arbus G. 1985. Sensitive method for detecting low numbers of verotoxinproducing Escherichia coli in mixed cultures by use of colony sweeps and polymyxin extraction of verotoxin. J Clin Microbiol 22: 614-619.
13. Koneman EW, Allen SD, Janda WM, Schreckenberger PC, Winn WC. 2004. Diagnóstico microbiológico. 5ª ed. Buenos Aires: Ed Médica Panamericana. 1413 p.
14. Konowalchuck, J, Speirs J, Stavric S. 1977. Vero response to a cytotoxin of Escherichia coli. Infect Immun 18: 775- 779.
15. Margall N, Domínguez A, Prats G, Salleras L. 1997. Escherichia coli enterohemorrágica. Rev Esp Salud Pública 71: 437-433.
16. Mercado EC, Rodríguez S, Elizondo A, Marcoppido G, Parreño V. 2004. Isolation of shiga toxin-producing Escherichia coli from a South American camelid (Lama guanicoe) with diarrhea. J Clin Microbiol 42: 4809-4811.
17. Mouricot M. 1991. Swine and cattle enterotoxigenic Escherichia colimediated diarrhea. Development of therapies based on inhibition of bacteria- host interactions. Eur J Epidemiol 7: 588-604.
18. Nataro J, Kaper J. 1998. Diarrheagenic Escherichia coli. Clin Microbiol Rev 11: 142-201.
19. Pass M, Odedra R, Batt RM. 2000. Multiplex PCRs for identification of Escherichia coli virulence genes. J Clin Microbiol 38: 2001-2004.
20. Ramírez A. 1990. Colibacilosis entérica en crías de alpacas. En: Avances sobre investigación en salud animal camélidos sudamericanos. UNMSM. Bol Div Nº 23. 64 p.
21. Rosadio R, Maturrano L, Pérez D, Llanco L, Castillo H, Véliz A, Yaya K, Londoñe P. 2008. Enterotoxemia: new evidence on pathogenesis and prevention of the number one cause of neonatal alpaca mortality in South America. In: Proceedings of the World Alpaca Conference, Sydney, Australia. p 50-55.
22. Sacsaquispe R, Velásquez J. 2002. Manual de procedimientos para la prueba de sensibilidad antimicrobiana por el método de disco difusión. Serie de Normas Técnicas N.º 30. Lima: Ministerio de Salud, Instituto Nacional de Salud. 68 p.
23. Sambrook J, Fritsch E, Maniatis T. 1989. Molecular cloning: A laboratory manual. 2nd ed. Vol. 3. New York, USA: Cold Spring Harbor Laboratory Press. 551 p.