Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú v.23 n.3 Lima ago. 2012
Optimización de una técnica para la extracción de ADN de heces de vicuña (vicugna vicugna mensalis)
DNA extraction methods from vicuña fecal samples (vicugna vicugna mensalis)
Lenin Maturrano H.1,2,3, Juan M. Aguilar L.2, Paloma Krüger D.2, Jesús Chávez I.2, Raúl Rosadio A.1,2, Jane C. Wheeler2
1 Unidad de Biología y Genética Molecular, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima
2 CONOPA – Instituto de Investigación y Desarrollo de Camélidos Sudamericanos, Lima
3 E-mail: lenin.maturrano@gmail.com
RESUMEN
Actualmente es posible determinar el estatus de especies silvestres mediante el análisis de ADN extraído de muestras no invasivas como las heces. Aunque dichos análisis suelen ser dificultosos debido principalmente a la composición y conservación de las heces, se ha demostrado en muchas especies que con el desarrollo de métodos adecuados de extracción es posible maximizar la fiabilidad y eficacia de este procedimiento. Por tanto, el objetivo del presente estudio fue optimizar un método de extracción de ADN de heces de vicuña Vicugna vicugna mensalis, especie clasificada como casi amenazada en el Perú, con la finalidad de obtener ADN de suficiente calidad para análisis moleculares. Se recolectaron 51 muestras de heces durante los meses de agosto y septiembre de 2008 en dos comunidades vicuñeras ubicadas a más de 4600 msnm en los departamentos de Junín y Moquegua. Luego de observar la defecación, las muestras fueron recolectadas en forma inmediata, conservadas en etanol y transportadas al laboratorio para la extracción del ADN. El kit comercial QIAamp® DNA Stool Mini Kit (QIAGEN) fue modificado para incluir un vigoroso y extenso proceso de agitación y los resultados fueron comparados con dos protocolos comúnmente utilizados: fenol/cloroformo/alcohol isoamílico y el mismo kit QIAGEN. La cantidad del ADN extraído fue observado en electroforesis horizontal en geles de agarosa, y la calidad evaluada mediante la amplificación y visualización de los tamaños esperados de los microsatélites YWLL46 y LCA19. Aunque se logró extraer ADN con los tres métodos, solamente el método modificado permitió obtener ADN de suficiente calidad para ser utilizado en pruebas moleculares.
Palabras clave: extracción de ADN, heces, vicuña
ABSTRACT
At present, the use of DNA extracted from non-invasive samples, especially feces, is common practice in the discipline of conservation genetics. Even though DNA analysis is often difficult mainly due to the composition and preservation of feces has been shown in many species with the development of suitable extraction methods is possible to maximize the reliability and effectiveness of such procedures. The objective of this study has been optimization of a protocol for the extraction of DNA from vicuña feces (Vicugna vicugna mensalis), species classified as Near Threatened in Peru, in order to obtain DNA of sufficient quality for molecular analysis. A total of 51 samples were taken from vicuña dung piles in two communities located 4600 meters above sea level in the dry Puna ecosystems of the regions of Junin and Moquegua between August and September 2008. After observing defecation, samples were collected immediately, preserved in ethanol and transported to the laboratory for DNA extraction. Modification of the protocol of the QIAamp ® DNA Stool Mini Kit (QIAGEN) to include an extended period of vigorous shaking was found to producer higher quality DNA than was obtained using both the kit protocol and PCI (Phenol/chloroform/isoamyl alcohol). The amount of extracted DNA was visualized using horizontal agarose gel electrophoresis, and the quality was tested analyzing microsatellite markers LCA 19 and YWLL 46 on polyacrylamide gels. Although DNA was obtained by all three methods, only the modified protocol produced DNA of sufficient quality for use in molecular analyses.
Key words: fecal DNA extraction, feces, vicuña
INTRODUCCIÓN
Históricamente, la colecta de muestras para estudios de genética en animales silvestres ha sido a través de la captura de los animales, implicando la generación de estrés, alteración del comportamiento individual o social y, algunas veces, la muerte del animal. En la actualidad, con el avance de la ciencia, el monitoreo no invasivo de poblaciones de animales es una herramienta de importancia en los estudios de conservación y manejo de especies amenazadas o elusivas (Kohn y Wayne, 1999; Waits y Paetkau, 2005). Dichos estudios se ven facilitados con la obtención y análisis de ADN a partir de muestras no invasivas como las heces, pelos y restos de piel, entre otros. La extracción y análisis de ADN de heces y otras muestras no invasivas (Panasci et al., 2011) permite obtener información detallada sobre la estructura genética (Quéméré et al., 2010) y genómica (Ouborg et al., 2010; Perry et al., 2010), así como sobre la abundancia (Luikart et al., 2010; Brinkman et al., 2011; Poole et al., 2011) de las poblaciones sin perturbar a los animales; información esencial para un adecuado manejo de especies amenazadas.
Aunque las muestras de heces se caracterizan por la poca cantidad y pobre calidad de ADN que contienen y pueden ser extraídos (Bosch et al., 2005; Panasci et al., 2011), es necesario la adecuación de los protocolos de extracción para cada especie, a fin de que permita obtener ADN de suficiente calidad para la realización de estudios genéticos (Taberlet et al., 1996), inclusive a nivel genómico (Ouborg et al., 2010; Perry et al., 2010). Estudios en heces de rumiantes como la cabra de montaña Oreamnos americanus (Poole et al., 2011), el cérvido Sitka de cola negra Odocoileus bemionus sitkensis (Brinkman et al., 2011) y la oveja de las Montañas Ovis canadensis (Wehausen et al., 2004, Luikart et al., 2006), han demostrado muy buena recuperación de ADN en estas especies que, como la vicuña, depositan sus heces en forma de bolitas o pellets.
Por tanto, con la finalidad de optimizar el análisis genético de muestras no invasivas como las heces, se modificó un método de extracción de ADN para heces de vicuña con la finalidad de obtener ADN de suficiente calidad para posteriores análisis moleculares. Dicho método modificado fue comparado con dos métodos comúnmente usados en estudios de diversidad genética en poblaciones de camélidos sudamericanos domésticos y silvestres (Kadwell et al., 2001; Wheeler et al., 2001).
MATERIALES Y MÉTODOS
Toma de Muestras
Se recolectaron 51 muestras de heces en las comunidades de Villa Junín (provincia y departamento de Junín) e Ichuña (provincia de Sánchez Cerro, departamento de Moquegua), durante la época seca entre los meses de agosto y setiembre de 2008. Estas comunidades se encuentran ubicadas en puna seca a más de 4600 msnm. Las muestras fueron colectadas mediante Autorización N.° 057-2008-INRENA-IFFS-DCB.
Una vez localizadas las tropas de vicuñas, se observó a la distancia, vía monocular, el momento de defecación. Se recolectó cerca de 40 g de heces frescas por animal en tubos falcón de 50 ml estériles conteniendo etanol absoluto (Fig. 1). Los tubos rotulados y herméticamente cerrados fueron transportados a temperatura ambiente al laboratorio donde se almacenaron a -20 ºC entre 14 a 20 días antes de su procesamiento.
Extracción del ADN
Se extrajo entre 180 a 200 mg de la superficie de las bolitas de heces (Fig. 1) para maximizar la cantidad de células descamadas del colon del animal durante la defecación y reducir contaminantes de plantas y otras contenidas en su interior.
El material fecal de todos los animales fue utilizado para obtener el ADN empleando tres métodos de extracción en cada muestra: a) PCI (fenol/cloroformo/alcohol isoamílico, 25:24:1); b) un kit estándar comercial (QIAamp® DNA Stool Mini Kit, QIAGEN), y c) Protocolo modificado del kit comercial.
El proceso de extracción del ADN utilizando el protocolo de PCI, se inició incubando las muestras en buffer QLB (Queen Lysis Buffer) y proteinasa K durante 12 horas a 56 ºC, donde se agregó volúmenes similares de PCI. El ADN fue precipitado con isopropanol en proporciones de v/v, y resuspendido en buffer TE (10 mM Tris-HCl, 1 mM EDTA, pH 8.0).
El segundo protocolo utilizó el kit comercial QIAamp® DNA Stool Mini Kit (QIAGEN), siguiendo las instrucciones del fabricante. En el tercer protocolo se modificó el procedimiento de este kit comercial: se agregó 3-4 ml de buffer de lisado ASL a 200 mg de material fecal, y se le sometió a un proceso de agitación severo y extenso. La agitación original de 15 minutos se modificó para agitar las muestras vigorosamente con ayuda de un vortex por cuatro días consecutivos.
Calidad del ADN
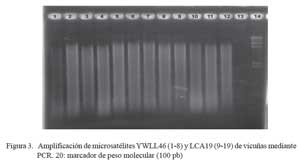
La calidad del ADN, en todas las muestras, fue visualizada mediante electroforesis en un gel de agarosa al 0.8% en buffer TBE 0.5X, y fue corroborada amplificando segmentos de microsatélites por PCR utilizando el kit QIAGEN Multiplex PCR, siguiendo las recomendaciones del fabricante. Las condiciones de amplificación contemplaron temperaturas de 95 ºC por 15 minutos, 95 ºC por 30 segundos, 60 ºC por 30 segundos y 72 ºC por 1 minuto en 35 ciclos con una extensión final de 72 ºC durante 10 minutos. Los productos de PCR fueron analizados mediante electroforesis en geles de agarosa.
Resolución de Fragmentos Amplificados
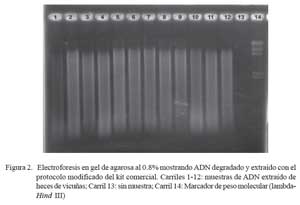
Los productos de cada PCR fueron analizados en gel de agarosa al 2% en solución tampón TBE 0.5X (Tris, ácido bórico, EDTA), usando una cámara de electroforesis horizontal junto a un marcador de peso molecular (Fago lambda digerido con Hind III), para determinar el tamaño aproximado de los fragmentos amplificados. Los productos de PCR fueron teñidos con bromuro de etidio, visualizados en un transiluminador y fotografiados con una cámara Polaroid (DS 34).
Tamaño de los Microsatélites
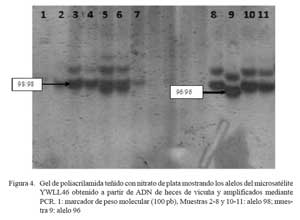
Para la determinación del tamaño de los alelos amplificados, se realizó una electroforesis en geles de poliacrilamida al 10% en buffer TBE 0.5X, a 150 V, durante 20 horas y revelados con tinción de nitrato de plata. Los tamaños de los microsatélites fueron determinados por migración respecto a un marcador de referencia (50 pb).
RESULTADOS Y DISCUSIÓN
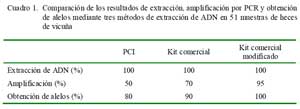
El proceso de extracción de ADN de heces de vicuña con los tres métodos ensayados fue exitoso. Se logró extraer ADN en todas las muestras (Cuadro 1) pero evidentemente degradados (Fig. 2), lo cual generó dificultades en análisis posteriores. El ADN obtenido mediante las técnicas de PCI y el protocolo estándar del kit comercial, mostraron las mayores contaminaciones o dificultades para su análisis, pues terminaron inhibiendo el 50 y 70% de las reacciones de PCR, respectivamente (Cuadro 1). Sin embargo, el protocolo modificado del kit comercial, independientemente del estado de la muestra, y a pesar de que tomó más tiempo, permitió obtener en el 95% de los casos, ADN de buena calidad, pues los alelos de los microsatélites evaluados se amplificaron mediante PCR en todas las muestras analizadas (Cuadro 1). Consecuentemente, el protocolo modificado permitió ahorrar muchas horastrabajo, reactivos y, sobre todo, el material de las muestras, pues no hubo necesidad de repetir la extracción como sucedió generalmente con los otros dos protocolos.
El análisis genético a partir de ADN extraído de heces es complicado y engorroso, pues generalmente el ADN obtenido se encuentra contaminado con restos propios del material fecal, de la dieta del animal o suele estar en bajas cantidades o degradado (Deuter et al., 1995; Kohn et al., 1995; Mak et al., 2004; Panasci et al., 2011). El uso de un método adecuado de extracción es fundamental, no solamente para eliminar impurezas, pero sobre todo, para evitar la inhibición en la amplificación de productos en pruebas de PCR.
El método de extracción con PCI es muy recomendado para la extracción de ADN a partir de tejidos o sangre, donde el número de células es mayor y los contaminantes o inhibidores de la PCR son casi inexistentes (Sambrook y Russell, 2001); sin embargo, no se obtuvieron buenos resultados al utilizarse muestras de heces de vicuña (Cuadro 1). Por otro lado, el kit comercial es comúnmente empleado para la extracción de ADN a partir de muestras de heces de diferentes especies, y especialmente acondicionado para la extracción de ADN de muestras fecales de humanos (Vanderberg y van Oorschot, 2002; Johnson et al., 2005).
La modificación en el protocolo del kit comercial consistió básicamente en prolongar la fase de lisis hasta cuatro días antes de iniciar el proceso de extracción. Aunque se desconoce la composición de los reactivos del kit comercial, prologando la acción física (agitación) del reactivo productor de lisis celular (4 días) causaría una mayor liberación de ADN y mejor degradación de los componentes de las heces (disminución de contaminantes e inhibidores de la PCR). Las modificaciones realizadas al protocolo del kit comercial permitieron obtener ADN de suficiente calidad para realizar análisis moleculares, incluyendo pruebas exitosas de PCR en el 95% de las muestras evaluadas. Asimismo, se pudo determinar los alelos de los microsatélites en el 100% de los casos. Esto superó lo que normalmente se obtenía con dicho kit siguiendo el protocolo del fabricante (70% de amplificación y 90% de determinación de alelos de las muestras amplificadas).
El uso de muestras fecales en estudios moleculares de especies silvestres tiene múltiples ventajas. A diferencia de las capturas, requiere poco personal en el campo, las muestras pueden obtenerse sin perturbar al animal y se pueden transportar fácilmente en etanol a temperatura ambiente (Broquet et al., 2007; Green et al., 2007). Aunque las dificultades técnicas de análisis están siendo superadas, no ocurre lo mismo con la reglamentación para la colecta de las muestras. Fuera del Perú, no se requiere ningún permiso de la autoridad CITES para la colección, transporte o estudio de heces de especies amenazadas (Luikart et al., 2006), pero actualmente en el Perú, según el Decreto Supremo 003- 2009-MINAM – Reglamento de Acceso a los Recursos Genéticos, no es posible recolectar muestras de heces de especies silvestres ni realizar análisis genéticos de su ADN, sin autorización previa, dificultando su estudio y por ende su conservación.
En el presente estudio, el ADN proveniente de heces de vicuña extraído por el protocolo modificado amplificó satisfactoriamente dos microsatélites (Cuadro 1, Fig. 3 y Fig. 4), comprobando su calidad para la realización de otros estudios genéticos. En el Perú, el análisis de variabilidad genética de los camélidos sudamericanos (Kadwell et al., 2001) ha proporcionado importante información, sobre todo para el manejo de la vicuña (Wheeler et al., 2001). La posibilidad de aplicar esta herramienta en forma masiva, utilizando ADN extraído de las heces, será muy importante para la toma de decisiones ade cuadas sobre la conservación de la vicuña. La adaptación de este protocolo a otras especies podría aplicarse en el estudio de especies en peligro de extinción, tales como el guanaco andino, el oso de anteojos u otras especies difíciles de capturar o muestrear.
CONCLUSIÓN
El protocolo de extracción comercial modificado permitió la extracción de ADN en suficiente cantidad y calidad para amplificar dos microsatélites utilizados en estudios de variabilidad genética de la vicuña.
Agradecimientos
El presente estudio se realizó gracias al apoyo financiero de FINCyT – Fondo para la Innovación, Ciencia y Tecnología, Contrato 006-FINCyT-PIBAP-2007 Estudio de la variabilidad genética de la vicuña como herramienta para la conservación de la especie en el Perú.
LITERATURA CITADA
1. Bosch M, Marmi J, Ferrando A, Lopez-Giraldez F, Andrés O, García E, Ponsa M, et al. 2005. Genotipar sin capturar. Galemys 17: 81:102.
2. Brinkman TJ, Person DK, Chapin III FS, Smith W, Hundertmark KJ. 2011. Estimating abundance of Sitka blacktailed deer using DNA from fecal pellets. J Wildlife Manage 75: 232-242.
3. Broquet T, Ménard N, Petit E. 2007. Noninvasive population genetics: a review of simple source, diet, fragment length and microsatellite motif effects on amplification success and genotyping error rates. Conserv Genetic 8: 249-260.
4. Deuter R, Pietsch S, Hertel S, Muller O. 1995. A method for preparation of fecal DNA suitable for PCR. Nucleic Acids Res 23: 3800-380.
5. Green ML, Herzing DL, Baldwin JD. 2007. Noninvasive methodology for the sampling and extraction of DNA from free-ranging Atlantic spotted dolphins (Stenella frontalis). Mol Ecol 7: 1287- 1292.
6. Johnson D, Martin L, Roberts K. 2005. STR-typing of human DNA from human fecal matter using the QIAGEN QIAamp stool mini kit. J Forensic Sci 50(4): 1-7.
7. Kadwell M, Fernandez M, Stanley H, Baldi R, Wheeler JC, Rosadio R, Brufor M. 2001. Genetic analysis reveals the wild ancestors of the llama and the alpaca. P Roy Soc Lon B Bio 268: 2575-2584.
8. Kohn MH, Wayne RK. 1999. Facts from feces revisited. Trends Ecol Evol 12: 223-227.
9. Kohn M, Knauer F, Stoffella A, Schröeder W, Pääbo S. 1995. Conservation genetics of the European brown bear – a study using excremental PCR of nuclear and mitochondrial sequences. Mol Ecol 4: 95-103.
10. Luikart G, Zundel S, Rioux D, Miquel C, Keating KA, Hogg JT, Steele B, et al. 2006. Low genotyping error rates and noninvasive sampling in Bighorn sheep. J Wildlife Manage 72: 299-304.
11. Luikart G, Ryman N, Tallmon DA, Schwartz MK, Allendorf FW. 2010. Estimation of census and effective population sizes: the increasing usefulness of DNA-based approaches. Conserv Genetic 11: 355-373.
12. Mak T, Lalloo F, Evans DGR, Hill J. 2004. Molecular stool screening for colorectal cancer. Brit J Surg 91: 790- 800.
13. Ouborg NJ, Pertoldi C, Loeschcke V, Bljlsma R, Hedrick PW. 2010. Conservation genetics in transition to conservation genomics. Trends Genet 26: 177-187.
14. Panasci M, Ballard WB, Breck S, Rodriguez D, Densmore III LD, Webster DB, Baker RJ. 2011. Evaluation of fecal DNA preservation techniques and effects of sample age and diet on genotyping success. J Wildlife Manage 74: 1616-1624.
15. Perry GH, Marioni JC, Melsted P, Gilad Y. 2010. Genomic scale capture and sequencing of endogenous DNA from feces. Mol Ecol 19: 5332-5344.
16. Poole KG, Reynolds DM, Mowat G, Paetkau D. 2011. Estimating mountain goat abundance using DNA from fecal pellets. J Wildlife Manage 75: 1527-1534.
17. Quéméré E, Louis EE, Ribéron A, Chikhi L, Crouau-Roy B. 2010. Noninvasive conservation genetics of the critically endangered golden-crowned sifaka (Propithecus tattersalli): high diversity and significant genetic differentiation over a small range. Conserv Genetic 11: 675-687.
18. Sambrook J, Russell D. 2001. Molecular cloning: A laboratory manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press.2368p.
19. Taberlet P, Griffin S, Goosens B, Questiau S, Manceau V, Escaravage N, Waits LP, Bouvet J. 1996. Reliable genotyping of samples with very low DNA quantites using PCR. Nucleic Acids Res 24: 3189-3194.
20. Vanderberg N, Van Oorschot R. 2002. Extraction of human nuclear DNA from feces samples using the QIAamp DNA Stool Mini Kit. J Forensic Sci 47: 1-3.
21. Waits LP, Paetkau D. 2005. Noninvasive genetic sampling tools for wildlife biologists: A review of applications and recommendations for accurate data collection. J Wildlife Manage 69: 1419-1433.
22. Wehausen JD, Ramey II RR, Epps CW. 2004. Experiments in DNA extraction and PCR amplification from Bighorn sheep feces: the importance of DNA extraction method. J Hered 95: 503-509.
23. Wheeler JC, Fernández M, Rosadio R, Hoces D, Kadwell M, Bruford MW. 2001. Diversidad genética y manejo de poblaciones de vicuñas en el Perú. Rev Inv Vet, Perú Supl 1: 170-183.