Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú v.23 n.4 Lima dez./dic. 2012
Estandarización de la técnica RT-PCR tiempo real para la detección del virus de la necrosis pancreática infecciosa (VNPI) en truchas arco iris (oncorhynchus mykiss)
Standardization of real time RT-PCR technique for infectious pancreatic necrosis virus (IPNV) detection in rainbow trout (oncorhynchus mykiss)
Gina Castro S.1, Alberto Manchego S.1,3, Kim-Lam Chiok C.1, Nieves Sandoval C.2, Juan More B.1, Mercy Ramírez V.1, Hermelinda Rivera G.1,4
1 Laboratorio de Microbiología y Parasitología Veterinaria,
2 Laboratorio de Histología, Embriología y Patología Animal, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima
3 E-mail: amanchegos@unmsm.edu.pe
4 E-mail: hriverag2005@yahoo.es
RESUMEN
El objetivo del presente estudio fue estandarizar y validar la técnica de RT-PCR tiempo real para el diagnóstico del virus de Necrosis Pancreática Infecciosa (VNPI) en truchas arco iris (Oncorhynchus mykiss) de la sierra central del Perú. Se colectaron muestras de riñón y bazo de truchas con aparentes signos clínicos de necrosis pancreática infecciosa (NPI) (n= 61) y truchas aparentemente sanas (n= 60) de dos piscigranjas del valle del Mantaro, Junín. Previo a la estandarización, las muestras fueron procesadas para la detección de antígeno del VNPI por inmunofluorescencia indirecta (IFI). Diez muestras de riñón y bazo de truchas negativas al VNPI por IFI fueron inoculados con una cepa del VNPI con título de 103 UFP/ml. La extracción del ácido nucleico del VNPI de las 121 muestras de riñón y bazo y de las 10 muestras de riñón y bazo inoculadas con el VNPI, la síntesis del ADN complementario y la RT-PCR tiempo real se realizaron utilizando kits comerciales. Se usaron los cebadores WB1 y WB2 correspondientes a un segmento del gen que codifica la proteína VP2 de los Aquabirnavirus. Una cepa del VNPI fue utilizada como control positivo y cepa de Rotavirus A, virus de Gumboro, virus de diarrea viral bovina, parainfluenza 3 como controles negativos. Las 121 muestras de riñón y bazo de las truchas arco iris resultaron negativas al VNPI por IFI. Los resultados de la RT-PCR en tiempo real fueron determinados en función a los valores del ciclo de amplificación que fue de 31.7 y temperatura de disociación de 80.4 °C de los productos del virus control positivo. La técnica de RT-PCR en tiempo real fue capaz de detectar hasta una concentración de 102 DI50UFP/ml del VNPI en las muestras de riñón y bazo inoculadas, mientras que las 121 muestras de truchas resultaron negativas al VNPI. La técnica de RT-PCR en tiempo real tuvo 100% de sensibilidad y especificidad en la detección del virus de la necrosis pancreática infecciosa en truchas arco iris.
Palabras clave: virus de la necrosis pancreática infecciosa, trucha arco iris, inmunofluorescencia, RT-PCR tiempo real
ABSTRACT
The objective of this study was to standardize and validate the real-time RT-PCR technique for diagnosis of Infectious Pancreatic Necrosis virus (IPNV) in rainbow trout (Oncorhynchus mykiss) from Central Sierra of Peru. Samples of kidney and spleen (n=61) of rainbow trout with apparent clinical signs of IPN and from rainbow trout apparently healthy (n=60) were collected from two fish farms located in the central sierra. Before standardization of the real-time RT-PCR technique, the kidney and spleen samples of 121 rainbow trout were tested for antigen of IPNV by indirect immunofluorescent (IIF) test. In addition, kidney and spleen samples of 10 rainbow trout negative to IPNV by IIF were inoculated with a serial dilution of IPNV strain (from initial concentration of 103 PFU/ml). The RNA extraction of the 121 samples and of the 10 samples inoculated with IPNV strain, the cDNA and the real-time RT-PCR were carried out by commercial kits. The WB1 and WB2 primers were used to amplify a segment that codifies the protein VP2 of the Aquabirnavirus. A strain of IPNV as a positive control and Rotavirus A strain, Gumboro disease virus, bovine viral diarrhea virus, and parainfluenza-3 were used as negative controls. The 121 kidney and spleen samples were negative to IPNV by IIF test. The results of real time RT-PCR technique were evaluated considering the cycle threshold values of 31.7 and temperature melting of 80.4 °C of the amplicons. The real time RT-PCR technique was able to detect a concentration of 102 PFU/ml of IPNV in the kidney and spleen samples inoculated and the 121 of kidney and spleen samples were negative to IPNV. This technique had 100% of sensibility and specificity for detection of IPNV.
Key words: rainbow trout, Infectious Pancreatic Necrosis virus, IPNV, RT-PCR, real time RT-PCR
INTRODUCCIÓN
En estas últimas décadas la acuicultura continental ha alcanzado un significante desarrollo en el Perú, lo cual se refleja en el incremento de derechos otorgados a los productores y en hectáreas de espejos de agua aptas para el desarrollo de la acuicultura (PRODUCE, 2008). A pesar del desarrollo de esta industria, la sanidad acuícola – un importante componente de la producción – no está teniendo un similar avance por muchos factores, siendo uno de ellos la falta de metodologías diagnósticas para una eficiente y rápida identificación de las enfermedades infecciosas como la Necrosis Pancreática Infecciosa (NPI).
La NPI es una enfermedad infecciosa aguda y muy contagiosa, causante de alta mortalidad en diversas especies de peces salmónidos jóvenes, especialmente menores de seis meses. La NPI es causada por el virus de la necrosis pancreática infecciosa (VNPI), perteneciente al género Aquabirnavirus de la familia Birnaviridae (Dobos, 1995). La transmisión viral es de tipo horizontal y vertical, donde la transmisión a través del agua es la principal vía de transmisión entre alevines. Los peces enfermos por NPI presentan cambios de comportamiento como anorexia, movimientos natatorios circulares con ataxia e hinchazón abdominal, entre otros. En la necropsia se observa a menudo el bazo, corazón, hígado y riñones aumentados de tamaño y pálidos (Rodríguez Saint-Jean et al., 2003); sin embargo, los signos clínicos y lesiones macroscópicas no son característicos de la NPI, ya que pueden ser ocasionados también por otros patógenos.
En el Perú, el VNPI representa un riesgo para la crianza de truchas arco iris, debido a la constante importación de ovas con las que podría ingresar este y otros patógenos y propagarse en una explotación comercial ocasionando severas pérdidas económicas. Existen escasos estudios patológicos que sugieren la presencia del VNPI en truchas arco iris (Oncorhynchus mykiss) en el sur del país (Mora, 2006). La identificación y confirmación del VNPI en el país requiere de técnicas con mayor sensibilidad y especificidad como las técnicas moleculares.
La técnica de RT-PCR tiempo real posee alta sensibilidad y especificidad para la detección de VNPI frente a otras pruebas diagnósticas (Williams et al., 1999; Rodríguez et al., 2001; Bowers et al., 2008), por lo que el objetivo del presente estudio fue estandarizar y validar la técnica de RT-PCR tiempo real para el diagnóstico de VNPI a fin de apoyar los programas de control sanitario y vigilancia epidemiológica acuícola en el país.
MATERIALES Y MÉTODOS
Lugar de Colección y Tamaño de Muestra
La colección de las truchas arco iris (O. mykiss) fue realizada en dos piscigranjas del valle del Mantaro, Huancayo, a 3400 msnm. La temperatura, contenido de oxígeno y pH del agua de las dos piscigranjas estuvieron dentro de los rangos normales para la crianza intensiva de la trucha arco iris.
Para el tamaño de muestra se consideró lo recomendado en el Manual de Animales Acuáticos del Organismo Internacional de Salud Animal (OIE, 2006). Para una población de más de 2000 peces y una prevalencia de 5% de la enfermedad, el tamaño de muestra es de 60 truchas. Se colectaron 61 (30 y 31/piscigranja) truchas arco iris con aparentes signos clínicos del VNPI y 60 de apariencia normal entre juveniles y adultos (4 y 12 meses de edad).
Luego del examen externo e interno, se colectaron muestras de la porción craneal del riñón y bazo de cada trucha. Al momento de la colecta se hizo una impronta de riñón y bazo en una lámina portaobjeto. Las láminas fueron fijadas en acetona fría por 10 min y conservadas hasta el momento del examen por inmunofluorescencia indirecta (IFI). Las muestras fueron mantenidas a temperatura de refrigeración y transportadas al Laboratorio de Virología de la Facultad de Medicina Veterinaria de la Universidad Nacional Mayor de San Marcos, Lima.
Detección de Antígeno del Virus NPI
La detección del antígeno del VNPI se realizó en las improntas de riñón y bazo fijados previamente mediante la prueba de IFI utilizando el kit IPNV-FluoroTest IndirectoTM (BIOS Chile) y según el manual disponible del kit.
Extracción del ARN del VNPI de las Muestras
Las muestras de riñón y bazo de las 121 truchas y las 10 muestras de riñón y bazo inoculadas con VNPI fueron machacadas para convertirlas en suspensión al 20% (P/V). Se centrifugaron a 800 g y el sobrenadante fue alicuotado en viales de 2 ml y conservadas a -35 ºC.
La extracción del ARN viral fue realizada utilizando el kit comercial PureLink RNA Purification System (Invitrogen, USA) y según lo indicado en el manual del kit, el cual utiliza 2-mercaptoetanol e isotiocianato de guanidina (buffer de lisis) y posteriormente etanol. Así mismo, utiliza un sistema de cartuchos de centrifugación con membrana de sílice donde el ARN se adhiere y es purificado tras varios procesos de lavado y centrifugación, y finalmente es eludido con agua libre de nucleasas. Los productos obtenidos se conservaron a -70 °C.
RT-PCR de Dos Pasos
Para la técnica de RT-PCR tiempo real se utilizó el kit comercial de dos pasos SYBR® GreenER Two-Step qRT-PCR Kit Universal (Invitrogen, USA), según el manual disponible en el kit, y los cebadores WB1 y WB2, correspondientes a un segmento genómico que codifica a la proteína VP2 de los Aquabirnavirus (Williams et al., 1999).
El ADN complementario (ADNc) se obtuvo por retrotranscripción incubando 1 µl de la preparación de los ARN extraídos con 10 µl de la mezcla de RT [2.5 µM de oligo d(T)20, 2.5 ng/µl de hexámeros al azar, 10 mM de MgCl2 y dNTPs], 2 µl de mezcla de enzima RT (RT M-MLV modificada) y 7 µl de agua libre de nucleasas proveído en el kit y en un volumen de reacción de 20 µl por cada muestra, a 25 °C por 10 min, 50 °C por 30 min, 85 °C por 5 min y mantenido a 4 °C; posteriormente, se adicionó 1 µl de ARNasa H y se incubó a 37 °C por 20 min. Los productos se conservaron a -20 °C hasta su posterior uso.
El proceso de PCR se realizó en un volumen de reacción de 20 µl por muestra utilizando 1µl de ADNc de las muestras, 10 µl de la mezcla comercial (que contiene ADN polimerasa Taq Hot Start, fluorocromo Sybr Green I, MgCl2, dNTPs, UDG), 0.4 µl de cada cebador (a 10 µM), 1 µl de colorante de referencia ROX (50 nM) y 7.2 µl de agua libre de nucleasas.
Se evaluaron dos protocolos de temperatura. En el protocolo 1, se siguió las indicaciones del manual del kit para un PCR en tiempo real: 50 °C por 2 min, 95 °C por 10 min, seguido de 40 ciclos de 95 °C por 10 s y 60 °C por un min. En el protocolo 2, se siguió los parámetros de un PCR convencional: 50 °C por 2 min, 95 °C por 10 min, seguido de 40 ciclos de 95 °C por 10 s, 60 °C por 1 min y 72 °C por 30 s. El proceso fue realizado utilizando el termociclador en tiempo real PTC 200 (MJ Research, UK) y el equipo lector Chromo-4 (MJ Research, UK). Los resultados se analizaron en función a los valores del ciclo de amplificación (Ct) y temperatura de disociación (Tm) obtenidos con el software Opticon Monitor 2 v2.0.3.
Estandarización y Validación
En la estandarización de la RT-PCR tiempo real se determinó la especificidad de los cebadores utilizados, así como optimización de la técnica empleando dos protocolos respecto a la temperatura. Como control positivo se empleó una cepa del VNPI y como controles negativos a una cepa de Rotavirus A y virus de la enfermedad de Gumboro (cepas de virus relacionados al VNPI), así como a los virus de la diarrea viral bovina y parainfluenza 3 (cepas de virus no relacionados al VNPI).
La sensibilidad de la RT-PCR tiempo real se determinó utilizando como muestras positivas a las 10 muestras de riñón y bazo de truchas inoculadas con 10 µl de diluciones seriadas del VNPI (a partir de una concentración inicial conocida de 103 UFP/ml) y agua libre de nucleasas; y como muestras negativas las 121 muestras de riñón y bazo de truchas arco iris, previamente diagnosticadas por IFI.
Los parámetros de sensibilidad, especificidad, valor predictivo positivo y negativo fueron evaluados mediante una tabla de contingencia 2x2 a partir de las muestras inoculadas y las de campo, con un intervalo de confianza del 95%, comparando los resultados de las pruebas de IFI y la RT-PCR tiempo real para el diagnóstico de la necrosis pancreática infecciosa en truchas.
RESULTADOS
Las 121 muestras de riñón y bazo de las truchas de las dos piscigranjas estudiadas resultaron negativas a antígeno del VNPI mediante la prueba de IFI.
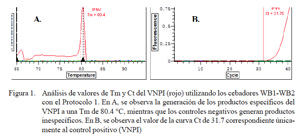
La RT-PCR tiempo real utilizando el protocolo 1 dio los siguientes resultados: el control positivo (VNPI) presentó un valor Ct de 31.7 dando un producto específico a una Tm de 80.4 °C, los controles negativos (Rotavirus A y virus de Gumboro y los virus no relacionados) no marcaron valores Ct y los valores Tm fueron inespecíficos (65 a 72 °C) (Fig. 1).
Utilizando el protocolo 2, se observaron resultados similares con la amplificación de productos específicos obtenidos con el control positivo (VNPI) a una Tm de 80.4 °C; pero hubo un mayor ruido o fondo. La amplificación de productos específicos se realizó en el ciclo 35 (Ct = 35.020) (datos no mostrados).
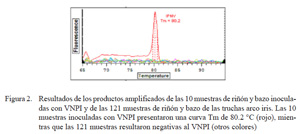
Las 10 muestras de riñón y bazo inoculadas con el VNPI resultaron positivas hasta una concentración de 102 UFP/ml y las 121 muestras de riñón y bazo resultaron negativas al VNPI mediante la RT-PCR tiempo real (Fig. 2).
El análisis de los resultados obtenidos mediante la tabla de contingencia de 2x2 determinó una sensibilidad y una especificidad, así como los valores predictivos positivo y negativo de 100%.
DISCUSIÓN
La técnica de RT-PCR tiempo real está siendo utilizada para el diagnóstico del VNPI por su alta sensibilidad y especificidad (Bowers, et al., 2008). En el Perú, la NPI ha sido reportada en truchas arco iris en base a lesiones histopatológicas; sin embargo, las lesiones histopatológicas no son características de la enfermedad de la NPI, ya que otros patógenos también pueden producir similares signos clínicos, por lo que es necesaria una prueba confirmatoria como el aislamiento viral o detección del ácido nucleico mediante técnicas moleculares.
Los resultados del presente estudio indican que los cebadores WB1 y WB2 permiten detectar el ARN del VNPI con 100% de sensibilidad y especificidad, diferenciándolo incluso de cepas virales pertenecientes a la misma familia viral como los Rotavirus y el virus de la enfermedad de Gumboro, que contienen genomas ARN doble hebra segmentados (Fig. 1A). Williams et al. (1999), utilizando estos mismos cebadores en RT-PCR convencional, obtuvo una sensibilidad del 100% al detectar una concentración de 101 TCDI50/ml del VNPI. Los cebadores WB1 y WB2 amplifican una secuencia de un segmento del gen que codifica la proteína VP2 del VNPI generando un producto no mayor de 400 pb, que es requerido para un PCR en tiempo real (Williams et al., 1999; Bustin, 2000; Dorak, 2006).
En los kits de PCR tiempo real empleados, se utiliza la tecnología de Taq polimerasa Hot-start y la actividad uracil-ADN-glicosilasa (Longo et al., 1990; Chou et al., 1992) para prevenir la reamplificación de los productos de ARN presentes después del primer paso (Retro Transcriptasa) y para inhibir la actividad de la polimerasa a temperatura ambiente resultando productos más específicos (Chou et al., 1992).
De los dos protocolos utilizados, el protocolo 1 permitió obtener productos más es- pecíficos comparado con los productos del protocolo 2, ya que las modificaciones propias de la polimerasa utilizada permitió que actúe eficientemente a la temperatura de hibridación de los cebadores con el ADNc viral, pues los cebadores utilizados tienen una Tm de 67 y 68 °C, optándose por utilizar la temperatura de hibridación de 60 °C (Innis et al., 1990; Rychlik et al., 1990).
La temperatura de extensión de 72 °C por 30 s en los ciclos del protocolo 2, que normalmente se usa para la extensión y actividad de una polimerasa convencional, permite la generación de productos inespecíficos debido posiblemente a la amplificación de contaminantes del ADN genómico. La tasa de extensión de Taq polimerasa es entre 30 y 70 bases/segundo (Jeffreys et al. 1988; Bustin, 2000), por lo que al utilizar una temperatura de hibridación de 60 °C se evita o se reduce la amplificación de contaminantes de ADN (Bustin, 2000).
La positividad de una muestra es determinada asociando los resultados del valor Ct con los valores de Tm, ya que todos los ADNc y cebadores presentes en la mezcla generan una Tm, pero solo los positivos dan un valor Ct en un ciclo determinado, dependiente de la cantidad de molde o templado, como ocurrió en el presente estudio. Sin embargo, al utilizar el fluoróforo SYBR Green I se debe considerar que el fluoróforo se une a la totalidad del ADN formado durante la PCR, incluyendo productos inespecíficos como los dímeros de cebadores. Por esta razón, es importante realizar un adecuado análisis de la curva de disociación, teniendo en cuenta que el valor dependerá del contenido de GC y del tamaño y secuencia del producto (Ririe et al., 1997; Giglio et al., 2003; Bustin, 2005; Valasek y Repa, 2005), y de esa forma solo los productos específicos se disocian a una Tm más alta (Dorak, 2006) (Fig. 1 y Fig. 2).
La técnica de IFI es considerada una prueba útil en el diagnóstico rutinario de la NPI por su rapidez, fácil ejecución y alta sensibilidad. Rodríguez et al. (2001) reportaron valores de sensibilidad de IFI del 100% cuando el título viral fue mayor a 103 UFP/ml indicando que la sensibilidad es directamente proporcional a la cantidad de virus presente en la muestra (Rodríguez et al., 2001). La dificultad para detectar títulos bajos del virus es una de las principales desventajas de esta prueba, a diferencia de esta, la técnica de RT-PCR tiempo real es capaz de detectar títulos bajos como 101 DITC50/ml del virus (Williams et al., 1999; Bowers et al., 2008), siendo comparable con el aislamiento viral en cultivos celulares; sin embargo, la desventaja de RT-PCR es que no puede evaluar la viabilidad de las partículas virales.
La técnica validada de RT-PCR tiempo real para el diagnóstico de la NPI demostró ser altamente específica, sensible, fácil de reproducir y rápida en la detección del virus, requiriendo de al menos 5 a 6 horas para obtener un resultado. Sin embargo, los procesos previos como son el procesamiento de la muestra y la extracción de ARN deben realizarse con el máximo cuidado para evitar los riesgos de contaminación al manejar numerosas muestras.
CONCLUSIONES
-
La técnica de RT-PCR tiempo real validada en el presente estudio, posee una sensibilidad del 100% para la detección del virus de la necrosis pancreática infecciosa en truchas arco iris.
-
La técnica de RT-PCR tiempo real utilizando el juego de cebadores WB1-WB2 posee una especificidad del 100% para la detección del virus de la necrosis pancreática infecciosa en truchas arco iris.
Agradecimiento
Los autores agradecen a los dueños de las piscigranjas por el apoyo brindado para la colección de las truchas arco iris. El trabajo fue financiado por el Vicerrectorado de Investigación de la Universidad Nacional Mayor de San Marcos. Código: 080801081, 2008.
LITERATURA CITADA
1. Bowers RM, LaPatra SE, Dhar AK. 2008. Detection and quantification of infectious pancreatic necrosis virus by real-time reverse transcriptase-polyme-rase chain reaction using lethal and non-lethal tissue sampling. J Virol Methods 147: 226-234.
2. Bustin SA. 2000. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays J Mol Endocrinol 25: 169-193.
3. Bustin SA. 2005. Real-Time PCR. In: Podda M, Fuchs J (eds). Encyclopedia of diagnostic genomics and proteomics. New York: Marcel Dekker. p 1117-1125.
4. Chou Q, Russell M, Birch DE, Raymond J, Bloch W. 1992. Preven-tion of pre-PCR mis-priming and primer dimerization improves low-copy-number amplifications. Nucleic Acids Res 20: 1717-1723.
5. Dobos P. 1995. The molecular biology of infectious pancreatic necrosis virus (IPNV). Annu Rev Fish Dis 5: 25-54
6. Dorak T. 2006. Real Time PCR (Advanced Methods Series). Oxford: Taylor & Francis. [Internet], [10 setiembre 2009]. Disponible en: http://dorakmt.tripod.com/genetics/realtime.html
7. Giglio S, Monis PT, Saint CP. 2003. Demonstration of preferential binding of SYBR Green I to specific DNA fragments in real-time multiplex PCR. Nucleic Acids Res 31: e136.
8. Innis MA, Gelfand DH, Sninsky JJ, White TJ. 1990. PCR protocols: A guide to methods and applications. San Diego, CA: Academic Press. 482 p.
9. Jeffreys AJ, Wilson V, Neumann R, Keyte J. 1988. Amplification of human mini satellities by the polymerase chain reaction: towards DNA fingerprinting of single cells. Nucleic Acids Res 16: 10953-10971.
10. Longo MC, Berninger MS, Hartley JL. 1990. Use of uracil DNA glycosy-lase to control carry-over contamination in polymerase chain reactions. Gene 93:125-128.
11. Mora CP. 2006. Diagnóstico de enfermedad viral en la cría de trucha arco iris Oncorhynchus mykiss en piscigranjas de la región Cusco. En: II Congreso Nacional de Acuicultura. Lima: Universidad Nacional Agraria La Molina.
12. [OIE] Organización Mundial de Sanidad Animal. 2006. Manual de pruebas de diagnóstico para los animales acuáticos. 5° ed. París: OIE. 499 p.
13. PRODUCE. 2008. La Acuicultura en el Perú. Dirección General de Acuicultura. Lima, Perú: Ministerio de Producción. [Internet], [10 julio 2008]. Disponible en: http://www.produ-ce.gob.pe/pesqueria
14. Ririe KM, Rasmussen RP, Wittwer CT. 1997. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction. Annal Biochem 245: 154-160.
15. Rodríguez S, Borrego JJ, Pérez Prieto SI. 2001. Comparative evaluation of five serological methods and RT-PCR assay for the detection of aquabirnavi-ruses in fish. J Virol Methods 97: 23-31.
16. Rodríguez-Saint-Jean S, Pérez Prieto SI, Vilas Minondo MP. 2003. Infectious Pancreatic Necrosis Virus: biology, pathogenesis, and diagnostic methods. Adv Virus Res 62: 113-165.
17. Rychlik W, Spencer WJ, Rhoads RE. 1990. Optimization of the annealing temperature for DNA amplification in vitro. Nucleic Acids Res 18: 6409-6412.
18. Williams K, Blake S, Sweeney A, Sin-ger JT, Nicholson BL. 1999. Multiplex Reverse Transcriptase PCR assay for simultaneous detection of three fish viruses. J Clin Microbiol 37: 4139-4141.
19. Valasek MA, Repa JJ. 2005. The power of real-time PCR. Adv Physiol Educ 29: 151-159.