Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.27 no.2 Lima abr./jun. 2016
http://dx.doi.org/10.15381/rivep.v27i2.11641
ARTÍCULOS PRIMARIOS
Sexaje molecular a partir de heces en osos de anteojos (Tremarctos ornatus)
Molecular sexing from stools in spectacled bear (Tremarctos ornatus)
Cristina Caselli S.1, Lenin Maturrano H.2,3
1 Laboratorio de Microbiología y Parasitología Veterinaria,
2 Laboratorio de Zootecnia y Producción Agropecuaria, Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
3 E-mail: lenin.maturrano@gmail.com
RESUMEN
El oso de anteojos (Tremarctos ornatus) se encuentra vulnerable debido a la caza y destrucción de su medio ambiente y es de difícil observación en vida libre, lo que obstaculiza su manejo e investigación, por lo que el uso de muestras no invasivas como heces se convierte en una gran herramienta. El objetivo del presente estudio fue adaptar una técnica de sexado mediante la reacción en cadena de la polimerasa (PCR) de ADN extraído de células exfoliadas del colon presentes en muestras de heces. Se utilizaron 17 muestras (10 de hembras y 7 de machos) de osos de sexo conocido y criados en cautiverio en cuatro instituciones de la zona de Lima, Perú. Para la extracción de ADN se utilizó un kit comercial específico para heces. Se logró extraer de 20 a 30 ng de ADN de cada muestra. Mediante PCR se evaluaron dos secuencias de genes: amelogenina con los cebadores SE47-SE48 y la región determinante del sexo en el cromosoma Y (SRY) con los cebadores SRYB3- SRYB5. Con el gen de amelogenina se logró sexar a los 17 osos (100%), mientras que con el gen SRY no se logró el sexado. Se concluye que con el gen amelogenina hay una coincidencia de 100% entre el sexo conocido y el sexo hallado mediante PCR de ADN extraído de heces.
Palabras clave: oso de anteojos, sexado, gen de amelogenina, SRY, PCR, heces
ABSTRACT
The spectacled bear (Tremarctos ornatus) is a vulnerable species because of hunting and destruction of its environment. Also, it is difficult to monitor in the wild hampering the management and research efforts and therefore, the use of non-invasive samples such as faeces becomes a useful tool. The aim of this study was to develop a technique for sexing by the Polymerase Chain Reaction (PCR) using DNA extracted from exfoliated colon cells present in faecal samples. Seventeen samples (from 10 females and 7 males) were collected from bears of known sex reared in captivity in four institutions in Lima Peru. DNA extraction was done by using a specific kit for faecal samples. The concentration of extracted DNA varied between 20 to 30 ng. Using PCR, two gene sequences were evaluated: amelogenin with primers SE47-SE48 and sex-determining region in the Y chromosome (SRY) with primers SRYB5-SRYB3. Sex determination by the amelogenin gene was achieved in all bears (100%), whereas failed in all samples when using the SRY gene primers. It is concluded that there is 100% match with the amelogenin gene between the known and detected sex using the PCR of DNA extracted from faeces.
Key words: spectacled bear, sexing, amelogenin gene, SRY, PCR, faeces
INTRODUCCIÓN
El oso de anteojos (Tremarctos ornatus), también conocido como el oso andino, vive en la región andino amazónica de América del Sur y es una de las ocho especies de osos que existen en el planeta. Habita en ecosistemas que van desde los 250 msnm en el desierto costero hasta los páramos a 4750 msnm, mostrando preferencia por bosques húmedos entre 1990 y 2350 msnm, llegando a ocupar una área de 260 000 km2. Dos tercios de la población de osos de anteojos se encuentran en el Perú (Peyton et al., 1998).
El número de oso de anteojos está disminuyendo rápidamente, quedando en el Perú, según el último censo, una población cercana a 2000 (Orejuela y Jorgenson, 1996); aunque su número es aún incierto. La conservación de esta especie se encuentra amenazada por la disminución de su hábitat debido a la ganadería migratoria, incendios forestales, tala de bosques y la fragmentación por colonización; asimismo, por el tráfico ilícito como mascota de circo y por la creencia que sus garras tienen propiedades curativas. La ausencia de información actualizada hace difícil desarrollar planes adecuados para su conservación (IUCN, 2015).
La visualización de estos animales en vida libre es difícil (Castellanos et al., 2005), de allí que se deben utilizar rastros indirectos como sus heces (Peyton, 1980). Estas muestras pueden ser sometidas a pruebas moleculares debido a que contienen células exfoliadas del colon de las que se puede extraer material genético (Maturrano et al., 2012). No obstante, no existe una técnica para extraer ADN de las heces del oso de anteojos en suficiente cantidad y calidad que permita realizar análisis moleculares, como la determinación del sexo.
El objetivo del presente estudio fue acondicionar una prueba molecular que permita sexar al oso de anteojos utilizando ADN extraído de muestras de heces mediante PCR. Para ello se evaluaron parámetros de temperatura y concentración de ADN y se usaron dos genes como candidatos a marcador genético para el sexado. Se espera que con esta técnica se pueda, a futuro, determinar la proporción hembra/macho en animales de vida libre y estimar su población en una determinada región, así como conocer la estructura y la dinámica de una población (Maturrano et al., 2012), a fin de desarrollar mejores estrategias de conservación (Griffiths y Tiwari, 1995).
MATERIALES Y MÉTODOS
Tomas de Muestras
Se recolectaron muestras de heces de 17 osos en cautiverio (10 hembras y 7 machos). Los animales fueron del zoocriadero del Colegio La Inmaculada (n=2), Parque Zoológico de Huancayo - CRCLL (n=3), zoológico del Parque de Las Leyendas (n=6) y Parque Zoológico Huachipa (n=6). Las muestras fueron tomadas con la autorización N.° 0194-2014-MINAGRI-DGFFS/DGEFFS.
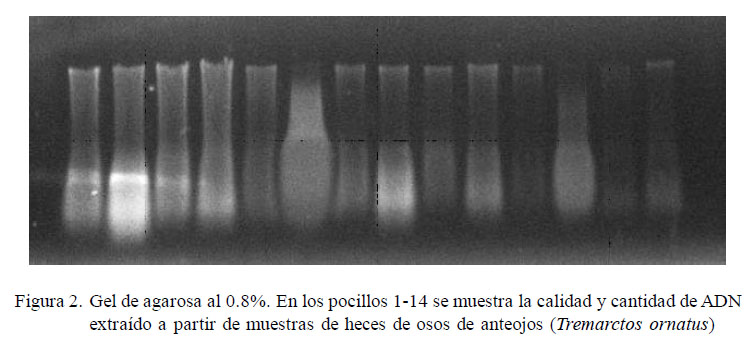
Las muestras fueron conservadas con etanol absoluto y se rotularon con el sexo, nombre, ubicación y fecha de colecta (Fig. 1).
Extracción de ADN
Se tomaron entre 180 y 200 mg de la superficie de las heces (Fig. 1), eliminando los residuos de alimentos y tomando las áreas más brillosas con una hoja de bisturí. Para la extracción de ADN se utilizó el kit comercial QIAamp® DNA Stool Mini Kit (Qiagen) con una modificación en el protocolo. Para esto, se colocó el material fecal extraído con 3-4 ml de buffer de lisado (ASL) y se agitó con un vortex por 15 minutos durante cuatro días consecutivos. Luego se continuó con el proceso de extracción descrito por el fabricante del kit.
Las extracciones fueron realizadas con autorización R.D. N° 037-2015 SERFORDGGSPFFS- DGSPFS.
Amplificación del Gen Amelogenina (SE47-SE48)
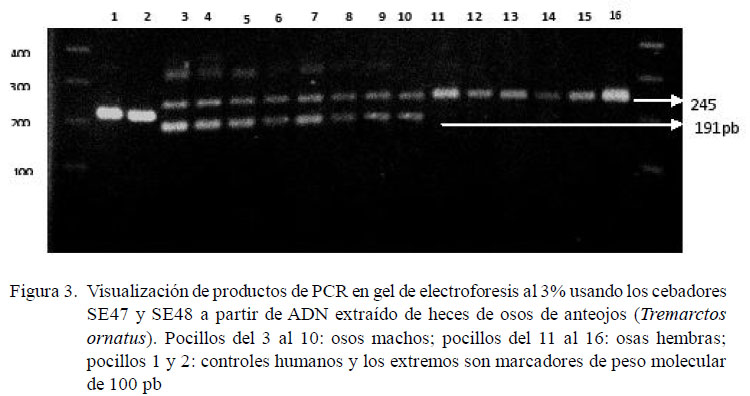
Se usaron los cebadores SE47 (5’CAGCCAAACCTCCCTCTGC) y SE48 (5’CCCGCTTGGTCTTGTCTGTTGC) (Yamamoto et al., 2002).
Para la PCR se usó un volumen final de 20 μl conteniendo 10 μl de Master-Mix (Taq, dNTPs) y 0.4 μl de cada cebador a una concentración de trabajo de 20 pM. Las condiciones fueron: 95 °C por 9 min, seguido por 35 ciclos de 94 °C por 30 s, 58 °C por 30 s, 72 °C por 1 min y, finalmente, 72 °C por 5 min.
Para la visualización de los productos del PCR, se diluyeron con Buffer Loading a razón de 5:1, que se depositaron en un volumen final de 20 μl en un gel de agarosa al 3% durante 2 h y 30 min a 100 voltios. Se utilizó un marcador de 100 bp para corroborar los tamaños de los alelos.
Amplificación del Gen SRY (SRYB3- SRYB5)
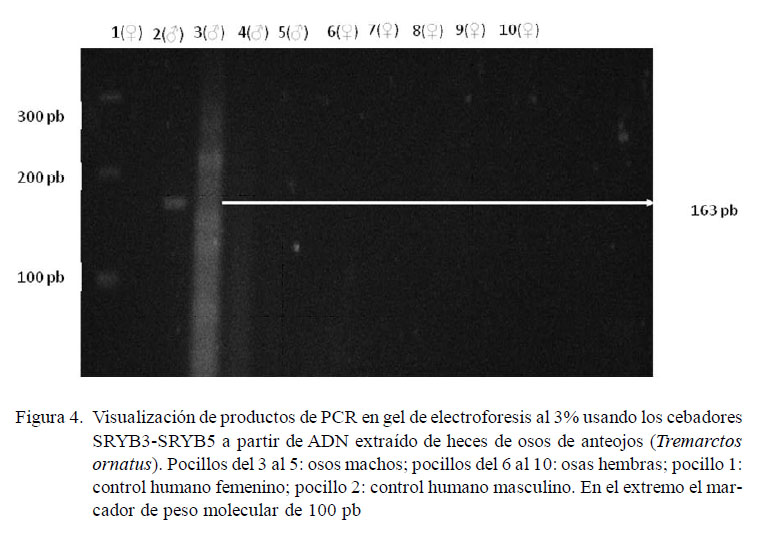
Se emplearon los cebadores SRYB3 (5’GCCAGTAGTCTCTGTGCCTCCT) y SRYB5 (5’TGAACGCTTTCATTGTGTGGTC), segmentos del gen SRY presente en el cromosoma Y, los cuales han sido usados en varias especies de mamíferos (Pomp et al., 1995).
Para la PCR se uso un volumen final de 20 μl, con aproximadamente 20 a 30 ng ADN, 10 μl de Master-Mix (Taq, dNTPs) y 0.4 μl de cada cebador a una concentración de trabajo de 20 pM. Las condiciones fueron: 95 °C por 4 min, seguido por 35 ciclos de 95 °C por 15 s, 55 °C por 45 s, 72 °C por 30 s y, finalmente, 72 °C por 5 min.
Para la visualización de los productos del PCR se siguió el procedimiento indicado para el gen amelogenina.
RESULTADOS
En las 17 muestras fecales hubo heces semilíquidas y sólidas, algunas con abundante material fibroso (cáscara de choclo, restos de camote y otros); sin embargo, en todas se obtuvo una concentración de 20 a 30 ng de ADN. En la electroforesis, algunas muestras no presentaron bandas definidas de ADN, sino un barrido (ADN degradado), por lo que se estima que el ADN obtenido era de baja calidad (Fig. 2).
El sexo (10 hembras y 7 machos) fue identificado en forma correcta en las 17 muestras. En el genotipo hembra se vio una doble banda de 245 pb correspondientes a los cromosomas XX, mientras que en machos de visualizaron dos bandas, una de 245 pb y otra de 191 pb correspondientes a los cromosomas X e Y, respectivamente. Como controles se usó ADN humano femenino y masculino (Fig. 3).
En ninguna de las muestras analizadas con los cebadores SRY específicos se logró determinar el sexo. Aquí se usó un control de ADN masculino, el cual, como era de esperar, amplificó una sola banda de 163 pb (Fig. 4).
DISCUSIÓN
El empleo de técnicas no invasivas para caracterizar el sexo de individuos de una población ha aumentado enormemente en los últimos años, debido al desarrollo de técnicas cada vez más eficientes para la extracción de ADN a partir de muestras muy pequeñas y degradadas como las heces. Para ello la toma y conservación de muestras es de vital importancia.
La calidad y consistencia de las heces es dependiente del estado de salud del animal y de la dieta que reciben en cada zoológico. En este estudio, se aceptó todo tipo de heces (semilíquidas, liquidas, patosas y en bloques) y fueron conservadas en etanol absoluto para la extracción de ADN. Esto es importante pues en condiciones de vida libre se puede encontrar estos tipos de heces y deberían ser usadas sin inconvenientes. Las muestras conservadas en etanol absoluto mostraron tan igual desempeño que muestras colectadas en frascos con los silicagel utilizados en el estudio del oso pardo (Castellanos et al., 2005), lo cual disminuye el costo de la prueba, además de ser de fácil adquisición.
En este estudio, la extracción de ADN fue un paso crítico para obtener buenos resultados, siendo el método fenol-cloroformo el más común para obtener ADN de buena calidad y cantidad; además, permite que el ADN se mantenga estable por más tiempo (Castellanos et al., 2005).
El método de la sílica para extraer ADN a partir de heces es muy efectivo (Taberlet et al., 1997; Frantzen et al., 1998; Marshall y Ritland, 2002). En la actualidad, las casas comerciales están desarrollando nuevos sistemas de extracción de ADN a partir de muestras de mala calidad, como heces y tejidos muy degradados. Este tipo de técnicas se presenta a veces como la única alternativa posible en especies muy amenazadas; sin embargo, el método a partir de excrementos no es fácil, debido a que el ADN obtenido suele ser escaso y muy fragmentado, además de contener restos de plantas. Por este motivo, en el presente estudio se modificó el protocolo del fabricante del kit en la fase de lisis celular empleada con éxito en un estudio previo en heces de vicuñas (Maturrano et al., 2012). Con esta modificación se extrajo cantidades óptimas de ADN sin necesidad de realizar múltiples réplicas de cada análisis (Taberlet et al., 1996).
Los resultados muestran que solo se pudo amplificar adecuadamente con el gen de amelogenina, permitiendo identificar correctamente el sexo de los animales. El sexo en el oso de anteojos ha llegado a ser identificado por ambos genes pero empleando pelos como fuente de ADN (Page’s et al., 2009). La extracción de ADN a partir de pelos resulta en una mayor cantidad de ADN pues contienen menos inhibidores de PCR que las muestras de heces. Asimismo, la utilización de cebadores no específicos para la especie en muestras tan degradadas como heces suelen no ser fiables ya que es posible que no amplifiquen (Page’s et al., 2009).
A través del presente trabajo se pudo demostrar que es posible sexar al oso de anteojos mediante PCR usando el gen de la amelogenina (SE47 y SE48) a partir de muestras no invasivas como las heces. Se espera que en el futuro esta prueba sea usada en osos de vida libre proporcionando información de vital importancia para su conservación.
CONCLUSIONES
-
Se logró adaptar una prueba molecular capaz de sexar al oso de anteojos (Tremarctos ornatus) usando el gen amelogenina (SE47 y SE48) a partir de ADN extraído de heces.
-
El gen SRY (SRYB3-SRYB5) no permite determinar el sexo en estos animales a partir de ADN extraído de heces. Agradecimientos Los autores agradecen al Vicerrectorado de Investigación de la UNMSM por el financiamiento del estudio, a la Sección de Biología y Genética Molecular de la FMV-UNMSM por brindar las facilidades para los análisis de ADN, y a los zoológicos y zoocriaderos que facilitaron el uso de las muestras.
LITERATURA CITADA
1. Castellanos A, Altamirano MA, Tapia G. 2005. Ecología y comportamiento de osos andinos reintroducidos en la reserva biológica Maquipucuna, Ecuador: implicaciones en la conservación. Politécnica 26(1): 54-82. [ Links ]
2. Frantzen MA, Silk JB, Ferguson JW, Wayne RK, Kohn MH. 1998. Empirical evaluation of preservation methods for faecal DNA. Mol Ecol 7: 1423-1428. [ Links ]
3. Griffiths R, Tiwari B. 1995. Sex of the last wild Spix’s macaw. Nature 375: 454- 455. doi:10.1038/375454a0 [ Links ]
4. Hänni C, Laudet V, Stehelin D, Taberlet P. 1994. Tracking the origins of the cave bear (Ursus spelaeus) by mitochondrial DNA sequencing. Proc Natl Acad Sci USA 91: 12336-12340. [ Links ]
5. [IUCN] International Union for Conservation of Nature. 2015. The IUCN Red List of Threatened Species. [Internet]. Disponible en http:// ww.iucnredlist.org/details/22066/0
6. Marshall HD, Ritland K. 2002. Genetic diversity and differentiation of Kermode bear populations. Mol Ecol 11: 685-697. [ Links ]
7. Maturrano L, Aguilar JM, Krüger DP, Chávez IJ, Rosadio AR, Wheeler JC. 2012. Optimización de una técnica para la extracción de ADN de heces de vicuña (Vicugna vicugna mensalis). Rev Inv Vet Perú 23: 369-376. doi: 10.15381/ rivep.v23i3.918 [ Links ]
8. Orejuela J, Jorgenson J. 1996. Plan de acción del oso andino. Encuentro nacional sobre conservación y manejo del oso andino. Colombia: Ministerio del Medio Ambiente. Colombia. 27 p. [ Links ]
9. Page’s M, Maudet C, Bellemain E, Taberlet P, Hughes S, Hänni C. 2009. A system for sex determination from degraded DNA: a useful tool for palaeogenetics and conservation genetics of ursids. Conserv Genet 10: 897-907. doi: 10.1007/s10592-008-9650-x [ Links ]
10. Peyton B. 1980. Ecology, distribution, and food habits of spectacled bears, Tremarctos ornatus, in Peru. J Mammal 61: 639-652. doi: 10.2307/1380309 [ Links ]
11. Peyton B, Yerena E, Rumiz DI Jorgenson J, Orejuela J. 1998. Status of wild Andean bears and policies for their management. Ursus 10: 87-100. [ Links ]
12. Pomp D, Good BA, Geisert RD, Corbin CJ, Conley AC. 1995. Sex identification in mammals with polymerase chain reaction and its use to examine sex effects on diameter of day- 10 or -11 pig embryos. J Anim Sci 73: 1408-1415. [ Links ]
13. Taberlet P, Camarra JJ, Griffin S, Uhrès E, Hanotte O, Waits LP, et al. 1997. Noninvasive genetic tracking of the endangered Pyrenean brown bear population. Mol Ecol 6: 869-876. [ Links ]
14. Taberlet P, Griffin S, Goossens B, Questiau S, Manceau V, Escaravage N, et al. 1996. Reliable genotyping of samples with very low DNA quantities using PCR. Nucleic Acids Res 24: 3189- 3194. [ Links ]
15. Yamamoto K, Tsubota T, Komatsu T, Katayama A, Murase T, Kita I, Kudo T. 2002. Sex identification of Japanese black bear, Ursus thibetanus japonicus, by PCR on amelogenin gene. J Vet Med Sci 64: 505-508. doi: 10.1292/ jvms.64.505 [ Links ]
Recibido: 17 de marzo de 2015
Aceptado para publicación: 20 de febrero de 2016