Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.27 no.3 Lima jul./set. 2016
10.15381/rivep.v27i3.11476
Diversidad genética de palomas domésticas (Columba livia) en Lorica, Colombia, utilizando genes que codifican la coloración del plumaje
Genetic diversity of the domestic pigeon (Columba livia) in Lorica, Colombia using genes that encode plumage colour
Luis Alfonso Causil Vargas1,3, Adrián Rodríguez De La Barrera1, Orlando Causil Vargas2
1 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Colombia
2 Departamento de Ciencias Naturales y Educación Ambiental, Facultad de Educación y Ciencias Humanas, Universidad de Córdoba, Colombia
3 E-mail: luiscausilvargas@gmail.com
RESUMEN
El objetivo de este trabajo fue evaluar la diversidad genética de las poblaciones de palomas domésticas (Columba livia) en Lorica, Colombia, utilizando genes que codifican la coloración y diseño del plumaje. Se realizaron muestreos aleatorios en seis colonias entre noviembre y diciembre de 2015. Mediante excursiones urbanas, observación directa y registros fotográficos se estudiaron 356 palomas. Se utilizaron los marcadores autosómicos que codifican la coloración y diseño del plumaje: Grizzle (G), Spread (S), Checker (C), y el locus ligado al sexo Ash-Red (B). Los parámetros genéticos de frecuencia alélica, diversidad genética, equilibrio Hardy-Weinberg y estructura poblacional fueron calculados a través del programa PopGene 1.31, la estructura genética y la distancia genética mediante el programa FSTAT v. 2.9.3.2, y el dendrograma se realizó con el programa MEGA 5. Los marcadores Spread y Checker fueron los de mayor frecuencia, mientras el marcador Ash-Red presentó los valores más bajos. Se obtuvo escasa diferenciación genética entre las poblaciones y un elevado flujo génico, por lo que se asume ausencia de consanguinidad. Además, se observó un exceso de heterocigotos y ausencia de equilibrio Hardy-Weinberg, y se evidenció posible selección natural para el marcador Spread.
Palabras clave: frecuencias alélicas, diversidad genética, flujo génico, equilibrio Hardy- Weinberg
ABSTRACT
The aim of this study was to evaluate the genetic diversity of populations of domestic pigeons (Columba livia) in Lorica, Colombia using genes that encode plumage color. Random sampling were made in six colonies between November and December 2015. Through urban excursions, direct observation and photographic records, 356 pigeons were studied. Autosomal markers that encode plumage coloration and design were used: Grizzle (G), Spread (S), Checker (C), and sex-linked locus Ash-Red (B). The genetic parameters allele frequencies, genetic diversity, Hardy-Weinberg equilibrium and population structure were calculated by PopGene 1.31, genetic structure and genetic distance by FSTAT v.2.9.3.2 and the dendrogram was made using the program MEGA 5. Spread and Checker markers were the most frequent, while Ash-Red marker presented the lowest values. Low genetic differentiation was obtained among populations and high gene flow and therefore, it is assumed absence of inbreeding. Besides, an excess of heterozygotes and absence of Hardy-Weinberg equilibrium was observed, and possible natural selection for the Spread marker was evident.
Key words: allele frequencies, genetic diversity, gene flow, Hardy-Weinberg equilibrium
INTRODUCCIÓN
Las poblaciones naturales, dentro o entre especies, tienen implicaciones importantes, debido a su potencial en cripsis, especiación, adaptación y selección sexual. Los cambios en la coloración se producen en escalas de tiempo evolutivas sorprendentemente cortos, mientras que la repetición de patrones de colores similares a través de grandes distancias filogenéticas es común (Poelstra et al., 2015).
Estudios de mecanismos selectivos que permiten el mantenimiento de polimorfismo en poblaciones naturales de palomas domésticas han demostrado cómo un espectro de codificación y mutaciones reguladoras en un pequeño número de genes puede interactuar para generar diversidad fenotípica sustancial en un modelo clásico de evolución darwiniana (Domyan et al., 2014).
La paloma es probablemente originaria del sur de Asia, desde donde inició su migración a todo el continente, África, Europa y resto del mundo. Inicialmente vivían en acantilados rocosos inaccesibles, donde anidaban en las oquedades de las paredes y se desplazaban a los campos cercanos en busca de alimento, razón por la cual posiblemente desarrollaron un mecanismo especial de orientación, basado en la vista y en una gran memoria. La paloma se encuentra entre las aves que forman colonias de mayor éxito de las ciudades, donde viven en estado de semidomesticación (Giunchi et al., 2012; Pardo et al., 2015b).
La paloma doméstica (Columba livia) puede alcanzar los 36 cm y pesar hasta 360 g. El color de su plumaje es muy diverso, siendo más común el color gris en cabeza, cola y alas, con dos franjas negras al final de las alas, una franja negra y ancha en la cola y las patas rojas. La cabeza es generalmente oscura y con frecuencia presenta una iridiscencia verde-púrpura (Olalla et al., 2009). No obstante, el plumaje es muy variable entre individuos, observándose animales de color blanco, negro, café, manchados y variaciones de estos colores. Además, presenta pico oscuro con cara blanca en la base, patas rojizas y no presenta dimorfismo sexual (Casanova, 2013).
Las bases genéticas responsables del color y diseño del plumaje en las poblaciones de palomas domésticas han sido descritas (Haag,1987; Hetmañski y Jarosiewicz, 2008; Ferman et al., 2010; Čanády y Mošanský, 2013).
En Colombia, Pardo et al. (2015a) realizaron la caracterización genotípica de la paloma doméstica de la ciudad de Bogotá, utilizando genes que codifican la coloración y diseño del plumaje. Asimismo, se dispone de un estudio donde se determinó la abundancia y distribución de la paloma doméstica en Montería, Córdoba (Begambre y Pardo, 2015), y otro donde se determinó la densidad poblacional en el antiguo mercado de la ciudad de Sincelejo, Sucre (Sánchez, 2015).
Los marcadores fenotípicos relacionados con la coloración constituyen una valiosa herramienta en estudios donde se analiza la estructura genética de las poblaciones debido a su gran contenido informativo, bajo costo, fácil manipulación e identificación en campo y rápida obtención de resultados (Pardo et al., 2015b). Por esta razón, dada la escasa información existente sobre estos estudios y la gran presencia de palomas en ambientes urbanos, el presente estudio tuvo como objetivo analizar la diversidad genética de la paloma doméstica (Columba livia) en el municipio de Lorica, Córdoba, Colombia, mediante genes que codifican la coloración y diseño del plumaje.
MATERIALES Y MÉTODOS
El estudio se realizó en la zona urbana de Lorica (09º13’54" Latitud Norte y 75º49’11" Longitud Oeste), con una temperatura promedio de 28 °C. Se recolectaron muestras de seis colonias localizadas en Plaza de Bolívar, Plaza de la Cruz, Mercado Público, La Troncal, Estadio 3 de Mayo y Estadio Santa Teresita.
Se realizaron muestreos aleatorios entre noviembre y diciembre de 2015 mediante excursiones urbanas, observación directa y registros fotográficos. Se realizó una clasificación fenotípica de cada individuo de las bandadas de palomas (n=356). Se estudió toda el área urbana del municipio (7.3 km²), y se hicieron transeptos entre 500 m2 y 1 km2, demarcándose una ruta.. Cada ruta fue utilizada una sola vez con la finalidad de evitar el remuestreo. Se registró la presencia o ausencia de los marcadores autosómicos que codifican para el diseño y coloración de plumaje, siguiendo el modelo y metodología propuesto por Pardo et al. (2015a): Grizzle (G), Spread (S), Checker (C), y el locus ligado al sexo Ash-Red (B).
Las frecuencias alélicas de cada marcador a nivel poblacional y global, así como las medidas de diversidad genética establecidas por Nei (1973) correspondientes a la heterocigosidad esperada (He), heterocigosidad esperada de la población total (HT), coeficiente de diferenciación genética (GST), flujo génico (Nm), equilibrio Hardy-Weinberg y la distancia genética de Nei (1972) entre las poblaciones fueron estimadas a través del programa PopGene 1.31 (Yeh et al., 1999). La estructura genética de las poblaciones, atendiendo a los índices de fijación propuestos por Wright (FIS, FIT y FST), fue calculada mediante el programa FSTAT v. 2.9.3.2 (Goudet, 1995).
La elaboración del dendrograma que representa los valores estimados de distancia genética se realizó a través del método UPGMA (Unweighted Pair Group Methodwith Arithmetic Mean), empleando el programa MEGA 5.2 (Tamura et al., 2011).
RESULTADOS
Frecuencias Alélicas
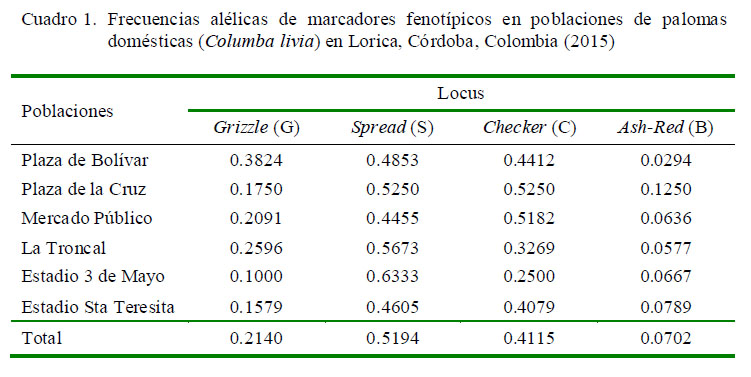
Las frecuencias alélicas para cada subpoblación (Cuadro 1) mostraron que el gen Spread fue el más frecuente, principalmente en las subpoblaciones Estadio 3 de Mayo (p=0.6333), La Troncal (p=0.5673) y Plaza de la Cruz (p=0.5250).
En segundo lugar, la mayor frecuencia la evidenció el marcador Checker en las colonias de Plaza de la Cruz (p=0.5250) y Mercado Público (p= 0.5182). Por su parte, el marcador Ash-Red, responsable de la presencia de producir palomas de plumaje rojo, mostró las menores frecuencias tanto a nivel global (p=0.0702) como en las subpoblaciones.
Diversidad Genética
El nivel medio de variabilidad genética de cada marcado fue relativamente bajo (Cuadro 2). La heterocigosidad esperada más alta se encontró para el marcador Spread (0.5005) y la más baja para el marcador Ash- Red (0.1259). Asimismo, los valores de heterocigosidad observada fueron bajos, siendo entre un máximo de 0.0169 para el marcador Checker y un mínimo de 0.0000 para el gen Ash-Red.
Las poblaciones en el test de equilibrio Hardy-Weinberg a nivel global reportaron valores altos para Chi cuadrado evidenciando ausencia de equilibrio.
Diferenciación Genética y Flujo Génico
El nivel de diferenciación genética en la población de palomas domésticas de Lorica fue muy bajo (GST=0.027), lo cual indica que aproximadamente el 2.7% de la variación detectada se debe a diferencias entre las poblaciones. Es así que las seis subpoblaciones no fueron significativamente diferentes para los marcadores en estudio, de allí que se debería realizar una subestructura de la población para un mejor análisis.
El valor de flujo génico (Nm=7.654) permite suponer que las poblaciones mantienen un grado de intercambio genético, asumiéndose un total de aproximadamente ocho migrantes por generación. Asimismo, la cifra obtenida resultó ser mayor que 1, lo que indica que las subpoblaciones se comportan como una sola población.
Estructura Poblacional
La correlación entre los genes de los individuos y los de la subpoblación se representa por FIS (Cuadro 3). Los valores negativos de FIS para todos los marcadores evidencian un exceso de heterocigotos de los individuos con respecto a cada población y, por lo tanto, se asume ausencia de consanguinidad.
La correlación entre los genes de los individuos y los de la población total se representa por FIT, y corresponde a la endogamia total. Este índice mostró, en forma similar, exceso de heterocigotos de los individuos con respecto a la población total, abarcando valores de 0.0001 a -0.0613 para los marcadores Checker y Spread, respectivamente (Cuadro 3).
El índice FST mide la reducción en la heterocigosis debida a diferenciación genética entre poblaciones. El valor promedio de FST resultó bajo (-0.0322; Cuadro 3), indicando poca diferenciación génica existente entre las poblaciones, lo que sugiere que las frecuencias alélicas son estadísticamente similares en todas las subpoblaciones; es decir, con poca diferenciación entre ellas.
Distancia Genética
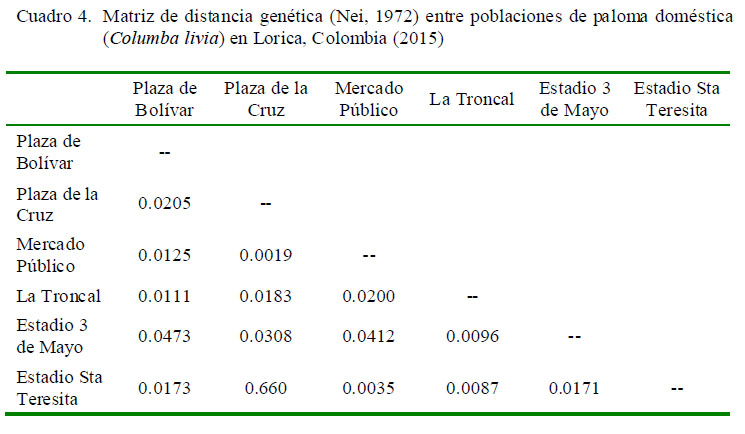
La distancia genética entre las poblaciones fue baja, siendo Plaza de la Cruz y Mercado Público las poblaciones más cercanas (Cuadro 4), mientras que la colonia Plaza de la Cruz y la colonia Estadio Santa Teresita resultaron ser las de mayor diferencia génica.
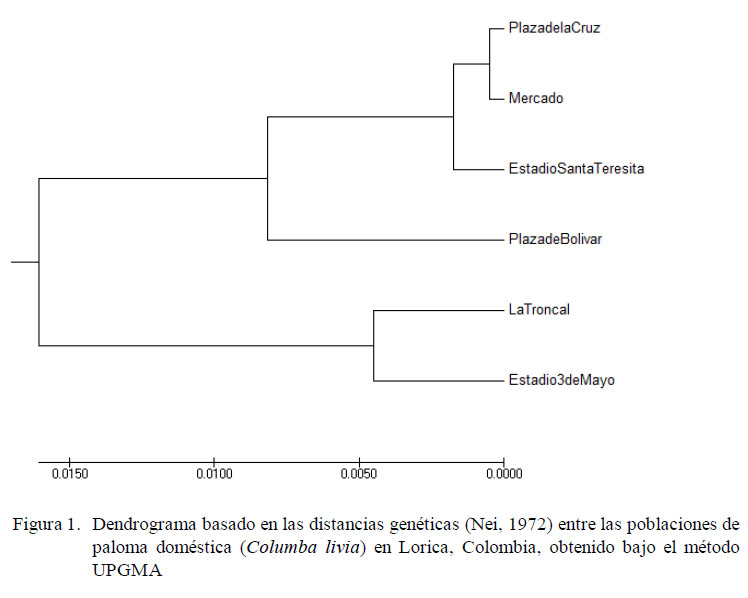
El dendrograma (Figura 1) evidencia la similaridad genética entre las colonias de Plaza de la Cruz y Mercado Público a las cuales se asocia la colonia Estadio Santa Teresita, y estas tres con Plaza de Bolívar. Por otro lado, las colonias La Troncal y Estadio 3 de Mayo mostraron similitud. No obstante, los valores de la distancias resultaron ser poco significativos, lo que permite deducir que las subpoblaciones se encuentran genéticamente muy relacionadas.
DISCUSIÓN
La alta frecuencia de los marcadores melánicos Spread y Checker en esta investigación coinciden con otros estudios (Èanády y Mošanský, 2013; Pardo et al., 2015). La prevalencia de fenotipos oscuros en aves es generalmente coherente con fluctuaciones constantes de presión natural como la selección, que mantienen la heterogeneidad de coloración, basada en el grado de melanina, entre y dentro de poblaciones. Es así que la coloración puede variar con la historia de vida y las características morfológicas, fisiológicas y de comportamiento en el espacio y en el tiempo (Hõrak y Männiste, 2016).
Bajo restricción de alimentos, las hembras de fenotipos más oscuros tienden a tener una producción más alta de huevos y, por otra parte, las crías bajo condiciones limitadas de alimentos, tienen una masa corporal superior al nacer cuando son descendientes de progenitores ‘oscuros’ (Jacquin et al., 2012). Esto sugiere un efecto genético positivo de individuos más oscuros, en condiciones de alimentación limitada. En contraste, cuando la comida es abundante, hembras de fenotipos claros y oscuros tienen producciones similares de huevos, pero los adultos más oscuros pierden significativamente menos masa corporal que sus congéneres más claros (Jacquin et al., 2011).
Otro factor que estaría relacionado con elevada frecuencia de marcadores melánicos tiene que ver con la susceptibilidad a parásitos. La paloma doméstica tiene usualmente alta carga parasitaria (Naupay et al., 2015). Los individuos melánicos reflejan menor susceptibilidad a endoparásitos, y tienen una mayor respuesta inmune frente a ellos, que individuos de fenotipos más claros (Jacquin et al., 2011). Esta «asociación genética» traería como consecuencia que el parasitismo podría desempeñar un papel fundamental en el mantenimiento del polimorfismo de color en las poblaciones naturales (Jacquin et al., 2011).
En otro estudio se encontró que los fenotipos más claros estaban menos parasitados que fenotipos oscuros en ambientes suburbanos, pero se vieron afectados negativamente por el aumento de la urbanización, en tanto que en fenotipos oscuros no hubo diferencia significativa, mostrando un comportamiento por igual de cargas parasitarias en todos los ambientes (Jacquin et al., 2013). Por lo tanto, esos resultados sugieren que la coloración podría reflejar estrategias alternativas para hacer frente a la urbanización a través de diferentes exposiciones o susceptibilidad a parásitos (Naupay et al., 2015). En el presente estudio era de esperarse que las mayores frecuencias alélicas las mostrarían los marcadores melanóticos como Spread y Checker, indicando que la distribución espacial de las palomas podría estar condicionadas a las presiones parasitarias, conllevando a una relación de variabilidad de fenotipos según el grado de urbanización.
La desviación del equilibrio de Hardy- Weinberg para los marcadores estudiados puede obedecer a diversas causas. El exceso de heterocigotos podría atribuirse a facto res evolutivos como la selección natural, pues los individuos melánicos confieren ventaja selectiva sobre fenotipos claros. Otro aspecto sería la cercanía geográfica entre las subpoblaciones, lo que ocasiona un aumento considerable del flujo génico entre ellas, dado que la existencia de un alto intercambio de genes previene eventos de endogamia al interior de las poblaciones (Correa et al., 2015). Esto conllevaría a un aumento de genotipos heterocigotos en la población.
El grado de flujo génico permite inferir que las subpoblaciones se encuentran muy relacionadas genéticamente y se comportan como una metapoblación. Si la tasa efectiva de migración Nm es mayor que 1, está ligada al apareamiento aleatorio entre individuos, la deriva génica es despreciable y el flujo génico hace que todas las poblaciones evolucionen de manera cohesiva como un conjunto, situación a la cual se atribuye la aproximación de todas las poblaciones desde el punto de vista estructural (Rodríguez et al., 2013).
El exceso de heterocigotos obtenidos a través de los distintos índices de fijación (FIS y FIT) en cada una de las subpoblaciones estudiadas evidenciaron que se comportan como una estructura casi homogénea, pues un alto intercambio genético supera los efectos de la deriva génica y evita la distinción local, mostrando bajos índices de diversidad genética, de allí que el grado de diferenciación genética entre las subpoblaciones fue bajo (GST= 0.027). Los niveles de diferenciación genética se incrementan significativamente con la distancia geográfica que separa las poblaciones (Jacob et al., 2015). En el caso del presente estudio, este hecho no solo podría estar relacionado por la cercanía geográfica de las subpoblaciones, sino por factores evolutivos como la selección natural y la densidad poblacional antrópica, pues existe una correlación entre las densidades poblacionales antrópicas y las densidades poblacionales de palomas (Przybylska et al., 2012).
Las distancias génicas encontradas entre las subpoblaciones podrían justificarse por el alto flujo génico entre ellas debido a la cercanía geográfica. Otra posibilidad es que los procesos selectivos sean unificantes y ejerzan presiones similares en todas las subpoblaciones y la similitud sea simplemente un fenómeno de convergencia evolutiva. Sin embargo, la homogeneización por flujo genético parece muy factible y sería la causa principal de que la subestructura poblacional se deprecie, y se hagan los respectivos análisis como una metapoblación.
En los resultados del dendograma se debe considerar que la cercanía genética de las poblaciones es directamente proporcional a la cercanía geográfica (Cruz-Salazar et al., 2014).
CONCLUSIONES
El marcador fenotípico de mayor frecuencia fue Spread, posiblemente favorecido por las condiciones ecológicas y factores pleiotrópicos que evidencian una posible selección natural.
La diversidad genética total fue moderada. La mayor parte de la diversidad génica se encontró dentro de poblaciones y poca entre poblaciones, evidenciando que las poblaciones locales comparten una gran proporción de la diversidad total, comportándose como una sola población.
Hubo ausencia del equilibrio de Hardy- Weinberg para todos los marcadores, posiblemente debido a la selección natural y presiones del ambiente en la dinámica poblacional.
LITERATURA CITADA
1. Begambre M, Pardo E. 2015. Abundancia y distribución de las palomas caseras (Columba livia) Bistua Rev Fac Cienc Básicas 13(2): 57-62. [ Links ]
2. Čanády A, Mošanský L. 2013. Population size and plumage polymorphism of feral pigeon (Columba livia forma urbana) from urban environment of Košice city (Slovakia). Zool Ecol 23: 104-110. doi:10.1080/ 21658005.2013.797149 [ Links ]
3. Casanova PM. 2013. Morphological similarities between Spanish pigeon breeds. Turk J Vet Anim Sci 37: 346-351. doi: 10.3906/vet-1111-22 [ Links ]
4. Correa L, Reyes C, Pardo E, Cavadia T. 2015. Genetic diversity detection of the domestic horse (Equus caballus) by genes associated with coat color. Rev MVZ Córdoba 20: 4779-4789. [ Links ]
5. Cruz-Salazar B, Ruiz-Montoya L, Navarrete-Gutiérrez D, Espinoza- Medinilla EE, Vázquez-Domínguez E, Vázquez LB. 2014. Diversidad genética y abundancia relativa de Didelphis marsupialis y Didelphis virginiana en Chiapas, México. Rev Mex Biodiv 85: 251-261. doi: 10.7550/rmb.36116 [ Links ]
6. Domyan ET, Guernsey MW, Kronenberg Z, Krishnan S, Boissy RE., Vickrey AI, Rodgers C, et al. 2014. Epistatic and combinatorial effects of pigmentary gene mutations in the domestic pigeon. Curr Biol 24: 459-464. doi: 10.1016/j.cub.2014.01.020 [ Links ]
7. Ferman L, Peter H, Montalti D. 2010. A study of feral pigeon Columba livia var. in urban and suburban areas in the city of Jena, Germany. Zoologica 8: 1-8. [ Links ]
8. Giunchi D, Albores-Barajas Y, Baldaccini NM, Vanni L, Soldatini S. 2012. Feral pigeons: problems, dynamics and control methods. In: Larramendy M, Soloneski S (eds). Integrated Pest Management and Pest Control – Current and Future Tactics. INTECH. [Internet]. Available in: http://www.intechopen.com/ books/howtoreference/integrated-pestmanagement- and-pest-control-currentand- future-t actics/feral-pigeonsproblems -dynamics-and-cont rolmethods [ Links ]
9. Goudet J. 1995. FSTAT (Version 1.2): a computer program to calculate Fstatistics. J Heredity 86: 485-486. [ Links ]
10. Haag D. 1987. Regulationsmechanismen bei der Strassentaube Columba livia forma domestica (Gmelin 1798). Verh Natur-forsch Ges Basel 97: 31-41. [ Links ]
11. Hetmañski T, Jarosiewicz A. 2008. Plumage polymorphism and breeding parameters of various feral pigeon (Columba livia gm.) morphs in urban area (Gdañsk, North Poland). Pol J Ecol 56: 683-691. [ Links ]
12. Hõrak P, Männiste M. 2016. Viability selection affects black but not yellow plumage colour in greenfinches. Oecologia 180: 23-32. doi: 10.1007/ s00442-015-3451-y [ Links ]
13. Jacob G, Prévot A-C, Baudry E. 2015. The geographic scale of genetic differentiation in the feral pigeon (Columba livia): implications for management. Biol Invasions 17: 23-29. doi: 10.1007/s10530-014-0713-2 [ Links ]
14. Jacquin L, Lenouvel P, Haussy C, Ducatez S, Gasparini J. 2011. Melanin-based coloration is related to parasite intensity and cellular immune response in an urban free living bird: the feral pigeon Columba livia. J Avian Biol 42: 11-15. doi: 10.1111/j.1600-048X. 2010.05120.x [ Links ]
15. Jacquin L, Récapet C, Bouche P, Leboucher G, Gasparini J. 2012. Melanin-based coloration reflects alternative strategies to cope with food limitation in pigeons. Behav Ecol 23: 907- 915. doi: 10.1093/beheco/ars055 [ Links ]
16. Jacquin L, Récapet C, Prévot-Julliard AC, Leboucher G, Lenouvel P, Erin N, Corbel H, et al. 2013. A potential role for parasites in the maintenance of color polymorphism in urban birds. Oecologia 173: 1089-1099. doi: 10.1007/s00442-013-2663-2 [ Links ]
17. Naupay A, Castro J, Caro J, Sevilla L, Hermosilla J, Larraín K, et al. 2015. Ectoparásitos en palomas Columba livia comercializadas en un mercado del distrito de San Martín de Porres, Lima, Perú. Rev Inv Vet Perú 26: 259-265. doi: 10.15381/rivep.v26i2. 11094 [ Links ]
18. Nei M. 1972. Genetic distance between populations. Am Nat 106: 283-292. [ Links ]
19. Nei M. 1973. Analysis of gene diversity in subdivided populations. P Nat Acad Sci 70: 3321-3323. [ Links ]
20. Olalla A, Ruiz G, Ruvalcaba I, Mendoza R. 2009. Palomas, especies invasoras. Biodiversitas 82: 7-10. [ Links ]
21. Pardo E, Cavadía T, Alvarino G. 2015a. Análisis de la diversidad genética de la paloma doméstica (Columba livia) en Bogotá, Colombia utilizando genes que codifican la coloración y diseño del plumaje. Bistua Rev Fac Cienc Básicas 13: 35-45. [ Links ]
22. Pardo E, Causil L, Rodríguez A. 2015b. Estudio de la diversidad genética de gato doméstico (Felis catus) mediante genes asociados al color del pelaje en Lorica-Córdoba, Colombia. Arch Zootec 64: 389-395. [ Links ]
23. Poelstra J, Vijay N, Hoeppner M, Wolf J. 2015. Transcriptomics of colour patterning and coloration shifts in crows. Mol Ecol 24: 4617-4628. doi: 10.1111/ mec.13353 [ Links ]
24. Przybylska K, Haidt A, Myczko L, Ekner-Grzyb A, Rosin ZM, Kwiecinski Z, Tryjanowsk P, et al. 2012. Local and landscape-level factors affecting the density and distribution of the feral pigeon Columba livia var. domestica in an urban environment. Acta Ornithol 47: 37-45. doi: 10.3161/000164512X653908 [ Links ]
25. Rodríguez N, López A, Echeverri J. 2013. Estructura genética poblacional del gen lactoferrina bovino en vacas Holstein del departamento de Antioquia. Rev MVZ Córdoba 18: 3355-3361. [ Links ]
26. Sánchez CV. 2015. Densidad de paloma doméstica (Columbia livia domestica GMELIN, 1789) en el antiguo mercado público de Sincelejo, Sucre, Colombia. Rev Asoc Col Cienc Biol 27: 72-79. [ Links ]
27. Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol 28: 2731-2739. doi: 10.1093/molbev/msr121 [ Links ]
28. Yeh F, Yang R, Boyle T. 1999. Microsoft Windows-based free ware for population genetic analysis (Release 1.31). University of Alberta, Edmonton. [Internet]. Available in: https:// www.ualberta.ca/~fyeh/popgene.pdf [ Links ]
Recibido: 13 de enero de 2016
Aceptado para publicación: 22 de abril de 2016