Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.27 no.3 Lima jul./set. 2016
http://dx.doi.org/10.15381/rivep.v27i3.11989
ARTÍCULOS PRIMARIOS
Desarrollo de un PCR para la Identificación del Parásito Dawestrema (Trematoda: Monogenea) en el Pez Arapaima gigas
Development of a PCR Assay for the Identification of the Parasite Dawestrema (Trematoda: Monogenea) in Arapaima Gigas Fish
Enrique Serrano-Martínez1,3, Manuel Tantaleán V.1, Marco Quispe H.1, Gina Casas V.1, Pablo Londoñe B.2,4
1 Grupo SALUVET-UPCH, Facultad de Medicina Veterinaria y Zootecnia, Universidad Peruana Cayetano Heredia, Lima, Perú
2 Instituto Tecnológico de la Producción, Lima, Perú
3 E-mail: enrique.serrano@upch.pe
4 E-mail: plondone@itp.gob.pe
RESUMEN
Estudios realizados en la especie amazónica Arapaima gigas («paiche») han demostrado la presencia del parásito monogeneo Dawestrema cycloancistrium en altas cargas parasitarias, principalmente en los estadios comprendidos entre los 180 y 365 días de edad. D. cycloancistrium se adhiere principalmente a las branquias, ocasionando una disminución del grado de oxigenación y estrés en el pez, predisponiéndolo a la presentación de enfermedades. El presente estudio tuvo por objetivo desarrollar una técnica de diagnóstico molecular frente a D. cycloancistrium para su aplicación como medida de control en el manejo de la biodiversidad del paiche. Se recolectaron 200 especímenes de D. cycloancistrium, realizándose la extracción de ADN, cuantificación, diseño de cebadores y elaboración del PCR. Se logró la amplificación de la secuencia 236 pb del monogeneo Dawestrema.
Palabras clave: PCR, biología molecular, paiche, parásitos, D. cycloancistrium, sanidad acuícola, biodiversidad
ABSTRACT
Studies in the Amazonian species Arapaima gigas («paiche») have demonstrated the presence of Dawestrema cycloancistrium monogenean parasite in high parasite loads, mainly in stages between 180 and 365 days of age. D. cycloancistrium adheres to the gills causing a decrease in the degree of oxygenation and stress on the fish predisponing to disease presentation. This study aimed to develop a molecular diagnostic technique for D. cycloancistrium in order to aply it in the management of the fish biodiversity. For this purpose, 200 specimens of D. cycloancistrium were collected, DNA was extracted and quantified, primers were designed and a PCR was developed. The amplification of a sequence of 236 bp of the Dawestrema monogenean was obtained.
Key words: PCR, molecular biology, paiche, parasites, D. cycloancistrium, aquatic health, biodiversity
INTRODUCCIÓN
El paiche «Arapaima gigas» es una especie de gran importancia para el poblador amazónico, por ser fuente de alimentación y generador de ingresos a través de la comercialización de sus productos. Sin embargo, años atrás, esta condición propició una intensa captura en su medio natural, causando una disminución de las poblaciones naturales, reflejado en menores desembarques en las últimas dos décadas (de 700 a 144 TM) (Alcántara et al., 2006). Esta especie está considerada en peligro de extinción y se encuentra incluida en el Apéndice II de la Convención sobre el Comercio Internacional de Especies Amenazadas de Fauna y Flora Silvestres (CITES). Debido a ello, los gobiernos regionales de la amazonía peruana promueven la investigación en esta especie, a fin de contribuir a mejorar las condiciones de vida de las poblaciones humanas y generar fuentes de trabajo sin mermar el hábitat ni el tamaño poblacional del paiche.
Las medidas de protección y de incentivo a su crianza deben ser complementadas con estudios sanitarios que determinen la presencia de agentes patógenos en esta especie, así como la prevención y control de enfermedades. Por ello, el grupo de investigación SALUVET-UPCH de la Facultad de Medicina Veterinaria y Zootecnia de la Universidad Peruana Cayetano Heredia, mediante el apoyo del Programa de Ciencia y Tecnología (FINCyT), optó por determinar la presencia de agentes patógenos en paiches criados en cautiverio que podrían estar afectando su productividad. En estos estudios se encontró la presencia del parásito tremátodo monogeneo Dawestrema cycloancistrium en todos los especímenes muestreados (n=30), cuyas edades se encontraban entre los 180 y 365 días (estadio juvenil, principalmente) (E. Serrano-Martínez, Lima, comunicación personal).
D. cycloancistrium se localiza en piel y branquias y ha sido reportado en estadios juveniles de paiches criados en cautiverio (Kritsky et al., 1985; Mathews y Chu, 2007, Araújo et al., 2009), hasta en un 100% de los especímenes estudiados. Al respecto, un estudio asoció su alta carga a la ocurrencia de mortalidad en paiches juveniles, dado que el parásito se adhiere principalmente a las branquias, disminuyendo el grado de oxigenación del hospedero, ocasionando situaciones de estrés con la predisposición a la presentación de enfermedades e incremento de la mortalidad (Buchmann et al., 1994).
La identificación de los monogeneos requiere recuperarlos de las branquias de los peces y evaluar la morfología del parásito, especialmente el tamaño de las estructuras quitinosas del opistohaptor y del complejo genital masculino (Kritsky et al., 1985, Smith et al., 1992). El examen parasitológico es considerado como la prueba «Gold Standard», pero requiere del sacrificio de los peces y de la experiencia de un profesional bien entrenado. Se dispone de pruebas serológicas a base de ELISA que identifican anticuerpos IgM específicos para Cichlidogyrus sp (Sandoval-Gío et al., 2008), monogeneo que infesta a la tilapia (Oreochromis niloticus), pero requiere de altos títulos de anticuerpos. y por otro lado no es muy sensible ni específico. En la actualidad, se viene empleando la técnica de la Reacción en Cadena de la Polimerasa (PCR, Polymerase Chain Reaction), la cual emplea muy poco material parasitario para poder realizar el diagnóstico (Harris et al., 1999; Li et al., 2005; Collins et al., 2010; Freema y Shinn, 2011; Verma et al., 2012).
Ante esto, el objetivo del presente estudio fue desarrollar una técnica de diagnóstico molecular frente a D. cycloancistrium, para su aplicación como medida de control en el manejo de la biodiversidad del paiche.
MATERIALES Y MÉTODOS
Lugar de Estudio y Peces
La fase de campo fue realizada en un centro de cultivo de paiches, localizado en la carretera Iquitos-Nauta, provincia de Maynas, región Loreto. Las muestras obtenidas fueron remitidas al Laboratorio de Parasitología Animal de la Facultad de Medicina Veterinaria y Zootecnia (FAVEZ) de la Universidad Peruana Cayetano Heredia (UPCH), en Lima, Perú.
Se trabajó con 10 paiches menores de 180 días de edad y positivos a la presencia de D. cycloancistrium mediante el examen directo por raspado de las branquias de paiches. Los peces eran parte de un centro de cultivo y estaban destinados al beneficio para consumo humano.
Obtención de Parásitos
Se recolectaron especímenes de D. cycloancistrium mediante raspado de las branquias en 10 paiches del centro de cultivo y que estaban destinados al beneficio para consumo humano. Se recolectaron 200 especímenes que fueron colocados en crioviales de 1.5 ml con cierre hermético conteniendo buffer de lisis (200 ml de buffer ATL del kit DNeasy Tissue®, Qiagen). Los crioviales fueron mantenidos a temperatura ambiente hasta su traslado a los laboratorios de FAVEZ-UPCH.
Del mismo modo, se colocaron 10 especímenes de D. cycloancistrium en una luna de reloj conteniendo 1 ml de suero fisiológico estéril. Se dejó reposar por una hora con la finalidad de obtener antígeno de secreción-excreción, el cual fue almacenado en crioviales a -20 °C.
Desarrollo de la Técnica de Reacción en Cadena de la Polimerasa (PCR)
Extracción de ADN
El ADN de las muestras se extrajo empleando el DNeasy Blood & Tissue Kit (Qiagen), que usa una tecnología avanzada que comprende una membrana de gel de sílice para una rápida y eficiente purificación del ADN celular total sin extracción orgánica o precipitación de etanol. La extracción de ADN se llevó a cabo realizando modificaciones al protocolo del fabricante, empleando 5 min de incubación y 50 µl del buffer AE en dos ocasiones para lograr una mayor concentración de ADN parasitario.
Cuantificación de ADN
El ADN fue cuantificado mediante el uso del equipo Nanodrop 2000. Además, se sembró en geles de agarosa al 1% en buffer TBE 0.5X, con las siguientes condiciones: 150 V, 100 W, 9 A y 45 min. En la corrida se colocó 5 µl de un marcador de peso molecular de 100 pb en un pozo, un control negativo (Agua DEPC [Dietilpirocarbonato]) y un control positivo (ADN de tejido de paiche); estos últimos tres en un volumen de 8 µl. Asimismo, se colocó 8 µl de cada muestra y se les adicionó 2 µl de buffer de corrida.
Diseño de cebadores del gen ribosomal 18S de Dawestrema cycloancistrum
Se ubicaron las secuencias presentes en el GenBank (http://www.ncbi.nlm.nih.gov/genbank/) del gen ribosomal 18S de los monogeneos de la familia Ancyrocephalidae, y se les llevó al formato FASTA, donde se alinearon por intermedio del programa Clustalw2 (http://www.ebi.ac.uk/Tools/msa/clustalw2/).
Una vez obtenida la secuencia consenso se procedió a diseñar los cebadores con la ayuda del programa Primer3 (http://frodo.wi.mit.edu/). Los cebadores diseñados fueron:
-
DAW182F: CAGTCTCCGGGAAACCTGTA
-
DAW182R: CAGACAGCTCGTACCACGAA
-
DAW183F: CCGTTTGTTGCCTGAAGATT
-
DAW183R: GGCAGATGCTTTCGCTTTAG
-
DAW184F: CCGTTTGTTGCCTGAAGATT
-
DAW184R: GCAGATGCTTTCGCTTTAGG
Elaboración del PCR para la detección de Dawestrema y visualización del producto aislado
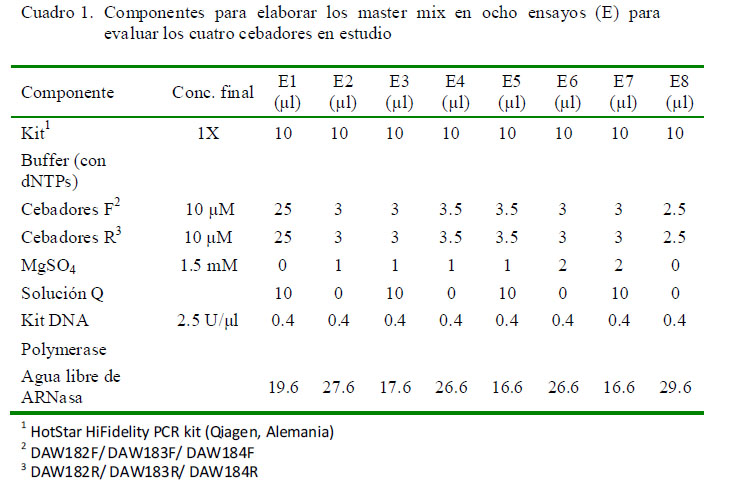
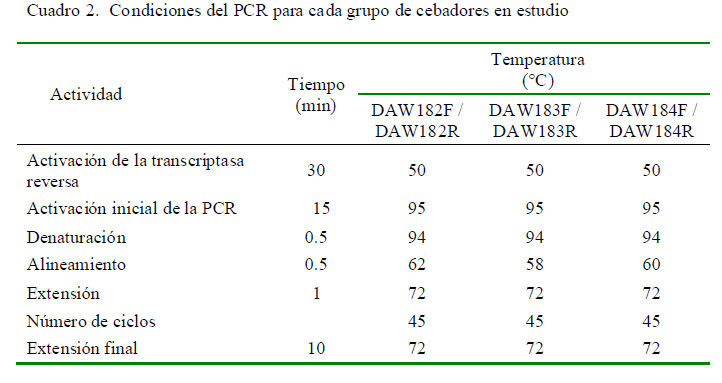
Se diseñaron ensayos con diferentes concentraciones de los cebadores sintetizados anteriormente, MgSO4 utilizó el «HotStar HiFidel y solución Q. Se Polymerase Kit» (Qiagen, Alemania), procediéndose según las indicaciones del fabricante. La disposición de los componentes del master mix para ocho análisis diseñados para cada par de cebadores se presenta en el Cuadro 1, en tanto que las condiciones del PCR se presentan en el Cuadro 2.
Corrida electroforética del producto de PCR
Los productos de PCR fueron corridos en gel de agarosa al 1.0%, con buffer TBE 0.5X a 150 V, 100 A, 150 W y por 45 min. En los pocillos, el marcador de peso molecular de 100 pb se colocó en un volumen de 5 µl y las muestras fueron de 8 µl más 2 µl de buffer de corrida. Al preparar cada gel, se adicionó 0.5 µl de bromuro de etidio por cada 100 µl de agarosa TBE 0.5X. Al finalizar la corrida electroforética los productos de PCR fueron visualizados mediante el sistema de captura de imágenes GelCapture que está integrado al equipo Bio-Imaging Systems MiniBis Pro.
RESULTADOS
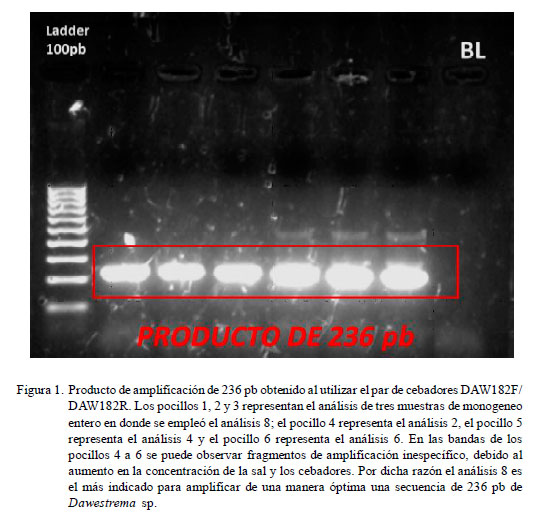
Las corridas electroforéticas de los productos de PCR de los cebadores DAW183F/ DAW183R y DAW184F/DAW184R no amplificaron productos. Asimismo, las corridas de los productos de PCR de los cebadores DAW182F/DAW182R con temperatura de alineamiento de 60 °C tampoco amplificaron productos, mientras que con la temperatura de alineamiento a 62 °C se logra amplificar un producto de PCR de 236 pb (Figura 1).
Las seis bandas del gel de la Figura 1 se guardaron a -20 °C para su posterior purificación con el QIAquick Gel Extraction Kit (Qiagen), siguiendo las especificaciones del fabricante y serán remitidas para su secuenciamiento.
Todos los resultados de la PCR positivos de las muestras de raspado de las agallas fueron confirmados por la presencia del parásito en la necropsia.
DISCUSIÓN
El hallazgo del parásito monogeneo Dawestrema cycloancistrium en todos los especímenes juveniles muestreados de paiche reiteran los resultados hallados por este grupo de investigación (Serrano-Martínez et al., 2015) y que concuerdan con estudios previos (Cotrina y Rebaza, 2008; Dos Santos et al., 2008; Araújo et al., 2009).
La presencia de monogeneos ha sido un problema recurrente para las granjas de crianza de peces de cultivo como la tilapia (Sandoval-Gío et al., 2008), trucha (RubioGodoy et al., 2003; Rubio-Godoy y Tinsley, 2004) y paiche (Cotrina y Rebaza, 2008; Dos Santos et al., 2008; Araújo et al., 2009). El control de Dawestrema sp se basa en sustancias químicas que son ineficientes y los métodos actuales para la detección de este parásito requieren el sacrificio del pez. En este estudio, se ha estandarizado una prueba de PCR que permite detectar ADN aislado directamente de D. cycloancistrum de las branquias del paiche. La PCR implementada en el presente documento mostró que el par DAW182F/DAW182R amplificó una región de 236 pb.
Una de las principales ventajas de esta prueba es que permite detectar bajos niveles de infestación de parásitos; es decir, una infestación temprana, sin tener que sacrificar al pez. Asimismo, no se presentaron falsos positivos ni falsos negativos con la prueba del PCR. El utilizar un gen altamente conservado, gen ribosomal 18S en la PCR, tiene la ventaja que podría ser utilizado en paiches, tanto criado en condiciones naturales como de cultivo, donde las dos especies de Dawestrema están siempre presentes y son morfológicamente muy parecidas.
Dada la alta tasa de infestación por D. cycloancistrium, el desarrollo de la técnica molecular para su diagnóstico con uso de una secuencia específica del gen 18S ADNr de Dawestrema sp, obtenido en el presente estudio por primera vez en el país, será de gran importancia para el diagnóstico por PCR de este parásito.
CONCLUSIONES
Se logró desarrollar una técnica de diagnóstico molecular (PCR) frente a D. cycloancistrium para su posterior aplicación como medida de control en el manejo de la biodiversidad del paiche.
Agradecimientos
Agradecemos al Sr. Wenseslao Solsol, paichicultor de la provincia de Maynas, del departamento de Loreto, Perú, por su apoyo en el presente estudio, y a la Dirección Regional de la Producción de Loreto, por su apoyo en los trabajos de campo. A la doctora Manuela Verástegui, de los Laboratorios de Investigación y Desarrollo (LID), Facultad de Ciencias de la Universidad Peruana Cayetano Heredia por su apoyo con el equipo Nanodrop 2000.
El trabajo fue realizado con el financiamiento del Consejo Nacional de Ciencia y Tecnología – CONCYTEC, mediante contrato N° ° 314-2011-CONCYTEC-OAJ.
LITERATURA CITADA
1. Alcántara B, Wust W, Tello M, Rebaza M, Del Castilo D. 2006. Paiche el gigante del Amazonas. Loreto: Instituto de Investigaciones de la Amazonía Peruana. 69 p. [ Links ]
2. Araújo CSO, Gomes AL, TavaresDías M, Andrade SMS, Belem-Costa A, Borges JT, et al. 2009. Parasitic infections in pirarucu fry, Arapaima gigas Schinz, 1822 (Arapaimatidae) kept in a semi-intensive fish farm in Central Amazon, Brazil. Veterinarski Archiv 79: 499-507. [ Links ]
3. Buchmann K, Uldal A, Mellergaard S. 1994. Mortality of captive Arapaima gigas (Osteoglossidae) heavily infected with the gill monogenean Dawestrema cycloancistrium. Bull Eur Ass Fish Pathol 14(5): 171-173. [ Links ]
4. Collins C, Kerr R, McIntosh R, Snow M. 2010. Development of a real-time PCR assay for the identification of Gyrodactylus parasites infecting salmonids in northern Europe. Dis Aquat Organ 90: 135-142. doi: 10.3354/dao02201 [ Links ]
5. Cotrina M, Rebaza C. 2008. Caracterización de la fauna parasitaria en juveniles de paiche «Arapaima gigas» criados en cautiverio. En: Memoria Institucional 2008. Programa de Investigación en Ecosistemas Acuáticos del IIAP. Perú [ Links ].
6. Dos Santos S, Ceccarelli P, Luque J. 2008. Helmintos parasitos do pirarucu, Arapaima gigas (Schinz, 1822) (Osteoglossiformes: Arapaimidae), no Rio Araguaia, Estado de Mato Grosso, Brasil. Rev Bras Parasitol Vet 17: 171173. doi: 10.1590/S1984-29612008-000300012 [ Links ]
7. Freeman M, Shinn A. 2011. Myxosporean hyperparasites of gill monogeneans are basal to the Multivalvulida. Parasit Vectors 4: 220. doi: 10.1186/1756-3305-4-220 [ Links ]
8. Harris D, Cable J, Tinsley R, Lazarus C. 1999. Combined ribosomal DNA and morphological analysis of individual Gyrodactylid monogeneans. J Parasitol 85: 188-191. [ Links ]
9. Kritsky DC, Boeger WA, Thatcher VE. 1985. Neotropical monogenea. Parasites of the pirarucu, Arapaima gigas (Cuvier), with descriptions of two new species and redescriptions of Dawestrema cycloancistrium Prince and Nowlin, 1967 (Dactylogyridae: Ancyrocephaline). Proc Biol Soc Wash 98: 321-331. [ Links ]
10. Li A, Wu X, Ding X, Lin R, Xie M, Lun Z, Zhu X. 2005. PCR-SSCP as a molecular tool for the identification of Benedeniinae (Monogenea: Capsalidae) from marine fish. Mol Cell Probes 19: 35-39. [ Links ]
11. Mathews P, Chu F. 2007. Parásitos asociados a alevinos de paiche Arapaima gigas en el C.I. de Quistococha. En: Memoria Institucional 2007. Programa de Investigación en Ecosistemas Acuáticos del IIAP. Perú [ Links ].
12. Rubio-Godoy M, Sigh J, Buchmann K, Tinsley R. 2003. Antibodies against Discocotyle sagittata (Monogenea) in farmed trout. Dis Aquat Org 56: 181-184. doi: 10.3354/dao056181 [ Links ]
13. Rubio-Godoy M, Tinsley R. 2004. Comparative susceptibility of brown trout and rainbow trout to Discocotyle sagittata (Monogenea). J Parasitol 90: 900-901. [ Links ]
14. Sandoval-Gío JJ, Rodríguez-Canul R, Vidal-Martínez VM. 2008. Humoral antibody response of the tilapia Oreochromis niloticus against Cichlidogyrus spp. J Parasitol 94: 404-409. doi: 10.1645/GE-1382.1 [ Links ]
15. Serrano-Martínez E, Tantaleán M, Leguía G, Quispe M, Casas G. 2015. Parásitos em Arapaima gigas de la Amazonía Peruana según grupo etario. Rev Inv Vet Peru 26: 303-309. doi: 10.15381/rivep.v26i2.11014 [ Links ]
16. Smith S, Levy M, Noga E. 1992. Development of an enzyme-linked inmunosorbent assay (ELISA) for the detection of antibody to the parasitic dinoflagellate Amyloodinium ocellatum in Oreochromis aureus. Vet Parasitol 42: 145-155. [ Links ]
17. Verma C, Chaudhary A, Singh H. 2012. PCR-based molecular characterization, phylogenetic analysis and secondary structure of the 28S rDNA of Thaparocleidus wallagonius (Monogenea: Dactylogyridae) – the most primitive species of this genus from India. Bioinformation 8: 816-819. doi: 10.6026/97320630008816 [ Links ]
Recibido: 2 de mayo de 2015
Aceptado para publicación: 29 de enero de 2016