Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú vol.28 no.2 Lima abr./jun. 2017
http://dx.doi.org/10.15381/rivep.v28i2.13074
http://dx.doi.org/10.15381/rivep.v28i2.13074
ARTÍCULOS PRIMARIOS
Identificación de Salmonella Enteritidis y Salmonella Typhimurium en cuyes mediante la técnica de PCR múltiple
Identification of Salmonella Enteritidis and Salmonella Typhimurium in Guinea pigs by the multiplex PCR
Geraldine Marcelo M.1, Raúl Rosadio A.1, Ana Chero O.1, Gerardo Díaz O.1, Aldo Ciprian C.2, Lenin Maturrano H.1,3
1 Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú
2 Laboratorio de Patología Clínica, Histología y Embriología, Universidad Nacional de San Cristóbal de Huamanga, Ayacucho, Perú
3 E-mail: lenin.maturrano@gmail.com
RESUMEN
El objetivo del presente estudio fue identificar mediante PCR múltiple la posible existencia de los serovares Salmonella Typhimurium y Enteritidis en 25 cepas de Salmonella spp previamente aisladas de cuyes e identificadas por sus características metabólicas. Mediante el análisis molecular se identificaron todas las cepas como Salmonella Typhimurium, evidenciando la amplificación de los cebadores específicos para los genes invAy fliC pertenecientes al género Salmonella y Salmonella Typhimurium, respectivamente. El presente estudio permitió establecer una metodología rápida para la identificación molecular de Salmonella Typhimurium y Enteritidis aislados de cuyes.
Palabras clave: cuyes, Salmonella Typhimurium, Salmonella Enteritidis, PCR múltiple
ABSTRACT
The aim of this study was to identify by mutiplex PCR-based assays the possible presence of serovars Salmonella Typhimurium and Enteritidis in 25 strains of Samonella spp that were previously isolated from guinea pigs and identified by their metabolic characteristics. The molecular analysis identified all 25 strains as Salmonella Typhimurium, evidencing the primer amplification of genes invA and fliC of Salmonella spp and Salmonella Typhimurium, respectively. This study established a rapid methodology for molecular identification of Salmonella Typhimurium and Enteritidis isolated from guinea pigs.
Key words: guinea pigs, Salmonella Typhimurium, Salmonella Enteritidis, multiplex PCR
INTRODUCCIÓN
La salmonelosis es la principal enfermedad infecciosa que afecta la explotación de cuyes, provocando graves pérdidas en la producción debido a los altos índices de mortalidad (Morales et al., 2007; Matsuura, 2010). El género Salmonella presenta más de 2500 serovares, siendo Salmonella Typhimurium y Salmonella Enteritidis los notificados con mayor frecuencia a nivel mundial en animales de sangre caliente (García, 2011; Grimont y Weill, 2007; WHO, 2013).
S. Typhimurium ha sido el serovar de mayor frecuencia en cuyes criados para consumo en el Perú (Garmendia et al., 2000; Matsuura et al, 2010); sin embargo, existen otros serovares como S. Enteritidis que fueron aislados de cuyes de laboratorio o criados como mascotas (Richardson 2000; Garmendia et al., 2000; Parra et al., 2002).
La salmonelosis se presenta en cuyes de cualquier edad, mayormente en las etapas de lactación y destete (Matsuura et al., 2010). La enfermedad se presenta en forma crónica o aguda, siendo esta última la más letal debido a que no se suelen observar signos clínicos aparentes que permitan realizar algún tratamiento (Matsuura et al., 2010). El diagnóstico en cuyes se realiza asociando las manifestaciones clínicas, hallazgos en la necropsia y el aislamiento bacteriano. Los órganos de elección para recuperar la bacteria en animales enfermos son principalmente el hígado y el bazo, pero también se puede aislar de pulmón, ganglio mesentérico, intestino, útero, vesícula biliar y glándula mamaria (Bustamante, 1993; Matsuura, 2010).
Entre los métodos para el diagnóstico de Salmonella spp, el cultivo microbiológico es la prueba más comúnmente empleada para el aislamiento de la bacteria a partir de tejidos y heces (Pachón, 2009), para luego observar las características metabólicas y antigénicas mediante pruebas bioquímicas y serológicas. Actualmente se prefiere el uso de técnicas de diagnóstico molecular debido a su gran rapidez y eficacia, siendo una de ellas la reacción en cadena de la polimerasa (PCR) (Myint et al., 2006; Grimont y Weill, 2007). La PCR múltiple, variante de la PCR convencional, es empleada para la detección y amplificación simultánea de distintas secuencias de ADN (en una única reacción). Asimismo, se han desarrollado diversos protocolos de PCR para la detección de Salmonella spp utilizando cebadores específicos para la detección de genes blanco del género Salmonella y los serovares S. Enteritidis y Typhimurium en productos de origen animal destinados al consumo humano (Soumet et al., 1999; Jamshidi et al., 2010).
Uno de los genes utilizados para la identificación del género Salmonella es el invA, que contiene secuencias únicas del género y ha sido probado como un blanco apropiado de PCR, con una potencial aplicación diagnóstica (Rahn et al., 1992). Así mismo, la identificación de la bacteria hasta el nivel de serovar es aún limitada, debido a la gran cantidad de serovares conocidos (Grimont y Weil, 2007). Los cebadores diseñados que permiten identificar a los serovares Typhimurium y Enteritidis son los más utilizados por el problema que causan en la salud humana y animal (CFSPH, 2005).
El gen fliC de la flagelina codifica el mayor componente del flagelo de S. Typhimurium, utilizándose para la identificación de este serovar (Aldridge et al., 2006). Por otro lado, el serovar Enteritidis alberga en su genoma un plásmido único de virulencia de 60 kb (Chu et al., 1999) que posee el gen Prot6E, el cual probablemente codifique una única secuencia de superficie fimbrial específica de S. Enteritidis (Clavijo et al., 2006). De esta manera, el objetivo del presente estudio fue identificar mediante PCR múltiple la posible existencia de los serovares S. Enteritidis y Typhimurium en 25 cepas de Salmonella spp previamente aisladas de cuyes e identificadas por sus características metabólicas.
MATERIALES Y MÉTODOS
Lugar de Estudio
Las muestras fueron obtenidas a partir de casos sospechosos de salmonelosis ocurridas en en un trabajo de investigación previo en explotaciones comerciales de cuyes en el departamento de Lima. El procesamiento y análisis de las muestras fueron realizados en la Sección de Biología y Genética Molecular del Laboratorio de Microbiología y Parasitología Veterinaria de la Facultad de Medicina Veterinaria, Universidad Nacional Mayor de San Marcos, Lima, Perú.
Material Experimental
Cepas en estudio
Se evaluaron 25 cepas previamente caracterizadas, morfológica y metabólicamente, como Salmonella spp, provenientes de animales con signos clínicos y lesiones anatomopatológicas de salmonelosis. Estas cepas fueron aisladas de hígado, vesícula biliar y médula ósea de 25 cuyes, seleccionando indistintamente solo una muestra por animal. Las muestras se encontraban conservadas en caldo cerebro corazón (BHI, por sus siglas en inglés) con glicerol al 20% en tubos de 2 ml a una temperatura de -80 °C.
Cepas de referencia
Se emplearon las cepas de referencia S. TyphimuriumATCC 14028 y S. Enteritidis ATCC 13076. Además, se utilizaron dos cepas control negativas de Escherichia coli y Proteus spp aisladas en este laboratorio. Las muestras se encontraban conservadas en BHI con glicerol al 20% en tubos de 2 ml a una temperatura de -80 °C.
Reactivación de Cepas
Las 25 cepas de cuyes y las cepas de referencia control negativo y control positivo fueron reactivadas empleando el protocolo descrito por Sánchez y Corrales (2005), con algunas modificaciones:
- Las cepas en glicerol se llevaron a una temperatura de -20 °C y luego de 4°C.
- Se sembraron por agotamiento en pla cas de agar sangre con la ayuda de un asa de siembra y se incubaron a 37 °C por 18 h.
- En paralelo al paso anterior, se sembra ron en placas de agar XLD (agar xilosa, lisina, desoxicolato), para observar el crecimiento típico de las colonias de Salmonella (colonias circulares transpa rentes con un centro negro por la pro ducción de sulfuro de hidrógeno).
- Luego, cada cepa fue inoculada en 1 ml de BHI e incubada a 37 °C por 24 h.
Análisis de Muestras
Para la extracción de ADN se utilizó el Thermo Scientific GeneJET Genomic DNA Purification (ThermoFisher Scientific), siguiendo las instrucciones del fabricante. El ADN extraído se colocó en tubos microcentrífuga de 1.5 ml y se almacenó a -20 °C.
Para la identificación de las cepas obtenidas de cuyes con salmonelosis, se realizó una PCR múltiple basada en el protocolo de Jamshidi et al., (2010), con algunas modificaciones, empleando tres pares de cebadores que codifican secuencias específicas de los genes invA, fliCy Prot6E, que identifican al género Salmonella, S. Typhimurium y S. Enteritidis, respectivamente (Cuadro 1 ).
La PCR múltiple fue realizada en un volumen final de 20 µl consistiendo en 2.5 µl de 10X PCR buffer (500 mM KCl, 200 mM Tris–HCl), 1 µl de desoxinucleotido trifosfato (0.5 mM), 0.5 µl MgCl2 (2.5 mM), 0.4 µl por cada cebador, 0.3 µl de Taq DNApolimerasa y 2 µl de ADN. Las condiciones para la amplificación de los genes fueron de una incubación inicial a 95 °C por 5 min, seguida por 35 ciclos con una desnaturalización a 95 °C por 45 s, hibridación a 58 °C por 45 s, una extensión a 72 °C por 45 s, y una extensión final a 72 °C por 7 min.
Para la verificación de la prueba se utilizaron cepas control de S. Enteritidis (ATCC 13076), S. Typhimurium (ATCC 14028) y como controles negativos a Proteus spp y Escherichia coli. Los productos de la PCR múltiple fueron separados mediante electroforesis en gel al 2% con buffer TBE 0.5X durante 2 h. El tamaño de las distintas bandas se determinó mediante marcadores de peso molecular de 100 pb (DNA ladder, Gene RulerTR, Fermentas).
RESULTADOS
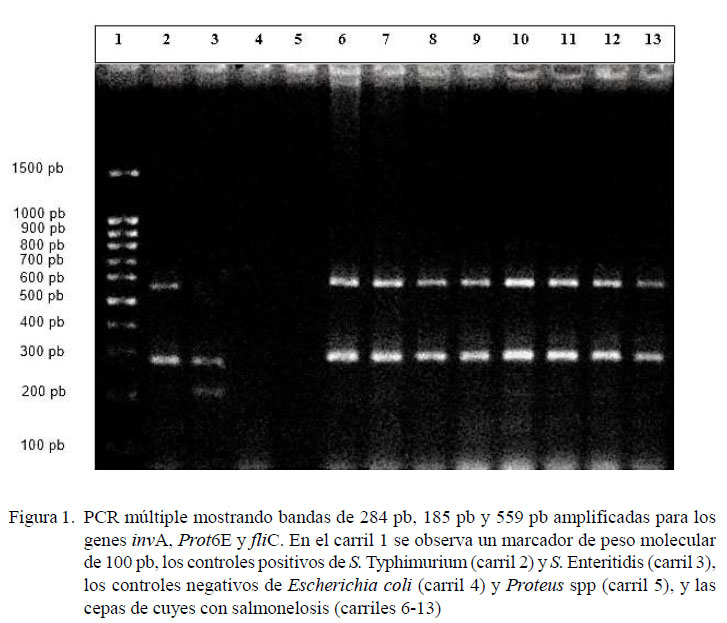
El ADN de las 25 cepas de Salmonella spp fue identificado mediante la técnica de PCR múltiple, evidenciándose en todas las muestras dos amplicones de 559 pb y 284 pb correspondientes a los genes fliCe invA, respectivamente. La presencia y visualización de estos amplicones indican que las cepas pertenecen al género S. serovar Typhimurium (Figura 1 ).
DISCUSIÓN
La técnica de PCR múltiple evidenció amplicones de 284 pb (gen invA) y 185 pb (gen fliC) en las 25 cepas de cuyes, coincidentes con el control positivo de S. Typhimurium. Estos datos corroboran los resultados de las pruebas bioquímicas realizadas previamente. Así mismo, debido a la obtención de amplicones de 559 pb se identificó como S. Typhimurium al total de las muestras evaluadas, diagnóstico que generalmente requeriría de una identificación serológica completa para la determinación de serotipo.
Esos resultados confirman estudios anteriores que indican que S. Typhimurium se encuentra en frecuencias que superan el 95% en relación a otros serovares hallados en cuyes (Bustamante, 1993; Garmendia et al., 2000), ya que S. Enteritidis también se encuentra implicado en cuadros de salmonelosis en cuyes, aunque en una mucho menor frecuencia (Garmendia et al., 2000; Richardson, 2000; Parra et al., 2002).
Los cebadores que amplifican secuencias específicas de genes de virulencia de invA, fliCy Prot6E del género Salmonella y los serovares S. Typhimurium y S. Enteritidis, respectivamente, han sido empleados con éxito en muestras de cuyes y otras especies (Soumet et al., 1999; Atyabi et al., 2012; Marcelo et al., 2015; Moosavy et al., 2015).
CONCLUSIONES
-
Se logró identificar S. Typhimurium en todas las cepas aisladas de cuyes con signos de salmonelosis mediante la técnica de PCR múltiple debido la amplificación de los genes invA (género Salmonella) y fliC (serovar S. Typhimurium) de 284 pb y 559 pb, respectivamente.
-
Se implementó un protocolo para la identificación específica de Salmonella spp y los serovares Typhimurium y Enteritidis.
Agradecimientos
Los autores expresan su agradecimiento al Programa Nacional de Innovación para la Competitividad y Productividad – Innóvate Perú, fuente financiadora del Proyecto «Desarrollo de una vacuna para el control y prevención de la salmonelosis en la producción de cuyes», Contrato N.° 362PNICP-PIAP-2014.
LITERATURA CITADA
1. Aldridge P, Gnerer J, Karlinsey JE, Hughes KT. 2006. Transcriptional and translational control of the Salmonella fliC gene. J Bacteriol 188: 4487-4496. [ Links ]
2. Atyabi N, Zahraei T, Ghazisaeedi F, Ashrafi I. 2012. The molecular investigation of widespread Salmonella serovars, S. Typhimurium and S. Enteritidis, involved in salmonellosis of cattle and sheep in farms around Tehran, Iran. Iranian J Vet Res 13: 126-133. [ Links ]
3. Bustamante J. 1993. Producción de cuyes. Lima: Universidad Nacional Mayor de San Marcos. 259 p. [ Links ]
4. [CFSPH] Center for Food Security and Public Health. 2005. Animal disease factsheets. Salmonellosis. Ames, IA, USA: Iowa State University. 8 p. [Internet]. Available in: http://www. cfsph.iastate.edu/Factsheets/pdfs/ nontyphoidal_salmonellosis.pdf [ Links ]
5. Chu C, Hong S, Tsai C, Lin W, Liu T, Ou J. 1999. Comparative physical and genetic maps of the virulence plasmids of Salmonella enterica serovars Typhimurium, Enteritidis, Choleraesuis, and Dublin. Infect Immun 67: 2611-2614. [ Links ]
6. Clavijo R, Loui C, Andersen G, Riley L, Lu S. 2006. Identification of genes associated with survival of Salmonella enterica serovar Enteritidis in chicken egg albumen. Appl Environ Microbiol 72: 1055-1064. doi: 10.1128/AEM.72.2. 1055-1064.2006 [ Links ]
7. Garmendia M, Selgrad S, Alezones F. 2000. Salmonelosis en animales de laboratorio. FONAIAP Divulga 68: 32-33 [Internet]. Disponible en: http://sian.inia. gob.ve/repositorio/revistas_tec/Fonaiap Divulga/fd68/texto/mgarmendia.htm [ Links ]
8. García F. 2011. Salmonelosis porcina en España: prevalencia, factores de riesgo y resistencia antimicrobiana. Tesis Doctoral. España: Univ de León. 197 p. [ Links ]
9. Grimont P, Weill F. 2007. Antigenic formulae of the Salmonella serovars. 9th ed. Paris, France: World Health Organization Collaborating Centre for Reference and Research on Salmonella. Institut Pasteur. 166 p. [ Links ]
10. Jamshidi A, Kalidari G, Hedayati M. 2010. Isolation and identification of Salmonella Enteritidis and Salmonella Typhimurium from the eggs of retail stores in Mashhad, Iran using conventional culture method and multiplex PCR assay. J Food Safety 30: 558-568. doi: 10.1111/j.1745-4565.2010.-00225.x [ Links ]
11. Marcelo G, Chero A, Díaz G, Rímac R, Hurtado R, Ciprian A, Rosadio R, Maturrano L. 2015. Evaluación de un protocolo de PCR múltiple para la identificación de Salmonella Typhimurium en cobayos. En: XXXVIII Reunión Científica Anual de la Asociación Peruana de Producción Animal. Ayacucho, Perú: APPA. [ Links ]
12. Matsuura A, Morales S, Calle S, Ara M. 2010. Susceptibilidad a antibacterianos in vitro de Salmonella enterica aislada de cuyes de crianza familiar-comercial en la provincia de Carhuaz, Ancash. Rev Inv Vet Perú 21: 93-99. doi: 10.15381/rivep.v21i1.355 [ Links ]
13. Moosavy M, Esmaelli S, Amiri F, Mostafavi E, Salehi Z. 2015. Detection of Salmonella spp in commercial eggs in Iran. Iran J Microbiol 7: 50-54. [ Links ]
14. Morales S, Mattos J, Calle S. 2007. Efecto de la muña (Satureja parvifolia) en la dinámica de la infección por Salmonella enterica en cobayos. En: XXX Reunión Científica Anual de la Asociación Peruana de Producción Animal. Cusco, Perú: APPA. [ Links ]
15. Morales S. 2012. Patógenos oportunistas por transmisión fecal-oral en cuyes reproductores introducidos al distrito de San Marcos. Científica 9(1): 33-38. [Internet]. Disponible en: https:// issuu.com/bibliotecacientifica/docs/ cientifica-v9n1/33 [ Links ]
16. Myint MS, Johnson YJ, Tablante NL, Heckert RA. 2006. The effect of preenrichment protocol on the sensitivity and specificity of PCR for detection of naturally contaminated Salmonella in raw poultry compared to conventional culture. Food Microbiol 23: 599-604. [ Links ]
17. Pachón DA. 2009. Aislamiento, identificación y serotipificación de enterobacterias del género Salmonella en una población de Crocodylus intermedius y Testudines mantenidos en cautiverio en la Estación de Biología Tropical Roberto Franco EBTRF de la Facultad de Ciencias, Universidad Nacional de Colombia en Villavicencio-Meta. Tesis de Biología. Bogotá: Pontificia Universidad Javeriana. 115 p. [ Links ]
18. Parra M, Durango J, Mattar S. 2002. Microbiología, patogénesis, epidemiología, clínica y diagnóstico de las infecciones producidas por salmonella. MVZCórdoba 7: 187-200. [ Links ]
19. Rahn K, DeGrandis S, Clarke R, Mcewen S, Galán JE, Ginocchio C, Curtiss III R, Gyles CL. 1992. Amplification of an invA gene sequence of Salmonella typhimurium by polymerase chain reaction as a specific method of detection of Salmonella. Mol Cell Probes 6: 271-279. doi: 10.1016/ 0890-8508(92)90002-F [ Links ]
20. Richardson VC. 2000. Diseases of domestic guinea pigs. 2nd ed. Oxford: Blackwell Science. 145 p. [ Links ]
21. Sánchez L, Corrales R. 2005. Evaluación de la congelación para conservación de especies autóctonas bacterianas. NOVA 3(4): 21-29. [ Links ]
22. Soumet C, Ermel G, Rose V, Rose N, Drouin P, Salvat G, Collin P. 1999. Identification by a multiplex PCR-based assay of Salmonella Typhimurium and Salmonella Enteritidis strains from environmental swabs poultry houses. Lett App Microbiol 29: 1-6. [ Links ]
23. [WHO] World Health Organization. 2013. Salmonella (non-typhoidal). Fact sheet N.° 139. [Internet]. Disponible en: ht tp:/ /www.who.i nt /mediacentr e/ factsheets/fs139/en/ [ Links ]
Fuente financiera: Innovate Perú, Proyecto «Desarrollo de una vacuna para el control y prevención de la salmonelosis en la producción de cuyes». Contrato N°362-PNICP-PIAP-2014
Recibido: 27 de abril de 2016
Aceptado para publicación: 31 de enero de 2017