Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
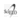 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú vol.28 no.3 Lima jul./set. 2017
http://dx.doi.org/10.15381/rivep.v28i3.13353
ARTÍCULOS PRIMARIOS
Diversidad Genética del Caballo Criollo (Equus caballus) mediante Genes Asociados al Pelaje en Valencia, Colombia
Genetic Diversity of the Criollo Horse (Equus caballus) by Genes Associated to the Skin Coat in Valencia, Colombia
Wilson Yepes1, Enrique Pardo Pérez1,2, Luis Alfonso Causil Vargas1
1 Departamento de Biología, Facultad de Ciencias Básicas, Universidad de Córdoba, Colombia
2 E-mail: epardop@correo.unicordoba.edu.co
RESUMEN
El objetivo de este trabajo fue estudiar la variabilidad genética de caballos criollos (Equus caballus) mediante genes asociados al pelaje. Se realizaron muestreos entre enero y mayo de 2016 en cinco poblaciones de la ciudad de Valencia, Colombia. Se realizó la caracterización fenotípica de los animales adultos presentes en las fincas, atendiendo a los marcadores autosómicos de codificación morfológica Extension, Agouti, Cream, Gris, White, Tobiano, Overo y Roan. Los resultados mostraron ausencia de los marcadores White y Overo, mientras que los marcadores Extension y Agouti fueron los de mayores frecuencias, posiblemente favorecidas por selección artificial, debido a temperamento y astucia, respectivamente. Las poblaciones se encontraron en equilibrio de HardyWeinberg, exhibieron exceso de homocigotos y altos valores de flujo genético.
Palabras clave: caballo; frecuencia alélica; heterocigosidad; equilibrio Hardy-Weinberg; hábitat
ABSTRACT
The objective of this study was to investigate the genetic variability of criollo horses (Equus caballus) by genes associated with the coat. Fields visits were done between January and May 2016 in five populations of the municipality of Valencia, Colombia. The phenotypic characterization of the adult animals present in the farms was performed in relation to the autosomal markers of morphological codification Extension, Agouti, Cream, Gris, White, Tobiano, Overo and Roan. The results showed absence of the markers White and Overo, whereas the markers Extension and Agouti had the largest frequencies, possibly favored by artificial selection due to temperament and cunning respectively. Populations were found in equilibrium Hardy-Weinberg and showed excess of homozygotes and high levels of gene flow.
Key words: horse; allelic frequency; heterozygosity; Hardy-Weinberg equilibrium; habitat
INTRODUCCIÓN
Poblaciones actuales de caballo criollo americano (Equus caballus) constituyen las poblaciones remanentes introducidas al Nuevo Mundo por los conquistadores europeos, donde se asume que la fuente original fue la Península Hispánica (Jansen et al., 2002). Es por esta razón que sería de esperar que los caballos criollos estuvieran genéticamente relacionados con las razas andaluzas, españolas de origen celta, berberiscas y árabes (Giovambattista et al., 2010).
El interés sobre el patrón de herencia del color del manto en caballos fue uno de los primeros rasgos que demuestran la validez de las leyes de Mendel en los mamíferos. La variación del color depende principalmente de la melanina, la cual se produce en forma de gránulos de pigmento en los melanocitos, que son células que se encuentran en los folículos pilosos en piel, diafragma, y algunos tejidos internos (Finn et al., 2016). La herencia del color en caballos ha sido estudiada principalmente desde una perspectiva cualitativa y son controlados por unos pocos genes, donde su expresión es el resultado de muchas interacciones a lo largo de la historia (Toth et al., 2006).
Los distintos patrones del pelaje en general, expresados como marcadores fenotípicos, han sido de gran importancia para el estudio de poblaciones, debido a que son fácilmente reconocibles; razón por la cual es relativamente sencillo seguir el patrón de herencia de generación en generación (Thiruvenkadan et al., 2008). Por lo tanto, estos marcadores se han convertido en modelo para el estudio de las acciones e interacciones genéticas, no solo para explicar la estructura poblacional, sino que también podrían explicar algunas relaciones etológicas como fenotipo-comportamiento (Finn et al., 2016).
Se requiere evaluar la variabilidad genética y el flujo de genes dentro de la población antes de la puesta en práctica de programas de selección, a fin de establecer una adecuada gestión del patrimonio genético. En la cría doméstica de equinos, algunos parámetros demográficos simples, que dependen en gran medida de la política de gestión y el apareamiento, tienen un gran impacto de la variabilidad genética. Además, el estudio de la estructura de la población y la demografía puede poner de relieve las circunstancias importantes que afectan la historia genética de la población (Valera et al., 2005).
Un estudio reciente reveló la estructura genética de poblaciones de caballos (Equus caballus) en el departamento de Córdoba (Correa et al., 2015), donde se analizaron 341 individuos en las siete poblaciones estudiadas, siendo el marcador Extensión el de mayor frecuencia, mientras los genes Overo y Tobiano presentaron los menores valores. Se registraron cifras poco significativas de variabilidad genética a nivel global y poblacional; así mismo, se obtuvo una escasa diferenciación genética entre las poblaciones, acompañado de un elevado flujo génico. Estos resultados corroboran la importancia de este tipo de estudios, razón por el cual el objetivo de esta investigación fue evaluar la variabilidad genética del caballo doméstico (Equus caballus), usando genes asociados al pelaje en Valencia, Colombia.
MATERIALES Y MÉTODOS
Sitio de Estudio
El estudio se realizó en el municipio de Valencia, ubicado al suroccidente del departamento de Córdoba, Colombia. Presenta una temperatura promedio anual de 30 °C, altura promedio de 55 msnm, y una extensión de 914 km2. Se muestrearon cinco corregimientos: Valencia, Villa Nueva, Jaraguay, El Reposo y San Rafael del Pirú.
Obtención de Datos
El muestreo se realizó entre enero y mayo de 2016 mediante excursiones rurales. Se muestrearon animales adultos presentes en las fincas de cada población, donde se caracterizó fenotípicamente cada animal atendiendo a los marcadores autosómicos de codificación morfológica: Extension, Agouti, Cream, Gris, White, Tobiano, Overo y Roan, siguiendo la metodologÃa propuesta por Bartolomé et al. (2008).
Diseño Estadístico
Se estimaron las frecuencias alélicas por marcador a nivel poblacional y global, así como las medidas de diversidad genética de Nei (1972) como diversidad genética total (HT), diversidad dentro de las poblaciones (HS), diversidad entre poblaciones (DST), coeficiente de diversidad genética (GST), flujo génico (Nm), equilibrio Hardy-Weinberg y la distancia genética entre las poblaciones, utilizando el programa PopGene 1.31 (Yeh et al., 1999). La estructura genética de las poblaciones, atendiendo los índices de fijáción propuestos por Wright (FIS, FIT, FST) se calculó mediante el programa FSTAT v. 2.9.3.2(Goudet, 2001).
El dendograma se obtuvo a partir de la matriz distancia genéticas mediante el programa MEGA7 (Kumar et al., 2016) y el análisis de componentes principales (PCA) se realizó utilizando el programa Past 3.11 (Hammer et al., 2001).
RESULTADOS
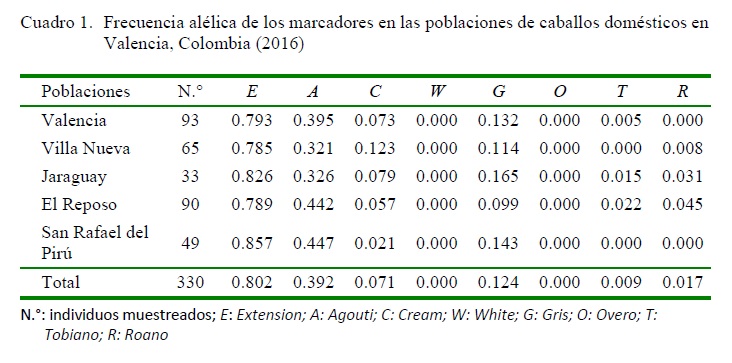
Se muestrearon 330 individuos distribuidos en cinco poblaciones: Valencia (n=93), Villa Nueva (n=65), Jaraguay (n=33), El Reposo (n=90) y San Rafael del Pirú (n=49). Solo 6 de los 8 marcadores utilizados en esta investigación estuvieron presentes en las poblaciones en estudio.
Las frecuencias alélicas para cada población (Cuadro 1) mostró que el marcador Extension (p=0.802) fue el más frecuente, seguido de Agouti (p=0.392), mientras que Cream evidenció los valores más bajos (p=0.071). Hubo ausencia de los marcadores White (W) y Overo (O), mientras que Tobiano (T) y Roano (R) solo se hallaron en 3 de las 5 poblaciones.
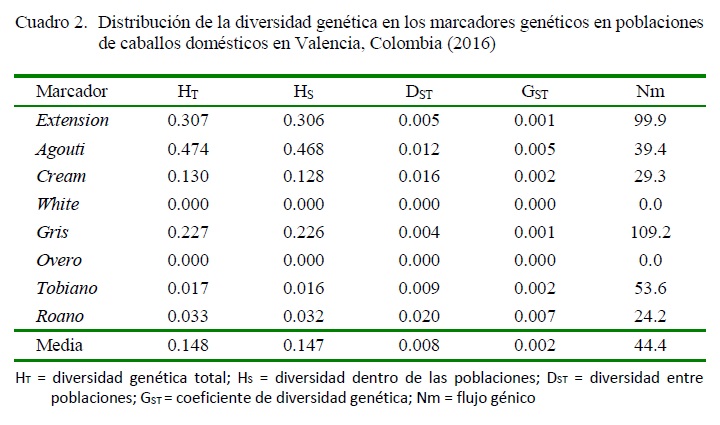
El índice de variabilidad genética a nivel global fue bajo (HT=0.148) (Cuadro 2). El marcador Agouti mostró el índice de diversidad más alta (HT=0.474), seguido de Extension (HT=0.2401), a diferencia de Tobiano que presentó la menor heterocigosidad. La diversidad dentro de las poblaciones (HS=0.147) fue mayor que entre las poblaciones (DST =0.008). El nivel de diferenciación genética entre las poblaciones de caballos domésticos criollos fue bajo(GST=0.002). El valor de flujo génico (Nm=44.4) evidenció que las poblaciones mantienen un alto grado de intercambio genético, dado que valores de flujo génico mayores a 1 sugieren que las poblaciones se comportan como una metapoblación.
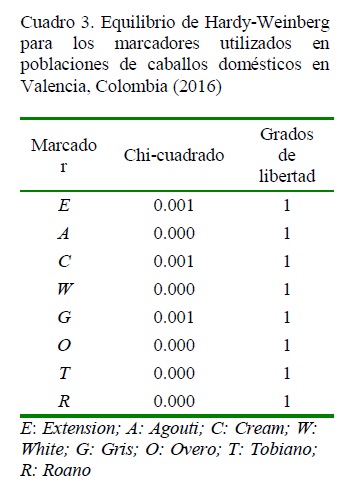
El Cuadro 3 muestra que todos los marcadores reportaron la existencia de equilibrio de Hardy-Weinberg.
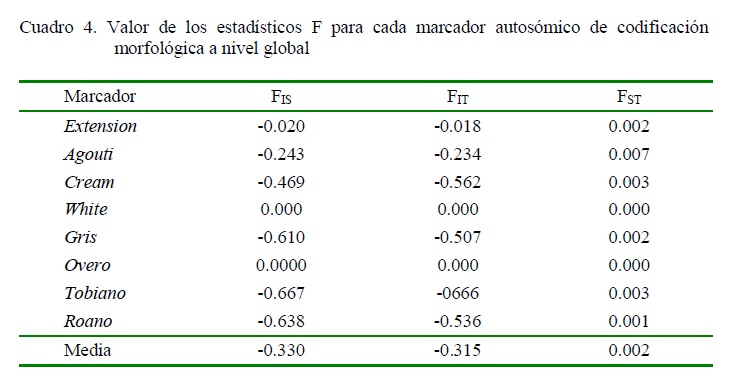
Los valores negativos obtenidos para el estadístico FIS en todos los marcadores revela un exceso de heterocigotos de los individuos con respecto a cada población (Cuadro 4) y, por lo tanto, hace suponer la ausencia de consanguinidad dentro de las poblaciones, con valores que oscilan entre -0.667 para el marcador Tobiano y -0.020, para el marcador Extension. El estadístico FIT, de igual forma, refleja un exceso de heterocigotos de los individuos con respecto a la población total. Por otro lado, el valor promedio de FST resultó ser bajo (0.002); lo que podría indicar que no existe diferenciación génica significativa entre las poblaciones.
La distancia genética obtenida entre las poblaciones fue baja, siendo Valencia y El Reposo las poblaciones más cercanas (Cuadro 5), mientras que Villa Nueva y San Rafael del Pirú fueron las de mayor diferencia génica, cifra poco significativa, pues no superó el 10%. Es así, que la población de Jaraguay presentó los mayores valores de distancia genética en comparación con el resto de las poblaciones.
El dendograma de la Figura 1 evidencia la similitud genética entre las poblaciones de Valencia y El reposo, a las cuales se asocia la población de San Rafael. Por otro lado, la población de Villa Nueva revela una cercanÃa con Jaraguay, pero con un valor de distancia poco significativo, que permite concluir que las poblaciones están muy relacionadas.
Mediante el Análisis de Componentes Principales (ACP) realizado a partir de las frecuencias alélicas, las componentes principales CP1 y CP2 explican el 87.5% de la variabilidad total entre las poblaciones, agrupando las poblaciones del norte y separándolas de las poblaciones del centro y sur de la ciudad, y la población de Valencia se ubica como punto medio entre las zona norte y sur (Figura 2).
DISCUSIÓN
La falta de caballos con alelos White y Overo puede obedecer a que su expresión se da en individuos heterocigotos, ya que condición homocigótica expresan letalidad, produciendo la muerte en estado embrionario (Thiruvenkadan et al., 2008).
Los genes responsables del color del pelaje controlan la cantidad y distribución de pigmentos de melanina en la piel y el pelo. En muchas especies de mamíferos, estos mismos genes expresan fenotipos que a menudo tienen efectos pleiotrópicos sobre el comportamiento; habiéndose encontrado recientemente esta relación entre fenotipos del pelaje y comportamiento en caballos (Finn et al., 2016). Esos resultados sugieren que los altos valores de las frecuencias encontrados en este estudio en los marcadores Extension y Agouti, se podrían explicar por esta relación, ya que se ha demostrado que los caballos castaños son más propensos a acercarse a objetos y animales en su ambiente, independientemente de su familiaridad. Esto sugiere que la selección para el fenotipo castaño podría verse favorecido por factores como la audacia y la forma de cómo interactúan con su entorno. Jacobs et al. (2016) encontraron una asociación entre el genotipo y el temperamento, que puede influir en la forma como los seres humanos interactúan con los caballos.
Los resultados de este estudio concuerdan con una investigación realizada en Ciénaga de Oro, Córdoba (Correa et al., 2015), donde el marcador Extension fue el más frecuente y sus resultados se fundamentan en el hecho de que las variantes alélicas responsables de las coloraciones oscuras se ven favorecidas en comparación con aquellas que expresan tonalidades claras; además, la capacidad de absorción de radiación calórica es más eficiente en animales de capa oscura que en aquellos de tonalidades claras, lo que permite suponer que los caballos oscuros, están mejor adaptados a las condiciones imperantes en el clima tropical.
La diversidad de las especies domésticas es considerada como un importante componente de la biodiversidad global. El pool génico de las razas criollas puede haber conservado genes o combinaciones de genes que se han perdido en aquellas razas donde los seres humanos tienden a seleccionar ciertos fenotipos. Las poblaciones criollas son consideradas como un valioso patrimonio cultural, histórico y genético de los diferentes países americanos; sin embargo, la mayoría de las razas criollas vienen pasando por una drástica reducción en el tamaño poblacional, estando muchas de ellas en riesgo de extinción (Giovambattista et al., 2010); hecho que podría explicar el bajo valor de diversidad genética total (HT) encontrada en la población.
En el presente estudio, el valor de diversidad genética total (HT = 0.1488) fue menor a lo reportado por Kelly et al. (2002) en el análisis serológico de siete grupos sanguíneos y nueve polimorfismos bioquímicos en 145 caballos criollos de Uruguay (0.295 a 0.443). Asimismo, presenta poca diferencia con el reporte de Correa et al. (2015), quienes registran para el municipio de Ciénaga de Oro, Córdoba, una heterocigosidad total de 0.1542 en el análisis de ocho genes morfológicos. Aunque la heterocigosidad esperada total es directamente proporcional al número de alelos por marcador, el bajo valor de HT en los caballos del municipio de Valencia puede ser causado por la poca variación entre las poblaciones analizadas (GST = 0.0085).
El índice de diferenciación génica (GST) indicaría que el 0.2% de la variación detectada se debe a diferencias entre poblaciones, mostrando la ausencia de subestructuración poblacional, debido a la homogeneización por el elevado valor de flujo genético encontrado (Nm = 44.4). Puesto que todas las poblaciones se comportan como una sola, es conveniente que se traten los datos como una metapoblación (Correa et al., 2015)
Se ha reportado que el rango de heterocigosidad presentado para razas equinas domésticas va de 0.295 a 0.443 (Kelly et al., 2002). En nuestro estudio, este valor fue bastante bajo, dado que todas las poblaciones se comportan como una sola. Al no existir una subestructura en la población, se podría percibir que la homogeneización por flujo genético parece muy factible y sería la causa principal de que la subestructura poblacional se deprecie, y se hagan los respectivos análisis como una metapoblación (Causil et al., 2016).
Los resultados de los índices de fijación FIS y FIT en este estudio, revelan el exceso de heterocigotos, lo cual estaría en concordancia con un elevado flujo genético, mostrando la existencia de un elevado intercambio de genes, lo cual evita eventos de endogamia dentro de las poblaciones (Pardo et al., 2015).
Los bajos valores de las distancias gen éticas obtenidas entre las poblaciones estudiadas podrían obedecer a la cercanía geográfica, como ha sido demostrado previamente (Cruz-Salazar et al., 2014). Por otro lado, el proceso de domesticación ha podido fijar algunos genes debido a preferencias antrópicas, por lo que caballos con un objetivo de cría concreto (trabajo doméstico) no estarían genéticamente muy diferenciados con respecto a los demás (Hyland, 2003). Este hecho podría explicar las relaciones existentes encontradas, evidenciados en el dendograma, puesto que todas las poblaciones se comportan como una sola, y de allí la cercanía genética entre todas.
CONCLUSIONES
- Los marcadores fenotípicos Extension y Agouti fueron los de mayor frecuencia en las poblaciones de caballos domésticos en Valencia, Colombia, posiblemente favorecidos por selección artificial, debido a temperamento y astucia, respectivamente.
- Los bajos valores de variabilidad genética y el alto grado de flujo génico sugieren que las subpoblaciones se encuentran muy relacionadas genéticamente y se comportan como una metapoblación.
- Todos los marcadores estudiados se encontraron en equilibrio de HardyWeinberg.
LITERATURA CITADA
1. Aranguren-Méndez J, Román R, Isea W, Villasmil Y, Jordana J. 2005. Los microsatélites (STR’s), marcadores moleculares de ADN por excelencia para programas de conservación: una revisión. Arch Latinoam Prod Anim.13: 1-6.
2. Bartolomé E, Azor P, Gómez M, Peña F. 2008. La determinación genética del color de la capa en el caballo: bases y aplicación al caballo de la raza Pottoka. Departamento de Genética, Universidad de Córdoba, España. [Internet]. Disponible en: http://pottoka.info/files/galeria/Genetica_color_capa_pottoka.pdf [ Links ]
3. Causil L, De La Barrera A, Causil O. 2016. Diversidad genética de palomas domésticas (Columba livia) en Lorica, Colombia, utilizando genes que codifican la coloración del plumaje. Rev Inv Vet Perú 27: 448-457. doi:10.15381rivep.v27i3.11476 [ Links ]
4. Cruz-Salazar B, Ruiz-Montoya L, Navarrete-Gutiérrez D, EspinozaMedinilla EE, Vázquez-Domínguez E, Vázquez LB. 2014. Diversidad genética y abundancia relativa de Didelphis marsupialis y Didelphis virginiana en Chiapas, México. Rev Mex Biodiv 85: 251-261. doi: 10.7550/rmb.36116 [ Links ]
5. Finn JL, Haase B, Willet CE, van Rooy D, Chew T, Wade CM, Hamilton N, Velie BD. 2016. The relationship between coat colour phenotype and equine behaviour: a pilot study. Appl Anim Behav Sci 174: 66-69. doi:10.1016/j.applanim.2015.11.004 [ Links ]
6. Giovambattista G, Rogberg-Muñoz A, Ripoli MV, Villegas-Castagnasso EE, Díaz S, Posik DM, et al. 2010. La genética molecular de bovinos y equinos criollos en los albores del siglo XXI. BAG J Basic Appl Gen 21(Supl 1): 1-14. [ Links ]
7. Goudet J. 2001. FSTAT, a program to estimate and test gene diversities and fixation indices. Version 2.9.3. 2001. [Internet]. Available in: http://www.unil.ch/izea/softwares/fstat.html [ Links ]
8. Hammer O, Harper DAT, Ryan PD. 2001. PAST: Paleontological statistics software package for education and data analysis. Palaeont Electr 4(1): 9. [ Links ]
9. Hyland A. 2003. The horse in the ancient world. Gloucestershire, England: Sutton Pub. 210 p. [ Links ]
10. Jacobs LN, Staiger EA, Albright JD, Brooks SA. 2016. The MC1R and ASIP coat color loci may impact behavior in the horse. J Hered 107: 214-219. doi:10.1093/jhered/esw007 [ Links ]
11. Jansen T, Forster P, Levine MA, Oelke H, Hurles M, Renfrew C, Weber J, Olek, K. 2002. Mitochondrial DNA and the origins of the domestic horse. Proc Natl Acad Sci 99: 10905-10910. doi: 10.1073/pnas.152330099 [ Links ]
12. Kelly L, Postiglioni A, De Andres D, Gagliardi R, Biagetti R, Franco J. 2002. Variabilidad genética de los caballos Criollos del Uruguay. Arch Med Vet 34: 13-23. doi: 10.4067/S0301732X2002000100002 [ Links ]
13. Kumar S, Stecher G, Tamura K. 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol Biol Evol 33: 1870-1874. doi: 10.1093/molbev/msw054 [ Links ]
14. Correa LS, Reyes A, Pardo E, Cavadía M. 2015. Genetic diversity detection of the domestic horse (Equus caballus) by genes associated with coat color. Rev MVZ Córdoba 20: 4779- [ Links ]4789.
15. Nei M. 1972. Genetic distance between populations. Am Nat 106: 283-292. [ Links ]
16. Pardo E, Causil L, Rodríguez A. 2015. Estudio de la diversidad genética de gato doméstico (Felis catus) mediante genes asociados al color del pelaje en Lorica-Córdoba, Colombia.Arch Zootec 64: 389-395. [ Links ]
17. Thiruvenkadan A, Kandasamy N, Panneerselvam S. 2008. Coat colour inheritance in horses. Livest Sci 117: 109129. doi: 10.1016/j.livsci.2008.05.008 [ Links ]
18. Toth Z, Kaps M, Soelkner J, Bodo I, Curik I. 2006. Quantitative genetic aspects of coat color in horses. J Anim Sci 84: 2623-2628. doi:10.2527/jas.2005-704 [ Links ]
19. Valera M, Molinab A, Gutiérrez J, Gómez J, Goyache F. 2005. Pedigree analysis in the Andalusian horse: population structure, genetic variability and influence of the Carthusian strain. Livest Prod Sci 95: 57-66. doi:10.1016/j.livprodsci.2004.12.004 [ Links ]
20. Yeh F, Yang R, Boyle T. 1999. POPGENE: Microsoft Windows-based freeware for population genetic analysis, version 1.31. University of Alberta, Edmonton, Canada. [Internet]. Available in: https://sites.ualberta.ca/~fyeh/popgene.pdf [ Links ]
Recibido: 19 de octubre de 2016
Aceptado para publicación: 24 de febrero de 2017