Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO  uBio
uBio
Compartilhar
Revista de Investigaciones Veterinarias del Perú
versão impressa ISSN 1609-9117
Rev. investig. vet. Perú vol.28 no.3 Lima jul./set. 2017
http://dx.doi.org/10.15381/rivep.v28i3.13350
ARTÍCULOS PRIMARIOS
Identificación de la microbiota levaduriforme en canal auditivo de porcinos con y sin secreción ótica
Determination of yeast microbiota in swine ear canal with and without ear secretion
Adriana Pulido-Villamarín1,3, Sabine Damme-Pedraza1, Rubiela Castañeda-Salazar1, Melva Linares-Linares1, Angélica Barbosa-Buitrago2
1 Línea de Epidemiología y Salud Animal, Unidad de Investigaciones Agropecuarias (UNIDIA), Departamento de Microbiología, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá, Colombia
2 Facultad de Ciencias Pecuarias, Universidad de Ciencias Aplicadas y Ambientales (UDCA), Bogotá, Colombia
3 E-mail: adriana.pulido@javeriana.edu.co
RESUMEN
Las levaduras de los géneros Malassezia y Candida forman parte de la microbiota normal en piel y mucosas de humanos y otros animales; sin embargo, han sido poco estudiadas en el canal auditivo porcino. El objetivo del presente estudio fue identificar la microbiota levaduriforme aislada a partir de hisopados del canal auditivo porcino. Se realizaron aislamientos primarios de 25 muestras (16 y 9 cerdos con y sin secreción serosa marrón,respectivamente)enAgar Dixon yAgar Sabouraud con cloranfenicol. Sedescribieron las características morfológicas macroscópicas y microscópicas de las colonias obtenidas; además, se realizaron pruebas bioquímicas, fisiológicas y moleculares para su identificación. Se recuperaron 55 aislados, cuya identificación molecular evidenció la presencia de M. sympodialis (42.8%), M. slooffiae (28.6%), M. furfur (18.4%) y M. pachydermatis (8.2%).
Palabras clave: Malassezia spp; Candida spp; porcinos; canal auditivo
ABSTRACT
Malassezia and Candida yeasts are considered part of the normal skin and mucosal microbiota in humans and other animals; however, there have been few studies on the pig’s ear canal microbiota. The aim of this study was to identify the yeast microbiota isolated from swabs of porcine ear canal. Primary isolation from 25 samples (16 and 9 pigs with and without brownish serous secretion) in Dixon and Sabouraud medium. Macroscopic and microscopic morphological features were described. Besides, biochemical, physiological and molecular tests were performed to identify the isolated yeasts. Fifty-five isolates were recovered and the molecular identification showed the presence of M. sympodialis (42.8%), M. slooffiae (28.6%), M. furfur (18.4%) and M. pachydermatis (8.2%).
Key words: Malassezia spp; Candida spp; swine; ear canal
INTRODUCCIÓN
Las patologías auditivas y la microbiota levaduriforme del canal auditivo porcino han sido poco estudiadas; sin embargo, clínicamente se reporta acúmulo de cerumen, leve a moderada inflamación en el canal auditivo y movimientos bruscos de cabeza en animales mayores de dos años de edad y en cerdos mascota (Pinter et al., 2002; Han y Na, 2009). Por otro lado, el análisis citológico de secreción ótica marrón en cerdos sanos reporta la presencia de levaduras y células epiteliales y, en algunos casos, microbiota mixta de levaduras y bacterias con células epiteliales (Harris et al., 2012). Entre las levaduras reportadas se encuentran las pertenecientes a los géneros Malassezia y Candida.
Las levaduras del género Malassezia son reconocidas como parte de la microbiota normal en piel de animales, aunque también han sido implicadas en procesos patológicos como patógenos oportunistas (Sugita et al., 2010). Se caracterizan por ser un grupo de levaduras lipofílicas, habiéndose reportado 13 especies lipodependientes: Malassezia furfur, M. globosa, M. obtusa, M. restricta, M. slooffiae, M. sympodialis (Guého et al., 1996), M. dermatis (Sugita et al., 2002), M. japonica (Sugita et al., 2003), M. nana (Hirai et al., 2004), M. yamatoensis (Sugita et al., 2004), M. equina, M. caprae (Cabañes et al., 2007) y M. cuniculi (Cabañes et al., 2011) y una no lipodependiente, M. pachydermatis (Guého et al., 1996). Recientemente, han sido propuestas dos nuevas especies lipodependientes: Malassezia brasiliensis sp. nov. y Malassezia psittaci sp. nov. (Cabañes et al., 2016).
Por otro lado, las levaduras del género Candida, que han sido descritas como microbiota comensal, causan alteraciones digestivas en porcinos, y se relacionan con factores predisponentes como el tratamiento con antibióticos (García y Blanco, 2000). En casos de inmunosupresión, ocasionan candidiasis mucocutánea y sistémica (Zlotowski et al., 2006).
La mayoría de estudios acerca de aisladosdeespeciesdelosgénerosMalassezia y Candida en el canal auditivo están referidos a caninos, por lo que teniendo en cuenta los pocos reportes en la literatura (Granados et al., 2014), el objetivo del presente estudio fue identificar la microbiota levaduriforme aislada a partir del canal auditivo externo porcino.
MATERIALES Y MÉTODOS
Población de Estudio
Se hizo una selección al azar de 25 cerdos adultos (hembras de cría, hembras de reemplazo y machos) con (n=16) o sin secreción (n=9) ótica de dos granjas semitecnificadas (cada una con menos de 200 hembras), sin historial clínico de afecciones óticas, ubicadas en los municipios de Tena y Ubaque, departamento de Cundinamarca (Colombia). El estudio fue de tipo descripti-vo, de corte transversal, con un solo muestreo al azar.
Obtención y Procesamiento de Muestras
Se obtuvieron hisopados del canal auditivo externo y se conservaron a temperatura ambiente en tween 20 al 0.05% para mantener el ambiente lipofílico (Pulido et al., 2010), durante un tiempo no mayor a 4 h. Las muestras fueron transportadas hasta los laboratorios de la Pontificia Universidad Javeriana, Bogotá, donde fueron cultivadas en Agar Dixon modificado y Agar Sabouraud suple-mentado con cloranfenicol, e incubadas a 32 °C. Las colonias morfológicamente compatibles con levaduras fueron purificadas para su posterior identificación (Cafarchia y Otranto, 2004; Ashbee, 2007; Crespo et al., 2008; Guého-Kellermann et al., 2010).
Para realizar la descripción macroscópica, se evaluaron los parámetros morfológicos de las colonias y se determinó el diámetro promedio (mm) de no menos de 10 colonias. Para la descripción microscópica, con aumento de 100X, se midió el largo y ancho de 30 células levaduriformes teñidas con Gram, utilizando el software Leica Microsystems®.
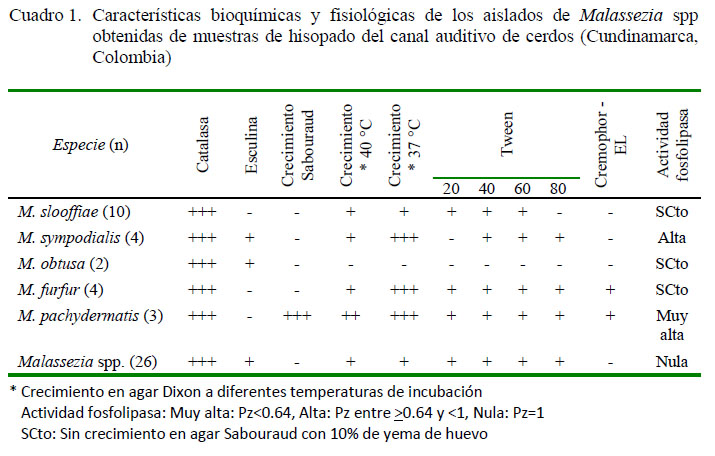
A las colonias presuntivas de Malassezia spp (urea positiva), se les realizaron pruebas bioquímicas y fisiológicas como asimilación de suplementos lipídicos (Cremophor-EL y varios Tween), crecimiento a temperaturas de 37 y 40 °C y pruebas enzimáticas como catalasa, β-glucosidasa (Cafarchia y Otranto, 2004; Ashbee, 2007; Crespo et al., 2008; Guého-Kellermann et al., 2010). Adicionalmente, se evaluó la actividad fosfolipasa, utilizando agar Sabouraud con 10% de yema de huevo, en cuatro puntos de siembra. La lectura se realizó al día 21 promediando los valores Pz, considerando una actividad nula aquellas con valor Pz=1, alta con valores Pz entre 0.64 y 0.99 y muy alta con Pz menor de 0.64 (Cafarchia y Otranto, 2004; Coutinho, 2005, Hurtado-Suarez et al., 2016).
Las colonias presuntivas de Candida spp (urea negativa) fueron identificadas usando medio cromogénico Chromagar Candida®.
Cepas de Referencia
Se utilizaron las cepas del Centraalbureau voor Schimmelcultures (CBS), Países Bajos: Malassezia furfur CBS 7019, Malassezia pachydermatis CBS 1879, Malassezia sympodialis CBS 7222, Malassezia slooffiae CBS 7956 y Malassezia globosa CBS 7966. Las cepas control positivo para la prueba de actividad fosfolipasa fueron Candida albicans ATCC 90028, Candida krusei ATCC 6258 y Candida parapsilosis ATCC 22019.
Identificación Molecular de Levaduras del Género Malassezia
La extracción del ADN se realizó con el estuche ADN genómico de hongos/levaduras (Norgen®). Estas extracciones se sometieron a tratamiento con ARNasa (Sigma®) 2 mg/ml a 37 °C por 4 h (Gemmer et al., 2002). Se realizó amplificación de la región 5.8S ADNr-ITS, utilizando los iniciadores ITS3 (5’-GCATCGATGAAGAACGCAGC-3’) e ITS4 (5’-TCCTCCGCTTATTGATATGC-3’) (Gaitanis et al., 2002; Hernández, 2005). La amplificación se llevó a cabo mediante PCR en volúmenes de reacción de 50 µl, conteniendo 45 µl de PCR SuperMix (1.1X) (Invitrogen), 1 µl de cada uno de los iniciadores (10 pmol/µl) y 3 µl de ADN genómico (20 ng/ml) bajo las siguientes condiciones: un ciclo de denaturación inicial a 95 °C por 5 min, 30 ciclos de 95 °C por 1 min, 55 °C por 1 min y 72 °C por 1.5 min, y un ciclo de extensión final a 72 °C por 5 min (Gaitanis et al., 2002; Hernández, 2005). Los productos de la amplificación obtenidos fueron evidenciados mediante electroforesis en geles de agarosa al 1.5% con buffer TBE 1X, teñidos con bromuro de etidio. Los productos de amplificación de la región 5.8S ADNr-ITS fueron enviados para su purificación y secuenciación a Macrogen® Inc. (Corea del Sur). Las secuencias fueron analizadas mediante BLASTn (Identities %: 9799%; E-value 0.0).
Análisis de Datos
Los datos obtenidos fueron analizados mediante estadística descriptiva.
RESULTADOS
De los 25 cerdos seleccionados al azar, nueve no presentaban secreción ótica y 16 tuvieron secreción serosa marrón. Del total de animales, se lograron 55 aislados, 49 de ellos compatibles con Malassezia spp (ureasa positiva) y 6 con Candida spp (ureasa negativa). Asimismo, de cada animal se aislaron desde uno hasta cuatro morfotipos.
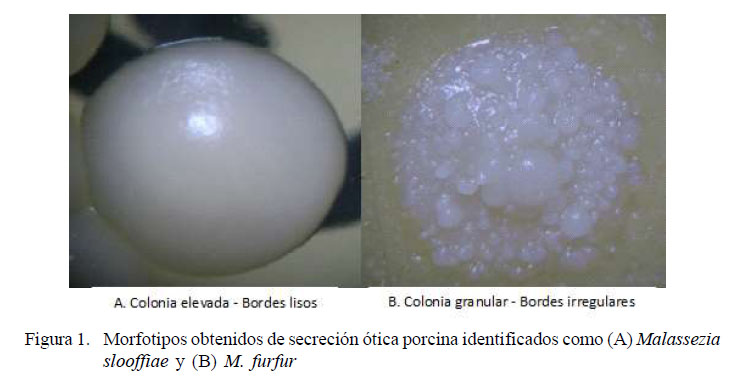
La caracterización morfológica macroscópica de Malassezia spp mostró diversidad de morfotipos (Figura 1). Desde colonias lisas de bordes regulares hasta colonias granulares, rugosas de bordes irregulares, con diámetros entre 1 y 5.5 mm. Microscópicamente, el tamaño de las células fue igualmente variable, detectando células madre con longitudes entre 2.2 y 3.8 µm y anchos de 1.6 y 3.2 µm en promedio.
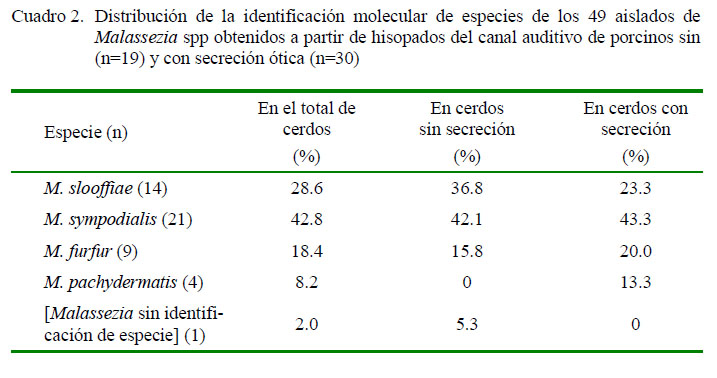
Con base en las pruebas bioquímicas, algunos aislados compatibles con Malassezia spp pudieron ser identificados hasta especie (Cuadro 1). Las especies identificadas mediante las pruebas moleculares se presentan en el Cuadro 2, donde se observa que se pudo determinar la especie de los aislados que fenotípicamente se identificaron como Malassezia spp. Las pruebas fenotípicas solo permitieron identificar hasta especie el 46.9% (n=23) de los aislados, mientras las pruebas moleculares determinaron la especie en el 98% de los aislados. El porcentaje de coincidencia entre las dos técnicas fue de 22.4%, dado que la identificación de la misma coincidió en 11 aislados.
Con respecto a los aislamientos de Candida spp, las colonias se caracterizaron por presentar bordes enteros, textura lisa, brillantes y de color blanco, con diámetros entre 2.5 y 5 mm. Microscópicamente, las longitudes fueron de 2.3 a 3.8 µm con anchos de 1.9 a 3.2 µm. Todos los aislamientos compatibles con Candida se obtuvieron únicamente a partir de cerdos con secreción ótica. Las especies fenotípicamente identificadas fueron Candida glabrata (n=2), C albicans, C. tropicalis, C. krusei y C. catenulata.
DISCUSIÓN
En el presente estudio, al igual que en otros reportes relacionados con Malassezia spp, se evidenciaron diferencias, tanto en las características macroscópicas de las colonias como en el comportamiento metabólico, especialmente para las pruebas de asimilación de los tween y el Cremophor-EL. De acuerdo con las características macroscópicas descritas en estudios previos y con lo evidenciado en las cepas de referencia, estas levaduras se caracterizan por presentar una textura lisa de bordes regulares (Guého-Kellermann et al., 2010; Hurtado-Suarez et al., 2016); sin embargo, esto difiere de lo observado para algunos de los aislamientos del presente estudio, donde se evidenciaron colonias rugosas de bordes irregulares, lo que podría atribuirse a la amplia variabilidad morfológica de los diferentes aislados de Malassezia spp (Hernández, 2005).
Teniendo en cuenta las pruebas metabólicas para la identificación de cada especie, M. pachydermatis es la única con capacidad de crecer sin suplemento lípidico, mientras que M. furfur se caracteriza por asimilar Cremophor-EL, conjuntamente con algunos aislamientos de M. pachydermatis (Mayser et al., 1997; Hossain et al., 2007; Guého-Kellermann et al., 2010; Guého et al., 2011); aspecto evidenciado en los aislamientos procedentes de porcinos. Por otro lado, se ha reportado que cada especie tiene ciertas preferencias para la asimilación de los tween; por ejemplo M. furfur y M. pachydermatis pueden asimilar tween 20, 40, 60 y 80, mientras M. sympodialis no asimila tween 20, pero si los demás (40, 60 y 80) (Guého et al., 1996; Cabañes et al., 2007; Crespo et al., 2008; Nardoni et al., 2010). Este mismo comportamiento fue observado, tanto para las cepas de referencia como para las especies identificadas a partir de los aislamientos obtenidos. Adicionalmente, otros estudios hacen referencia a que la actividad fosfolipasa es considerada como un factor de virulencia importante; específicamente para M. pachyder-matis (Juntachai et al., 2009), tal y como fue detectada en la mayoría de los aislamientos hallados en los cerdos con secreción del presente estudio (Cuadro 1), lo que sugiere su posible implicación en procesos patológicos.
Dada la dificultad en la identificación a nivel de especie mediante las pruebas fenotípicas, debido a la variabilidad de reacciones entre las mismas especies, la secuenciación de la región 5.8S ADNr-ITS permitió identificar el 98% de los aislamientos obtenidos. Esta misma región ha sido utilizada por otros autores, quienes también evidenciaron que mediante esta herramienta se obtiene una identificación más precisa (Cabañes et al., 2005; Gaitanis et al., 2006) y permite una aproximación epidemiológica real.
Estudios previos reportan la presencia de M. pachydermatis, M. furfur, M. sympodialis y M. sloofiae en el canal auditivo de porcinos. Los autores también hacen referencia a la dificultad en la clasificación, por lo que suelen ser reportadas como Malassezia spp (Pinter et al., 2002; Nardoni et al., 2010; Sugita et al., 2010). Aunque son pocos los reportes específicos, M. pachydermatis se ha reportado en cerdos con acúmulo de cerumen ótico e inflamación en el canal auditivo (Pinter et al., 2002), como ocurrió en el presente estudio con la presencia del 13.3% de esta especie en ejemplares con secreción ótica; sin embargo, Garau et al. (2005) reportaron la presencia de M. sympodialis (58%) y de M. slooffiae (30%) en cerdos sanos, siendo estos porcentajes cercanos a los encontrados en el presente estudio (Cuadro 2). Aunque M. furfur fue hallado tanto en porcinos sanos como con secreción, ha sido reportado como agente etiológico de otitis externa en un cerdo mascota (Han y Na, 2009).
Por otro lado, la experiencia del grupo de investigación, señala que generalmente predominaba una sola especie de Malassezia por muestra clínica; sin embargo, en el presente estudio, se evidenció que fueron obtenidas hasta cuatro especies a partir de una muestra de hisopado ótico, lo que sugiere que estas levaduras corresponderían a microbiota normal auditiva.
A nivel latinoamericano, solamente se encuentra un reporte en Costa Rica, donde analizaron cerdos con abundante secreción serosa en canal auditivo, encontrando 46% de prevalencia de M. pachydermatis, 21% de Trichosporon spp, 5% de Candida famata, 2% de C. parapsilosis, 3% de Aspergillus terreus y 23% de levaduras sin identificar (Granados et al., 2014); resultados que difieren de los hallados en el presente estudio.
De manera específica, levaduras como Candida glabrata y C. albicans, halladas en los cerdos con secreción ótica, han sido asociadas a lesiones mucocutáneas en dos casos de síndromes de debilitamiento posdestete (Zlotowski et al., 2006) y en un cuadro septicémico de un lechón (Zlotowski et al., 2014). Estos casos, aunque epidemiológicamente raros, podrían sugerir que la presencia de estas levaduras en canal auditivo de un paciente inmunosuprimido, podrían ser un factor de riesgo para que se presenten cuadros diseminados que podrían afectar la producción porcina.
Aunque es necesario profundizar en los antecedentes clínicos, citológicos y microbiológicos de los animales, teniendo en cuenta los resultados obtenidos, es posible concluir que la identificación a nivel de especie mediante pruebas moleculares determinó que M. sympodialis predominó en la microbiota levaduriforme del canal auditivo de los cerdos. Adicionalmente, la presencia de M. pachydermatis y las especies de Candida encontradas en porcinos con secreción ótica, podría sugerir su capacidad patogénica en esta especie animal. Este es el primer reporte en Colombia sobre la microbiota levaduriforme presente en el canal auditivo de porcinos.
Agradecimientos
Los autores agradecen a la Vicerrectoría de Investigación de la Pontificia Universidad Javeriana por el apoyo recibido mediante proyectos de investigación financiados con recursos propios de las Facultades e Institutos no adscritos a Facultad (2014, ID PTA 00006373), y a la Dra.Adriana Marcela Celis (Universidad de los Andes), por su contribución con las cepas de referencia.
LITERATURA CITADA
1. Ashbee H. 2007. Update on the genus Malassezia. Med Mycol 45: 287-303. doi: 10.1080/13693780701191373 [ Links ]
2. Cabañes FJ, Coutinho SDA, Puig L, Bragulat MR, Castellá G. 2016. New lipid-dependent Malassezia species from parrots. Rev Iberoam Micol 33: 92-99. doi: 10.1016/j.riam.2016.03.003 [ Links ]
3. Cabañes FJ, Hernández JJ, Castella G. 2005. Molecular analysis of Malassezia sympodialis-related strains from domestic animals. J Clin Microbiol 43: 277-283. doi: 10.1128/JCM.43.1.277- 283.2005 [ Links ]
4. Cabañes FJ, Theleen B, Castellá G, Boekhout T. 2007. Two new lipiddependent Malassezia species from domestic animals. FEMS Yeast Res 7: 1064-1076. doi: 10.1111/j.1567- 1364.2007.00217.x [ Links ]
5. Cabañes FJ, Vega S, Castellá G. 2011. Malassezia cuniculi sp. nov., a novel yeast species isolated from rabbit skin. Med Mycol 49: 40-48. doi: 10.3109/ 13693786.2010.493562 [ Links ]
6. Cafarchia C, Otranto D. 2004. Association between phospholipase production by Malassezia pachydermatis and skin lesions. J Clin Microbiol 42: 4868-4869. doi: 10.1128/JCM.42.10. 4868-4869.2004 [ Links ]
7. Coutinho S. 2005. Malassezia pachydermatis: enzymes production in isolates from external ear canal of dogs with and without otitis. Arq Bras Med Vet Zootec 57: 149-153. doi: 10.1590/ S0102-09352005000800003 [ Links ]
8. Crespo V, Crespo M, Gómez E. 2008. Diagnóstico de laboratorio de las levaduras del género Malassezia. Piel 23: 570-576. doi: 10.1016/S0213-9251(08) 75801-5 [ Links ]
9. Gaitanis G, Robert V, Velegraki A. 2006. Verifiable single nucleotide polymorphisms of the internal transcribed spacer 2 region for the identification of 11 Malassezia species. J Dermatol Sci 43: 214-217. doi: 10.1016/j.jdermsci.- 2006.03.013 [ Links ]
10. Gaitanis G, Velegraki A, Frangoulis E, Mitroussia A, Tsigonia A, Katsambas A, Legakis NJ. 2002. Identification of Malassezia species from patient skin scales by PCR-RFLP. Clin Microbiol Infect 8: 162-173. doi: 10.1046/j.1469-0691.2002.00383.x [ Links ]
11. Garau M, del Palacio A, Garcia J. 2005. Prevalence of Malassezia spp in healthy pigs. Mycoses 48: 17-20. doi: 10.1111/j.1439-0507.2004.01048.x [ Links ]
12. García ME, Blanco JL. 2000. Principales enfermedades fúngicas que afectan a los animales domésticos. Rev Iberoam Micol 17: S2-S7. [ Links ]
13. Gemmer C, DeAngelis Y, Theelen B, Boekhout T, Dawson T. 2002. Fast, noninvasive method for molecular detection and differentiation of Malassezia yeast species on human skin and application of the method to dandruff microbiology. J Clin Microbiol 40: 3350- 3357. doi: 10.1128/JCM.40.9.3350- 3357.2002 [ Links ]
14. Granados L, Calderón A, Urbina A. 2014. Aislamiento e identificación de levaduras en el conducto auditivo externo de cerdos en una granja porcina en Santa Bárbara de Heredia, Costa Rica. En: XXI Congreso Nacional de Médicos Veterinarios de Costa Rica. doi: 10.13140/2.1.1517.7286 [ Links ]
15. Guehó E, Batra R, Boekhout T. 2011. Malassezia Baillon (1889). In: Kurtzman CP, Fell JW, Boekhout T (eds). The yeast a taxonomic study. New York: Elsevier. p 1807-1832. [ Links ]
16. Guého E, Midgley G, Guillot J. 1996. The genus Malassezia with description of four new species. Antonie Van Leeuwenhock 69: 337-355. [ Links ]
17. Guého-Kellermann E, Boekhout T, Begerow D. 2010. Biodiversity, phylogeny and ultrastructure. En: Boekhout T, Guého E, Mayser P, Velegraki A (eds). Malassezia and the skin. Science and clinical practice: Germany: Springer- Verlag. p 17-63. [ Links ]
18. Han JI, Na KJ. 2008. Otitis externa caused by Malassezia furfur in a miniature pig. J Vet Clin 26: 303-305. [ Links ]
19. Harris T, Sott D, Smith M. 2012. The cytology of the external ear canal in the normal pig. Jpn J Vet Dermatol 18: 103-106. [ Links ]
20. Hernández J. 2005. Caracterización molecular de especies del género Malassezia. Tesis de Doctorado. España: Univ Autónoma de Barcelona. 139 p. [ Links ]
21. Hirai A, Kano R, Makimura K, Duarte RE, Hamdan JS, Lachance MA, Yamaguchi H, Hasegawa A. 2004. Malassezia nana sp. nov., a novel lipiddependent yeast species isolated from animals. Int J Syst Evol Microbiol 54: 623- 627. doi: 10.1099/ijs.0.02776-0 [ Links ]
22. Hossain H, Landgraf V, Weiss R, Mann M, Hayatpour J, Chakraborty T, Mayser P. 2007. Genetic and biochemical characterization of Malassezia pachydermatis with particular attention to pigment-producing subgroups. Med Mycol 45: 41-49. doi: 10.1080/13693780601003827 [ Links ]
23. Hurtado-Suárez A, Pulido-Villamarín A, Linares-Linares M, Suárez- Fernández L, Castañeda-Salazar R, Rodríguez-Bocanegra MX. 2016. Caracterización fenotípica de aislamientos de Malassezia spp de origen canino. Rev MVZ Córdoba 21: 5535-5546. [ Links ]
24. Juntachai W, Oura T, Yamagata M, Kajiwara S. 2009. The lipolytic enzymes activities of Malassezia species. Med Mycol 47: 477-484. doi: 10.1080/ 13693780802314825 [ Links ]
25. Mayser P, Haze P, Papavassilis C, Pickel M, Gruender K, Guehó E. 1997. Differentiation of Malassezia species: selectivity of Cremophor-EL, castor oil and ricinoleic acid for M. furfur. Brit J Dermatol 137: 208-213. doi: 10.1046/j.1365-2133.1997.18071890.x [ Links ]
26. Nardoni S, Merildi V, Frangioni S, Ariti G, Verin R, Vannucci P, Mancianti F. 2010. Isolation and characterization of Malassezia spp in healthy swine of different breeds. Vet Microbiol 141: 155- 158. doi: 10.1016/j.vetmic.2009.07.033 [ Links ]
27. Pinter L, Anthony RM, Glumac N, Hajsig D, Pogacnik M, Drobnic- Kosorok M. 2002. Apparent crossinfection with a single strain of Malassezia pachydermatis on a pig farm. Acta Vet Hung 50: 151-156. doi: 10.1556/AVet.50.2002.2.3 [ Links ]
28. Pulido A, Castañeda R, Linares M, Mercado M. 2010. Diagnóstico clínicomicrobiológico de otitis externa en caninos de Bogotá-Colombia. Rev MVZ Córdoba 15: 2215-2222. [ Links ]
29. Sugita T, Boekhout T, Velegraki A, Guillot J, Haðina S, Cabañes FJ. 2010. Epidemiology of Malasseziarelated skin diseases. In: Boekhout T, Guého E, Mayser P, Velegraki A (eds). Malassezia and the skin. Science and clinical practice. Germany: Springer- Verlag. p 65-119. [ Links ]
30. Sugita T, Tajima M, Takashima M, Amaya M, Saito M, Tsuboi R, Nishikawa A. 2004. A new yeast, Malassezia yamatoensis, isolated from a patient with seborrheic dermatitis and its distribution in patients and healthy subjects. Microbiol Immunol 48: 579-583. doi: 10.1111/j.1348-0421.2004.tb03554.x [ Links ]
31. Sugita T, Takashima M, Kodama M, Tsuboi R, Nishikawa A. 2003. Description of a new yeast species, Malassezia japonica, and its detection in patients with atopic dermatitis and healthy subjects. J Clin Microbiol 41: 4695-4699. doi: 10.1128/JCM.41.10. 4695-4699.2003 [ Links ]
32. Sugita T, Takashima M, Shinoda T, Suto H, Unno T, Tsuboi R, Ogawa H, Nishikawa A. 2002. New yeast species, Malassezia dermatis, isolated from patients with atopic dermatitis. J Clin Microbiol 40: 1363-1367. doi: 10.1128/ JCM.40.4.1363-1367.2002 [ Links ]
33. Zlotowski P, Amaral de CL, Spanamberg A, Cavallini SEM, Hein HE, Corbellini LG, et al. 2014. Candida glabrata septicemia in a piglet. Acta Sci Vet 41(Suppl 1): 27. [Internet]. Disponible en: http://revistas.bvs-vet.org.br/ actascivet/article/view/32258/pdf [ Links ]
34. Zlotowski P, Rozza DB, Pescador CA, Barcellos DE, Ferreiro L, Sanches EMC, Driemeier D. 2006. Mucocutaneous candidiasis in two pigs with postweaning multisystemic wasting síndrome. Vet J 171: 566-569. doi: 10.1016/ j.tvjl.2004.12.010 [ Links ]
Recibido: 11 de octubre de 2016
Aceptado para publicación: 2 de febrero de 2017