Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Veterinarias del Perú
versión impresa ISSN 1609-9117
Rev. investig. vet. Perú vol.28 no.3 Lima jul./set. 2017
http://dx.doi.org/10.15381/rivep.v28i3.13372
ARTÍCULOS PRIMARIOS
Aislamiento de Bacterias Ácido Lácticas a partir del Tracto Digestivo del Lechón
Isolation of Lactic Acid Bacteria from the Digestive Tract of the Piglet
Héctor Sánchez Súarez1,3, Fredy Fabián Domínguez1, Gloria Ochoa Mogollón1, Rubén Alfaro Aguilera2
1 Departamento Académico de Sanidad Vegetal y Producción Pecuaria, Facultad de Ciencias Agrarias,Universidad Nacional de Tumbes, Perú
2 Departamento de Biología y Bioquímica, Facultad de Salud, Universidad Nacional de Tumbes, Perú
3 E-mail: hsanchezs@untumbes.edu.pe
RESUMEN
El estudio tuvo como objetivo el aislar e identificar microorganismos nativos lactobacilos del tracto digestivo de lechones con la posibilidad de utilizarlos como inóculos para la mejora de los niveles productivos de lechones. Las muestras fueron sembradas y purificadas en agar Man, Rogosa y Sharpe (MRS). La identificación molecular de las bacterias se hizo mediante secuenciación del gen 16S ARNr. Se idientificaron cuatro especies: Lactobacillus johnsonii, Lactobacillus brevis, Enterococcus hirae y Pediococcus pentosaceus.
Palabra clave: bacterias ácido lácticas; lechón; inóculo
ABSTRACT
The aim of the study was to isolate and identify native lactobacillus microorganisms from the digestive tract of piglets with the possibility of using them for the improvement of the productive performance of piglets. Samples were cultured and purified on Man, Rogosa and Sharpe (MRS) agar. Molecular identification of the bacteria was done by the sequencing the 16S rRNA gene. Four species were identified: Lactobacillus johnsonii, Lactobacillus brevis, Enterococcus hirae and Pediococcus pentosaceus.
Key words: bacteria lactic acid; piglet; inoculum
INTRODUCCIÓN
El predestete y destete en los cerdos son etapas críticas, donde la alimentación líquida cambia drásticamente a una alimentación sólida (Castillo, 2000), predisponiendo al cerdo a factores del entorno que pueden incidir negativamente en su respuesta inicial al uso eficiente del alimento (Díaz, 2003).
El uso de promotores de crecimiento, entre otros aditivos inocuos, en las etapas críticas de la crianza de cerdos permite superar los problemas que se presentan; pero, por otro lado, aumentan los costos pudiendo limitar el proceso de producción (Bhandari et al., 2010). Debido a esto, se ha estudiado la influencia de diversos microorganismos, en especial de bacterias nativas del género Lactobacillus, así como de sus productos extracelulares, con efecto benéficos sobre el lechón (Cueto-Vigil et al., 2010; Kim et al., 2012). Bajo este contexto, en el presente estudio se aisló e identificó microorganismos nativos lactobacilos del tracto digestivo de lechones con la posibilidad de utilizarlos como inóculos benéficos para lechones.
MATERIALES Y MÉTODOS
Aislamiento de Bacterias Nativas
En una granja porcina de la ciudad de Tumbes, Perú, se seleccionó un lechón de 28 días de edad (destetado a los 21 días), con buen estado de salud y alimentado con concentrado comercial sin antibióticos. Cumpliendo con los estándares de ética de manejo de animales, el lechón fue anestesiado vía endovenosa, con dosis de 3 mg/k pv de ketamina a fin de realizar una laparotomía. Se tomaron muestras con hisopos estériles a partir de la mucosa epitelial del estómago, intestino delgado, intestino grueso y ciego. Las muestras fueron conservadas en tubos cónicos conteniendo caldo de Man, Rogosa y Sharpe (MRS) pH 6.5, y fueron trasladas en cadena de frío al laboratorio.
Se realizaron diluciones seriadas hasta 20-2 y fueron sembradas por superficie en placas Petri con agar MRS pH 6.5 e incubadas a 37 °C por 48 h. Posteriormente, se realizaron purificaciones consecutivas en agar MRS de las colonias bacterianas aisladas, los que fueron confirmados con tinción Gram.
Se aislaron un total de 19 cepas bacterianas. La evaluación de las características fenotípicas (bacilos Gram positivos sin presencia de esporas, catalasa negativa) permitió seleccionar cuatro cepas para su identificación molecular. Estas cepas fueron conservadas en tubos con agar MRS inclinado a 4 °C y congeladas a -20 °C en caldo MRS suplementado con 30% de glicerol.
Identif icación Molecular mediante Secuenciación del Gen 16S ARNr
Extracción de ADN
Se utilizó el método rápido de ebullición para plásmidos pequeños de Escherichia coli (basado en Holmes y Quigley [1981], modificado por Riggs y Mc Lachlan [1986]) con modificaciones). Para esto, las cepas seleccionadas fueron sembradas en tubos de vidrio con 10 ml de caldo MRS e incubadas a 37 ºC por 24 h. Se tomó 1.2 ml de la suspensión bacteriana en un microtubo de 1.5 ml y se centrifugó a 6160 g durante 2 min, se eliminó el sobrenadante y se resuspendió el pellet de células con 500 µl de solución PBS 1X estéril y, luego, centrifugado a 6160 g por 1 min. Se eliminó el sobrenadante y se agregó 200 µl de solución STET (sacarosa 8%, Tritón X-100 5%, EDTA 50 mM, Tris-HCl 50 mM pH 8.0) y 10 µl de lisozima (20 mg/ml, disuelto en agua destilada). Se incubó en baño de agua térmico a 100 °C por 45 a 55 s y se colocó sobre hielo. Se centrifugó a 10 410 g por 10 min y se transfirió el sobrenadante a otro microtubo que contenía 400 µl de isopropanol frío, dejándose reposar en congelación por 30 min. Luego se centrifugó a 10 410 g por 10 min y se eliminó el sobrenadante. El pellet obtenido fue lavado con etanol al 75% y secado a temperatura ambiente. Finalmente, el pellet de ADN fue disuelto con 200 µl de solución TE (Tris 1M y EDTA 0.1mM) a 65 °C. Las muestras fueron conservadas a -20 °C hasta su utilización.
Análisis por PCR
Se utilizó la técnica de PCR simple para amplificar la región 16S ARNr. Para esto, se utilizaron los cebadores universales 16S ARNr F518 (CCAGCAGCCGCGGTAATACG) y 16S ARNr R800 (TACCAG-GGTATCTA-ATCC), descritos para estudios filogenéticos bacterianos (Lane, 2001). Las reacciones de PCR se realizaron en mezclas de 25 µl, constituida por 2.5 µl de buffer Taq 10X, 1.5 mM de MgCl , 0.2 mM de cada dNTPs (10 mM), 0.5 U de Taq ADN polimerasa (Invitrogen), 0.36 pmol de cada cebador y 2 µl de ADN extraído. El perfil de la PCR se realizó en un termociclador (Thermo Scientific) y consta de un ciclo de 94 °C por 6 min, seguido de 40 ciclos de 94°C por 30 s, 58 °C por 45 s y 72 °C por 1 min, y una extensión final a 72 °C por 4 min.
Los productos de amplificación fueron verificados por electroforesis en geles de agarosa al 1.5%, teñidos con bromuro de etidio (0.5 µg/ml) y por electroforesis en TAE 1X.
Secuenciación de los productos
Se utilizaron 10 µl de los productos obtenidos por amplificación en la PCR, que fueron colocados en microtubos de 0.2 ml. Además, se prepararon microtubos de 0.2 ml con porciones de 5 µl de cada cebador universal para el gen 16S ARNr. Estos fueron empacados y la secuenciación de las dos cadenas de cada producto amplificado fue hecha por Macrogen, EEUU.
Las secuencias de ADN fueron alineadas con el software libre MEGA 5 y comparadas con las secuencias de 16S ARNr que se encuentra en la base de datos de acceso público del GenBank mediante el software libre BLAST (Basic Local Alignment Search Tool).
RESULTADOS
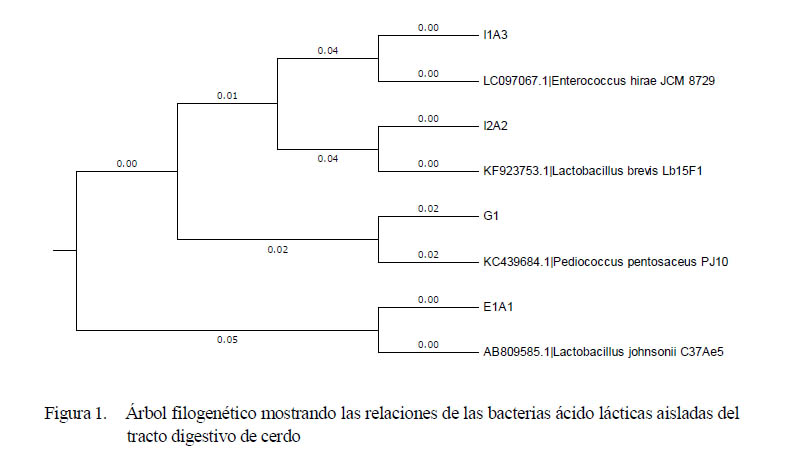
Las cuatro cepas molecularmente identificadas fueron Lactobacillus brevis, Lactobacillus johnsonii, Enterococcus hirae y Pediococcus pentosaceus, clasificadas dentro del orden Lactobacillales, las que también son denominadas bacterias ácido lácticas (Cuadro 1, Figura 1).
DISCUSIÓN
La flora microbiana natural del tracto digestivo proporciona las condiciones necesarias para el eficiente uso de los nutrientes recibidos con los alimentos (Cranwell, 1985; Rosmini et al., 2004). Dentro de esta flora microbiana, las bacterias son las más predominantes y colonizan específicamente cada porción del tracto intestinal, permitiendo una asociación simbiótica con el hospedero (Hong et al., 2011). Esto podría permitir el uso de bacterias benéficas aisladas de la misma especie productiva, debido a su alta especificidad para su hospedero, resultando en una mejora de la salud general de los animales.
En el presente estudio se logró aislar e identificar molecularmente mediante el gen 16S a cuatro especies de bacterias ácido lácticas, que comprenden entre otros, a los géneros Lactococcus, Streptococcus y Leuconostoc (Partanen y Mroz, 1999; Collado y Sanz, 2007; Cueto et al., 2010; Mallo et al., 2010). Diversos estudios demuestran que la presencia de lactobacilos en el tracto digestivo del cerdo mejora su rendimiento productivo (Konstantinov et al., 2008; Lamendela et al., 2011; Loft et al., 2014; Deusch et al., 2015). Para este fin, estas bacterias utilizan diferentes mecanismos de acción relacionados con la estimulación de la inmunidad, producción de sustancias inhibitorias, así como competencia por la adhesión en los receptores del epitelio intestinal y por nutrientes (Castillo, 2000; Pluske, 2003; Champagne et al., 2005; de Lange et al., 2010; Klose et al., 2010; Blajman et al., 2015).
Estudios dirigidos por Gebert et al. (2011) demuestran que la colonización temprana del tracto gastrointestinal con la cepa 1E1 de Lactobacillus brevis mejora el sistema inmune y reduce el establecimiento de patógenos en lechones lactantes. Pediococcus pentosaceus DPC6006 fue aislada, seleccionada in vitro con base a criterios probióticos y ensayada in vivo en lechones, demostrando que reduce la cantidad de microorganismos indeseables del tracto digestivo (Gardiner et al., 2004). Asimismo, se reporta a Enterococcus sp como una bacteria comensal del intestino de muchos mamíferos, entre ellos, gatos, perros, ratas, cerdos y terneros (Etheridge et al., 1988; Jergens et al., 1991). En forma similar, Larsson et al. (2014) mencionan que E. hirae coloniza específicamente el epitelio intestinal de lechones, generando diarreas. En otros casos, aislados bacterianos de E. hirae a partir de fluidos del rumen de Bos primigenius, presentaron propiedades bactericidas y antiinflamatorias (Arokiyaraj et al., 2014).
Wang et al. (2014) demostraron que la administración de L. johnsonii XS4 en dietas para cerdas al final de la gestación y durante la lactancia tuvieron efectos positivos sobre el rendimiento, en tanto que BuhnikRosenblau et al. (2012) sugieren que esta bacteria tiene una alta especificidad por sus hospederos. Esta especie bacteriana es considerada potencialmente probiótica y de gran interés para la industria alimentaria y farmacéutica (La Ragione et al., 2004; Pridmore et al., 2004).
Considerando la información obtenida, estos inóculos podrían ser evaluados experimentalmente en lechones para determinar su potencial en la mejora productiva porcina.
LITERATURA CITADA![]()
1. Arokiyaraj S, Islam H, Bharanidharan R, Raveendar S, Lee J, Kim do H, Oh Y, et al. 2014. Antibacterial, antiinflammatory and probiotic potential of Enterococcus hirae isolated from the rumen of Bos primigenius. World J Microbiol Biotechnol 30: 2111-2118. doi: 10.1007/s11274-014-1625-0 [ Links ]
2. Bhandari S, Opapeju F, Krause D, Nyachoti C. 2010. Dietary protein level and probiotic supplementation effects on piglet response to Escherichia coli K88 challenge: performance and gut microbial population. Livestock Sci 133: 185-188. doi: 10.1016/j.livsci.2010.06.060 [ Links ]
3. Blajman J, Zbrun M, Astesana D, Berisvil A, Romero Scharpen A, Fusari M, Soto L, et al. 2015. Probióticos en pollos parrilleros: una estrategia para los modelos productivos intensivos. Rev Argent Microbiol 47: 360-367. doi:10.1016/j.ram.2015.08.002 [ Links ]
4. Buhnik-Rosenblau K, Matsko-Efimov V, Jung M, Shin H, Danin-Poleg Y, Kashi Y. 2012. Indication for coevolution of Lactobacillus johnsonii with its host. BMC Microbiology 12: 149. doi: 10.1186/1471-2180-12-149 [ Links ]
5. Castillo W. 2000. Alteraciones en el sistema digestivo de lechones destetados precozmente. En: XXIII Reunión Científica Anual de la Asociación Peruana de Producción Animal. Huánuco, Perú [ Links ].
6. Collado M, Sanz Y. 2007. Characterization of the gastrointestinal mucosa-associated microbiota of pigs and chickens using culture-based and molecular methodologies. J Food Prot 70: 2799-2804. [ Links ]
7. Cranwell P. 1985. The development of acid and pepsin (EC 3.4.23.1) secretory capacity in the pig; the effects of age and weaning. 1. Studies in anaesthetized pigs. Br J Nutr 54: 305-320. doi: 10.1079/BJN19850113 [ Links ]
8. Cueto-Vigil M, Acuña-Monsalve Y, Valenzuela-Riaño J. 2010. Evaluación in vitro del potencial probiótico de bacterias ácido lácticas aisladas del suero costeño. Actu Biol 32: 129-138. [ Links ]
9. Champagne C, Gardner N, Roy D. 2005. Challenges in the addition of probiotic cultures to foods. Crit Rev Food Sci Nutr 45: 61-84. [ Links ]
10. de Lange C, Pluske J, Gong J, Nyachoti N. 2010. Strategic use of feed ingredients and feed additives to stimulate gut health and development in young pigs. Livestock Sci 134: 124-134. doi: 10.1016/j.livsci.2010.06.117 [ Links ]
11. Deusch S, Tilocca B, Camarinha-Silva A, Seifert J. 2015. News in livestock research use of Omics-technologies to study the microbiota in the gastrointestinal tract of farm animals. Comput Struc Biotechnol 13: 55-63. doi: 10.1016/j.csbj.2014.12.005 [ Links ]
12. Díaz W. 2003. Uso de flavorizante en dietas preiniciadoras y de recría para lechones destetados precozmente. Tesis de Doctorado. Tingo María, Perú: Univ Nacional Agraria de la Selva. 45 p. [ Links ]
13. Etheridge M, Yolken R, Vonderfecht S. 1988. Enterococcus hirae implicated as a cause of diarrhea in suckling rats. J Clin Microbiol 26: 1741-1744. [ Links ]
14. Gebert S, Davis E, Rehberger T, Maxwell C. 2011. Lactobacillus brevis strain 1E1 administered to piglets through milk supplementation prior to weaning maintains intestinal integrity after the weaning event. Benef Microbes 2(1): 35-45. doi: 10.3920/BM2010.0043 [ Links ]
15. Gardiner G, Casey P, Casey G, Lynch P, Lawlor P, Hill C, Fitzgerald G, et al. 2004. Relative ability of orally administered Lactobacillus murinus to predominate and persist in the porcine gastrointestinal tract. Appl Environ Microbiol 70: 1895-1906. doi: 10.1128/AEM.70.4.1895-1906.2004 [ Links ]
16. Holmes D, Quigley M. 1981. A rapid boiling method for the preparation of bacterial plasmids. Anal Biochem 114: 193-197. doi: 10.1016/0003-2697(81)90473-5 [ Links ]
17. Hong T, Passoth V, Lindberg J. 2011. Bacterial diversity at different sites of the digestive tract of weaned piglets fed liquid diets. Asian Austral J Anim Sci 24: 834-843. doi: 10.5713/ajas.2011.10291 [ Links ]
18. Jergens A, Moore F, Prueter J, Yankauskas P. 1991. Adherent grampositive cocci on the intestinal villi of two dogs with gastrointestinal disease. J Am Vet Med Assoc 198: 1950-1952. [ Links ]
19. Kim H, Borewicz K, White B, Singer R, Sreevatsan S, Tu Z, Isaacson R. 2012. Microbial shifts in the swine distal gut in response to the treatment with antimicrobial growth promoter, tylosin. Proc Natl Acad Sci USA 109: 1548515490. doi: 10.1073/pnas.1205147109 [ Links ]
20. Klose V, Bayer K, Bruckbeck R, Schatzmayr G, Loibner A. 2010. In vitro antagonistic activities of animal intestinal strains against swine-associated pathogens. Vet Microbial 144: 515-521. doi: 10.1016/j.vetmic.2010.02.025 [ Links ]
21. Konstantinov S, Smidt H, Akkermans A, Casini L, Trevisi P, Mazzoni M, De Filippi S, et al. 2008. Feeding of Lactobacillus sobrius reduces Escherichia coli F4 levels in the gut and promotes growth of infected piglets. FEMS Microbiol Ecol 66: 599-607. doi: 10.1111/j.1574-6941.2008.00517.x [ Links ]
22. Lane D. 1991. 16S/23S rRNA sequencing. In: Stackebrandt E, Goodfellow M (eds). Nucleic acid techniques in bacterial systematics. New York: Wiley. p 115-175. [ Links ]
23. Lamendela R, Santo Domingo J, Ghosh S, Martinson J, Oerther D. 2011. Comparative fecal metagenomics unveils unique functional capacity of the swine gut. BMC Microbiology 11: 103. doi: 10.1186/1471-2180-11-103 [ Links ]
24. Larsson J, Lindberg R, Aspan A, Grandon R, Westergren E, Jacobson M. 2014. Neonatal piglet diarrhoea associated with enteroadherent Enterococcus hirae. J Comp Path 151: 137-147. doi: 10.1016/j.jcpa.2014.04.003 [ Links ]
25. La Ragione R, Narbad A, Gasson M, Woodward M. 2004. In vivo characterization of Lactobacillus johnsonii FI9785 for use as a defined competitive exclusion agent against bacterial pathogens in poultry. Lett Appl Microbiol 38: 197-205. [ Links ]
26. Loft T, Allen H, Cantarel B, Levine U, Bayles D, Alt D, Henrissat B, Stanton T. 2014. Bacteria, phages and pigs: the effects of in-feed antibiotics on the microbiome at different gut locations. ISME J 8: 1566-1576. doi: 10.1038/ ismej.2014.12 [ Links ]
27. Mallo J, Rioperez J, Honrubia P. 2010. The addition of Enterococcus faecium to diet improves piglet’s intestinal microbiota and performance. Livestock Sci 133: 176-178. doi: 10.1016/j.livsci.2010.06.057
28. Partanen K, Mroz Z. 1999. Organic acid for performance enhancement in pig diets. Nutr Res Rev 12: 117-145. doi: 10.1079/095442299108728884 [ Links ]
29. Pluske J. 2013. Feedand feed additives-related aspects of gut health and development in weanling pigs. J Anim Sci Biotechnol 4: 1. doi:10.1186/2049-1891-4-1 [ Links ]
30. Pridmore R, Berger P, Desiere F, Vilanova D, Barretto C, Pittet A, Zwahlen M, et al. 2004. The genome sequence of the probiotic intestinal bacterium Lactobacillus johnsonii NCC 533. Proc Nat Acad Sci 101: 25122517. doi:10.1073/pnas.0307327101 [ Links ]
31. Riggs M, Mc Lachlan A. 1986. A simplified screening procedure for large numbers of plasmid mini-preparation. Biotech Tech 4: 310-313. [ Links ]
32. Rosmini M, Sequeira G, Guerrero I, Martí L, Dalla R, Frizzo L, Bonazza L. 2004. Producción de prebióticos para animales de abasto: importancia del uso de la microbiota intestinal indígena. Rev Mex Ing Quím 3: 181-191. [ Links ]
Recibido: 19 de octubre de 2016
Aceptado para publicación: 3 de abril de 2017